Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Evaluierung der Caspase-Aktivierung zur Beurteilung des angeborenen Immunzelltods
In diesem Artikel
Zusammenfassung
Dieses Protokoll beschreibt eine umfassende Methode zur Beurteilung der Caspase-Aktivierung (Caspase-1, Caspase-3, Caspase-7, Caspase-8, Caspase-9 und Caspase-11) als Reaktion auf In-vitro - und In-vivo-Modelle (in Mäusen) von Infektionen, sterilen Beleidigungen und Krebs, um die Initiierung von Zelltodwegen wie Pyroptose, Apoptose, Nekroptose und PANoptose zu bestimmen.
Zusammenfassung
Die angeborene Immunität stellt die entscheidende erste Verteidigungslinie als Reaktion auf Krankheitserreger und sterile Beleidigungen dar. Eine wichtige mechanistische Komponente dieser Reaktion ist die Einleitung des angeborenen immunprogrammierten Zelltods (PCD), um infizierte oder beschädigte Zellen zu eliminieren und Immunantworten zu verbreiten. Eine übermäßige PCD ist jedoch mit Entzündungen und Pathologien verbunden. Daher ist das Verständnis der Aktivierung und Regulation von PCD ein zentraler Aspekt bei der Charakterisierung der angeborenen Immunantwort und der Identifizierung neuer therapeutischer Ziele im gesamten Krankheitsspektrum.
Dieses Protokoll bietet Methoden zur Charakterisierung der PCD-Aktivierung des angeborenen Immunsystems durch Überwachung von Caspasen, einer Familie von Cystein-abhängigen Proteasen, die häufig mit verschiedenen PCD-Signalwegen assoziiert sind, einschließlich Apoptose, Pyroptose, Nekroptose und PANoptose. Erste Berichte charakterisierten Caspase-2, Caspase-8, Caspase-9 und Caspase-10 als Initiator-Caspasen und Caspase-3, Caspase-6 und Caspase-7 als Effektor-Caspasen bei Apoptose, während spätere Studien die entzündlichen Caspasen Caspase-1, Caspase-4, Caspase-5 und Caspase-11 fanden, die Pyroptose auslösen. Es ist nun bekannt, dass es eine umfangreiche Wechselwirkung zwischen den Caspasen und anderen angeborenen Immun- und Zelltodmolekülen über die zuvor definierten PCD-Signalwege gibt, was eine wichtige Wissenslücke im mechanistischen Verständnis der angeborenen Immunität und der PCD identifiziert und zur Charakterisierung der PANoptose führt. PANoptose ist ein einzigartiger angeborener immuninflammatorischer PCD-Signalweg, der durch PANoptosom-Komplexe reguliert wird, die Komponenten, einschließlich Caspasen, aus anderen Zelltodwegen integrieren.
Hier werden Methoden zur Beurteilung der Aktivierung von Caspasen als Reaktion auf verschiedene Reize bereitgestellt. Diese Methoden ermöglichen die Charakterisierung von PCD-Signalwegen sowohl in vitro als auch in vivo, da aktivierte Caspasen eine proteolytische Spaltung durchlaufen, die durch Western Blot unter Verwendung optimaler Antikörper und Blotting-Bedingungen sichtbar gemacht werden kann. Es wurden ein Protokoll und ein Western-Blotting-Workflow etabliert, die es ermöglichen, die Aktivierung mehrerer Caspasen aus derselben Zellpopulation zu bewerten und eine umfassende Charakterisierung der PCD-Prozesse zu ermöglichen. Diese Methode kann in allen Forschungsbereichen in den Bereichen Entwicklung, Homöostase, Infektion, Entzündung und Krebs angewendet werden, um PCD-Signalwege in zellulären Prozessen in Gesundheit und Krankheit zu bewerten.
Einleitung
Das angeborene Immunsystem fungiert als erste Verteidigungslinie während einer Infektion und als Reaktion auf sterile Reize wie Gewebeverletzungen und Veränderungen der Homöostase. Angeborene Immunsensoren auf der Zelloberfläche und im Zytoplasma reagieren auf pathogen- oder schädigungsassoziierte molekulare Muster (PAMPs bzw. DAMPs), um entzündliche Signalwege und zelluläre Reaktionen auszulösen. Einer der Schlüsselprozesse der angeborenen Immunantwort ist die Induktion des Zelltods, um infizierte oder beschädigte Zellen zu entfernen und weitere angeborene und adaptive Immunantworten voranzutreiben. Der programmierte Zelltod (PCD) ist ein hochkonservierter Prozess über Spezies hinweg, was seine evolutionäre Bedeutung als angeborener Immunmechanismus unterstreicht.
Es gibt mehrere angeborene Immun-PCD-Signalwege, die in allen Zelltypen aktiviert werden können. Caspasen sind eine Schlüsselfamilie hochkonservierter, intrazellulärer, Cystein-abhängiger Proteasen, die für viele PCD-Signalwege, einschließlich des traditionell nicht-inflammatorischen Apoptosewegs sowie entzündlicher PCD-Signalwege wie Pyroptose, Nekroptose und PANoptosekritisch sind 1,2,3,4,5 . Es gibt 11 humane und 10 murine Caspasen, die gut definiert sind, sowie Pseudo-Caspasen, die funktionell sein können, und die meisten werden konstitutiv als inaktive monomere oder dimere Pro-Caspasen exprimiert, die eine Spaltung zur Aktivierung erfordern 6,7. Caspasen enthalten auch wichtige Domänen für die Rekrutierung und Bildung von Multiproteinkomplexen. Dazu gehören die Caspase-Aktivierungs- und Rekrutierungsdomäne (CARD), die in Caspase-1, Caspase-2, Caspase-4, Caspase-5, Caspase-9 und Caspase-11 zu finden ist, oder die Todeseffektordomäne (DED), die in Caspase-8 und Caspase-10 zu finden ist. Sowohl durch ihre proteolytische Aktivität als auch durch ihre Fähigkeit, Multiproteinkomplexe zu bilden, sind Caspasen entscheidende Treiber der angeborenen Immun-PCD.
Die Rolle von Caspasen bei der angeborenen Immun-PCD wurde erstmals in der Apoptose identifiziert, wo die Initiator-Caspasen Caspase-2, Caspase-8, Caspase-9 und Caspase-10 die Henker-Caspasen Caspase-3, Caspase-6 und Caspase-7 aktivieren, um den Zelltodzu beschleunigen 8,9,10,11,12. Initiator-Caspasen können durch verschiedene Signalkaskaden aktiviert werden; Der extrinsische Signalweg aktiviert Caspase-8 durch extrazelluläre Liganden-induzierte Todesrezeptor-Signalgebung, und der intrinsische Signalweg aktiviert Caspase-9 durch die Störung der mitochondrialen Integrität13. Aktivierte Initiator-Caspasen spalten den Linker und trennen die großen und kleinen katalytischen Untereinheiten von Henker-Caspasen, um ihre aktiven Formen zu erzeugen. Die Henker-Caspasen spalten dann ihre Substrate, um die Zelle zu zerlegen, was zu DNA-Abbau, Membranblasen, Kernfragmentierung und der Freisetzung apoptotischer Körper führt14,15. Dieser Prozess endet typischerweise in einer nicht-lytischen und nicht-entzündlichen Form des Zelltods, wenn er mit der sofortigen Beseitigung der sterbenden Zellen durch Efferozytosegekoppelt ist 16. Defekte in der Efferozytose oder ein Mangel an phagozytischen Zellen können jedoch zur Anhäufung von apoptotischen Zellen führen, die dann einen lytischen und entzündlichen Zelltod durchlaufen17,18.
Es wurde entdeckt, dass die entzündlichen Caspasen, einschließlich Caspase-1 (Mensch und Maus), Caspase-4 und Caspase-5 (Mensch) und Caspase-11 (Maus), während einer Form der entzündlichen angeborenen Immun-PCD (III-PCD) aktiviert werden, die als Pyroptose bezeichnet wird. Die Caspase-1-Aktivierung ist mit der Bildung von Inflammasomen verbunden, bei denen es sich um Multiproteinkomplexe handelt, die einen zytosolischen angeborenen Immunsensor, ein Adaptermolekül (Apoptose-assoziiertes speckartiges Protein, das eine CARD [ASC] enthält) und Caspase-1 enthalten. Die Bildung dieses Komplexes ermöglicht es Caspase-1, eine Proximity-vermittelte Autoproteolyse zu durchlaufen, um seine aktive Form freizusetzen, die Zielsubstrate spalten kann, einschließlich der proinflammatorischen Zytokine Interleukin (IL)-1β und IL-18 und des porenbildenden Moleküls Gasdermin D (GSDMD)19,20,21,22,23 . Caspase-11, Caspase-4 und Caspase-5 können GSDMD auch aktivieren, ohne dass das Inflammasom stromaufwärts gebildet wird, nachdem PAMPs wie Lipopolysaccharid (LPS) erkannt wurden19,20. Diese Caspasen durchlaufen eine Dimerisierung, gefolgt von einer Oligomerisierung und Selbstspaltung zur Aktivierung bei Bindung an zytosolisches LPS, was zu einer nicht-kanonischen Inflammasom-Aktivierung 24,25,26 und einer Caspase-1-Aktivierung in zellintrinsischer Weise führt, um die IL-1β- und IL-18-Reifung zu induzieren 20. Die Reifung und Freisetzung dieser entzündungsfördernden Zytokine charakterisieren diese Caspasen als "entzündlich". Darüber hinaus wurde festgestellt, dass die apoptotische Caspase-8 auf dem Inflammasom lokalisiert ist, was eine Verbindung zwischen apoptotischen und pyroptotischen Prozessen herstellt. Studien haben gezeigt, dass die apoptotische Caspase-8 auch für die Regulierung einer anderen Form der PCD, der Nekroptose, entscheidend ist. Der Verlust von Caspase-8 führt zu einer spontanen Rezeptor-interagierenden Serin-Threonin-Kinase 3 (RIPK3)-vermittelten Mixed-Lineage-Kinase-Domänen-ähnlichen Pseudokinase (MLKL)-Aktivierung, um den III-PCD-Weg der Nekroptose 27,28,29,30,31,32,33,34,35 anzutreiben.
Während Caspasen in der Vergangenheit aufgrund der Art des Zelltods, den sie auslösen, als "apoptotisch" oder "entzündlich" eingestuft wurden, deuten immer mehr Hinweise darauf hin, dass es durch Caspasen ein umfangreiches Übersprechen zwischen den angeborenen immunen PCD-Signalwegen gibt 3,4. Zum Beispiel spaltet die inflammatorische Caspase-1 aus Inflammasomen die apoptotische Caspase-7 an ihrer kanonischen Aktivierungsstelle34. Die Caspase-1-Aktivierung kann auch zur Spaltung von apoptotischen Substraten wie der Poly(ADP-Ribose)-Polymerase 1 (PARP1)36 führen. In Zellen, denen GSDMD fehlt, kann Caspase-1 auch Caspase-3 spalten37,38. Zusätzlich kann die kanonisch apoptotische Caspase-3 Gasdermin E (GSDME) spalten, um PCD17,18 zu induzieren, und verarbeitet auch GSDMD in eine inaktive Form40. Darüber hinaus wurde eine Caspase-8-Rekrutierung für den Inflammasom-Komplex beobachtet 39,40,41,42,43,44,45, und Caspase-8 ist ein Schlüsselregulator der kanonischen und nicht-kanonischen Inflammasom-Aktivierung 39. Es gibt auch überlappende und redundante Rollen für Caspase-8 und Caspase-1 bei vielen entzündlichen Erkrankungen, und die angeborene Immun-PCD, die durch die Aktivierung von pyroptotischen, apoptotischen und nekrototischen Komponenten gekennzeichnet ist, tritt im gesamten Krankheitsspektrumauf 39,46,47,48,49,50.
Basierend auf dieser Wechselwirkung zwischen entzündlichen und apoptotischen Caspasen wurde eine Schlüssellücke im mechanistischen Verständnis der angeborenen Immunität und der PCD identifiziert, die zur Entdeckung der PANoptose führte. PANoptose ist eine einzigartige Form der III-PCD, die als Reaktion auf Krankheitserreger, PAMPs, DAMPs und Veränderungen der Homöostase aktiviert wird und durch PANoptosomen reguliert wird - facettenreiche makromolekulare Komplexe, die Komponenten aus anderen Zelltodwegen integrieren 44,50,51,52,53,54,55 . Die Gesamtheit der biologischen Wirkungen bei der PANoptose kann nicht allein durch Pyroptose, Apoptose oder Nekroptose allein erklärt werden 3,4,35,36,39,46,47,48, da die PANoptose durch die Aktivierung mehrerer Caspasen gekennzeichnet ist, einschließlich Caspase-1, Caspase-11, Caspase-8, Caspase-9, Caspase-3 und/oder Caspase-7, je nach Kontext 44,48,49,50,51,52,53,54,56,57,58,59,60,61,62 . PANoptose wird zunehmend mit Infektions- und Entzündungskrankheiten sowie mit Krebs und Krebstherapien in Verbindung gebracht 3,4,35,36,39,44,46,47,48,49,50,51,52,53 ,54,56
,57,58,59,60,61,62,63,64,65,66.
Angesichts der essentiellen Rolle von Caspasen in Zelltodwegen, einschließlich Apoptose, Pyroptose, Nekroptose und PANoptose, ist es wichtig, Techniken zu entwickeln, um ihre Aktivierung zu charakterisieren und die volle Komplexität der PCD-Signalwege zu verstehen. Das Protokoll beschreibt hier eine Methode zur Stimulation von Zellen und zur Messung der anschließenden Aktivierung von Caspasen (Abbildung 1). Diese Methode nutzt die proteolytische Spaltung von Caspasen, die im Allgemeinen für ihre Aktivierung erforderlich ist, um sie zu untersuchen. Durch Western Blot können die Proteingrößen bestimmt werden, was eine klare Visualisierung und Differenzierung von inaktiven Pro-Caspasen und ihren aktivierten, gespaltenen Formen ermöglicht.
Die Hauptvorteile dieses Protokolls sind 1) seine Fähigkeit, die Aktivierung mehrerer Caspasen parallel aus einer einzigen Population endogener Zellen zu beurteilen, um die PCD-Aktivierung genauer zu bestimmen, und 2) die Verwendung relativ einfacher Labortechniken, die keine umfangreiche Schulung oder teure Ausrüstung erfordern. Frühere Protokolle haben Western Blotting, Fluoreszenzreporter oder Antikörperfärbung verwendet, um die Caspase-Aktivierung in Kulturüberständen, Zell- und Gewebelysaten, ganzen Zellen durch Mikroskopie und in vivo 67,68,69,70,71 zu überwachen, aber diese Techniken überwachen im Allgemeinen nur ein oder zwei Caspasen in einer Probe. Während synthetische Peptidsubstrate, die Caspase-Spaltstellen enthalten, die bei der Spaltung fluoreszieren, zur Überwachung der Caspase-Aktivierung in Zell- oder Gewebelysaten69 verwendet wurden, können diese Substrate oft von mehr als einer Caspase gespalten werden, was es schwierig macht, die spezifische Aktivierung einzelner Caspasen in diesem System zu bestimmen. Darüber hinaus ermöglicht die Verwendung von Western Blot anstelle der Verwendung von fluoreszierenden Reportern oder anderen Tag-basierten Methoden den Forschern, endogene Zellen zu verwenden, anstatt spezifische Zelllinien mit Reportergenen zu erzeugen. Die Verwendung endogener Zellen hat mehrere Vorteile, einschließlich der Tatsache, dass viele immortalisierte Zelllinien einen Mangel an Schlüsselmolekülen für den Zelltodaufweisen 72,73, was die Ergebnisse beeinflussen könnte. Darüber hinaus ermöglicht die Verwendung endogener Zellen die Bewertung verschiedener Zelltypen wie Makrophagen, Epithelzellen und Endothelzellen anstelle einer einzelnen Linie. Western Blot ist auch eine relativ einfache und kostengünstige Technik, die in Labors auf der ganzen Welt durchgeführt werden kann, ohne dass große, teure Geräte oder komplizierte Setups erforderlich sind.
Dieses Protokoll ist in der gesamten Biologie weit verbreitet, um sowohl die zelltodabhängigen als auch die zelltodunabhängigen Funktionen von Caspasen zu verstehen, einschließlich ihrer Gerüstrollen und Funktionen in anderen entzündlichen Signalwegen74. Die Anwendung dieser Methode ermöglicht einen einheitlichen Ansatz bei der Untersuchung von PCD-Signalwegen des angeborenen Immunsystems und der Entzündungssignalisierung über Krankheiten und Zustände hinweg, und dieses Protokoll kann verwendet werden, um kritische regulatorische Prozesse und mechanistische Verbindungen zu identifizieren, die die Entwicklung zukünftiger therapeutischer Strategien beeinflussen werden.
Protokoll
Die Verwendung und die Verfahren der Tiere wurden vom St. Jude Children's Research Hospital Committee on the Use and Care of Animals genehmigt.
1. Vorbereitung der Lösungen
- Bereiten Sie L929-konditionierte Medien vor.
- Platte 1 × 106 L929-Zellen (siehe Materialtabelle) in einem 182cm2 Gewebekulturkolben mit 50 ml L929-Nährmedien (siehe Tabelle 1 zur Herstellung des Mediums).
- Züchten Sie die Zellen in einem befeuchteten Inkubator bei 37 °C mit 5% CO2.
- Nach 7 Tagen den Überstand auffangen und mit einem 0,45-μm-Filter filtrieren. Bereiten Sie 50 ml Aliquote vor (lagern Sie gefrorene Aliquote bis zu 1 Jahr bei −80 °C).
- Bereiten Sie 500 ml aus Knochenmark gewonnene Makrophagen (BMDM) -Nährmedien vor (Tabelle 1).
- Bereiten Sie 500 ml BMDM-Stimulationsmedien vor (Tabelle 1).
- Bereiten Sie 500 ml Medien für die Infektion vor (Tabelle 1).
- Bereiten Sie 100 ml 1 m Tris-Puffer vor (Tabelle 1).
- Bereiten Sie 4x Natriumdodecylsulfat (SDS)-Puffer vor (Tabelle 1).
- Bereiten Sie 1 ml 1 M 1,4-Dithiothreitol (DTT, Tabelle 1) vor.
- Bereiten Sie 40 ml Caspase-Lysepuffer vor (Tabelle 1).
- Bereiten Sie 100 ml 1,5 m Tris-Puffer vor (Tabelle 1).
- Bereiten Sie 100 ml einer 10%igen (Gew./vol) SDS-Lösung vor (Tabelle 1).
- Bereiten Sie 50 ml eines 2-fachen Radioimmunpräzipitations-Assays (RIPA)-Puffers vor (Tabelle 1).
- Bereiten Sie eine 5 mg/ml LPS-Lösung vor (Tabelle 1).
- Bereiten Sie eine 0,5 M ATP-Lösung vor (Tabelle 1).
- Bereiten Sie die Western-Blotting-Lösungen vor.
- Bereiten Sie 1 l 5x laufenden Pufferstock vor (Tabelle 1).
- Bereiten Sie 1 l 10x Transferpufferlager vor (Tabelle 1).
- Bereiten Sie 1 l Tris-gepufferte Kochsalzlösung mit Tween 20 vor (TBST, Tabelle 1).
- Bereiten Sie 100 ml einer 5%igen Magermilchblockierlösung vor (Tabelle 1).
- Bereiten Sie die primären Antikörperlösungen vor.
- Bereiten Sie 10 ml Caspase-1-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml Caspase-11-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml Caspase-3-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml Caspase-7-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml Caspase-8-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml Caspase-9-Primärantikörper vor (Tabelle 1).
- Bereiten Sie 10 ml HRP-konjugierten β-Aktin-Primärantikörper vor (Tabelle 1).
- Bereiten Sie die sekundären Antikörperlösungen vor.
- Bereiten Sie 10 ml sekundären Anti-Kaninchen-Antikörper vor (Tabelle 1).
- Bereiten Sie 10 ml sekundären Anti-Maus-Antikörper vor (Tabelle 1).
- Bereiten Sie 10 ml sekundären Anti-Ratten-Antikörper vor (Tabelle 1).
2. Isolierung von Makrophagen aus dem Knochenmark
HINWEIS: Für dieses Protokoll können 6-10 Wochen alte Wildtyp-Mäuse mit intakten PCD-Signalwegen oder mutierte Mäuse verwendet werden, bei denen die PCD-Regulatoren, Effektoren oder interessierenden Moleküle gelöscht oder verändert wurden.
- Euthanasieren Sie eine Maus in einer CO 2 -Kammer mit einer Durchflussrate, die 10% -30% des Käfigvolumens pro Minute für2-3 Minuten verdrängt. Führen Sie dann eine sekundäre Euthanasiemethode durch, z. B. eine Zervixluxation. Befolgen Sie gegebenenfalls alle zusätzlichen einrichtungs-, institutions- und regierungsspezifischen Richtlinien und Vorschriften.
- Sezieren Sie die Maus, um die Hinterbeinknochen zu erhalten.
- Stecken Sie die Maus auf den Rücken, so dass der Bauch freigelegt ist. Sprühen Sie mit 70% (vol/vol) Ethanol ein, um die Hinterbeine und den Bauch zu sterilisieren.
ACHTUNG: Ethanol ist brennbar. Halten Sie es von offenen Flammen fern. - Verwenden Sie eine Schere, um einen Schnitt an der Mittellinie des Bauches zu machen; Schneiden Sie weiter in Richtung der Beine, um die Oberschenkelknochen sichtbar zu machen.
- Nehmen Sie das rechte hintere Bein und ziehen Sie die Haut vom Körper weg in Richtung Mittellinie. Lösen Sie das Bein vom Körper, indem Sie die Adduktorenmuskeln in Richtung Mittellinie durchtrennen. Schneiden Sie dann das Bein zwischen dem Hüftgelenk und der Wirbelsäule ab. Als nächstes schneiden Sie die Pfote distal zum Knöchel ab und entfernen Sie überschüssiges Gewebe vom Knochen, indem Sie die Haut abziehen und das Wadengewebe mit einer leicht geöffneten Schere entfernen.
- Legen Sie das Bein auf ein mit 70% (vol/vol) Ethanol getränktes Handtuch und präzieren Sie das Schienbein und den Oberschenkelknochen.
- Fassen Sie die Tibia und den Femur jeweils mit einer separaten Pinzette. Drücken Sie das Schienbein sanft gegen die natürliche Richtung des Kniegelenks. Dies führt dazu, dass die Tibia am Knie bricht.
- Verwenden Sie bei Bedarf die Pinzette und die Sezierschere, um das verbleibende hängende Gewebe zu entfernen. Bewahren Sie das Schienbein für die spätere Verwendung auf, indem Sie es auf das mit Ethanol getränkte Handtuch legen.
- Sammeln Sie den Oberschenkelknochen auf die gleiche Weise, indem Sie das Knie abbrechen.
- Wiederholen Sie die Schritte 3 und 4 oben für das linke Bein, um es aus dem Körper zu entfernen und die Tibia und den Femur zu präparieren.
- Besprühen Sie die Knochen mit 70% (vol/vol) Ethanol.
- Reinigen Sie die Knochen, indem Sie sie auf ein sauberes, mit 70% (vol/vol) Ethanol getränktes Handtuch legen, den fleischigen Teil des Handtuchs zusammendrücken und das Handtuch gegen den Knochen reiben, um überschüssiges Gewebe zu entfernen.
- Stecken Sie die Maus auf den Rücken, so dass der Bauch freigelegt ist. Sprühen Sie mit 70% (vol/vol) Ethanol ein, um die Hinterbeine und den Bauch zu sterilisieren.
- Sobald beide Oberschenkelknochen und beide Schienbeine sauber sind, besprühen Sie alle vier Knochen mit 70% (vol/vol) Ethanol. Sammeln Sie die Knochen in einer sterilen Petrischale und spülen Sie sie mit 10 ml BMDM-Nährmedien ab, indem Sie das Medium vorsichtig in der Schale schwenken.
- Füllen Sie eine 10-ml-Spritze mit 10 ml frischem BMDM-Nährmedium und setzen Sie eine 25-g-Nadel ein.
- Nehmen Sie die Tibia mit einer Pinzette auf; Schneiden Sie dann das Sprunggelenk in einem Winkel von ~45° ab.
- Spülen Sie das Knochenmark von der Tibia.
- Halten Sie das Schienbein über ein 50-ml-Röhrchen, wobei das schmale Ende des Schienbeins nach unten zeigt. Geben Sie das Medium aus der gefüllten Spritze über das Schienbein ab.
- Führen Sie die Nadel (zunächst vorsichtig) am oberen Ende des Knochenmarks ein und geben Sie das Medium ab.
- Entfernen Sie die Nadel und führen Sie sie erneut ein. Verwenden Sie kurze Hochdruckstöße, um das Medium zu dosieren und die Nadel in das Mark zu bewegen.
- Sobald das Medium aus der Unterseite des Knochens herausfließt, verwenden Sie kurze, Hochdruckstöße, um die Zellen weiter auszuspülen. Überwachen Sie die Farbe des Knochens während dieses Vorgangs und entsorgen Sie ihn, wenn der Knochen weiß ist.
- Tun Sie dies für beide Tibias.
- Wiederholen Sie die Schritte 5 und 6 oben für die Oberschenkelknochen und schneiden Sie am Hüftgelenk, wie die Tibia am Sprunggelenk geschnitten wurde. Verwenden Sie das gleiche 50-ml-Röhrchen, um das BMDM-Nährmedium zu sammeln, während das Knochenmark gespült wird.
- Sobald alle vier Knochen gespült wurden, saugen Sie das Knochenmark und das Medium aus dem 50-ml-Röhrchen 3x durch eine 18-g-Nadel auf einer 10-ml-Spritze auf und ab und spülen Sie jedes Mal die Seiten des Röhrchens ab, um das Mark zu dispergieren.
- Stellen Sie das Endvolumen im 50-ml-Röhrchen mit BMDM-Nährmedien auf 30 ml ein und stellen Sie sicher, dass die Zellen gründlich suspendiert sind.
- Verwenden Sie ein 70-μm-Zellsieb, um die BMDM-Nährmedien aus dem 50-ml-Röhrchen zu filtern.
- Die resultierende Zellsuspension, die die Vorläuferzellen des Knochenmarks enthält, wird in drei 150-mm-Gewebekulturschalen gegeben, indem jeweils 10 ml (oder ~20 × 106 Zellen) hinzugefügt werden. Fügen Sie dann weitere 10 ml BMDM-Nährmedien zu jeder Schüssel hinzu. In einem befeuchteten Inkubator bei 37 °C inkubieren.
3. Differenzierung der BMDMs und Plattierung für die Experimente
- Die plattierten Knochenmark-Vorläuferzellen werden 3 Tage lang bei 37 °C inkubiert. Entfernen Sie dann jede Schale und fügen Sie weitere 5-8 ml BMDM-Nährmedien hinzu (Tabelle 1). Kehren Sie bei 37 °C in den Inkubator zurück.
- Entfernen Sie am 5. Tag nach dem ersten Anrichten jede Schale und fügen Sie weitere 5 ml BMDM-Nährmedien hinzu. Kehren Sie bei 37 °C in den Inkubator zurück.
- Entfernen Sie an Tag 6 jede Schüssel und entsorgen Sie die Medien. Fügen Sie dann 10 ml kaltes (bei 4 °C gelagertes) PBS hinzu, um es einmal zu waschen. Entsorgen Sie die 10 ml kalte PBS-Wäsche. Fügen Sie dann 10 ml frisches, kaltes PBS zu jedem Gericht hinzu und inkubieren Sie jedes Gericht 5 Minuten lang auf Eis.
- Kratzen Sie die Zellen aus allen drei Schalen vorsichtig mit einem Zellschaber in ein 50-ml-Röhrchen. Die Zellen werden bei 270 × g bei 4 °C vorsichtig 5 Minuten lang heruntergeschleudert; Dann den Überstand verwerfen.
- Fügen Sie 20 ml BMDM-Nährmedien hinzu, pipettieren Sie auf und ab, um das Pellet zu resuspendieren, und zählen Sie die Zellen.
HINWEIS: Es wird erwartet, dass jede Maus ungefähr 60 × 10 6-100 × 106 Zellen liefert. - Planen Sie das 12-Well-Plattenlayout für den gewünschten In-vitro-Zelltod -/Entzündungsstimulations-Assay. Planen Sie, 1 × 106 Zellen pro Vertiefung zu platten. Fügen Sie für jede geplante Stimulation mindestens drei biologische Replikate hinzu und platten Sie einen Satz von Vertiefungen, die in Caspase-Lysepuffer geerntet werden sollen, und einen zweiten Satz von Vertiefungen, die in RIPA-Puffer geerntet werden sollen.
- Platte 1 × 106 Zellen in 1 ml BMDM-Nährmedien pro Vertiefung in 12-Well-Platten. Kultivieren Sie über Nacht in einem befeuchteten Inkubator bei 37 °C, bevor Sie mit der Bewertung des Zelltods/der Entzündung fortfahren. Entfernen Sie nach der Inkubation über Nacht das Medium und geben Sie 1 ml warmes (37 °C) PBS in jede Vertiefung, um die Zellen zu waschen.
- Entfernen Sie die PBS-Wäsche und fügen Sie 500 μl BMDM-Stimulationsmedien mit Antibiotika (bei nicht-bakteriellen Stimulationen) oder BMDM-Stimulationsmedien ohne Antibiotika (bei bakteriellen Stimulationen) hinzu (Tabelle 1). Inkubieren Sie 2 Stunden lang, bevor Sie mit Schritt 4 für die In-vitro-Stimulation /Infektion fortfahren.
4. Stimulation oder Infektion der Zellen
VORSICHT: Die in diesem Protokoll enthaltenen Wirkstoffe sind potenziell pathogen und sollten mit den entsprechenden Vorsichtsmaßnahmen in einer Einrichtung der Biosicherheitsstufe 2 (BSL2) mit Genehmigung der zuständigen institutionellen und staatlichen Behörden gehandhabt werden.
- Stimulieren Sie die BMDMs, um den Zelltod mit dem interessierenden Auslöser zu aktivieren.
HINWEIS: Für die Zwecke dieses Protokolls gelten das Influenza-A-Virus (IAV), das Herpes-simplex-Virus 1 (HSV1), Francisella novicidaund LPS + ATP werden zur Veranschaulichung verwendet, es können jedoch auch andere Trigger verwendet werden.- Beispiel Stimulation 1: Infektion mit IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (konstruiert nach Hoffmann et al.75; der Virustiter für die Bestimmung der Infektionsvielfalt [MOI] wird durch einen Plaque-Assay in MDCK-Zellen berechnet):
- Berechnen Sie das Virusvolumen, das für die Infektion bei einer Multiplizität der Infektion (MOI) von 20 plaquebildenden Einheiten (PFU) benötigt wird, indem Sie Gleichung (1) und Gleichung (2) verwenden:

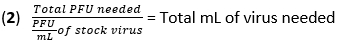

- Entfernen Sie das Medium aus den BMDMs und waschen Sie die Zellen einmal mit 500 μl PBS. Fügen Sie 450 μl IAV (20 MOI) in DMEM mit hohem Glukosegehalt ohne hitzeinaktiviertes (HI)-FBS zu jeder Vertiefung hinzu. Inkubieren Sie die Platten bei 37 °C für 1 h in einem befeuchteten Inkubator, um eine Absorption zu ermöglichen.
- Entfernen Sie die Platten und fügen Sie 50 μl HI-FBS hinzu. Legen Sie die Platten bei 37 °C wieder in den Inkubator. Inkubieren Sie insgesamt 12 Stunden.
- Berechnen Sie das Virusvolumen, das für die Infektion bei einer Multiplizität der Infektion (MOI) von 20 plaquebildenden Einheiten (PFU) benötigt wird, indem Sie Gleichung (1) und Gleichung (2) verwenden:
- Beispiel Stimulation 2: Infektion mit HSV1 (HF-Stamm; kultiviert wie zuvorbeschrieben 44; der Virustiter für die MOI-Bestimmung wird durch einen Plaque-Assay in Vero-Zellen berechnet):
- Berechnen Sie das Virusvolumen, das für die Infektion bei einem MOI von 10 PFU benötigt wird, indem Sie Gleichung (1) und Gleichung (2) aus Schritt 1 im obigen Abschnitt IAV-Infektion verwenden.

- Entfernen Sie die Medien aus den BMDMs. Geben Sie 450 μl HSV1 (MOI 10) in DMEM mit hohem Glukosegehalt ohne HI-FBS in jede Vertiefung. Inkubieren Sie die Platten bei 37 °C für 1 h in einem befeuchteten Inkubator, um eine Absorption zu ermöglichen.
- Entfernen Sie die Platten und fügen Sie 50 μl HI-FBS hinzu. Legen Sie die Platten bei 37 °C wieder in den Inkubator. Inkubieren Sie insgesamt 12 Stunden.
- Berechnen Sie das Virusvolumen, das für die Infektion bei einem MOI von 10 PFU benötigt wird, indem Sie Gleichung (1) und Gleichung (2) aus Schritt 1 im obigen Abschnitt IAV-Infektion verwenden.
- Beispiel Stimulation 3: Infektion mit F. novicida (U112-Stamm; kultiviert wie zuvorbeschrieben 44 unter aeroben Bedingungen bei 37 °C in BBL Trypticase Sojabrühe ergänzt mit 0,2% L-Cystein über Nacht. Anschließend werden die Bakterien im Verhältnis 1:10 bei 37 °C für weitere 4 h in frischem Medium subkultiviert, bevor die optische Dichte (OD) bei 600 nm unter Verwendung des frischen Mediums als Leerwert entnommen wird. Ein OD-Wert von 1 entspricht 1 × 109 koloniebildenden Einheiten (KBE) pro ml.)
- Berechnen Sie das Volumen von F. novicida , das für eine Infektion bei einem MOI von 50 KBE benötigt wird, indem Sie Gleichung (3) und Gleichung (4) verwenden:

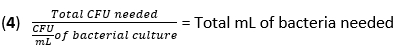
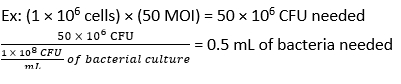
- Entfernen Sie das Medium aus den BMDMs. Geben Sie 500 μl F. novicida (MOI 50) in BMDM-Stimulationsmedien ohne Antibiotika in jede Vertiefung. Inkubieren Sie die Platten bei 37 °C für 4 h in einem befeuchteten Inkubator, um eine Absorption zu ermöglichen.
- Waschen Sie die Zellen dreimal mit erwärmtem (37 °C) PBS und fügen Sie 500 μl BMDM-Stimulationsmedien hinzu, die 50 μg/ml Gentamycin enthalten. Legen Sie die Platten bei 37 °C wieder in den Inkubator. Über Nacht inkubieren (16 h).
- Berechnen Sie das Volumen von F. novicida , das für eine Infektion bei einem MOI von 50 KBE benötigt wird, indem Sie Gleichung (3) und Gleichung (4) verwenden:
- Beispiel Stimulation 4: Stimulieren Sie mit LPS + ATP.
- Entfernen Sie das Medium aus den BMDMs. Geben Sie 500 μl BMDM-Stimulationsmedien mit Antibiotika (Tabelle 1) mit 100 ng/ml LPS in jede Vertiefung. Inkubieren Sie die Platten bei 37 °C für 3,5 h in einem befeuchteten Inkubator.
- 5 μl 0,5 M ATP-Stammlösung (Tabelle 1) in jede Vertiefung geben. Legen Sie die Platten bei 37 °C wieder in den Inkubator. 30 Minuten inkubieren.
- Beispiel Stimulation 1: Infektion mit IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (konstruiert nach Hoffmann et al.75; der Virustiter für die Bestimmung der Infektionsvielfalt [MOI] wird durch einen Plaque-Assay in MDCK-Zellen berechnet):
5. Sammeln des kombinierten Überstands und des Proteinlysats, das für Caspase-Western-Blots verwendet werden soll
- Entfernen Sie nach 4 h, 12 h oder 16 h Inkubation (der spezifische Zeitpunkt hängt vom verwendeten Auslöser ab) die Platte aus dem Inkubator.
- Entfernen Sie 150 μl des Überstandes; Verwerfen oder speichern Sie diese für andere überstehende Analysen (z. B. Enzyme-linked Immunosorbent Assay [ELISA]). Entfernen Sie nicht den restlichen Überstand.
- Stellen Sie die Proteinsammellösung her, indem Sie 50 μl Caspase-Lysepuffer + 100 μl 4x SDS-Puffer pro Well kombinieren (Tabelle 1). Geben Sie dann 150 μl der Mischung in jede Vertiefung.
- Pipettieren Sie die Mischung für jede Vertiefung auf und ab, um die lysierten Zellen und den Überstand zu sammeln. Kratzen Sie während des Pipettierens auch den Boden der Vertiefung mit der Pipettenspitze ab, um die Zellen aufzubrechen. Nach dem Schaben und Pipettieren wird das Proteinlysat in markierten 1,5-ml-Röhrchen gesammelt.
- Verwenden Sie einen Heizblock, um alle Rohre 12 Minuten lang auf 100 °C zu erhitzen.
- Pelletieren Sie alle unlöslichen Komponenten durch Zentrifugieren bei 14.500 × g für 30 s bei Raumtemperatur.
HINWEIS: Dies ist ein Pausenpunkt – das Protein aus dem kombinierten Überstand und den Proteinlysaten kann entweder sofort verwendet oder bis zu 2 Monate bei -20 °C oder bis zu 6 Monate bei -80 °C gelagert werden, bis es gebrauchsfertig ist.
6. Sammeln des Proteinlysats, das für Caspase-Western-Blots verwendet werden soll
- Entfernen Sie nach 4 h, 12 h oder 16 h Inkubation (der spezifische Zeitpunkt hängt vom verwendeten Auslöser ab) die Platte aus dem Inkubator. Entfernen Sie den gesamten Überstand; verwerfen oder für andere überstehende Analysen (z.B. ELISA) aufbewahren.
- Erzeugen Sie den 1x RIPA-Puffer, indem Sie die 2x RIPA-Stammlösung (Tabelle 1) in einem gleichen Volumen deionisiertem Wasser verdünnen. Fügen Sie dann eine Phosphatase-Inhibitor-Tablette und eine Proteasehemmer-Tablette hinzu und lassen Sie sie sich auflösen. Fügen Sie 150 μl 1x RIPA-Puffer und 50 μl 4x SDS zu jeder Vertiefung hinzu.
- Für jede Vertiefung pipettieren Sie die Mischung auf und ab, um die lysierten Zellen zu sammeln. Kratzen Sie während des Pipettierens auch den Boden der Vertiefung mit der Pipettenspitze ab, um die Zellen aufzubrechen. Nach dem Schaben und Pipettieren wird das Proteinlysat in markierten 1,5-ml-Röhrchen gesammelt.
- Verwenden Sie einen Heizblock, um alle Rohre 12 Minuten lang auf 100 °C zu erhitzen.
- Pelletieren Sie alle unlöslichen Komponenten durch Zentrifugieren bei 14.500 × g für 30 s bei Raumtemperatur.
HINWEIS: Dies ist ein Pausenpunkt – die Proteinlysate können entweder sofort verwendet oder bei -20 °C oder -80 °C gelagert werden, bis sie gebrauchsfertig sind.
7. Durchführung von Western Blot unter Verwendung der Lysate, die aus den BMDMs gemäß den obigen Schritten oder aus Gewebehomogenaten entnommen wurden
HINWEIS: Wenn Sie Gewebe verwenden, kann es von Hand oder durch einen kraftbetriebenen Gewebehomogenisator homogenisiert werden. Das Protokoll von Simpson76 bietet eine detaillierte Beschreibung der Gewebehomogenisierung.
- Bereiten Sie 1x Laufpuffer vor: Kombinieren Sie 200 ml des 5x laufenden Pufferlagers (Tabelle 1) und 800 ml deionisiertes Wasser. Machen Sie diesen 1x laufenden Puffer kurz vor jedem Experiment.
- Bereiten Sie das Elektrophoresegerät mit einem 12%igen (wt/vol) Polyacrylamid-Gel mit 10 Vertiefungen vor. Füllen Sie das Elektrophoresegerät mit 1x Laufpuffer. Entfernen Sie dann den Gelkamm.
HINWEIS: Um Caspase-1, Caspase-11, Caspase-3, Caspase-7, Caspase-8 und Caspase-9 für jede Probe zu analysieren, werden sechs Gele benötigt. - Planen Sie für Caspase-1-, Caspase-3-, Caspase-7- und Caspase-8-Blots 30 μl des kombinierten Überstands und des Proteinlysats in Caspase-Lysepuffer oder dem Gewebehomogenat. Für Caspase-11- und Caspase-9-Blots sollten 20 μl des Proteinlysats in RIPA-Puffer oder im Gewebehomogenat verwendet werden. Wenn die Proben bei −20 °C oder −80 °C gelagert wurden, tauen Sie sie zuerst auf Eis auf.
- Alle Proben werden 5 Minuten lang auf 100 °C erhitzt und vor dem Laden 30 s lang bei 4 °C bei 14.500 × g zentrifugiert. Laden Sie dann langsam 20 oder 30 μl der Probe in jede Spur. Vermeiden Sie, dass Proben in die anderen Fahrspuren überlaufen. Um alle sechs Caspasen gleichzeitig zu bewerten, verwenden Sie das gleiche Verfahren, um die entsprechenden Proben in jedes der sechs Gele zu laden.
- Schließen Sie das Elektrophoresegerät an die Stromquelle an. Stellen Sie dann die Leistung für 20 Minuten auf 80 V ein, um den Gellauf zu starten, und stellen Sie dann die Leistung für 45-60 Minuten auf 130 V ein.
- Beobachten Sie die Farbstofffront sorgfältig und schalten Sie das Gerät aus, wenn sich die Farbstofffront am unteren Rand des Gels befindet, aber noch nicht aus dem Gel herausgedrückt wurde.
- Während das Gel läuft, bereiten Sie 1x Transferpuffer vor, indem Sie 700 ml deionisiertes Wasser, 100 ml des 10-fachen Transferpufferlagers (Tabelle 1) und 200 ml Methanol kombinieren. Machen Sie die 1x-Lösung jedes Mal frisch.
HINWEIS: Seien Sie vorsichtig, da Methanol brennbar ist. Führen Sie die Vorbereitung des Transferpuffers nicht in der Nähe von offenen Flammen durch. - Entfernen Sie das Gel vorsichtig aus dem Elektrophoresegerät, indem Sie den Gelablöser verwenden.
- Richten Sie für jedes Gel einen Transferstapel ein.
- Aktivieren Sie eine PVDF-Membran, indem Sie sie 1 Minute lang in Methanol einweichen.
- Befeuchten Sie zwei Stücke Filterpapier, das Gel und die PVDF-Membran 5 Minuten lang im Transferpuffer. Bewahren Sie die PVDF-Membran und das Gel während dieser 5-minütigen Inkubation in getrennten Behältern auf.
- Montieren Sie den Transferstapel auf dem halbtrockenen System. Beginnen Sie auf der unteren Platinanodenseite und legen Sie ein Stück Filterpapier, die PVDF-Membran, das Gel und schließlich ein Stück Filterpapier ein. Rollen Sie die Luftblasen zwischen den Schichten vorsichtig aus oder drücken Sie sie heraus und schließen Sie die Oberseite des Systems. Stellen Sie sicher, dass die Sicherheitsabdeckung gesichert ist, bevor Sie fortfahren.
- Schließen Sie das Gerät an die Stromquelle an. Stellen Sie die Stromversorgung für 45 Minuten auf 25 V ein.
- Nachdem die Übertragung abgeschlossen ist, zerlegen Sie den Transferstapel und sammeln Sie die Membran. Legen Sie es in eine quadratische Petrischale (Inkubationsschale).
- Führen Sie eine Membranblockierung durch, indem Sie 15 ml einer 5%igen Magermilchlösung hinzufügen (Tabelle 1). Inkubieren Sie die Membran auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur für 1 h.
HINWEIS: Dies ist ein Pausenpunkt – die Membran kann entweder nach 1 h entfernt oder über Nacht in Blockierlösung bei 4 °C gelagert werden. - Entfernen Sie nach 1 Stunde oder Inkubation über Nacht die Blockierlösung. 10 ml der verdünnten Antikörperlösung (Anti-Caspase-1-Antikörper, Anti-Caspase-11-Antikörper, kombinierter Anti-Caspase-3- und Anti-gespaltener Caspase-3-Antikörper, kombinierter Anti-Caspase-7- und Anti-Cleaved-Caspase-7-Antikörper, kombinierter Anti-Caspase-8- und Anti-Cleaved-Caspase-8-Antikörper oder Anti-Caspase-9-Antikörper) (Tabelle 1). Bei 50 U/min bis 70 U/min auf einen Schaukelshaker legen, um bei Raumtemperatur 2 h oder bei 4 °C über Nacht (16 h) zu inkubieren.
- Sammeln Sie die Antikörperlösung (verwenden Sie sie bis zu 3x oder entsorgen Sie sie) und waschen Sie sie durch Zugabe von 15 ml TBST (Tabelle 1) zur Membran auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur für 10 Minuten. Entsorgen Sie die TBST.
- Wiederholen Sie den Waschgang mit 15 ml TBST nach Schritt 14 insgesamt 3x.
- 10 ml der verdünnten sekundären HRP-konjugierten Antikörperlösung (Anti-Kaninchen für Blots, die mit primären Antikörpern gegen Caspase-3, Caspase-7, Caspase-8 oder Caspase-9 gefärbt sind; Anti-Maus für Blots, die mit primärem Antikörper gegen Caspase-1 gefärbt sind; Anti-Ratte für Bplots, die mit primärem Antikörper gegen Caspase-11 gefärbt sind) (Tabelle 1). Auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur 1 h inkubieren.
- Entfernen Sie die Antikörperlösung und waschen Sie sie, indem Sie 15 ml TBST auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur für 10 Minuten auf die Membran geben. Entsorgen Sie die TBST.
- Wiederholen Sie den Waschgang mit 15 ml TBST nach Schritt 17 insgesamt 3x.
- Fügen Sie der Membran 10 ml des hochempfindlichen HRP-Substrats hinzu. 1 Minute bei Raumtemperatur im Dunkeln ruhen lassen.
- Entfernen Sie die Membran vom Substrat. Fahren Sie direkt mit der Bildgebung fort, indem Sie einen Chemilumineszenz-Imager verwenden, wobei das weiße Zubehörfach in der unteren Position eingesetzt ist. Belichten Sie die Membran im automatischen Belichtungsmodus (in der Regel ~1-2 min Belichtungszeit).
- Unter Verwendung der Membran aus dem Caspase-9- oder Caspase-11-Blotting (d. h. einer Membran mit RIPA-Lysatproben) 10 ml Stripping-Puffer hinzufügen und 5 Minuten lang auf einem Wippshaker bei 50 U/min bis 70 U/min bei Raumtemperatur inkubieren.
- Entsorgen Sie den Stripping-Puffer und waschen Sie ihn, indem Sie 15 ml TBST auf einem Wippshaker bei 50 U/min bis 70 U/min bei Raumtemperatur für 10 Minuten auf die Membran geben. Entsorgen Sie die TBST.
- Wiederholen Sie den Waschgang mit 15 ml TBST nach Schritt 22 insgesamt 3x.
- Führen Sie eine Membranblockierung durch, indem Sie 15 ml einer 5%igen Magermilchlösung hinzufügen. Inkubieren Sie die Membran auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur für 1 h.
HINWEIS: Dies ist ein Pausenpunkt – die Membran kann entweder nach 1 h entfernt oder über Nacht in Blockierlösung bei 4 °C gelagert werden. - Entfernen Sie nach 1 Stunde oder Inkubation über Nacht die Blockierlösung. Fügen Sie 10 ml der verdünnten Anti-β-Aktin-Antikörperlösung (HRP-konjugiert) hinzu. Bei 50 U/min bis 70 U/min auf einen Wippshaker legen, um 1,5 h bei Raumtemperatur zu inkubieren.
- Entfernen Sie die Antikörperlösung und waschen Sie sie, indem Sie 15 ml TBST auf einem Schaukelschüttler bei 50 U/min bis 70 U/min bei Raumtemperatur für 10 Minuten auf die Membran geben. Entsorgen Sie die TBST.
- Wiederholen Sie den Waschvorgang mit 15 ml TBST nach Schritt 26 insgesamt 3x.
- Fügen Sie der Membran 10 ml des HRP-Substrats mit Standardempfindlichkeit hinzu. 1 Minute bei Raumtemperatur im Dunkeln ruhen lassen.
- Fahren Sie direkt mit der Bildgebung fort, indem Sie einen Chemilumineszenz-Imager verwenden, wobei das weiße Zubehörfach in der unteren Position eingesetzt ist. Belichten Sie die Membran im automatischen Belichtungsmodus (in der Regel <1 Minute Belichtungszeit).
Ergebnisse
PANoptose wurde als Reaktion auf zahlreiche bakterielle, virale und Pilzinfektionen und andere entzündliche Reize sowie in Krebszellen beobachtet 44,48,49,50,51,52,53,54,56,57,58,60,61,62
Diskussion
Die Überwachung der Caspase-Spaltung und -Aktivierung liefert eines der umfassendsten Bilder der PCD-Aktivierung des angeborenen Immunsystems als Teil der angeborenen Immunantwort. Das hier beschriebene Protokoll demonstriert eine Strategie zur Überwachung der Caspase-Aktivierung als Reaktion auf IAV-, HSV1- und F. novicida-Infektionen und den sterilen Auslöser LPS + ATP, aber zahlreiche andere Stimuli können PCD induzieren und könnten in dieser Methode verwendet werden, wie in mehreren Veröffentlichungen ...
Offenlegungen
T.-D.K. ist Berater für Pfizer.
Danksagungen
Wir danken den Mitgliedern des Kanneganti-Labors für ihre Kommentare und Vorschläge, und wir danken J. Gullett, PhD, für die Unterstützung bei der wissenschaftlichen Redaktion. Die Arbeit in unserem Labor wird durch die Zuschüsse AI101935, AI124346, AI160179, AR056296 und CA253095 (an T.-D.K.) der National Institutes of Health (NIH) sowie durch die American Libanese Syrian Associated Charities (an T.-D.K.) unterstützt. Der Inhalt liegt ausschließlich in der Verantwortung der Autoren und spiegelt nicht unbedingt die offiziellen Ansichten der National Institutes of Health wider.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Millipore | SCHVU05RE | |
| 10 mL syringe | BD Biosciences | 309604 | |
| 12% polyacrylamide gel with 10 wells | Bio-Rad | 4561043 | |
| 12-well plate | Corning | 07-200-82 | |
| 18 G needle | BD Biosciences | 305195 | |
| 25 G needle | BD Biosciences | 305122 | |
| 50 mL tube | Fisher Scientific | 50-809-218 | |
| 70 μm cell strainer | Corning | 431751 | |
| 150 mm tissue culture dishes | Corning | 430597 | |
| 182-cm2 tissue culture flask | Genesee Scientific | 25-211 | |
| Accessory white trans tray | Cytiva | 29-0834-18 | |
| Anti–caspase-1 antibody | AdipoGen | AG-20B-0042-C100 | |
| Anti–caspase-11 antibody | Novus Biologicals | NB120-10454 | |
| Anti–caspase-3 antibody | Cell Signaling Technology | 9662 | |
| Anti–caspase-7 antibody | Cell Signaling Technology | 9492 | |
| Anti–caspase-8 antibody | Cell Signaling Technology | 4927 | |
| Anti–caspase-9 antibody | Cell Signaling Technology | 9504 | |
| Anti–cleaved caspase-3 antibody | Cell Signaling Technology | 9661 | |
| Anti–cleaved caspase-7 antibody | Cell Signaling Technology | 9491 | |
| Anti–cleaved caspase-8 antibody | Cell Signaling Technology | 8592 | |
| Anti-mouse HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 315-035-047 | |
| Anti-rabbit HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 111-035-047 | |
| Anti-rat HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 112-035-003 | |
| Anti–β-Actin antibody (C4) HRP | Santa Cruz | sc-47778 HRP | |
| ATP | InvivoGen | tlrl-atpl | |
| BBL Trypticase Soy Broth | BD Biosciences | 211768 | |
| Bead bath | Chemglass Life Sciences | CLS-4598-009 | |
| Biophotometer D30 | Eppendorf | 6133000010 | |
| BME | Sigma | M6250 | |
| Bromophenol blue | Sigma | BO126 | |
| Cell scrapers | CellTreat Scientific Products | 229315 | |
| Chemiluminescence imager (Amersham 600) | Cytiva | 29083461 | |
| CO2 chamber | VetEquip | 901703 | |
| Cuvettes | Fisher Scientific | 14-955-129 | |
| Dissecting scissors | Thermo Fisher Scientific | 221S | |
| DMEM | Thermo Fisher Scientific | 11995-073 | |
| DTT | Sigma | 43815 | |
| Eelectrophoresis apparatus | Bio-Rad | 1658004 | |
| Ethanol | Pharmco | 111000200 | |
| Fetal bovine serum | Biowest | S1620 | |
| Filter paper | Bio-Rad | 1703965 | |
| Forceps | Fisher Scientific | 22-327379 | |
| Francisella novicida (U112 strain) | BEI Resources | NR-13 | |
| Gel releaser | Bio-Rad | 1653320 | |
| Gentamycin | Gibco | 15750060 | |
| Glycerol | Sigma | G7893 | |
| Glycine | Sigma | G8898 | |
| HCl | Sigma | H9892 | |
| Heat block | Fisher Scientific | 23-043-160 | |
| Herpes simplex virus 1 (HF strain) | ATCC | VR-260 | |
| High glucose DMEM | Sigma | D6171 | |
| Human anti–caspase-1 antibody | R&D Systems | MAB6215 | |
| Human anti–caspase-8 antibody | Enzo | ALX-804-242 | |
| Humidified incubator | Thermo Fisher Scientific | 51026282 | |
| Image analysis software | ImageJ | v1.53a | |
| IMDM | Thermo Fisher Scientific | 12440-053 | |
| Influenza A virus (A/Puerto Rico/8/34, H1N1 [PR8]) | constructed per Hoffmann et al. | ||
| L929 cells | ATCC | CCL-1 | cell line for creating L929-conditioned media |
| L-cysteine | Thermo Fisher Scientific | BP376-100 | |
| Luminata Forte Western HRP substrate | Millipore | WBLUF0500 | standard-sensitivity HRP substrate |
| MDCK cells | ATCC | CCL-34 | cell line for determining IAV viral titer |
| Methanol | Sigma | 322415 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002401 | |
| Non-essential amino acids | Gibco | 11140050 | |
| Nonfat dried milk powder | Kroger | ||
| NP-40 solution | Sigma | 492016 | |
| PBS | Thermo Fisher Scientific | 10010023 | |
| Penicillin and streptomycin | Sigma | P4333 | |
| Petri dish | Fisher Scientific | 07-202-011 | |
| PhosSTOP | Roche | PHOSS-RO | |
| Power source | Bio-Rad | 164-5052 | |
| Protease inhibitor tablet | Sigma | S8820 | |
| PVDF membrane | Millipore | IPVH00010 | |
| Rocking shaker | Labnet | S2035-E | |
| SDS | Sigma | L3771 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium deoxycholate | Sigma | 30970 | |
| Sodium hydroxide | Sigma | 72068 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Square Petri dish | Fisher Scientific | FB0875711A | |
| Stripping buffer | Thermo Fisher Scientific | 21059 | |
| Super Signal Femto HRP substrate | Thermo Fisher Scientific | 34580 | high-sensitivity HRP substrate |
| Tabletop centrifuge | Thermo Fisher Scientific | 75004524 | |
| Trans-Blot semi-dry system | Bio-Rad | 170-3940 | |
| Tris | Sigma | TRIS-RO | |
| Tween 20 | Sigma | P1379 | |
| Ultrapure lipopolysaccharide (LPS) from E. coli 0111:B4 | InvivoGen | tlrl-3pelps | |
| Vero cells | ATCC | CCL-81 | cell line for determining HSV1 viral titer |
Referenzen
- Alnemri, E. S., et al. Human ICE/CED-3 protease nomenclature. Cell. 87 (2), 171 (1996).
- Man, S. M., Kanneganti, T. D. Converging roles of caspases in inflammasome activation, cell death and innate immunity. Nature Reviews Immunology. 16 (1), 7-21 (2016).
- Gullett, J. M., Tweedell, R. E., Kanneganti, T. D. It's all in the PAN: Crosstalk, plasticity, redundancies, switches, and interconnectedness encompassed by PANoptosis underlying the totality of cell death-associated biological effects. Cells. 11 (9), 1495 (2022).
- Pandian, N., Kanneganti, T. D. PANoptosis: A unique innate immune inflammatory cell death modality. Journal of Immunology. 209 (9), 1625-1633 (2022).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death & Differentiation. 25 (3), 486-541 (2018).
- Shi, Y. Caspase activation: Revisiting the induced proximity model. Cell. 117 (7), 855-858 (2004).
- Galluzzi, L., Lopez-Soto, A., Kumar, S., Kroemer, G. Caspases connect cell-death signaling to organismal homeostasis. Immunity. 44 (2), 221-231 (2016).
- Fernandes-Alnemri, T., Litwack, G., Alnemri, E. S. CPP32, a novel human apoptotic protein with homology to Caenorhabditis elegans cell death protein Ced-3 and mammalian interleukin-1 beta-converting enzyme. Journal of Biological Chemistry. 269 (49), 30761-30764 (1994).
- Tewari, M., et al. Yama/CPP32 beta, a mammalian homolog of CED-3, is a CrmA-inhibitable protease that cleaves the death substrate poly(ADP-ribose) polymerase. Cell. 81 (5), 801-809 (1995).
- Nicholson, D. W., et al. Identification and inhibition of the ICE/CED-3 protease necessary for mammalian apoptosis. Nature. 376 (6535), 37-43 (1995).
- Stennicke, H. R., et al. Pro-caspase-3 is a major physiologic target of caspase-8. Journal of Biological Chemistry. 273 (42), 27084-27090 (1998).
- Twiddy, D., Cohen, G. M., Macfarlane, M., Cain, K. Caspase-7 is directly activated by the approximately 700-kDa apoptosome complex and is released as a stable XIAP-caspase-7 approximately 200-kDa complex. Journal of Biological Chemistry. 281 (7), 3876-3888 (2006).
- Kesavardhana, S., Malireddi, R. K. S., Kanneganti, T. D. Caspases in cell death, inflammation, and pyroptosis. Annual Reviews of Immunology. 38, 567-595 (2020).
- Kerr, J. F., Wyllie, A. H., Currie, A. R. Apoptosis: a basic biological phenomenon with wide-ranging implications in tissue kinetics. British Journal of Cancer. 26 (4), 239-257 (1972).
- Taylor, R. C., Cullen, S. P., Martin, S. J. Apoptosis: Controlled demolition at the cellular level. Nature Reviews Molecular Cell Biology. 9 (3), 231-241 (2008).
- Morioka, S., Maueröder, C., Ravichandran, K. S. Living on the edge: Efferocytosis at the interface of homeostasis and pathology. Immunity. 50 (5), 1149-1162 (2019).
- Wang, Y., et al. Chemotherapy drugs induce pyroptosis through caspase-3 cleavage of a gasdermin. Nature. 547 (7661), 99-103 (2017).
- Rogers, C., et al. Cleavage of DFNA5 by caspase-3 during apoptosis mediates progression to secondary necrotic/pyroptotic cell death. Nature Communications. 8, 14128 (2017).
- Kayagaki, N., et al. Caspase-11 cleaves gasdermin D for non-canonical inflammasome signalling. Nature. 526 (7575), 666-671 (2015).
- Shi, J., Gao, W., Shao, F. Pyroptosis: Gasdermin-mediated programmed necrotic cell death. Trends in Biochemical Sciences. 42 (4), 245-254 (2017).
- Sborgi, L., et al. GSDMD membrane pore formation constitutes the mechanism of pyroptotic cell death. EMBO Journal. 35 (16), 1766-1778 (2016).
- Aglietti, R. A., et al. GsdmD p30 elicited by caspase-11 during pyroptosis forms pores in membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (28), 7858-7863 (2016).
- Hagar, J. A., Powell, D. A., Aachoui, Y., Ernst, R. K., Miao, E. A. Cytoplasmic LPS activates caspase-11: implications in TLR4-independent endotoxic shock. Science. 341 (6151), 1250-1253 (2013).
- Kayagaki, N., et al. Noncanonical inflammasome activation by intracellular LPS independent of TLR4. Science. 341 (6151), 1246-1249 (2013).
- Shi, J., et al. Inflammatory caspases are innate immune receptors for intracellular LPS. Nature. 514 (7521), 187-192 (2014).
- Lamkanfi, M., et al. Targeted peptidecentric proteomics reveals caspase-7 as a substrate of the caspase-1 inflammasomes. Molecular & Cellular Proteomics. 7 (12), 2350-2363 (2008).
- Kalai, M., et al. Tipping the balance between necrosis and apoptosis in human and murine cells treated with interferon and dsRNA. Cell Death & Differentiation. 9 (9), 981-994 (2002).
- Li, C., et al. Development of atopic dermatitis-like skin disease from the chronic loss of epidermal caspase-8. Proceedings of the National Academy of Sciences of the United States of America. 107 (51), 22249-22254 (2010).
- Kovalenko, A., et al. Caspase-8 deficiency in epidermal keratinocytes triggers an inflammatory skin disease. Journal of Experimental Medicine. 206 (10), 2161-2177 (2009).
- Kang, T. B., et al. Caspase-8 serves both apoptotic and nonapoptotic roles. Journal of Immunology. 173 (5), 2976-2984 (2004).
- Oberst, A., et al. Catalytic activity of the caspase-8-FLIP(L) complex inhibits RIPK3-dependent necrosis. Nature. 471 (7338), 363-367 (2011).
- Kaiser, W. J., et al. RIP3 mediates the embryonic lethality of caspase-8-deficient mice. Nature. 471 (7338), 368-372 (2011).
- Zhang, H., et al. Functional complementation between FADD and RIP1 in embryos and lymphocytes. Nature. 471 (7338), 373-376 (2011).
- Shi, J., et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature. 526 (7575), 660-665 (2015).
- Hitomi, J., et al. Identification of a molecular signaling network that regulates a cellular necrotic cell death pathway. Cell. 135 (7), 1311-1323 (2008).
- Malireddi, R. K., Ippagunta, S., Lamkanfi, M., Kanneganti, T. D. Cutting edge: Proteolytic inactivation of poly(ADP-ribose) polymerase 1 by the Nlrp3 and Nlrc4 inflammasomes. Journal of Immunology. 185 (6), 3127-3130 (2010).
- Tsuchiya, K., et al. Caspase-1 initiates apoptosis in the absence of gasdermin D. Nature Communications. 10 (1), (2019).
- Taabazuing, C. Y., Okondo, M. C., Bachovchin, D. A. Pyroptosis and apoptosis pathways engage in bidirectional crosstalk in monocytes and macrophages. Cell Chemical Biology. 24 (4), 507-514 (2017).
- Gurung, P., et al. FADD and caspase-8 mediate priming and activation of the canonical and noncanonical Nlrp3 inflammasomes. Journal of Immunology. 192 (4), 1835-1846 (2014).
- Man, S. M., et al. Inflammasome activation causes dual recruitment of NLRC4 and NLRP3 to the same macromolecular complex. Proceedings of the National Academy of Sciences of the United States of America. 111 (20), 7403-7408 (2014).
- Man, S. M., et al. Salmonella infection induces recruitment of Caspase-8 to the inflammasome to modulate IL-1beta production. Journal of Immunology. 191 (10), 5239-5246 (2013).
- Van Opdenbosch, N., et al. Caspase-1 engagement and TLR-induced c-FLIP expression suppress ASC/caspase-8-dependent apoptosis by inflammasome sensors NLRP1b and NLRC4. Cell Reports. 21 (12), 3427-3444 (2017).
- Pierini, R., et al. AIM2/ASC triggers caspase-8-dependent apoptosis in Francisella-infected caspase-1-deficient macrophages. Cell Death & Differentiation. 19 (10), 1709-1721 (2012).
- Lee, S., et al. AIM2 forms a complex with pyrin and ZBP1 to drive PANoptosis and host defence. Nature. 597 (7876), 415-419 (2021).
- Sagulenko, V., et al. AIM2 and NLRP3 inflammasomes activate both apoptotic and pyroptotic death pathways via ASC. Cell Death & Differentiation. 20 (9), 1149-1160 (2013).
- Lukens, J. R., et al. Dietary modulation of the microbiome affects autoinflammatory disease. Nature. 516 (7530), 246-249 (2014).
- Gurung, P., Burton, A., Kanneganti, T. D. NLRP3 inflammasome plays a redundant role with caspase 8 to promote IL-1beta-mediated osteomyelitis. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4452-4457 (2016).
- Kuriakose, T., et al. ZBP1/DAI is an innate sensor of influenza virus triggering the NLRP3 inflammasome and programmed cell death pathways. Science Immunology. 1 (2), (2016).
- Malireddi, R. K. S., et al. TAK1 restricts spontaneous NLRP3 activation and cell death to control myeloid proliferation. Journal of Experimental Medicine. 215 (4), 1023-1034 (2018).
- Malireddi, R. K. S., et al. Innate immune priming in the absence of TAK1 drives RIPK1 kinase activity-independent pyroptosis, apoptosis, necroptosis, and inflammatory disease. Journal of Experimental Medicine. 217 (3), (2020).
- Christgen, S., et al. Identification of the PANoptosome: A molecular platform triggering pyroptosis, apoptosis, and necroptosis (PANoptosis). Frontiers in Cellular and Infection Microbiology. 10, 237 (2020).
- Malireddi, R. K. S., et al. RIPK1 distinctly regulates Yersinia-induced inflammatory cell death, PANoptosis. Immunohorizons. 4 (12), 789-796 (2020).
- Zheng, M., Karki, R., Vogel, P., Kanneganti, T. D. Caspase-6 is a key regulator of innate immunity, inflammasome activation, and host defense. Cell. 181 (3), 674-687 (2020).
- Karki, R., et al. ADAR1 restricts ZBP1-mediated immune response and PANoptosis to promote tumorigenesis. Cell Reports. 37 (3), 109858 (2021).
- Wang, Y., et al. Single cell analysis of PANoptosome cell death complexes through an expansion microscopy method. Cellular and Molecular Life Sciences. 79 (10), 531 (2022).
- Malireddi, R. K. S., et al. Inflammatory cell death, PANoptosis, mediated by cytokines in diverse cancer lineages inhibits tumor growth. Immunohorizons. 5 (7), 568-580 (2021).
- Kesavardhana, S., et al. The Zα2 domain of ZBP1 is a molecular switch regulating influenza-induced PANoptosis and perinatal lethality during development. Journal of Biological Chemistry. 295 (24), 8325-8330 (2020).
- Banoth, B., et al. ZBP1 promotes fungi-induced inflammasome activation and pyroptosis, apoptosis, and necroptosis (PANoptosis). Journal of Biological Chemistry. 295 (52), 18276-18283 (2020).
- Karki, R., et al. Interferon regulatory factor 1 regulates PANoptosis to prevent colorectal cancer. JCI Insight. 5 (12), 136720 (2020).
- Zheng, M., et al. Impaired NLRP3 inflammasome activation/pyroptosis leads to robust inflammatory cell death via caspase-8/RIPK3 during coronavirus infection. Journal of Biological Chemistry. 295 (41), 14040-14052 (2020).
- Karki, R., et al. Synergism of TNF-α and IFN-γ triggers inflammatory cell death, tissue damage, and mortality in SARS-CoV-2 infection and cytokine shock syndromes. Cell. 184 (1), 149-168 (2021).
- Karki, R., et al. ZBP1-dependent inflammatory cell death, PANoptosis, and cytokine storm disrupt IFN therapeutic efficacy during coronavirus infection. Science Immunology. 7 (74), (2022).
- Jiang, W., Deng, Z., Dai, X., Zhao, W. PANoptosis: A new insight into oral infectious diseases. Frontiers in Immunology. 12, 789610 (2021).
- Chi, D., et al. Real-time induction of macrophage apoptosis, pyroptosis, and necroptosis by Enterococcus faecalis OG1RF and two root canal isolated strains. Frontiers in Cellular and Infection Microbiology. 11, 720147 (2021).
- Lin, J. F., et al. Phosphorylated NFS1 weakens oxaliplatin-based chemosensitivity of colorectal cancer by preventing PANoptosis. Signal Transduction and Targeted Therapy. 7 (1), 54 (2022).
- Song, M., et al. Self-assembled polymeric nanocarrier-mediated co-delivery of metformin and doxorubicin for melanoma therapy. Drug Delivery. 28 (1), 594-606 (2021).
- Boucher, D., Chan, A., Ross, C., Schroder, K. Quantifying caspase-1 activity in murine macrophages. Methods in Molecular Biology. 1725, 163-176 (2018).
- Boucher, D., Duclos, C., Denault, J. B. General in vitro caspase assay procedures. Methods in Molecular Biology. 1133, 3-39 (2014).
- Kaushal, V., Herzog, C., Haun, R. S., Kaushal, G. P. Caspase protocols in mice. Methods in Molecular Biology. 1133, 141-154 (2014).
- Swacha, P., Gekara, N. O., Erttmann, S. F. Biochemical and microscopic analysis of inflammasome complex formation. Methods in Enzymology. 625, 287-298 (2019).
- Talley, S., et al. A caspase-1 biosensor to monitor the progression of inflammation in vivo. Journal of Immunology. 203 (9), 2497-2507 (2019).
- Pelegrin, P., Barroso-Gutierrez, C., Surprenant, A. P2X7 receptor differentially couples to distinct release pathways for IL-1beta in mouse macrophage. Journal of Immunology. 180 (11), 7147-7157 (2008).
- Yu, J. W., et al. Cryopyrin and pyrin activate caspase-1, but not NF-kappaB, via ASC oligomerization. Cell Death & Differentiation. 13 (2), 236-249 (2006).
- Henry, C. M., Martin, S. J. Caspase-8 acts in a non-enzymatic role as a scaffold for assembly of a pro-inflammatory "FADDosome" complex upon TRAIL stimulation. Molecular Cell. 65 (4), 715-729 (2017).
- Hoffmann, E., Neumann, G., Kawaoka, Y., Hobom, G., Webster, R. G. A DNA transfection system for generation of influenza A virus from eight plasmids. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 6108-6113 (2000).
- Simpson, R. J. Homogenization of mammalian tissue. Cold Spring Harbor Protocols. (7), (2010).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten