É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Avaliação da ativação da caspase para avaliar a morte de células imunes inatas
Neste Artigo
Resumo
Este protocolo descreve um método abrangente para avaliar a ativação da caspase (caspase-1, caspase-3, caspase-7, caspase-8, caspase-9 e caspase-11) em resposta a modelos in vitro e in vivo (em camundongos) de infecção, insultos estéreis e câncer para determinar o início de vias de morte celular, como piroptose, apoptose, necroptose e PANoptose.
Resumo
A imunidade inata fornece a primeira linha crítica de defesa em resposta a patógenos e insultos estéreis. Um componente mecanicista chave dessa resposta é o início da morte celular imune programada inata (PCD) para eliminar células infectadas ou danificadas e propagar respostas imunes. No entanto, o excesso de PCD está associado à inflamação e patologia. Portanto, entender a ativação e a regulação da PCD é um aspecto central da caracterização das respostas imunes inatas e da identificação de novos alvos terapêuticos em todo o espectro da doença.
Este protocolo fornece métodos para caracterizar a ativação inata da PCD imune monitorando caspases, uma família de proteases dependentes de cisteína que são frequentemente associadas a diversas vias da PCD, incluindo apoptose, pirotose, necroptose e PANoptose. Os relatos iniciais caracterizaram caspase-2, caspase-8, caspase-9 e caspase-10 como caspases iniciadoras e caspase-3, caspase-6 e caspase-7 como caspases efetoras na apoptose, enquanto estudos posteriores encontraram as caspases inflamatórias, caspase-1, caspase-4, caspase-5 e caspase-11, impulsionam a piroptose. Sabe-se agora que há uma extensa conversa cruzada entre as caspases e outras moléculas inatas imunes e de morte celular através das vias PCD previamente definidas, identificando uma lacuna de conhecimento fundamental na compreensão mecanicista da imunidade inata e PCD e levando à caracterização da PANoptosis. A PANoptose é uma via inata de PCD inflamatória imune única regulada por complexos PANoptosome, que integram componentes, incluindo caspases, de outras vias de morte celular.
Aqui, métodos para avaliar a ativação de caspases em resposta a vários estímulos são fornecidos. Esses métodos permitem a caracterização das vias de PCD tanto in vitro quanto in vivo, uma vez que as caspases ativadas sofrem clivagem proteolítica que pode ser visualizada por western blotting usando anticorpos ideais e condições de blotting. Foi estabelecido um protocolo e um fluxo de trabalho de western blotting que permitem a avaliação da ativação de múltiplas caspases de uma mesma população celular, proporcionando uma caracterização abrangente dos processos de PCD. Este método pode ser aplicado em todas as áreas de pesquisa em desenvolvimento, homeostase, infecção, inflamação e câncer para avaliar as vias PCD em todos os processos celulares na saúde e na doença.
Introdução
O sistema imunológico inato atua como a primeira linha de defesa durante a infecção e em resposta a estímulos estéreis, como lesão tecidual e alterações na homeostase. Sensores imunes inatos na superfície celular e no citoplasma respondem a padrões moleculares associados a patógenos ou danos (PAMPs ou DAMPs, respectivamente) para desencadear vias de sinalização inflamatória e respostas celulares. Um dos principais processos da resposta imune inata é a indução da morte celular para remover células infectadas ou danificadas e impulsionar ainda mais respostas imunes inatas e adaptativas. A morte celular programada (PCD) é um processo altamente conservado em todas as espécies, destacando sua importância evolutiva como um mecanismo imunológico inato.
Existem várias vias inatas de PCD imunes que podem ser ativadas em todos os tipos de células. As caspases são uma família-chave de proteases altamente conservadas, intracelulares e dependentes de cisteína que são críticas em muitas vias de PCD, incluindo a via de apoptose tradicionalmente não inflamatória, bem como vias inflamatórias de PCD, como piroptose, necroptose e PANoptose 1,2,3,4,5 . Existem 11 caspases humanas e 10 murinas que estão bem definidas, bem como pseudocaspases que podem ser funcionais, e a maioria é expressa constitutivamente como pró-caspases monoméricas ou diméricas inativas que requerem clivagem para ativação 6,7. As caspases também contêm domínios importantes para o recrutamento e formação de complexos multiproteicos. Estes incluem o domínio de ativação e recrutamento da caspase (CARD), que pode ser encontrado na caspase-1, caspase-2, caspase-4, caspase-5, caspase-9 e caspase-11, ou o domínio efetor da morte (DED), que é encontrado na caspase-8 e caspase-10. Através de sua atividade proteolítica e sua capacidade de formar complexos multiproteicos, as caspases são fatores críticos da PCD imune inata.
O papel das caspases na PCD imune inata foi identificado pela primeira vez na apoptose, onde as caspases iniciadoras, caspase-2, caspase-8, caspase-9 e caspase-10, ativam as caspases carrasco, caspase-3, caspase-6 e caspase-7, para conduzir a morte celular 8,9,10,11,12. As caspases do iniciador podem ser ativadas por diversas cascatas de sinalização; a via extrínseca ativa a caspase-8 através da sinalização do receptor de morte induzida por ligantes extracelulares, e a via intrínseca ativa a caspase-9 através da ruptura da integridade mitocondrial13. As caspases iniciadoras ativadas clivam o ligador separando as grandes e pequenas subunidades catalíticas das caspases do carrasco para produzir suas formas ativas. As caspases do carrasco então clivam seus substratos para desmontar a célula, resultando em degradação do DNA, blebbing da membrana, fragmentação nuclear e liberação de corpos apoptóticos14,15. Esse processo geralmente termina em uma forma não lítica e não inflamatória de morte celular quando acoplado à depuração imediata das células moribundas por eferocitose16. No entanto, defeitos na eferocitose ou falta de células fagocíticas podem levar ao acúmulo de células apoptóticas, que então sofrem morte celular lítica e inflamatória17,18.
As caspases inflamatórias, incluindo caspase-1 (humana e rato), caspase-4 e caspase-5 (humana) e caspase-11 (rato), foram descobertas para serem ativadas durante uma forma de PCD IMUNE INATA INFLAMATÓRIA (III-PCD) chamada piroptose. A ativação da caspase-1 está associada à formação de inflamassomas, que são complexos multiproteicos contendo um sensor imune inato citosólico, uma molécula adaptadora (proteína speck-like associada à apoptose contendo um CARD [ASC]) e caspase-1. A formação desse complexo permite que a caspase-1 sofra autoproteólise mediada por proximidade para liberar sua forma ativa, que pode clivar substratos-alvo, incluindo as citocinas pró-inflamatórias interleucina (IL)-1β e IL-18 e a molécula formadora de poros gasdermina D (GSDMD)19,20,21,22,23 . A casspase-11, a caspase-4 e a caspase-5 também podem ativar o GSDMD sem a formação a montante do inflamassoma após a detecção de PAMPs como o lipopolissacarídeo (LPS)19,20. Essas caspases sofrem dimerização seguida de oligomerização e autoclivagem para ativação após a ligação ao LPS citosólico, o que leva à ativação do inflamassoma não canônico 24,25,26 e ativação da caspase-1 de forma intrínseca à célula para induzir a maturação da IL-1β e IL-18 20. A maturação e a liberação dessas citocinas pró-inflamatórias caracterizam essas caspases como "inflamatórias". Além disso, descobriu-se que a caspase-8 apoptótica localiza-se no inflamassoma, fornecendo uma ligação entre os processos apoptóticos e piropóticos. Estudos descobriram que a caspase-8 apoptótica também é crítica para regular outra forma de PCD chamada necroptose. A perda de caspase-8 resulta na ativação espontânea de serina-treonina quinase 3 (RIPK3) mediada por uma pseudoquinase de linhagem mista mediada por receptores (MLKL) para conduzir a via III-PCD da necroptose 27,28,29,30,31,32,33,34,35.
Embora as caspases tenham sido historicamente classificadas como "apoptóticas" ou "inflamatórias" com base no tipo de morte celular que iniciam, evidências crescentes sugerem que há uma extensa conversa cruzada entre as vias inatas da PCD imune através das caspases 3,4. Por exemplo, a caspase-1 inflamatória dos inflamassomos cliva a caspase-7 apoptótica em seu local de ativação canônica34. A ativação da caspase-1 também pode levar à clivagem de substratos apoptóticos, como a poli(ADP-ribose) polimerase 1 (PARP1)36. Em células sem GSDMD, a caspase-1 também pode clivar a caspase-337,38. Além disso, a caspase-3 canonicamente apoptótica pode clivar a gasdermina E (GSDME) para induzir a PCD17,18 e também processa a GSDMD em uma forma inativa40. Além disso, o recrutamento da caspase-8 para o complexo inflamassoma tem sido observado 39,40,41,42,43,44,45, e a caspase-8 é um regulador chave da ativação do inflamassoma canônico e não canônico 39. Há também papéis sobrepostos e redundantes para a caspase-8 e a caspase-1 em muitas condições inflamatórias, e a PCD imune inata caracterizada pela ativação de componentes piropóticos, apoptóticos e necroptóticos ocorre em todo o espectro da doença 39,46,47,48,49,50.
Com base nesse crosstalk entre caspases inflamatórias e apoptóticas, uma lacuna fundamental na compreensão mecanicista da imunidade inata e PCD foi identificada, levando à descoberta da PANoptosis. A PANoptose é uma forma única de III-PCD que é ativada em resposta a patógenos, PAMPs, DAMPs e alterações na homeostase e é regulada por PANoptossomos - complexos macromoleculares multifacetados que integram componentes de outras vias de morte celular 44,50,51,52,53,54,55 . A totalidade dos efeitos biológicos na PANoptose não pode ser individualmente explicada por piroptose, apoptose ou necroptose isoladamente 3,4,35,36,39,46,47,48, uma vez que a PANoptose é caracterizada pela ativação de múltiplas caspases, incluindo caspase-1, caspase-11, caspase-8, caspase-9, caspase-3 e/ou caspase-7, dependendo do contexto 44,48,49,50,51,52,53,54,56,57,58,59,60,61,62 . A PANoptose tem sido cada vez mais implicada em doenças infecciosas e inflamatórias, bem como em cânceres e terapias oncológicas 3,4,35,36,39,44,46,47,48,49,50,51,52,53 ,54,56
,57,58,59,60,61,62,63,64,65,66.
Dado o papel essencial das caspases nas vias de morte celular, inclusive na apoptose, piroptose, necroptose e PANoptose, é importante desenvolver técnicas para caracterizar sua ativação e compreender toda a complexidade das vias da PCD. O protocolo aqui detalha um método para estimular as células e medir a ativação subsequente das caspases (Figura 1). Este método aproveita a clivagem proteolítica das caspases, que geralmente é necessária para a sua ativação, como um meio de estudá-las. Através do western blotting, os tamanhos das proteínas podem ser determinados, permitindo a visualização clara e diferenciação de pró-caspases inativas e suas formas ativadas e clivas.
As principais vantagens deste protocolo são 1) sua capacidade de avaliar a ativação de múltiplas caspases em paralelo a partir de uma única população de células endógenas para determinar com mais precisão a ativação da PCD e 2) o uso de técnicas laboratoriais relativamente simples que não requerem treinamento extensivo ou equipamentos caros. Protocolos anteriores usaram western blotting, repórteres fluorescentes ou coloração de anticorpos para monitorar a ativação da caspase em sobrenadantes de cultura, lisados celulares e teciduais, células inteiras via microscopia e in vivo 67,68,69,70,71, mas essas técnicas geralmente monitoram apenas uma ou duas caspases em uma amostra. Além disso, enquanto substratos peptídicos sintéticos contendo sítios de clivagem da caspase que fluorescem sobre a clivagem têm sido usados para monitorar a ativação da caspase em lisados celulares ou teciduais69, esses substratos muitas vezes podem ser clivados por mais de uma caspase, dificultando a determinação da ativação específica de caspases individuais neste sistema. Além disso, o uso de western blotting em vez do uso de repórteres fluorescentes ou outros métodos baseados em tags permite que os pesquisadores usem células endógenas em vez de criar linhagens celulares específicas com genes repórteres. Existem múltiplas vantagens no uso de células endógenas, incluindo o fato de que muitas linhagens celulares imortalizadas são deficientes em moléculas-chave de morte celular72,73, o que poderia afetar os resultados. Além disso, o uso de células endógenas permite a avaliação de diversos tipos de células, como macrófagos, células epiteliais e células endoteliais, em vez de uma única linhagem. Western blotting também é uma técnica relativamente simples e econômica que pode ser realizada em laboratórios em todo o mundo sem a necessidade de equipamentos grandes e caros ou configurações complicadas.
Esse protocolo é amplamente aplicável em toda a biologia para entender as funções dependentes da morte celular e independentes da morte celular das caspases, incluindo seus papéis e funções de andaimes em outras vias de sinalização inflamatória74. A aplicação deste método permite uma abordagem unificada no estudo das vias inatas da PCD imune e da sinalização inflamatória entre doenças e condições, e este protocolo pode ser usado para identificar processos regulatórios críticos e conexões mecanicistas que informarão o desenvolvimento de futuras estratégias terapêuticas.
Protocolo
O uso e os procedimentos dos animais foram aprovados pelo Comitê de Pesquisa do Hospital Infantil St. Jude sobre o Uso e Cuidado dos Animais.
1. Preparação das soluções
- Prepare a mídia condicionada por L929.
- A placa 1 × 106 células L929 (ver Tabela de Materiais) num balão de cultura de tecidos de182 cm 2 contendo 50 ml de meios de cultura L929 (ver Tabela 1 para a preparação do meio).
- Cultivar as células em uma incubadora umidificada a 37 °C com 5% de CO2.
- Após 7 dias, colete o sobrenadante e filtre usando um filtro de 0,45 μm. Prepare alíquotas de 50 mL (armazene alíquotas congeladas a -80 °C por até 1 ano).
- Preparar 500 mL de meio de cultura de macrófagos derivados da medula óssea (BMDM) (Tabela 1).
- Preparar 500 mL de meio de estimulação da BMDM (Tabela 1).
- Preparar 500 mL de meio para infecção (Tabela 1).
- Preparar 100 mL de tampão Tris 1 M (Tabela 1).
- Preparar 4x tampão de dodecil sulfato de sódio (SDS) (Tabela 1).
- Preparar 1 ml de 1 M 1,4-ditiotreitol (TDT, Tabela 1).
- Preparar 40 mL de tampão de lise de caspase (Tabela 1).
- Preparar 100 mL de tampão Tris 1,5 M (Tabela 1).
- Preparar 100 mL de uma solução de SDS a 10% (m/vol) (Tabela 1).
- Preparar 50 mL de um tampão 2x ensaio de radioimunoprecipitação (RIPA) (Tabela 1).
- Preparar uma solução de LPS de 5 mg/ml (Tabela 1).
- Preparar uma solução de ATP 0,5 M (Tabela 1).
- Prepare as soluções de western blotting.
- Preparar 1 L de 5x corpóreo regulador em funcionamento (Tabela 1).
- Preparar 1 L de 10x de depósito regulador de transferência (Tabela 1).
- Preparar 1 L de solução salina tamponada com Tris com Tween 20 (TBST, Tabela 1).
- Preparar 100 ml de uma solução bloqueadora de leite desnatado a 5% (m/vol) (Tabela 1).
- Prepare as soluções primárias de anticorpos.
- Preparar 10 mL de anticorpo primário caspase-1 (Tabela 1).
- Preparar 10 mL de anticorpo primário caspase-11 (Tabela 1).
- Preparar 10 mL de anticorpo primário caspase-3 (Tabela 1).
- Preparar 10 mL de anticorpo primário caspase-7 (Tabela 1).
- Preparar 10 mL de anticorpo primário caspase-8 (Tabela 1).
- Preparar 10 mL de anticorpo primário caspase-9 (Tabela 1).
- Preparar 10 mL de anticorpo primário β-actina conjugado com HRP (Tabela 1).
- Prepare as soluções secundárias de anticorpos.
- Preparar 10 ml de anticorpo secundário anti-coelho (Tabela 1).
- Preparar 10 ml de anticorpo secundário anti-ratinho (Tabela 1).
- Preparar 10 mL de anticorpo secundário anti-rato (Tabela 1).
2. Macrófagos derivados da medula óssea isolados
NOTA: Para este protocolo, camundongos do tipo selvagem de 6 a 10 semanas de idade com vias PCD intactas ou camundongos mutantes com os reguladores, efetores ou moléculas de interesse do PCD excluídos ou alterados podem ser usados.
- Eutanasiar um rato em uma câmara de CO 2 com uma taxa de fluxo que desloca 10%-30% do volume da gaiola por minuto por2-3 min. Em seguida, realize um método secundário de eutanásia, como luxação cervical. Siga todas as diretrizes e regulamentos adicionais específicos de instalações, instituições e governos, sempre que aplicável.
- Dissecar o rato para recuperar os ossos da pata traseira.
- Prenda o rato nas costas para que o abdômen fique exposto. Pulverize com etanol a 70% (vol/vol) para esterilizar as patas traseiras e o abdômen.
CUIDADO: O etanol é inflamável. Mantenha-o longe de chamas abertas. - Use uma tesoura para fazer uma incisão na linha média do abdômen; continuar cortando em direção às pernas para tornar os fêmures visíveis.
- Pegue a perna traseira direita e puxe a pele para longe do corpo em direção à linha média. Desprenda a perna do corpo, cortando os músculos adutores em direção à linha média; em seguida, corte a perna entre a articulação do quadril e a coluna vertebral. Em seguida, corte a pata distal ao tornozelo e remova o excesso de tecido do osso descascando a pele e usando uma tesoura ligeiramente aberta para retirar o tecido da panturrilha.
- Coloque a perna em uma toalha embebida em etanol a 70% (vol/vol) e disseque a tíbia e o fêmur.
- Fecho a tíbia e o fêmur cada um com um conjunto separado de pinças. Pressione suavemente a tíbia contra a direção natural da articulação do joelho; isso fará com que a tíbia se quebre no joelho.
- Use a pinça e a tesoura dissecante, se necessário, para remover qualquer tecido pendurado restante. Guarde a tíbia para uso posterior, colocando-a na toalha embebida em etanol.
- Colete o fêmur da mesma maneira, arrancando o joelho.
- Repita os passos 3 e 4 acima para a perna esquerda para removê-la do corpo e dissecar a tíbia e o fêmur.
- Pulverize os ossos com etanol a 70% (vol/vol).
- Limpe os ossos colocando-os em uma toalha limpa de 70% (vol / vol) embebida em etanol, apertando a parte carnuda na toalha e esfregando a toalha contra o osso para remover o excesso de tecido.
- Prenda o rato nas costas para que o abdômen fique exposto. Pulverize com etanol a 70% (vol/vol) para esterilizar as patas traseiras e o abdômen.
- Uma vez que ambos os fêmures e ambas as tíbias estejam limpos, pulverize todos os quatro ossos com etanol a 70% (vol / vol). Colete os ossos em uma placa de Petri estéril e enxágue-os com 10 mL de meio de cultura BMDM, balançando suavemente o meio no prato.
- Encha uma seringa de 10 ml com 10 ml de meio de cultura BMDM fresco e coloque uma agulha de 25 G.
- Pegue a tíbia usando fórceps; em seguida, corte a articulação do tornozelo em um ângulo de ~ 45 °.
- Lave a medula óssea da tíbia.
- Segure a tíbia sobre um tubo de 50 mL, com a extremidade estreita da tíbia apontando para baixo. Dispense o meio sobre a tíbia da seringa cheia.
- Insira a agulha (suavemente no início) na extremidade superior da medula e dispense o meio.
- Retire a agulha e insira novamente. Use empurrões curtos e de alta pressão para dispensar o meio e mover a agulha para a medula.
- Uma vez que a mídia começa a fluir para fora do fundo do osso, use empurrões curtos e de alta pressão para continuar a liberar as células. Monitore a cor do osso durante esse processo e, quando o osso estiver branco, descarte-o.
- Faça isso para ambas as tíbias.
- Repita os passos 5 e 6 acima para os fêmures, cortando na articulação do quadril como a tíbia foi cortada na articulação do tornozelo. Use o mesmo tubo de 50 mL para coletar o meio de cultura BMDM à medida que a medula é lavada.
- Uma vez que todos os quatro ossos tenham sido lavados, aspirar a medula e o meio do tubo de 50 mL para cima e para baixo 3x através de uma agulha de 18 G em uma seringa de 10 mL, enxaguando os lados do tubo de cada vez para dispersar a medula.
- Ajuste o volume final no tubo de 50 mL para 30 mL com meio de cultura BMDM e certifique-se de que as células estejam completamente suspensas.
- Use um filtro celular de 70 μm para filtrar o meio de cultura BMDM do tubo de 50 mL.
- Placa a suspensão celular resultante, que contém as células progenitoras da medula óssea, em três placas de cultura de tecido de 150 mm, adicionando 10 mL (ou ~20 × 106 células) a cada uma. Em seguida, adicione mais 10 mL de meio de cultura BMDM a cada prato. Incubar numa incubadora humidificada a 37 °C.
3. Diferenciando as BMDMs e o chapeamento para os experimentos
- Incubar as células progenitoras da medula óssea chapeadas a 37 °C durante 3 dias. Em seguida, retire cada prato e adicione mais 5-8 mL de meio de cultura BMDM (Tabela 1). Regresso à incubadora a 37 °C.
- No dia 5 após o revestimento inicial, remova cada prato e adicione mais 5 mL de meio de cultura BMDM. Regresso à incubadora a 37 °C.
- No dia 6, retire cada prato e descarte a mídia. Em seguida, adicione 10 mL de PBS frio (armazenado a 4 °C) para lavar uma vez. Descarte os 10 mL de lavagem fria com PBS. Em seguida, adicione 10 mL de PBS fresco e frio a cada prato e incube cada prato no gelo por 5 min.
- Usando um raspador de células, raspe suavemente as células de todos os três pratos em um tubo de 50 mL. Girar suavemente as células a 270 × g a 4 °C durante 5 minutos; em seguida, descarte o sobrenadante.
- Adicione 20 mL de meio de cultura BMDM, pipete para cima e para baixo para ressuspender o pellet e conte as células.
NOTA: Espera-se que cada rato produza aproximadamente 60 × 10 6-100 × 106 células. - Planeje o layout da placa de 12 poços para o ensaio desejado de morte celular / estimulação da inflamação in vitro . Planeje chapar 1 × 106 células por poço. Para cada estimulação planejada, inclua pelo menos três replicações biológicas e pratique um conjunto de poços a serem colhidos em tampão de lise de caspase e um segundo conjunto de poços a serem colhidos em tampão RIPA.
- A placa 1 × 106 células em 1 mL de meio de cultura BMDM por poço em placas de 12 poços. Cultura durante a noite em incubadora umidificada a 37 °C antes de prosseguir para a avaliação da morte celular/inflamação. Após a incubação durante a noite, remova o meio e adicione 1 mL de PBS quente (37 °C) a cada poço para lavar as células.
- Remova a lavagem com PBS e adicione 500 μL de meio de estimulação BMDM com antibióticos (se realizar estimulações não bacterianas) ou meios de estimulação BMDM sem antibióticos (se realizar estímulos bacterianos) (Tabela 1). Incubar durante 2 h antes de passar para o passo 4 para estimulação/ infeção in vitro .
4. Estimular ou infectar as células
CUIDADO: Os agentes incluídos neste protocolo são potencialmente patogênicos e devem ser manuseados com as precauções apropriadas em uma instalação de nível de biossegurança 2 (BSL2) com aprovação das autoridades institucionais e governamentais relevantes.
- Estimular as BMDMs a ativar a morte celular com o gatilho de interesse.
NOTA: Para efeitos do presente protocolo, o vírus da gripe A (IAV), o vírus herpes simplex 1 (HSV1), Francisella novicidae LPS + ATP são usados para ilustração, mas outros gatilhos podem ser usados.- Exemplo de estimulação 1: Infectar com IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (construído de acordo com Hoffmann et al.75; o título viral para a determinação da multiplicidade de infecção [MOI] é calculado por um ensaio de placa em células MDCK):
- Calcular o volume de vírus necessário para a infecção em uma multiplicidade de infecção (MOI) de 20 unidades formadoras de placas (UFP) usando a equação (1) e a equação (2):

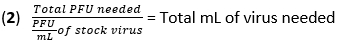

- Remova o meio dos BMDMs e lave as células uma vez com 500 μL de PBS. Adicione 450 μL de IAV (20 MOI) em DMEM de alta glicose sem FBS inativado por calor (HI) a cada poço. Incubar as placas a 37 °C durante 1 h numa incubadora humidificada para permitir a absorção.
- Retire as placas e adicione 50 μL de HI-FBS. Devolver as placas à incubadora a 37 °C. Incubar por um total de 12 h.
- Calcular o volume de vírus necessário para a infecção em uma multiplicidade de infecção (MOI) de 20 unidades formadoras de placas (UFP) usando a equação (1) e a equação (2):
- Exemplo de estimulação 2: Infectar com HSV1 (cepa HF; cultivada conforme descrito anteriormente44; o título viral para determinação de MOI é calculado por um ensaio de placa em células Vero):
- Calcule o volume de vírus necessário para a infecção em um MOI de 10 PFU usando a equação (1) e a equação (2) da etapa 1 na seção de infecção por IAV acima.

- Adicione 450 μL de HSV1 (MOI 10) em DMEM de alta glicose sem HI-FBS a cada poço. Incubar as placas a 37 °C durante 1 h numa incubadora humidificada para permitir a absorção.
- Remova as placas e adicione 50 μL de HI-FBS. Devolver as placas à incubadora a 37 °C. Incubar por um total de 12 h.
- Calcule o volume de vírus necessário para a infecção em um MOI de 10 PFU usando a equação (1) e a equação (2) da etapa 1 na seção de infecção por IAV acima.
- Exemplo de estimulação 3: Infectar com F. novicida (cepa U112; cultivada como descrito anteriormente44 em condições aeróbicas a 37 °C em caldo de soja BBL Trypticase suplementado com 0,2% de L-cisteína durante a noite. Em seguida, subcultura das bactérias a uma proporção de 1:10 a 37 °C por mais 4 h em meio fresco antes de tomar a densidade óptica (OD) a 600 nm usando o meio fresco como um espaço em branco. Um valor de DO de 1 equivale a 1 × 109 unidades formadoras de colônias (UFC) por mL.)
- Calcular o volume de F. novicida necessário para a infecção em um MOI de 50 UFC usando a equação (3) e a equação (4):

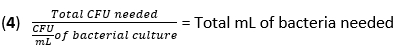
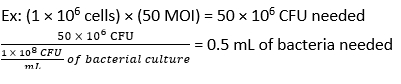
- Adicione 500 μL de F. novicida (MOI 50) em meios de estimulação BMDM sem antibióticos a cada poço. Incubar as placas a 37 °C durante 4 h numa incubadora humidificada para permitir a absorção.
- Lavar as células três vezes com PBS aquecido (37 °C) e adicionar 500 μL de meio de estimulação BMDM contendo 50 μg/mL de gentamicina. Devolver as placas à incubadora a 37 °C. Incubar durante a noite (16 h).
- Calcular o volume de F. novicida necessário para a infecção em um MOI de 50 UFC usando a equação (3) e a equação (4):
- Exemplo de estimulação 4: Estimular com LPS + ATP.
- Remova o meio dos BMDMs. Adicione 500 μL de meio de estimulação BMDM com antibióticos (Tabela 1) contendo 100 ng/mL LPS para cada poço. Incubar as placas a 37 °C durante 3,5 h numa incubadora humidificada.
- Adicionar 5 μL de solução-mãe de ATP 0,5 M (Tabela 1) a cada alvéolo. Devolver as placas à incubadora a 37 °C. Incubar por 30 min.
- Exemplo de estimulação 1: Infectar com IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (construído de acordo com Hoffmann et al.75; o título viral para a determinação da multiplicidade de infecção [MOI] é calculado por um ensaio de placa em células MDCK):
5. Recolha do sobrenadante combinado e do lisado proteico a utilizar nas manchas ocidentais da caspase
- Após 4 h, 12 h ou 16 h de incubação (o tempo específico depende do gatilho usado), remova a placa da incubadora.
- Remover 150 μL do sobrenadante; descartar ou salvá-lo para outras análises sobrenadantes (por exemplo, ensaio de imunoadsorção enzimática [ELISA]). Não remova o sobrenadante restante.
- Criar a solução de coleta de proteínas combinando 50 μL de tampão de lise de caspase + 100 μL de 4x tampão SDS por poço (Tabela 1). Em seguida, adicione 150 μL da mistura a cada poço.
- Para cada poço, pipete a mistura para cima e para baixo para coletar as células lisadas e o sobrenadante. Durante a pipetagnação, também raspe o fundo do poço com a ponta da pipeta para interromper as células. Após raspagem e pipetagem, colete o lisado de proteína em tubos de 1,5 mL marcados.
- Use um bloco de calor para aquecer todos os tubos a 100 °C por 12 min.
- Pellet quaisquer componentes insolúveis por centrifugação a 14.500 × g durante 30 s à temperatura ambiente.
NOTA: Este é um ponto de pausa – a proteína do sobrenadante combinado e dos lisados proteicos pode ser usada imediatamente ou armazenada a -20 °C por até 2 meses ou a -80 °C por até 6 meses até estar pronta para uso.
6. Coleta do lisado de proteína a ser usado para manchas ocidentais de caspase
- Após 4 h, 12 h ou 16 h de incubação (o tempo específico depende do gatilho usado), remova a placa da incubadora. Remova todo o sobrenadante; descartar ou salvá-lo para outras análises sobrenadantes (por exemplo, ELISA).
- Crie o tampão RIPA 1x diluindo a solução-mãe de RIPA 2x (Tabela 1) em um volume igual de água deionizada. Em seguida, adicione um comprimido inibidor de fosfatase e um comprimido inibidor de protease e deixe-os dissolver. Adicione 150 μL de 1x buffer RIPA e 50 μL de 4x SDS a cada poço.
- Para cada poço, pipete a mistura para cima e para baixo para coletar as células lisadas. Durante a pipetagnação, também raspe o fundo do poço com a ponta da pipeta para interromper as células. Após raspagem e pipetagem, colete o lisado de proteína em tubos de 1,5 mL marcados.
- Use um bloco de calor para aquecer todos os tubos a 100 °C por 12 min.
- Pellet quaisquer componentes insolúveis por centrifugação a 14.500 × g durante 30 s à temperatura ambiente.
NOTA: Este é um ponto de pausa – os lisados de proteína podem ser usados imediatamente ou armazenados a -20 °C ou -80 °C até estarem prontos para uso.
7. Realização de western blotting usando os lisados coletados dos BMDMs seguindo as etapas acima ou de homogeneizados teciduais
NOTA: Se estiver usando tecido, ele pode ser homogeneizado à mão ou através de um homogeneizador de tecido acionado por energia. O protocolo de Simpson76 fornece uma descrição detalhada da homogeneização tecidual.
- Preparar 1x tampão de corrida: Combine 200 mL do estoque regulador de 5x em funcionamento (Tabela 1) e 800 mL de água deionizada. Faça este buffer de execução 1x antes de cada experimento.
- Preparar o aparelho de eletroforese com um gel de poliacrilamida a 12% (wt/vol) com 10 poços. Encha o aparelho de eletroforese com 1x tampão de funcionamento. Em seguida, remova o pente de gel.
NOTA: Para analisar a caspase-1, caspase-11, caspase-3, caspase-7, caspase-8 e caspase-9 para cada amostra, serão necessários seis géis. - Para as bolha de caspase-1, caspase-3, caspase-7 e caspase-8, planeje usar 30 μL do sobrenadante combinado e do lisado proteico no tampão de lise da caspase ou no homogeneizado tecidual. Para as bolha de caspase-11 e caspase-9, planeje usar 20 μL do lisado proteico no tampão RIPA ou no homogeneizado tecidual. Se as amostras tiverem sido armazenadas a -20 °C ou -80 °C, descongele-as no gelo primeiro.
- Para todas as amostras, aquecer a 100 °C durante 5 min e centrifugar a 14 500 × g durante 30 s a 4 °C antes do carregamento. Em seguida, carregue lentamente 20 ou 30 μL da amostra em cada faixa. Evite que qualquer amostra transborde para as outras pistas. Para avaliar todas as seis caspases de uma só vez, use o mesmo procedimento para carregar as amostras apropriadas em cada um dos seis géis.
- Ligue o aparelho de electroforese à fonte de alimentação. Em seguida, defina a potência para 80 V por 20 minutos para iniciar a execução do gel e, em seguida, ajuste a potência para 130 V por 45-60 min.
- Observe a frente do corante com cuidado e desligue a energia quando a frente do corante estiver na parte inferior do gel, mas ainda não tiver sido empurrada para fora do gel.
- Enquanto o gel estiver em execução, prepare 1x tampão de transferência combinando 700 mL de água deionizada, 100 mL do estoque tampão de transferência 10x (Tabela 1) e 200 mL de metanol. Torne a solução 1x fresca de cada vez.
NOTA: Tenha cuidado, pois o metanol é inflamável. Execute a preparação do buffer de transferência longe das chamas abertas. - Remova o gel do aparelho de eletroforese suavemente usando o liberador de gel.
- Configure uma pilha de transferência para cada gel.
- Ative uma membrana de PVDF mergulhando-a em metanol por 1 min.
- Pré-molhado dois pedaços de papel de filtro, o gel e a membrana PVDF em tampão de transferência por 5 min. Mantenha a membrana e o gel PVDF em recipientes separados durante esta incubação de 5 minutos.
- Monte a pilha de transferência no sistema semi-seco. Começando no lado inferior do ânodo de platina, coloque um pedaço de papel de filtro, a membrana PVDF, o gel e, finalmente, um pedaço de papel de filtro. Estenda ou pressione suavemente as bolhas de ar entre as camadas e feche a parte superior do sistema. Certifique-se de que a tampa de segurança está presa antes de prosseguir.
- Conecte-se à fonte de alimentação. Defina a potência para 25 V por 45 min.
- Após a conclusão da transferência, desmonte a pilha de transferência e colete a membrana; coloque-o em uma placa de Petri quadrada (bandeja de incubação).
- Realizar o bloqueio da membrana adicionando 15 mL de uma solução de leite desnatado a 5% (em peso/vol) (Tabela 1). Incubar a membrana num agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente durante 1 h.
NOTA: Este é um ponto de pausa – a membrana pode ser removida após 1 h ou armazenada em solução de bloqueio a 4 °C durante a noite. - Após 1 h ou durante a incubação, retire a solução de bloqueio. Adicionar 10 mL da solução de anticorpos diluídos (anticorpo anticaspase-1, anticorpo anticaspase-11, anticorpo combinado anticaspase-3 e anticorpo anticaspase-3 clivado, anticorpo combinado anticaspase-7 e anticaspase-7 clivado, anticorpo combinado anticaspase-8 e anticaspase-8 clivrado ou anticorpo anticaspase-9) (Tabela 1). Colocar num agitador de balanço a 50 rpm a 70 rpm para incubar à temperatura ambiente durante 2 h ou a 4 °C durante a noite (16 h).
- Recolher a solução de anticorpos (reutilizar até 3x ou descartá-la) e lavar adicionando 15 ml de TBST (Tabela 1) à membrana num agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente durante 10 min. Descarte o TBST.
- Repita a lavagem com 15 mL de TBST seguindo o passo 14 um total de 3x.
- Adicionar 10 mL da solução secundária diluída de anticorpos conjugados com HRP (anticoelho para manchas coradas com anticorpos primários contra caspase-3, caspase-7, caspase-8 ou caspase-9; anti-camundongo para manchas coradas com anticorpo primário contra caspase-1; anti-rato para manchas coradas com anticorpo primário contra caspase-11) (Tabela 1). Incubar num agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente durante 1 h.
- Remova a solução de anticorpos e lave adicionando 15 mL de TBST à membrana em um agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente por 10 min. Descarte o TBST.
- Repita a lavagem com 15 mL de TBST seguindo o passo 17 um total de 3x.
- Adicione 10 mL do substrato HRP de alta sensibilidade à membrana. Deixe descansar à temperatura ambiente no escuro por 1 min.
- Remova a membrana do substrato. Prossiga diretamente para a imagem, usando um imageador de quimioluminescência com a bandeja trans branca acessória inserida na posição inferior. Exponha a membrana usando o modo de exposição automática (geralmente ~ 1-2 min de tempo de exposição).
- Usando a membrana da mancha de caspase-9 ou caspase-11 (ou seja, uma membrana com amostras de lisado RIPA), adicione 10 mL de tampão de decapagem e incube em um agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente por 5 min.
- Descarte o tampão de decapagem e lave adicionando 15 mL de TBST à membrana em um agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente por 10 min. Descarte o TBST.
- Repita a lavagem com 15 mL de TBST seguindo o passo 22 um total de 3x.
- Realizar o bloqueio da membrana adicionando 15 mL de uma solução de leite desnatado a 5% (em peso/vol). Incubar a membrana num agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente durante 1 h.
NOTA: Este é um ponto de pausa – a membrana pode ser removida após 1 h ou armazenada em solução de bloqueio a 4 °C durante a noite. - Após 1 h ou durante a incubação, retire a solução de bloqueio. Adicione 10 mL da solução diluída de anticorpo anti-β-actina (conjugada com HRP). Colocar num agitador de balanço a 50 rpm a 70 rpm para incubar à temperatura ambiente durante 1,5 h.
- Remova a solução de anticorpos e lave adicionando 15 mL de TBST à membrana em um agitador de balanço a 50 rpm a 70 rpm à temperatura ambiente por 10 min. Descarte o TBST.
- Repita a lavagem com 15 mL de TBST seguindo o passo 26 um total de 3x.
- Adicione 10 mL do substrato HRP de sensibilidade padrão à membrana. Deixe descansar à temperatura ambiente no escuro por 1 min.
- Prossiga diretamente para a imagem, usando um imageador de quimioluminescência com a bandeja trans branca acessória inserida na posição inferior. Expor a membrana usando o modo de exposição automática (geralmente <1 min de tempo de exposição).
Resultados
PANoptosis tem sido observada em resposta a numerosas infecções bacterianas, virais e fúngicas e outros estímulos inflamatórios, bem como em células cancerosas 44,48,49,50,51,52,53,54,56,57,58,60,61,62
Discussão
O monitoramento da clivagem e ativação da caspase fornece uma das imagens mais abrangentes da ativação inata do PCD imune como parte da resposta imune inata. O protocolo aqui descrito demonstra uma estratégia para monitorar a ativação da caspase em resposta às infecções por IAV, HSV1 e F. novicida e ao gatilho estéril LPS + ATP, mas inúmeros outros estímulos podem induzir PCD e poderiam ser utilizados nesse método, como tem sido demonstrado em várias publicações 44,48,49,50,51,52,53,54,56
Divulgações
T.-D.K. é consultor da Pfizer.
Agradecimentos
Agradecemos aos membros do laboratório Kanneganti por seus comentários e sugestões, e agradecemos a J. Gullett, PhD, pelo apoio à edição científica. O trabalho em nosso laboratório é apoiado pelos subsídios dos Institutos Nacionais de Saúde (NIH) AI101935, AI124346, AI160179, AR056296 e CA253095 (para T.-D.K.) e pelas Instituições de Caridade Associadas Sírias Libanesas Americanas (para T.-D.K.). O conteúdo é de responsabilidade exclusiva dos autores e não representa necessariamente as opiniões oficiais dos Institutos Nacionais de Saúde.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Millipore | SCHVU05RE | |
| 10 mL syringe | BD Biosciences | 309604 | |
| 12% polyacrylamide gel with 10 wells | Bio-Rad | 4561043 | |
| 12-well plate | Corning | 07-200-82 | |
| 18 G needle | BD Biosciences | 305195 | |
| 25 G needle | BD Biosciences | 305122 | |
| 50 mL tube | Fisher Scientific | 50-809-218 | |
| 70 μm cell strainer | Corning | 431751 | |
| 150 mm tissue culture dishes | Corning | 430597 | |
| 182-cm2 tissue culture flask | Genesee Scientific | 25-211 | |
| Accessory white trans tray | Cytiva | 29-0834-18 | |
| Anti–caspase-1 antibody | AdipoGen | AG-20B-0042-C100 | |
| Anti–caspase-11 antibody | Novus Biologicals | NB120-10454 | |
| Anti–caspase-3 antibody | Cell Signaling Technology | 9662 | |
| Anti–caspase-7 antibody | Cell Signaling Technology | 9492 | |
| Anti–caspase-8 antibody | Cell Signaling Technology | 4927 | |
| Anti–caspase-9 antibody | Cell Signaling Technology | 9504 | |
| Anti–cleaved caspase-3 antibody | Cell Signaling Technology | 9661 | |
| Anti–cleaved caspase-7 antibody | Cell Signaling Technology | 9491 | |
| Anti–cleaved caspase-8 antibody | Cell Signaling Technology | 8592 | |
| Anti-mouse HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 315-035-047 | |
| Anti-rabbit HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 111-035-047 | |
| Anti-rat HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 112-035-003 | |
| Anti–β-Actin antibody (C4) HRP | Santa Cruz | sc-47778 HRP | |
| ATP | InvivoGen | tlrl-atpl | |
| BBL Trypticase Soy Broth | BD Biosciences | 211768 | |
| Bead bath | Chemglass Life Sciences | CLS-4598-009 | |
| Biophotometer D30 | Eppendorf | 6133000010 | |
| BME | Sigma | M6250 | |
| Bromophenol blue | Sigma | BO126 | |
| Cell scrapers | CellTreat Scientific Products | 229315 | |
| Chemiluminescence imager (Amersham 600) | Cytiva | 29083461 | |
| CO2 chamber | VetEquip | 901703 | |
| Cuvettes | Fisher Scientific | 14-955-129 | |
| Dissecting scissors | Thermo Fisher Scientific | 221S | |
| DMEM | Thermo Fisher Scientific | 11995-073 | |
| DTT | Sigma | 43815 | |
| Eelectrophoresis apparatus | Bio-Rad | 1658004 | |
| Ethanol | Pharmco | 111000200 | |
| Fetal bovine serum | Biowest | S1620 | |
| Filter paper | Bio-Rad | 1703965 | |
| Forceps | Fisher Scientific | 22-327379 | |
| Francisella novicida (U112 strain) | BEI Resources | NR-13 | |
| Gel releaser | Bio-Rad | 1653320 | |
| Gentamycin | Gibco | 15750060 | |
| Glycerol | Sigma | G7893 | |
| Glycine | Sigma | G8898 | |
| HCl | Sigma | H9892 | |
| Heat block | Fisher Scientific | 23-043-160 | |
| Herpes simplex virus 1 (HF strain) | ATCC | VR-260 | |
| High glucose DMEM | Sigma | D6171 | |
| Human anti–caspase-1 antibody | R&D Systems | MAB6215 | |
| Human anti–caspase-8 antibody | Enzo | ALX-804-242 | |
| Humidified incubator | Thermo Fisher Scientific | 51026282 | |
| Image analysis software | ImageJ | v1.53a | |
| IMDM | Thermo Fisher Scientific | 12440-053 | |
| Influenza A virus (A/Puerto Rico/8/34, H1N1 [PR8]) | constructed per Hoffmann et al. | ||
| L929 cells | ATCC | CCL-1 | cell line for creating L929-conditioned media |
| L-cysteine | Thermo Fisher Scientific | BP376-100 | |
| Luminata Forte Western HRP substrate | Millipore | WBLUF0500 | standard-sensitivity HRP substrate |
| MDCK cells | ATCC | CCL-34 | cell line for determining IAV viral titer |
| Methanol | Sigma | 322415 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002401 | |
| Non-essential amino acids | Gibco | 11140050 | |
| Nonfat dried milk powder | Kroger | ||
| NP-40 solution | Sigma | 492016 | |
| PBS | Thermo Fisher Scientific | 10010023 | |
| Penicillin and streptomycin | Sigma | P4333 | |
| Petri dish | Fisher Scientific | 07-202-011 | |
| PhosSTOP | Roche | PHOSS-RO | |
| Power source | Bio-Rad | 164-5052 | |
| Protease inhibitor tablet | Sigma | S8820 | |
| PVDF membrane | Millipore | IPVH00010 | |
| Rocking shaker | Labnet | S2035-E | |
| SDS | Sigma | L3771 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium deoxycholate | Sigma | 30970 | |
| Sodium hydroxide | Sigma | 72068 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Square Petri dish | Fisher Scientific | FB0875711A | |
| Stripping buffer | Thermo Fisher Scientific | 21059 | |
| Super Signal Femto HRP substrate | Thermo Fisher Scientific | 34580 | high-sensitivity HRP substrate |
| Tabletop centrifuge | Thermo Fisher Scientific | 75004524 | |
| Trans-Blot semi-dry system | Bio-Rad | 170-3940 | |
| Tris | Sigma | TRIS-RO | |
| Tween 20 | Sigma | P1379 | |
| Ultrapure lipopolysaccharide (LPS) from E. coli 0111:B4 | InvivoGen | tlrl-3pelps | |
| Vero cells | ATCC | CCL-81 | cell line for determining HSV1 viral titer |
Referências
- Alnemri, E. S., et al. Human ICE/CED-3 protease nomenclature. Cell. 87 (2), 171 (1996).
- Man, S. M., Kanneganti, T. D. Converging roles of caspases in inflammasome activation, cell death and innate immunity. Nature Reviews Immunology. 16 (1), 7-21 (2016).
- Gullett, J. M., Tweedell, R. E., Kanneganti, T. D. It's all in the PAN: Crosstalk, plasticity, redundancies, switches, and interconnectedness encompassed by PANoptosis underlying the totality of cell death-associated biological effects. Cells. 11 (9), 1495 (2022).
- Pandian, N., Kanneganti, T. D. PANoptosis: A unique innate immune inflammatory cell death modality. Journal of Immunology. 209 (9), 1625-1633 (2022).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death & Differentiation. 25 (3), 486-541 (2018).
- Shi, Y. Caspase activation: Revisiting the induced proximity model. Cell. 117 (7), 855-858 (2004).
- Galluzzi, L., Lopez-Soto, A., Kumar, S., Kroemer, G. Caspases connect cell-death signaling to organismal homeostasis. Immunity. 44 (2), 221-231 (2016).
- Fernandes-Alnemri, T., Litwack, G., Alnemri, E. S. CPP32, a novel human apoptotic protein with homology to Caenorhabditis elegans cell death protein Ced-3 and mammalian interleukin-1 beta-converting enzyme. Journal of Biological Chemistry. 269 (49), 30761-30764 (1994).
- Tewari, M., et al. Yama/CPP32 beta, a mammalian homolog of CED-3, is a CrmA-inhibitable protease that cleaves the death substrate poly(ADP-ribose) polymerase. Cell. 81 (5), 801-809 (1995).
- Nicholson, D. W., et al. Identification and inhibition of the ICE/CED-3 protease necessary for mammalian apoptosis. Nature. 376 (6535), 37-43 (1995).
- Stennicke, H. R., et al. Pro-caspase-3 is a major physiologic target of caspase-8. Journal of Biological Chemistry. 273 (42), 27084-27090 (1998).
- Twiddy, D., Cohen, G. M., Macfarlane, M., Cain, K. Caspase-7 is directly activated by the approximately 700-kDa apoptosome complex and is released as a stable XIAP-caspase-7 approximately 200-kDa complex. Journal of Biological Chemistry. 281 (7), 3876-3888 (2006).
- Kesavardhana, S., Malireddi, R. K. S., Kanneganti, T. D. Caspases in cell death, inflammation, and pyroptosis. Annual Reviews of Immunology. 38, 567-595 (2020).
- Kerr, J. F., Wyllie, A. H., Currie, A. R. Apoptosis: a basic biological phenomenon with wide-ranging implications in tissue kinetics. British Journal of Cancer. 26 (4), 239-257 (1972).
- Taylor, R. C., Cullen, S. P., Martin, S. J. Apoptosis: Controlled demolition at the cellular level. Nature Reviews Molecular Cell Biology. 9 (3), 231-241 (2008).
- Morioka, S., Maueröder, C., Ravichandran, K. S. Living on the edge: Efferocytosis at the interface of homeostasis and pathology. Immunity. 50 (5), 1149-1162 (2019).
- Wang, Y., et al. Chemotherapy drugs induce pyroptosis through caspase-3 cleavage of a gasdermin. Nature. 547 (7661), 99-103 (2017).
- Rogers, C., et al. Cleavage of DFNA5 by caspase-3 during apoptosis mediates progression to secondary necrotic/pyroptotic cell death. Nature Communications. 8, 14128 (2017).
- Kayagaki, N., et al. Caspase-11 cleaves gasdermin D for non-canonical inflammasome signalling. Nature. 526 (7575), 666-671 (2015).
- Shi, J., Gao, W., Shao, F. Pyroptosis: Gasdermin-mediated programmed necrotic cell death. Trends in Biochemical Sciences. 42 (4), 245-254 (2017).
- Sborgi, L., et al. GSDMD membrane pore formation constitutes the mechanism of pyroptotic cell death. EMBO Journal. 35 (16), 1766-1778 (2016).
- Aglietti, R. A., et al. GsdmD p30 elicited by caspase-11 during pyroptosis forms pores in membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (28), 7858-7863 (2016).
- Hagar, J. A., Powell, D. A., Aachoui, Y., Ernst, R. K., Miao, E. A. Cytoplasmic LPS activates caspase-11: implications in TLR4-independent endotoxic shock. Science. 341 (6151), 1250-1253 (2013).
- Kayagaki, N., et al. Noncanonical inflammasome activation by intracellular LPS independent of TLR4. Science. 341 (6151), 1246-1249 (2013).
- Shi, J., et al. Inflammatory caspases are innate immune receptors for intracellular LPS. Nature. 514 (7521), 187-192 (2014).
- Lamkanfi, M., et al. Targeted peptidecentric proteomics reveals caspase-7 as a substrate of the caspase-1 inflammasomes. Molecular & Cellular Proteomics. 7 (12), 2350-2363 (2008).
- Kalai, M., et al. Tipping the balance between necrosis and apoptosis in human and murine cells treated with interferon and dsRNA. Cell Death & Differentiation. 9 (9), 981-994 (2002).
- Li, C., et al. Development of atopic dermatitis-like skin disease from the chronic loss of epidermal caspase-8. Proceedings of the National Academy of Sciences of the United States of America. 107 (51), 22249-22254 (2010).
- Kovalenko, A., et al. Caspase-8 deficiency in epidermal keratinocytes triggers an inflammatory skin disease. Journal of Experimental Medicine. 206 (10), 2161-2177 (2009).
- Kang, T. B., et al. Caspase-8 serves both apoptotic and nonapoptotic roles. Journal of Immunology. 173 (5), 2976-2984 (2004).
- Oberst, A., et al. Catalytic activity of the caspase-8-FLIP(L) complex inhibits RIPK3-dependent necrosis. Nature. 471 (7338), 363-367 (2011).
- Kaiser, W. J., et al. RIP3 mediates the embryonic lethality of caspase-8-deficient mice. Nature. 471 (7338), 368-372 (2011).
- Zhang, H., et al. Functional complementation between FADD and RIP1 in embryos and lymphocytes. Nature. 471 (7338), 373-376 (2011).
- Shi, J., et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature. 526 (7575), 660-665 (2015).
- Hitomi, J., et al. Identification of a molecular signaling network that regulates a cellular necrotic cell death pathway. Cell. 135 (7), 1311-1323 (2008).
- Malireddi, R. K., Ippagunta, S., Lamkanfi, M., Kanneganti, T. D. Cutting edge: Proteolytic inactivation of poly(ADP-ribose) polymerase 1 by the Nlrp3 and Nlrc4 inflammasomes. Journal of Immunology. 185 (6), 3127-3130 (2010).
- Tsuchiya, K., et al. Caspase-1 initiates apoptosis in the absence of gasdermin D. Nature Communications. 10 (1), (2019).
- Taabazuing, C. Y., Okondo, M. C., Bachovchin, D. A. Pyroptosis and apoptosis pathways engage in bidirectional crosstalk in monocytes and macrophages. Cell Chemical Biology. 24 (4), 507-514 (2017).
- Gurung, P., et al. FADD and caspase-8 mediate priming and activation of the canonical and noncanonical Nlrp3 inflammasomes. Journal of Immunology. 192 (4), 1835-1846 (2014).
- Man, S. M., et al. Inflammasome activation causes dual recruitment of NLRC4 and NLRP3 to the same macromolecular complex. Proceedings of the National Academy of Sciences of the United States of America. 111 (20), 7403-7408 (2014).
- Man, S. M., et al. Salmonella infection induces recruitment of Caspase-8 to the inflammasome to modulate IL-1beta production. Journal of Immunology. 191 (10), 5239-5246 (2013).
- Van Opdenbosch, N., et al. Caspase-1 engagement and TLR-induced c-FLIP expression suppress ASC/caspase-8-dependent apoptosis by inflammasome sensors NLRP1b and NLRC4. Cell Reports. 21 (12), 3427-3444 (2017).
- Pierini, R., et al. AIM2/ASC triggers caspase-8-dependent apoptosis in Francisella-infected caspase-1-deficient macrophages. Cell Death & Differentiation. 19 (10), 1709-1721 (2012).
- Lee, S., et al. AIM2 forms a complex with pyrin and ZBP1 to drive PANoptosis and host defence. Nature. 597 (7876), 415-419 (2021).
- Sagulenko, V., et al. AIM2 and NLRP3 inflammasomes activate both apoptotic and pyroptotic death pathways via ASC. Cell Death & Differentiation. 20 (9), 1149-1160 (2013).
- Lukens, J. R., et al. Dietary modulation of the microbiome affects autoinflammatory disease. Nature. 516 (7530), 246-249 (2014).
- Gurung, P., Burton, A., Kanneganti, T. D. NLRP3 inflammasome plays a redundant role with caspase 8 to promote IL-1beta-mediated osteomyelitis. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4452-4457 (2016).
- Kuriakose, T., et al. ZBP1/DAI is an innate sensor of influenza virus triggering the NLRP3 inflammasome and programmed cell death pathways. Science Immunology. 1 (2), (2016).
- Malireddi, R. K. S., et al. TAK1 restricts spontaneous NLRP3 activation and cell death to control myeloid proliferation. Journal of Experimental Medicine. 215 (4), 1023-1034 (2018).
- Malireddi, R. K. S., et al. Innate immune priming in the absence of TAK1 drives RIPK1 kinase activity-independent pyroptosis, apoptosis, necroptosis, and inflammatory disease. Journal of Experimental Medicine. 217 (3), (2020).
- Christgen, S., et al. Identification of the PANoptosome: A molecular platform triggering pyroptosis, apoptosis, and necroptosis (PANoptosis). Frontiers in Cellular and Infection Microbiology. 10, 237 (2020).
- Malireddi, R. K. S., et al. RIPK1 distinctly regulates Yersinia-induced inflammatory cell death, PANoptosis. Immunohorizons. 4 (12), 789-796 (2020).
- Zheng, M., Karki, R., Vogel, P., Kanneganti, T. D. Caspase-6 is a key regulator of innate immunity, inflammasome activation, and host defense. Cell. 181 (3), 674-687 (2020).
- Karki, R., et al. ADAR1 restricts ZBP1-mediated immune response and PANoptosis to promote tumorigenesis. Cell Reports. 37 (3), 109858 (2021).
- Wang, Y., et al. Single cell analysis of PANoptosome cell death complexes through an expansion microscopy method. Cellular and Molecular Life Sciences. 79 (10), 531 (2022).
- Malireddi, R. K. S., et al. Inflammatory cell death, PANoptosis, mediated by cytokines in diverse cancer lineages inhibits tumor growth. Immunohorizons. 5 (7), 568-580 (2021).
- Kesavardhana, S., et al. The Zα2 domain of ZBP1 is a molecular switch regulating influenza-induced PANoptosis and perinatal lethality during development. Journal of Biological Chemistry. 295 (24), 8325-8330 (2020).
- Banoth, B., et al. ZBP1 promotes fungi-induced inflammasome activation and pyroptosis, apoptosis, and necroptosis (PANoptosis). Journal of Biological Chemistry. 295 (52), 18276-18283 (2020).
- Karki, R., et al. Interferon regulatory factor 1 regulates PANoptosis to prevent colorectal cancer. JCI Insight. 5 (12), 136720 (2020).
- Zheng, M., et al. Impaired NLRP3 inflammasome activation/pyroptosis leads to robust inflammatory cell death via caspase-8/RIPK3 during coronavirus infection. Journal of Biological Chemistry. 295 (41), 14040-14052 (2020).
- Karki, R., et al. Synergism of TNF-α and IFN-γ triggers inflammatory cell death, tissue damage, and mortality in SARS-CoV-2 infection and cytokine shock syndromes. Cell. 184 (1), 149-168 (2021).
- Karki, R., et al. ZBP1-dependent inflammatory cell death, PANoptosis, and cytokine storm disrupt IFN therapeutic efficacy during coronavirus infection. Science Immunology. 7 (74), (2022).
- Jiang, W., Deng, Z., Dai, X., Zhao, W. PANoptosis: A new insight into oral infectious diseases. Frontiers in Immunology. 12, 789610 (2021).
- Chi, D., et al. Real-time induction of macrophage apoptosis, pyroptosis, and necroptosis by Enterococcus faecalis OG1RF and two root canal isolated strains. Frontiers in Cellular and Infection Microbiology. 11, 720147 (2021).
- Lin, J. F., et al. Phosphorylated NFS1 weakens oxaliplatin-based chemosensitivity of colorectal cancer by preventing PANoptosis. Signal Transduction and Targeted Therapy. 7 (1), 54 (2022).
- Song, M., et al. Self-assembled polymeric nanocarrier-mediated co-delivery of metformin and doxorubicin for melanoma therapy. Drug Delivery. 28 (1), 594-606 (2021).
- Boucher, D., Chan, A., Ross, C., Schroder, K. Quantifying caspase-1 activity in murine macrophages. Methods in Molecular Biology. 1725, 163-176 (2018).
- Boucher, D., Duclos, C., Denault, J. B. General in vitro caspase assay procedures. Methods in Molecular Biology. 1133, 3-39 (2014).
- Kaushal, V., Herzog, C., Haun, R. S., Kaushal, G. P. Caspase protocols in mice. Methods in Molecular Biology. 1133, 141-154 (2014).
- Swacha, P., Gekara, N. O., Erttmann, S. F. Biochemical and microscopic analysis of inflammasome complex formation. Methods in Enzymology. 625, 287-298 (2019).
- Talley, S., et al. A caspase-1 biosensor to monitor the progression of inflammation in vivo. Journal of Immunology. 203 (9), 2497-2507 (2019).
- Pelegrin, P., Barroso-Gutierrez, C., Surprenant, A. P2X7 receptor differentially couples to distinct release pathways for IL-1beta in mouse macrophage. Journal of Immunology. 180 (11), 7147-7157 (2008).
- Yu, J. W., et al. Cryopyrin and pyrin activate caspase-1, but not NF-kappaB, via ASC oligomerization. Cell Death & Differentiation. 13 (2), 236-249 (2006).
- Henry, C. M., Martin, S. J. Caspase-8 acts in a non-enzymatic role as a scaffold for assembly of a pro-inflammatory "FADDosome" complex upon TRAIL stimulation. Molecular Cell. 65 (4), 715-729 (2017).
- Hoffmann, E., Neumann, G., Kawaoka, Y., Hobom, G., Webster, R. G. A DNA transfection system for generation of influenza A virus from eight plasmids. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 6108-6113 (2000).
- Simpson, R. J. Homogenization of mammalian tissue. Cold Spring Harbor Protocols. (7), (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados