Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Évaluation de l’activation des caspases pour évaluer la mort des cellules immunitaires innées
Dans cet article
Résumé
Ce protocole décrit une méthode complète d’évaluation de l’activation des caspases (caspase-1, caspase-3, caspase-7, caspase-8, caspase-9 et caspase-11) en réponse à des modèles in vitro et in vivo (chez la souris) d’infection, d’agressions stériles et de cancer pour déterminer l’initiation des voies de mort cellulaire, telles que la pyroptose, l’apoptose, la nécroptose et la panoptose.
Résumé
L’immunité innée fournit la première ligne de défense critique en réponse aux agents pathogènes et aux insultes stériles. Un élément mécaniste clé de cette réponse est l’initiation de la mort cellulaire programmée immunitaire innée (DCP) pour éliminer les cellules infectées ou endommagées et propager les réponses immunitaires. Cependant, l’excès de DCP est associé à une inflammation et à une pathologie. Par conséquent, la compréhension de l’activation et de la régulation de la DCP est un aspect central de la caractérisation des réponses immunitaires innées et de l’identification de nouvelles cibles thérapeutiques à travers le spectre de la maladie.
Ce protocole fournit des méthodes pour caractériser l’activation immunitaire innée de la DCP en surveillant les caspases, une famille de protéases dépendantes de la cystéine qui sont souvent associées à diverses voies de PCD, y compris l’apoptose, la pyroptose, la nécroptose et la panoptique. Les rapports initiaux ont caractérisé la caspase-2, la caspase-8, la caspase-9 et la caspase-10 comme caspases initiatrices et la caspase-3, la caspase-6 et la caspase-7 comme caspases effectrices dans l’apoptose, tandis que des études ultérieures ont trouvé les caspases inflammatoires, caspase-1, caspase-4, caspase-5 et caspase-11, entraîner la pyroptose. On sait maintenant qu’il existe une diaphonie étendue entre les caspases et d’autres molécules immunitaires et de mort cellulaire innées à travers les voies de DCP précédemment définies, identifiant une lacune clé dans la compréhension mécaniste de l’immunité innée et de la DCP et conduisant à la caractérisation de PANoptosis. PANoptosis est une voie inflammatoire innée unique de PCD inflammatoire innée régulée par les complexes PANoptosomes, qui intègrent des composants, y compris des caspases, provenant d’autres voies de mort cellulaire.
Ici, des méthodes pour évaluer l’activation des caspases en réponse à divers stimuli sont fournies. Ces méthodes permettent de caractériser les voies de DCP in vitro et in vivo, car les caspases activées subissent un clivage protéolytique qui peut être visualisé par Western blot à l’aide d’anticorps et de conditions de buvard optimaux. Un protocole et un flux de travail de transfert Western ont été établis pour permettre l’évaluation de l’activation de plusieurs caspases de la même population cellulaire, fournissant une caractérisation complète des processus de DCP. Cette méthode peut être appliquée dans tous les domaines de recherche du développement, de l’homéostasie, de l’infection, de l’inflammation et du cancer pour évaluer les voies de la DCP tout au long des processus cellulaires de la santé et de la maladie.
Introduction
Le système immunitaire inné agit comme première ligne de défense pendant l’infection et en réponse à des stimuli stériles, tels que des lésions tissulaires et des altérations de l’homéostasie. Les capteurs immunitaires innés à la surface des cellules et dans le cytoplasme répondent aux schémas moléculaires associés aux agents pathogènes ou aux dommages (PAMP ou DAMP, respectivement) pour déclencher des voies de signalisation inflammatoires et des réponses cellulaires. L’un des processus clés de la réponse immunitaire innée est l’induction de la mort cellulaire pour éliminer les cellules infectées ou endommagées et stimuler d’autres réponses immunitaires innées et adaptatives. La mort cellulaire programmée (DCP) est un processus hautement conservé à travers les espèces, soulignant son importance évolutive en tant que mécanisme immunitaire inné.
Il existe plusieurs voies immunitaires innées de PCD qui peuvent être activées dans tous les types de cellules. Les caspases sont une famille clé de protéases hautement conservées, intracellulaires et dépendantes de la cystéine qui sont essentielles dans de nombreuses voies de DCP, y compris la voie de l’apoptose traditionnellement non inflammatoire, ainsi que les voies inflammatoires de DCP telles que la pyroptose, la nécroptose et la panoptique 1,2,3,4,5 . Il y a 11 caspases humaines et 10 caspases murines qui sont bien définies, ainsi que des pseudo-caspases qui peuvent être fonctionnelles, et la plupart sont constitutivement exprimées en pro-caspases monomères ou dimères inactives qui nécessitent un clivage pour l’activation 6,7. Les caspases contiennent également des domaines importants pour le recrutement et la formation de complexes multiprotéiques. Il s’agit notamment du domaine d’activation et de recrutement des caspases (CARD), qui peut être trouvé dans la caspase-1, la caspase-2, la caspase-4, la caspase-5, la caspase-9 et la caspase-11, ou le domaine effecteur de la mort (DED), qui se trouve dans la caspase-8 et la caspase-10. Grâce à leur activité protéolytique et à leur capacité à former des complexes multiprotéiques, les caspases sont des moteurs essentiels de la DCP immunitaire innée.
Le rôle des caspases dans la PCD immunitaire innée a été identifié pour la première fois dans l’apoptose, où les caspases initiatrices, caspases-2, caspases-8, caspases-9 et caspases-10, activent les caspases bourreaux, caspase-3, caspase-6 et caspase-7, pour conduire la mort cellulaire 8,9,10,11,12. Les caspases initiatrices peuvent être activées par diverses cascades de signalisation; La voie extrinsèque active la caspase-8 par la signalisation du récepteur de mort induite par un ligand extracellulaire, et la voie intrinsèque active la caspase-9 par la perturbation de l’intégrité mitochondriale13. Les caspases initiatrices activées clivent le linker séparant les grandes et les petites sous-unités catalytiques des caspases bourreaux pour produire leurs formes actives. Les caspases bourreaux clivent ensuite leurs substrats pour désassembler la cellule, ce qui entraîne une dégradation de l’ADN, un blebbing de la membrane, une fragmentation nucléaire et la libération de corps apoptotiques14,15. Ce processus se termine généralement par une forme non lytique et non inflammatoire de mort cellulaire lorsqu’il est associé à la clairance immédiate des cellules mourantes par efférocytose16. Cependant, des défauts d’efférocytose ou un manque de cellules phagocytaires peuvent entraîner l’accumulation de cellules apoptotiques, qui subissent alors la mort cellulaire lytique et inflammatoire17,18.
Les caspases inflammatoires, y compris la caspase-1 (humaine et souris), la caspase-4 et la caspase-5 (humaine) et la caspase-11 (souris), ont été découvertes pour être activées au cours d’une forme de PCD immunitaire innée inflammatoire (III-PCD) appelée pyroptose. L’activation de la caspase-1 est associée à la formation d’inflammasomes, qui sont des complexes multiprotéiques contenant un capteur immunitaire inné cytosolique, une molécule adaptatrice (protéine de type speck associée à l’apoptose contenant un CARD [ASC]) et de la caspase-1. La formation de ce complexe permet à la caspase-1 de subir une autoprotéolyse médiée par la proximité pour libérer sa forme active, qui peut cliver les substrats cibles, y compris les cytokines pro-inflammatoires interleukine (IL)-1β et IL-18 et la molécule formant des pores gasdermine D (GSDMD)19,20,21,22,23 . La caspase-11, la caspase-4 et la caspase-5 peuvent également activer la GSDMD sans la formation en amont de l’inflammasome après avoir détecté des PAMP tels que le lipopolysaccharide (LPS)19,20. Ces caspases subissent une dimérisation suivie d’une oligomérisation et d’un auto-clivage pour activation lors de la liaison au LPS cytosolique, ce qui conduit à l’activation non canonique de l’inflammasome 24,25,26 et à l’activation de la caspase-1 de manière cellulaire intrinsèque pour induire la maturation de l’IL-1β et de l’IL-18 20. La maturation et la libération de ces cytokines pro-inflammatoires caractérisent ces caspases comme « inflammatoires ». De plus, la caspase-8 apoptotique s’est localisée dans l’inflammasome, fournissant un lien entre les processus apoptotiques et pyroptotiques. Des études ont montré que la caspase-8 apoptotique est également essentielle pour réguler une autre forme de DCP appelée nécroptose. La perte de caspase-8 entraîne une activation spontanée de la pseudokinase mixte de type domaine de la lignée kinase 3 (RIPK3) médiée par la sérine-thréonine kinase (MLKL) pour piloter la voie III-PCD de nécroptose27,28,29,30,31,32,33,34,35.
Alors que les caspases ont toujours été classées comme « apoptotiques » ou « inflammatoires » en fonction du type de mort cellulaire qu’elles initient, de plus en plus de preuves suggèrent qu’il existe une diaphonie étendue entre les voies immunitaires innées de PCD à travers les caspases 3,4. Par exemple, la caspase-1 inflammatoire des inflammasomes clive la caspase-7 apoptotique à son site d’activation canonique34. L’activation de la caspase-1 peut également entraîner le clivage de substrats apoptotiques tels que la poly(ADP-ribose) polymérase 1 (PARP1)36. Dans les cellules dépourvues de GSDMD, la caspase-1 peut également cliver la caspase-337,38. De plus, la caspase-3 canoniquement apoptotique peut cliver la gasdermine E (GSDME) pour induire la DCP17,18 et transforme également la GSDMD en une forme inactive40. De plus, le recrutement de caspase-8 dans le complexe inflammasome a été observé 39,40,41,42,43,44,45, et la caspase-8 est un régulateur clé de l’activation canonique et non canonique de l’inflammasome 39. Il existe également des rôles chevauchants et redondants pour la caspase-8 et la caspase-1 dans de nombreuses conditions inflammatoires, et la PCD immunitaire innée caractérisée par l’activation de composants pyroptotiques, apoptotiques et nécroptotiques se produit dans tout le spectre de la maladie 39,46,47,48,49,50.
Sur la base de cette diaphonie entre les caspases inflammatoires et apoptotiques, une lacune clé dans la compréhension mécaniste de l’immunité innée et de la DCP a été identifiée, conduisant à la découverte de PANoptosis. PANoptosis est une forme unique de III-PCD qui est activée en réponse aux agents pathogènes, PAMPs, DAMP et altérations de l’homéostasie et est régulée par les PANoptosomes - complexes macromoléculaires multifacettes qui intègrent des composants d’autres voies de mort cellulaire 44,50,51,52,53,54,55 . La totalité des effets biologiques de PANoptosis ne peut pas être expliquée individuellement par la pyroptose, l’apoptose ou la nécroptose seule 3,4,35,36,39,46,47,48, car PANoptosis est caractérisée par l’activation de plusieurs caspases, y compris la caspase-1, la caspase-11, la caspase-8, la caspase-9, la caspase-3 et/ou la caspase-7, selon le contexte 44,48,49,50,51,52,53,54,56,57,58,59,60,61,62 . PANoptosis a été de plus en plus impliqué dans les maladies infectieuses et inflammatoires, ainsi que dans les cancers et les thérapies anticancéreuses 3,4,35,36,39,44,46,47,48,49,50,51,52,53 ,54,56
,57,58,59,60,61,62,63,64,65,66.
Compte tenu du rôle essentiel des caspases dans les voies de mort cellulaire, y compris dans l’apoptose, la pyroptose, la nécroptose et la panoptique, il est important de développer des techniques pour caractériser leur activation et comprendre toute la complexité des voies de DCP. Le protocole détaille ici une méthode pour stimuler les cellules et mesurer l’activation ultérieure des caspases (Figure 1). Cette méthode exploite le clivage protéolytique des caspases, qui est généralement nécessaire à leur activation, comme moyen de les étudier. Grâce au Western blotting, la taille des protéines peut être déterminée, ce qui permet de visualiser et de différencier clairement les pro-caspases inactives et leurs formes activées et clivées.
Les principaux avantages de ce protocole sont 1) sa capacité à évaluer l’activation de plusieurs caspases en parallèle à partir d’une seule population de cellules endogènes pour déterminer plus précisément l’activation des DCP et 2) l’utilisation de techniques de laboratoire relativement simples qui ne nécessitent pas de formation approfondie ou d’équipement coûteux. Les protocoles précédents ont utilisé le transfert Western, les rapporteurs fluorescents ou la coloration par anticorps pour surveiller l’activation des caspases dans les surnageants de culture, les lysats cellulaires et tissulaires, les cellules entières par microscopie et in vivo 67,68,69,70,71, mais ces techniques ne surveillent généralement qu’une ou deux caspases dans un échantillon. De plus, alors que des substrats peptidiques synthétiques contenant des sites de clivage de caspases fluorescentes lors du clivage ont été utilisés pour surveiller l’activation des caspases dans les lysats cellulaires ou tissulaires69, ces substrats peuvent souvent être clivés par plus d’une caspase, ce qui rend difficile la détermination de l’activation spécifique des caspases individuelles dans ce système. De plus, l’utilisation du Western blot plutôt que l’utilisation de rapporteurs fluorescents ou d’autres méthodes basées sur des marqueurs permet aux chercheurs d’utiliser des cellules endogènes plutôt que de créer des lignées cellulaires spécifiques avec des gènes rapporteurs. L’utilisation de cellules endogènes présente de multiples avantages, notamment le fait que de nombreuses lignées cellulaires immortalisées sont déficientes en molécules clés de mort cellulaire72,73, ce qui pourrait affecter les résultats. De plus, l’utilisation de cellules endogènes permet d’évaluer divers types de cellules, telles que les macrophages, les cellules épithéliales et les cellules endothéliales, plutôt qu’une seule lignée. Le Western blot est également une technique relativement simple et rentable qui peut être réalisée dans des laboratoires du monde entier sans avoir besoin de gros équipements coûteux ou de configurations compliquées.
Ce protocole est largement applicable à travers la biologie pour comprendre à la fois les fonctions dépendantes de la mort cellulaire et indépendantes de la mort cellulaire des caspases, y compris leurs rôles d’échafaudage et leurs fonctions dans d’autres voies de signalisation inflammatoires74. L’application de cette méthode permet une approche unifiée dans l’étude des voies immunitaires innées de la DCP et de la signalisation inflammatoire à travers les maladies et les affections, et ce protocole peut être utilisé pour identifier les processus de régulation critiques et les connexions mécanistes qui éclaireront le développement de futures stratégies thérapeutiques.
Protocole
L’utilisation et les procédures d’utilisation des animaux ont été approuvées par le comité sur l’utilisation et les soins des animaux de l’hôpital de recherche pour enfants St. Jude.
1. Préparer les solutions
- Préparez des supports conditionnés au L929.
- Plaque 1 × 106 cellules L929 (voir tableau des matières) dans une fiole de culture tissulaire de 182cm2 contenant 50 ml de milieu de culture L929 (voir tableau 1 pour la préparation des milieux).
- Cultiver les cellules dans un incubateur humidifié à 37 °C avec 5% de CO2.
- Après 7 jours, prélever le surnageant et filtrer à l’aide d’un filtre de 0,45 μm. Préparer des aliquotes de 50 ml (conserver les aliquotes congelées à −80 °C pendant une période maximale de 1 an).
- Préparer 500 mL de milieux de culture de macrophages dérivés de la moelle osseuse (BMDM) (tableau 1).
- Préparer 500 mL de milieu de stimulation BMDM (tableau 1).
- Préparer 500 mL de milieu pour l’infection (tableau 1).
- Préparer 100 mL de tampon Tris 1 M (tableau 1).
- Préparer 4x tampon de dodécylsulfate de sodium (SDS) (tableau 1).
- Préparer 1 mL de 1 M de 1,4-dithiothréitol (DTT, Tableau 1).
- Préparer 40 mL de tampon de lyse de caspase (tableau 1).
- Préparer 100 mL de tampon Tris 1,5 M (tableau 1).
- Préparer 100 mL d’une solution de FDS à 10 % (poids/volume) (tableau 1).
- Préparer 50 mL d’un tampon d’essai de radioimmunoprécipitation (RIPA) 2x (tableau 1).
- Préparer une solution de LPS à 5 mg/mL (tableau 1).
- Préparer une solution d’ATP 0,5 M (tableau 1).
- Préparer les solutions de transfert western.
- Préparer 1 L de 5x le stock régulateur courant (tableau 1).
- Préparer 1 L de 10x le stock régulateur de transfert (tableau 1).
- Préparer 1 L de solution saline tamponnée Tris avec Tween 20 (TBST, tableau 1).
- Préparer 100 mL d’une solution de blocage de lait écrémé à 5 % (poids/volume) (tableau 1).
- Préparer les solutions d’anticorps primaires.
- Préparer 10 mL d’anticorps primaire contre la caspase-1 (tableau 1).
- Préparer 10 mL d’anticorps primaire contre la caspase-11 (tableau 1).
- Préparer 10 mL d’anticorps primaire contre la caspase-3 (tableau 1).
- Préparer 10 mL d’anticorps primaire de caspase-7 (tableau 1).
- Préparer 10 mL d’anticorps primaires contre la caspase-8 (tableau 1).
- Préparer 10 mL d’anticorps primaire contre la caspase-9 (tableau 1).
- Préparer 10 mL d’anticorps primaire β-actine conjugué HRP (tableau 1).
- Préparer les solutions d’anticorps secondaires.
- Préparer 10 mL d’anticorps secondaire anti-lapin (tableau 1).
- Préparer 10 mL d’anticorps secondaire anti-souris (tableau 1).
- Préparer 10 mL d’anticorps secondaire anti-rat (tableau 1).
2. Isoler les macrophages dérivés de la moelle osseuse
REMARQUE: Pour ce protocole, des souris de type sauvage âgées de 6 à 10 semaines avec des voies PCD intactes ou des souris mutantes avec les régulateurs PCD, effecteurs ou molécules d’intérêt supprimés ou modifiés peuvent être utilisés.
- Euthanasier une souris dans une chambre de CO 2 avec un débit qui déplace 10% à 30% du volume de la cage par minute pendant2-3 min. Ensuite, effectuez une méthode d’euthanasie secondaire, telle que la luxation cervicale. Suivez toutes les autres lignes directrices et réglementations propres aux installations, aux institutions et au gouvernement, le cas échéant.
- Disséquez la souris pour récupérer les os de la patte postérieure.
- Épinglez la souris sur son dos afin que l’abdomen soit exposé. Vaporiser avec de l’éthanol à 70 % (vol/vol) pour stériliser les pattes postérieures et l’abdomen.
ATTENTION : L’éthanol est inflammable. Gardez-le à l’écart des flammes nues. - Utilisez des ciseaux pour faire une incision à la ligne médiane de l’abdomen; Continuez à couper vers les pattes pour rendre les fémurs visibles.
- Prenez la jambe arrière droite et retirez la peau du corps vers la ligne médiane. Détacher la jambe du corps en sectionnant les muscles adducteurs vers la ligne médiane; Ensuite, coupez la jambe entre l’articulation de la hanche et la colonne vertébrale. Ensuite, coupez la patte distale à la cheville et retirez l’excès de tissu de l’os en décollant la peau et en utilisant des ciseaux légèrement ouverts pour enlever le tissu du mollet.
- Placez la jambe sur une serviette imbibée d’éthanol à 70% (vol/vol) et disséquez le tibia et le fémur.
- Serrez le tibia et le fémur chacun avec un jeu séparé de forceps. Appuyez doucement le tibia contre la direction naturelle de l’articulation du genou; Cela provoquera la rupture du tibia au niveau du genou.
- Utilisez les pinces et les ciseaux à dissection si nécessaire pour enlever tout tissu suspendu restant. Conservez le tibia pour une utilisation ultérieure en le plaçant sur la serviette imbibée d’éthanol.
- Recueillez le fémur de la même manière, en arrachant le genou.
- Répétez les étapes 3 et 4 ci-dessus pour la jambe gauche afin de la retirer du corps et de disséquer le tibia et le fémur.
- Pulvériser les os avec de l’éthanol à 70% (vol/vol).
- Nettoyez les os en les plaçant sur une serviette propre imbibée d’éthanol à 70% (vol/vol), en pressant la partie charnue de la serviette et en frottant la serviette contre l’os pour enlever l’excès de tissu.
- Épinglez la souris sur son dos afin que l’abdomen soit exposé. Vaporiser avec de l’éthanol à 70 % (vol/vol) pour stériliser les pattes postérieures et l’abdomen.
- Une fois que les fémurs et les deux tibias sont propres, vaporiser les quatre os avec de l’éthanol à 70 % (vol/vol). Recueillir les os dans une boîte de Petri stérile et les rincer avec 10 ml de milieu de culture BMDM en remuant doucement le média dans la boîte.
- Remplissez une seringue de 10 mL avec 10 mL de milieu de culture BMDM frais et fixez une aiguille de 25 G.
- Ramasser le tibia à l’aide d’une pince; Ensuite, coupez l’articulation de la cheville à un angle de ~45 °.
- Rincer la moelle osseuse du tibia.
- Tenez le tibia au-dessus d’un tube de 50 mL, l’extrémité étroite du tibia pointant vers le bas. Distribuer le média sur le tibia de la seringue remplie.
- Insérez l’aiguille (doucement au début) à l’extrémité supérieure de la moelle et distribuez le média.
- Retirez l’aiguille, puis insérez-la à nouveau. Utilisez des poussées courtes et à haute pression pour distribuer le média et déplacer l’aiguille dans la moelle.
- Une fois que le média commence à s’écouler du bas de l’os, utilisez des poussées courtes et à haute pression pour continuer à rincer les cellules. Surveillez la couleur de l’os pendant ce processus, et lorsque l’os est blanc, jetez-le.
- Faites-le pour les deux tibias.
- Répétez les étapes 5 et 6 ci-dessus pour les fémurs, en coupant à l’articulation de la hanche lorsque le tibia a été coupé à l’articulation de la cheville. Utilisez le même tube de 50 ml pour recueillir le milieu de culture BMDM pendant le rinçage de la moelle.
- Une fois que les quatre os ont été rincés, aspirer la moelle et le média du tube de 50 ml de haut en bas 3x à l’aide d’une aiguille de 18 g sur une seringue de 10 ml, en rinçant les côtés du tube chaque fois pour disperser la moelle.
- Ajustez le volume final dans le tube de 50 mL à 30 mL avec le milieu de culture BMDM et assurez-vous que les cellules sont bien suspendues.
- Utilisez une passoire cellulaire de 70 μm pour filtrer le milieu de culture BMDM du tube de 50 mL.
- Plaquer la suspension cellulaire résultante, qui contient les cellules progénitrices de la moelle osseuse, dans trois boîtes de culture tissulaire de 150 mm en ajoutant 10 ml (ou ~20 × 106 cellules) à chacune. Ensuite, ajoutez 10 ml supplémentaires de milieu de culture BMDM à chaque boîte. Incuber dans un incubateur humidifié à 37 °C.
3. Différenciation des BMDM et du placage pour les expériences
- Incuber les cellules progénitrices de la moelle osseuse plaquées à 37 °C pendant 3 jours. Ensuite, retirez chaque plat et ajoutez 5 à 8 mL supplémentaires de milieu de culture BMDM (tableau 1). Retour à l’incubateur à 37 °C.
- Le jour 5 après le placage initial, retirer chaque plat et ajouter 5 ml supplémentaires de milieu de culture BMDM. Retour à l’incubateur à 37 °C.
- Le jour 6, retirez chaque plat et jetez le support. Ensuite, ajoutez 10 ml de PBS froid (conservé à 4 °C) à laver une fois. Jetez les 10 ml de lavage PBS froid. Ensuite, ajoutez 10 ml de PBS frais et froid à chaque plat et incuber chaque plat sur de la glace pendant 5 minutes.
- À l’aide d’un grattoir de cellules, gratter délicatement les cellules des trois boîtes dans un tube de 50 ml. Faire tourner doucement les cellules à 270 × g à 4 °C pendant 5 min; Ensuite, jetez le surnageant.
- Ajouter 20 ml de milieu de culture BMDM, pipeter de haut en bas pour remettre en suspension la pastille et compter les cellules.
REMARQUE : On s’attend à ce que chaque souris produise environ 60 × 10 6-100 × 106 cellules. - Planifiez la disposition des plaques à 12 puits pour le test de stimulation de la mort cellulaire / inflammation in vitro souhaité. Prévoyez de plaquer 1 × 106 cellules par puits. Pour chaque stimulation prévue, inclure au moins trois réplications biologiques et plaquer un ensemble de puits à récolter dans un tampon de lyse de caspase et un deuxième ensemble de puits à récolter dans un tampon RIPA.
- Plaque 1 × 106 cellules dans 1 mL de milieu de culture BMDM par puits dans des plaques de 12 puits. Culture pendant la nuit dans un incubateur humidifié à 37 °C avant de procéder à l’évaluation de la mort cellulaire / inflammation. Après une nuit d’incubation, retirez le média et ajoutez 1 mL de PBS chaud (37 °C) à chaque puits pour laver les cellules.
- Retirez le lavage PBS et ajoutez 500 μL de milieu de stimulation BMDM avec des antibiotiques (si vous effectuez des stimulations non bactériennes) ou un milieu de stimulation BMDM sans antibiotiques (si vous effectuez des stimulations bactériennes) (tableau 1). Incuber pendant 2 h avant de passer à l’étape 4 pour la stimulation / infection in vitro .
4. Stimuler ou infecter les cellules
ATTENTION : Les agents inclus dans ce protocole sont potentiellement pathogènes et doivent être manipulés avec les précautions appropriées dans une installation de niveau de biosécurité 2 (BSL2) avec l’approbation des autorités institutionnelles et gouvernementales compétentes.
- Stimuler les BMDM pour activer la mort cellulaire avec le déclencheur d’intérêt.
NOTA : Aux fins du présent protocole, virus de la grippe A (IAV), virus de l’herpès simplex 1 (HSV1), Francisella novicidaet LPS + ATP sont utilisés à titre d’illustration, mais d’autres déclencheurs peuvent être utilisés.- Exemple de stimulation 1 : Infecter par le IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (construit selon Hoffmann et al.75; le titre viral pour la détermination de la multiplicité de l’infection [MOI] est calculé par un dosage de la plaque dans les cellules MDCK) :
- Calculer le volume de virus nécessaire pour l’infection à une multiplicité d’infection (MOI) de 20 unités formant des plaques (UFP) à l’aide de l’équation (1) et de l’équation (2) :

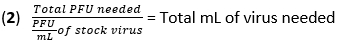

- Retirez le média des BMDM et lavez les cellules une fois avec 500 μL de PBS. Ajouter 450 μL d’IAV (20 MOI) dans du DMEM à haute teneur en glucose sans HFS-FBS inactivé par la chaleur à chaque puits. Incuber les plaques à 37 °C pendant 1 h dans un incubateur humidifié pour permettre l’absorption.
- Retirez les plaques et ajoutez 50 μL de HI-FBS. Remettre les plaques dans l’incubateur à 37 °C. Incuber pour un total de 12 h.
- Calculer le volume de virus nécessaire pour l’infection à une multiplicité d’infection (MOI) de 20 unités formant des plaques (UFP) à l’aide de l’équation (1) et de l’équation (2) :
- Exemple de stimulation 2 : Infecter avec HSV1 (souche HF; cultivée comme décrit précédemment44; le titre viral pour la détermination de MOI est calculé par un dosage de plaque dans les cellules Vero):
- Calculer le volume de virus nécessaire pour l’infection à un MO de 10 PFU en utilisant l’équation (1) et l’équation (2) de l’étape 1 de la section sur l’infection par le VAI ci-dessus.

- Retirez le support des BMDM. Ajouter 450 μL de HSV1 (MOI 10) dans du DMEM à haute teneur en glucose sans HI-FBS à chaque puits. Incuber les plaques à 37 °C pendant 1 h dans un incubateur humidifié pour permettre l’absorption.
- Retirez les plaques et ajoutez 50 μL de HI-FBS. Remettre les plaques dans l’incubateur à 37 °C. Incuber pour un total de 12 h.
- Calculer le volume de virus nécessaire pour l’infection à un MO de 10 PFU en utilisant l’équation (1) et l’équation (2) de l’étape 1 de la section sur l’infection par le VAI ci-dessus.
- Exemple de stimulation 3 : Infecter par F. novicida (souche U112; cultivé comme décrit précédemment44 dans des conditions aérobies à 37 °C dans un bouillon de soja BBL Trypticase supplémenté en L-cystéine à 0,2% pendant la nuit. Ensuite, sous-culture des bactéries dans un rapport de 1:10 à 37 °C pendant encore 4 heures dans un milieu frais avant de prélever la densité optique (DO) à 600 nm en utilisant le milieu frais comme blanc. Une valeur de DO de 1 équivaut à 1 × 109 unités formant colonies (UFC) par mL.)
- Calculer le volume de F. novicida nécessaire pour l’infection à une MOI de 50 UFC à l’aide de l’équation (3) et de l’équation (4) :

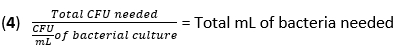
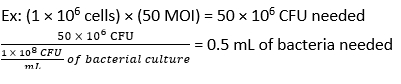
- Retirez le média des BMDM. Ajouter 500 μL de F. novicida (MOI 50) dans un milieu de stimulation BMDM sans antibiotiques à chaque puits. Incuber les plaques à 37 °C pendant 4 h dans un incubateur humidifié pour permettre l’absorption.
- Laver les cellules trois fois avec du PBS chauffé (37 °C) et ajouter 500 μL de milieu de stimulation BMDM contenant 50 μg/mL de gentamycine. Remettre les plaques dans l’incubateur à 37 °C. Incuber pendant la nuit (16 h).
- Calculer le volume de F. novicida nécessaire pour l’infection à une MOI de 50 UFC à l’aide de l’équation (3) et de l’équation (4) :
- Exemple de stimulation 4 : Stimuler avec LPS + ATP.
- Retirez le média des BMDM. Ajouter 500 μL de milieu de stimulation BMDM avec des antibiotiques (tableau 1) contenant 100 ng/mL de LPS à chaque puits. Incuber les plaques à 37 °C pendant 3,5 h dans un incubateur humidifié.
- Ajouter 5 μL de solution mère d’ATP 0,5 M (tableau 1) à chaque puits. Remettre les plaques dans l’incubateur à 37 °C. Incuber pendant 30 min.
- Exemple de stimulation 1 : Infecter par le IAV (A/Puerto Rico/8/34, H1N1 [PR8]) (construit selon Hoffmann et al.75; le titre viral pour la détermination de la multiplicité de l’infection [MOI] est calculé par un dosage de la plaque dans les cellules MDCK) :
5. Collecte du surnageant et du lysat protéique combinés à utiliser pour les transferts Western de caspase
- Après 4 h, 12 h ou 16 h d’incubation (le moment spécifique dépend de la gâchette utilisée), retirez la plaque de l’incubateur.
- Retirer 150 μL du surnageant; jeter ou conserver ceci pour d’autres analyses surnageantes (p. ex., dosage immunoenzymatique [ELISA]). Ne retirez pas le surnageant restant.
- Créer la solution de collecte de protéines en combinant 50 μL de tampon de lyse de caspase + 100 μL de 4x tampon SDS par puits (tableau 1). Ensuite, ajoutez 150 μL du mélange à chaque puits.
- Pour chaque puits, pipeter le mélange de haut en bas pour recueillir les cellules lysées et le surnageant. Pendant le pipetage, grattez également le fond du puits avec l’extrémité de la pipette pour perturber les cellules. Après le grattage et le pipetage, recueillir le lysat de protéines dans des tubes marqués de 1,5 mL.
- Utilisez un bloc de chaleur pour chauffer tous les tubes à 100 °C pendant 12 min.
- Ensemencer tous les composants insolubles par centrifugation à 14 500 × g pendant 30 s à température ambiante.
REMARQUE: Il s’agit d’un point de pause - la protéine du surnageant et des lysats protéiques combinés peut être utilisée immédiatement ou stockée à -20 ° C jusqu’à 2 mois ou à -80 ° C jusqu’à 6 mois jusqu’à ce qu’elle soit prête à être utilisée.
6. Collecte du lysat protéique à utiliser pour les transferts Western de caspase
- Après 4 h, 12 h ou 16 h d’incubation (le moment spécifique dépend de la gâchette utilisée), retirez la plaque de l’incubateur. Retirez tout le surnageant; jeter ou conserver ceci pour d’autres analyses surnageantes (par exemple, ELISA).
- Créez le tampon 1x RIPA en diluant la solution mère 2x RIPA (Tableau 1) dans un volume égal d’eau désionisée. Ensuite, ajoutez un comprimé inhibiteur de phosphatase et un comprimé inhibiteur de protéase et laissez-les se dissoudre. Ajouter 150 μL de 1x tampon RIPA et 50 μL de 4x SDS à chaque puits.
- Pour chaque puits, pipeter le mélange de haut en bas pour recueillir les cellules lysées. Pendant le pipetage, grattez également le fond du puits avec l’extrémité de la pipette pour perturber les cellules. Après le grattage et le pipetage, recueillir le lysat de protéines dans des tubes marqués de 1,5 mL.
- Utilisez un bloc de chaleur pour chauffer tous les tubes à 100 °C pendant 12 min.
- Ensemencer tous les composants insolubles par centrifugation à 14 500 × g pendant 30 s à température ambiante.
REMARQUE: Il s’agit d’un point de pause - les lysats de protéines peuvent être utilisés immédiatement ou stockés à -20 ° C ou -80 ° C jusqu’à ce qu’ils soient prêts à être utilisés.
7. Effectuer le Western blot en utilisant les lysats prélevés sur les BMDM en suivant les étapes ci-dessus ou sur des homogénats tissulaires
REMARQUE: Si vous utilisez du tissu, il peut être homogénéisé à la main ou à l’aide d’un homogénéisateur de tissu motorisé. Le protocole de Simpson76 fournit une description détaillée de l’homogénéisation tissulaire.
- Préparer 1x tampon de fonctionnement : Combiner 200 mL du stock tampon 5x (tableau 1) et 800 mL d’eau désionisée. Effectuez ce tampon 1x juste avant chaque expérience.
- Préparer l’appareil d’électrophorèse avec un gel de polyacrylamide à 12% (poids / vol) avec 10 puits. Remplissez l’appareil d’électrophorèse avec 1x tampon courant. Ensuite, retirez le peigne de gel.
REMARQUE: Pour analyser la caspase-1, la caspase-11, la caspase-3, la caspase-7, la caspase-8 et la caspase-9 pour chaque échantillon, six gels seront nécessaires. - Pour les taches de caspase-1, de caspase-3, de caspase-7 et de caspase-8, prévoyez d’utiliser 30 μL du surnageant combiné et du lysat protéique dans le tampon de lyse de la caspase ou l’homogénat tissulaire. Pour les taches de caspase-11 et de caspase-9, prévoyez d’utiliser 20 μL du lysat de protéine dans le tampon RIPA ou l’homogénat tissulaire. Si les échantillons ont été entreposés à -20 °C ou −80 °C, décongelez-les d’abord sur de la glace.
- Pour tous les échantillons, chauffer à 100 °C pendant 5 min et centrifuger à 14 500 × g pendant 30 s à 4 °C avant chargement. Ensuite, chargez lentement 20 ou 30 μL de l’échantillon dans chaque voie. Évitez tout débordement d’échantillon dans les autres voies. Pour évaluer les six caspases à la fois, utilisez la même procédure pour charger les échantillons appropriés dans chacun des six gels.
- Connectez l’appareil d’électrophorèse à la source d’alimentation. Ensuite, réglez l’alimentation sur 80 V pendant 20 minutes pour commencer le fonctionnement du gel, puis réglez la puissance à 130 V pendant 45 à 60 minutes.
- Surveillez attentivement le front de colorant et éteignez l’alimentation lorsque le front de colorant se trouve au fond du gel mais n’a pas encore été expulsé du gel.
- Pendant que le gel fonctionne, préparer 1x tampon de transfert en combinant 700 mL d’eau désionisée, 100 mL du stock tampon de transfert 10x (tableau 1) et 200 mL de méthanol. Préparez la solution 1x fraîche à chaque fois.
REMARQUE : Soyez prudent car le méthanol est inflammable. Effectuer la préparation du tampon de transfert loin des flammes nues. - Retirez doucement le gel de l’appareil d’électrophorèse à l’aide du libérateur de gel.
- Configurez une pile de transfert pour chaque gel.
- Activer une membrane PVDF en la trempant dans du méthanol pendant 1 min.
- Pré-mouiller deux morceaux de papier filtre, le gel et la membrane PVDF dans le tampon de transfert pendant 5 min. Conservez la membrane et le gel en PVDF dans des récipients séparés pendant cette incubation de 5 minutes.
- Assemblez la pile de transfert sur le système semi-sec. En commençant par le côté inférieur de l’anode en platine, placez un morceau de papier filtre, la membrane PVDF, le gel, et enfin un morceau de papier filtre. Étalez doucement ou pressez les bulles d’air entre les couches et fermez le haut du système. Assurez-vous que la couverture de sécurité est sécurisée avant de continuer.
- Connectez-vous à la source d’alimentation. Réglez l’alimentation sur 25 V pendant 45 min.
- Une fois le transfert terminé, démonter la pile de transfert et recueillir la membrane; placez-le dans une boîte de Petri carrée (plateau d’incubation).
- Effectuer le blocage de la membrane en ajoutant 15 mL d’une solution de lait écrémé à 5 % (poids/volume) (tableau 1). Incuber la membrane sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 1 h.
REMARQUE: Il s’agit d’un point de pause - la membrane peut être retirée après 1 h ou stockée dans une solution de blocage à 4 ° C pendant la nuit. - Après 1 h ou une nuit d’incubation, retirez la solution bloquante. Ajouter 10 mL de la solution d’anticorps dilués (anticorps anti-caspase-1, anticorps anti-caspase-11, anticorps combiné anti-caspase-3 et anti-caspase-3 clivée, anticorps combiné anti-caspase-7 et anti-caspase-7 clivé, anticorps combiné anti-caspase-8 et anti-caspase-8, ou anticorps anti-caspase-9) (tableau 1). Placer sur un agitateur à bascule à 50 rpm à 70 rpm pour incuber à température ambiante pendant 2 h ou à 4 °C pendant la nuit (16 h).
- Prélever la solution d’anticorps (réutiliser jusqu’à 3x ou la jeter) et laver en ajoutant 15 mL de TBST (tableau 1) à la membrane sur un agitateur à bascule à 50 tr/min à 70 tr/min à température ambiante pendant 10 min. Jetez le TBST.
- Répétez le lavage avec 15 mL de TBST après l’étape 14 un total de 3x.
- Ajouter 10 mL de la solution diluée d’anticorps conjugués HRP secondaire (anti-lapin pour les transferts colorés avec des anticorps primaires contre la caspase-3, la caspase-7, la caspase-8 ou la caspase-9; anti-souris pour les transferts colorés avec des anticorps primaires contre la caspase-1; anti-rat pour les transferts colorés avec des anticorps primaires contre la caspase-11) (tableau 1). Incuber sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 1 h.
- Retirer la solution d’anticorps et laver en ajoutant 15 mL de TBST à la membrane sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 10 min. Jetez le TBST.
- Répétez le lavage avec 15 mL de TBST après l’étape 17 un total de 3x.
- Ajouter 10 mL du substrat HRP haute sensibilité à la membrane. Laissez-le reposer à température ambiante dans l’obscurité pendant 1 min.
- Retirez la membrane du substrat. Passez directement à l’imagerie, à l’aide d’un imageur à chimiluminescence avec l’accessoire blanc trans tray inséré en position inférieure. Exposez la membrane en utilisant le mode d’exposition automatique (généralement ~1-2 min de temps d’exposition).
- À l’aide de la membrane du buvardage de caspase-9 ou de caspase-11 (c.-à-d. une membrane avec des échantillons de lysat RIPA), ajouter 10 ml de tampon de décapage et incuber sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 5 min.
- Jeter le tampon de décapage et laver en ajoutant 15 ml de TBST à la membrane sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 10 min. Jetez le TBST.
- Répétez le lavage avec 15 mL de TBST après l’étape 22 à un total de 3x.
- Effectuer le blocage de la membrane en ajoutant 15 mL d’une solution de lait écrémé à 5 % (poids/vol). Incuber la membrane sur un agitateur à bascule à 50 tr/min à 70 rpm à température ambiante pendant 1 h.
REMARQUE: Il s’agit d’un point de pause - la membrane peut être retirée après 1 h ou stockée dans une solution de blocage à 4 ° C pendant la nuit. - Après 1 h ou une nuit d’incubation, retirez la solution bloquante. Ajouter 10 mL de la solution d’anticorps anti-β-actine (conjuguée HRP) diluée. Placer sur un agitateur à bascule à 50 tr/min à 70 tr/min pour incuber à température ambiante pendant 1,5 h.
- Retirer la solution d’anticorps et laver en ajoutant 15 mL de TBST à la membrane sur un agitateur à bascule à 50 rpm à 70 rpm à température ambiante pendant 10 min. Jetez le TBST.
- Répétez le lavage avec 15 mL de TBST après l’étape 26 un total de 3x.
- Ajouter 10 mL du substrat HRP à sensibilité standard à la membrane. Laissez-le reposer à température ambiante dans l’obscurité pendant 1 min.
- Passez directement à l’imagerie, à l’aide d’un imageur à chimiluminescence avec l’accessoire blanc trans tray inséré en position inférieure. Exposez la membrane en utilisant le mode d’exposition automatique (généralement <1 min de temps d’exposition).
Résultats
PANoptosis a été observé en réponse à de nombreuses infections bactériennes, virales et fongiques et à d’autres stimuli inflammatoires, ainsi que dans des cellules cancéreuses 44,48,49,50,51,52,53,54,56,57,58,60,61,62 ...
Discussion
La surveillance du clivage et de l’activation des caspases fournit l’une des images les plus complètes de l’activation immunitaire innée de la DCP dans le cadre de la réponse immunitaire innée. Le protocole décrit ici démontre une stratégie de surveillance de l’activation des caspases en réponse aux infections IAV, HSV1 et F. novicida et au déclencheur stérile LPS + ATP, mais de nombreux autres stimuli peuvent induire la DCP et pourraient être utilisés dans cette méthode, comme cela a été ...
Déclarations de divulgation
T.-D.K. est consultant pour Pfizer.
Remerciements
Nous remercions les membres du laboratoire Kanneganti pour leurs commentaires et suggestions, et nous remercions J. Gullett, Ph.D., pour son soutien à l’édition scientifique. Le travail dans notre laboratoire est soutenu par les subventions AI101935, AI124346, AI160179, AR056296 et CA253095 (à T.-D.K.) des National Institutes of Health (NIH) et par les American Lebanese Syrian Associated Charities (à T.-D.K.). Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les opinions officielles des National Institutes of Health.
matériels
| Name | Company | Catalog Number | Comments |
| 0.45 μm filter | Millipore | SCHVU05RE | |
| 10 mL syringe | BD Biosciences | 309604 | |
| 12% polyacrylamide gel with 10 wells | Bio-Rad | 4561043 | |
| 12-well plate | Corning | 07-200-82 | |
| 18 G needle | BD Biosciences | 305195 | |
| 25 G needle | BD Biosciences | 305122 | |
| 50 mL tube | Fisher Scientific | 50-809-218 | |
| 70 μm cell strainer | Corning | 431751 | |
| 150 mm tissue culture dishes | Corning | 430597 | |
| 182-cm2 tissue culture flask | Genesee Scientific | 25-211 | |
| Accessory white trans tray | Cytiva | 29-0834-18 | |
| Anti–caspase-1 antibody | AdipoGen | AG-20B-0042-C100 | |
| Anti–caspase-11 antibody | Novus Biologicals | NB120-10454 | |
| Anti–caspase-3 antibody | Cell Signaling Technology | 9662 | |
| Anti–caspase-7 antibody | Cell Signaling Technology | 9492 | |
| Anti–caspase-8 antibody | Cell Signaling Technology | 4927 | |
| Anti–caspase-9 antibody | Cell Signaling Technology | 9504 | |
| Anti–cleaved caspase-3 antibody | Cell Signaling Technology | 9661 | |
| Anti–cleaved caspase-7 antibody | Cell Signaling Technology | 9491 | |
| Anti–cleaved caspase-8 antibody | Cell Signaling Technology | 8592 | |
| Anti-mouse HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 315-035-047 | |
| Anti-rabbit HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 111-035-047 | |
| Anti-rat HRP-conjugated secondary antibody | Jackson ImmunoResearch Laboratories | 112-035-003 | |
| Anti–β-Actin antibody (C4) HRP | Santa Cruz | sc-47778 HRP | |
| ATP | InvivoGen | tlrl-atpl | |
| BBL Trypticase Soy Broth | BD Biosciences | 211768 | |
| Bead bath | Chemglass Life Sciences | CLS-4598-009 | |
| Biophotometer D30 | Eppendorf | 6133000010 | |
| BME | Sigma | M6250 | |
| Bromophenol blue | Sigma | BO126 | |
| Cell scrapers | CellTreat Scientific Products | 229315 | |
| Chemiluminescence imager (Amersham 600) | Cytiva | 29083461 | |
| CO2 chamber | VetEquip | 901703 | |
| Cuvettes | Fisher Scientific | 14-955-129 | |
| Dissecting scissors | Thermo Fisher Scientific | 221S | |
| DMEM | Thermo Fisher Scientific | 11995-073 | |
| DTT | Sigma | 43815 | |
| Eelectrophoresis apparatus | Bio-Rad | 1658004 | |
| Ethanol | Pharmco | 111000200 | |
| Fetal bovine serum | Biowest | S1620 | |
| Filter paper | Bio-Rad | 1703965 | |
| Forceps | Fisher Scientific | 22-327379 | |
| Francisella novicida (U112 strain) | BEI Resources | NR-13 | |
| Gel releaser | Bio-Rad | 1653320 | |
| Gentamycin | Gibco | 15750060 | |
| Glycerol | Sigma | G7893 | |
| Glycine | Sigma | G8898 | |
| HCl | Sigma | H9892 | |
| Heat block | Fisher Scientific | 23-043-160 | |
| Herpes simplex virus 1 (HF strain) | ATCC | VR-260 | |
| High glucose DMEM | Sigma | D6171 | |
| Human anti–caspase-1 antibody | R&D Systems | MAB6215 | |
| Human anti–caspase-8 antibody | Enzo | ALX-804-242 | |
| Humidified incubator | Thermo Fisher Scientific | 51026282 | |
| Image analysis software | ImageJ | v1.53a | |
| IMDM | Thermo Fisher Scientific | 12440-053 | |
| Influenza A virus (A/Puerto Rico/8/34, H1N1 [PR8]) | constructed per Hoffmann et al. | ||
| L929 cells | ATCC | CCL-1 | cell line for creating L929-conditioned media |
| L-cysteine | Thermo Fisher Scientific | BP376-100 | |
| Luminata Forte Western HRP substrate | Millipore | WBLUF0500 | standard-sensitivity HRP substrate |
| MDCK cells | ATCC | CCL-34 | cell line for determining IAV viral titer |
| Methanol | Sigma | 322415 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002401 | |
| Non-essential amino acids | Gibco | 11140050 | |
| Nonfat dried milk powder | Kroger | ||
| NP-40 solution | Sigma | 492016 | |
| PBS | Thermo Fisher Scientific | 10010023 | |
| Penicillin and streptomycin | Sigma | P4333 | |
| Petri dish | Fisher Scientific | 07-202-011 | |
| PhosSTOP | Roche | PHOSS-RO | |
| Power source | Bio-Rad | 164-5052 | |
| Protease inhibitor tablet | Sigma | S8820 | |
| PVDF membrane | Millipore | IPVH00010 | |
| Rocking shaker | Labnet | S2035-E | |
| SDS | Sigma | L3771 | |
| Sodium chloride | Sigma | S9888 | |
| Sodium deoxycholate | Sigma | 30970 | |
| Sodium hydroxide | Sigma | 72068 | |
| Sodium pyruvate | Gibco | 11360-070 | |
| Square Petri dish | Fisher Scientific | FB0875711A | |
| Stripping buffer | Thermo Fisher Scientific | 21059 | |
| Super Signal Femto HRP substrate | Thermo Fisher Scientific | 34580 | high-sensitivity HRP substrate |
| Tabletop centrifuge | Thermo Fisher Scientific | 75004524 | |
| Trans-Blot semi-dry system | Bio-Rad | 170-3940 | |
| Tris | Sigma | TRIS-RO | |
| Tween 20 | Sigma | P1379 | |
| Ultrapure lipopolysaccharide (LPS) from E. coli 0111:B4 | InvivoGen | tlrl-3pelps | |
| Vero cells | ATCC | CCL-81 | cell line for determining HSV1 viral titer |
Références
- Alnemri, E. S., et al. Human ICE/CED-3 protease nomenclature. Cell. 87 (2), 171 (1996).
- Man, S. M., Kanneganti, T. D. Converging roles of caspases in inflammasome activation, cell death and innate immunity. Nature Reviews Immunology. 16 (1), 7-21 (2016).
- Gullett, J. M., Tweedell, R. E., Kanneganti, T. D. It's all in the PAN: Crosstalk, plasticity, redundancies, switches, and interconnectedness encompassed by PANoptosis underlying the totality of cell death-associated biological effects. Cells. 11 (9), 1495 (2022).
- Pandian, N., Kanneganti, T. D. PANoptosis: A unique innate immune inflammatory cell death modality. Journal of Immunology. 209 (9), 1625-1633 (2022).
- Galluzzi, L., et al. Molecular mechanisms of cell death recommendations of the Nomenclature Committee on Cell Death 2018. Cell Death & Differentiation. 25 (3), 486-541 (2018).
- Shi, Y. Caspase activation: Revisiting the induced proximity model. Cell. 117 (7), 855-858 (2004).
- Galluzzi, L., Lopez-Soto, A., Kumar, S., Kroemer, G. Caspases connect cell-death signaling to organismal homeostasis. Immunity. 44 (2), 221-231 (2016).
- Fernandes-Alnemri, T., Litwack, G., Alnemri, E. S. CPP32, a novel human apoptotic protein with homology to Caenorhabditis elegans cell death protein Ced-3 and mammalian interleukin-1 beta-converting enzyme. Journal of Biological Chemistry. 269 (49), 30761-30764 (1994).
- Tewari, M., et al. Yama/CPP32 beta, a mammalian homolog of CED-3, is a CrmA-inhibitable protease that cleaves the death substrate poly(ADP-ribose) polymerase. Cell. 81 (5), 801-809 (1995).
- Nicholson, D. W., et al. Identification and inhibition of the ICE/CED-3 protease necessary for mammalian apoptosis. Nature. 376 (6535), 37-43 (1995).
- Stennicke, H. R., et al. Pro-caspase-3 is a major physiologic target of caspase-8. Journal of Biological Chemistry. 273 (42), 27084-27090 (1998).
- Twiddy, D., Cohen, G. M., Macfarlane, M., Cain, K. Caspase-7 is directly activated by the approximately 700-kDa apoptosome complex and is released as a stable XIAP-caspase-7 approximately 200-kDa complex. Journal of Biological Chemistry. 281 (7), 3876-3888 (2006).
- Kesavardhana, S., Malireddi, R. K. S., Kanneganti, T. D. Caspases in cell death, inflammation, and pyroptosis. Annual Reviews of Immunology. 38, 567-595 (2020).
- Kerr, J. F., Wyllie, A. H., Currie, A. R. Apoptosis: a basic biological phenomenon with wide-ranging implications in tissue kinetics. British Journal of Cancer. 26 (4), 239-257 (1972).
- Taylor, R. C., Cullen, S. P., Martin, S. J. Apoptosis: Controlled demolition at the cellular level. Nature Reviews Molecular Cell Biology. 9 (3), 231-241 (2008).
- Morioka, S., Maueröder, C., Ravichandran, K. S. Living on the edge: Efferocytosis at the interface of homeostasis and pathology. Immunity. 50 (5), 1149-1162 (2019).
- Wang, Y., et al. Chemotherapy drugs induce pyroptosis through caspase-3 cleavage of a gasdermin. Nature. 547 (7661), 99-103 (2017).
- Rogers, C., et al. Cleavage of DFNA5 by caspase-3 during apoptosis mediates progression to secondary necrotic/pyroptotic cell death. Nature Communications. 8, 14128 (2017).
- Kayagaki, N., et al. Caspase-11 cleaves gasdermin D for non-canonical inflammasome signalling. Nature. 526 (7575), 666-671 (2015).
- Shi, J., Gao, W., Shao, F. Pyroptosis: Gasdermin-mediated programmed necrotic cell death. Trends in Biochemical Sciences. 42 (4), 245-254 (2017).
- Sborgi, L., et al. GSDMD membrane pore formation constitutes the mechanism of pyroptotic cell death. EMBO Journal. 35 (16), 1766-1778 (2016).
- Aglietti, R. A., et al. GsdmD p30 elicited by caspase-11 during pyroptosis forms pores in membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (28), 7858-7863 (2016).
- Hagar, J. A., Powell, D. A., Aachoui, Y., Ernst, R. K., Miao, E. A. Cytoplasmic LPS activates caspase-11: implications in TLR4-independent endotoxic shock. Science. 341 (6151), 1250-1253 (2013).
- Kayagaki, N., et al. Noncanonical inflammasome activation by intracellular LPS independent of TLR4. Science. 341 (6151), 1246-1249 (2013).
- Shi, J., et al. Inflammatory caspases are innate immune receptors for intracellular LPS. Nature. 514 (7521), 187-192 (2014).
- Lamkanfi, M., et al. Targeted peptidecentric proteomics reveals caspase-7 as a substrate of the caspase-1 inflammasomes. Molecular & Cellular Proteomics. 7 (12), 2350-2363 (2008).
- Kalai, M., et al. Tipping the balance between necrosis and apoptosis in human and murine cells treated with interferon and dsRNA. Cell Death & Differentiation. 9 (9), 981-994 (2002).
- Li, C., et al. Development of atopic dermatitis-like skin disease from the chronic loss of epidermal caspase-8. Proceedings of the National Academy of Sciences of the United States of America. 107 (51), 22249-22254 (2010).
- Kovalenko, A., et al. Caspase-8 deficiency in epidermal keratinocytes triggers an inflammatory skin disease. Journal of Experimental Medicine. 206 (10), 2161-2177 (2009).
- Kang, T. B., et al. Caspase-8 serves both apoptotic and nonapoptotic roles. Journal of Immunology. 173 (5), 2976-2984 (2004).
- Oberst, A., et al. Catalytic activity of the caspase-8-FLIP(L) complex inhibits RIPK3-dependent necrosis. Nature. 471 (7338), 363-367 (2011).
- Kaiser, W. J., et al. RIP3 mediates the embryonic lethality of caspase-8-deficient mice. Nature. 471 (7338), 368-372 (2011).
- Zhang, H., et al. Functional complementation between FADD and RIP1 in embryos and lymphocytes. Nature. 471 (7338), 373-376 (2011).
- Shi, J., et al. Cleavage of GSDMD by inflammatory caspases determines pyroptotic cell death. Nature. 526 (7575), 660-665 (2015).
- Hitomi, J., et al. Identification of a molecular signaling network that regulates a cellular necrotic cell death pathway. Cell. 135 (7), 1311-1323 (2008).
- Malireddi, R. K., Ippagunta, S., Lamkanfi, M., Kanneganti, T. D. Cutting edge: Proteolytic inactivation of poly(ADP-ribose) polymerase 1 by the Nlrp3 and Nlrc4 inflammasomes. Journal of Immunology. 185 (6), 3127-3130 (2010).
- Tsuchiya, K., et al. Caspase-1 initiates apoptosis in the absence of gasdermin D. Nature Communications. 10 (1), (2019).
- Taabazuing, C. Y., Okondo, M. C., Bachovchin, D. A. Pyroptosis and apoptosis pathways engage in bidirectional crosstalk in monocytes and macrophages. Cell Chemical Biology. 24 (4), 507-514 (2017).
- Gurung, P., et al. FADD and caspase-8 mediate priming and activation of the canonical and noncanonical Nlrp3 inflammasomes. Journal of Immunology. 192 (4), 1835-1846 (2014).
- Man, S. M., et al. Inflammasome activation causes dual recruitment of NLRC4 and NLRP3 to the same macromolecular complex. Proceedings of the National Academy of Sciences of the United States of America. 111 (20), 7403-7408 (2014).
- Man, S. M., et al. Salmonella infection induces recruitment of Caspase-8 to the inflammasome to modulate IL-1beta production. Journal of Immunology. 191 (10), 5239-5246 (2013).
- Van Opdenbosch, N., et al. Caspase-1 engagement and TLR-induced c-FLIP expression suppress ASC/caspase-8-dependent apoptosis by inflammasome sensors NLRP1b and NLRC4. Cell Reports. 21 (12), 3427-3444 (2017).
- Pierini, R., et al. AIM2/ASC triggers caspase-8-dependent apoptosis in Francisella-infected caspase-1-deficient macrophages. Cell Death & Differentiation. 19 (10), 1709-1721 (2012).
- Lee, S., et al. AIM2 forms a complex with pyrin and ZBP1 to drive PANoptosis and host defence. Nature. 597 (7876), 415-419 (2021).
- Sagulenko, V., et al. AIM2 and NLRP3 inflammasomes activate both apoptotic and pyroptotic death pathways via ASC. Cell Death & Differentiation. 20 (9), 1149-1160 (2013).
- Lukens, J. R., et al. Dietary modulation of the microbiome affects autoinflammatory disease. Nature. 516 (7530), 246-249 (2014).
- Gurung, P., Burton, A., Kanneganti, T. D. NLRP3 inflammasome plays a redundant role with caspase 8 to promote IL-1beta-mediated osteomyelitis. Proceedings of the National Academy of Sciences of the United States of America. 113 (16), 4452-4457 (2016).
- Kuriakose, T., et al. ZBP1/DAI is an innate sensor of influenza virus triggering the NLRP3 inflammasome and programmed cell death pathways. Science Immunology. 1 (2), (2016).
- Malireddi, R. K. S., et al. TAK1 restricts spontaneous NLRP3 activation and cell death to control myeloid proliferation. Journal of Experimental Medicine. 215 (4), 1023-1034 (2018).
- Malireddi, R. K. S., et al. Innate immune priming in the absence of TAK1 drives RIPK1 kinase activity-independent pyroptosis, apoptosis, necroptosis, and inflammatory disease. Journal of Experimental Medicine. 217 (3), (2020).
- Christgen, S., et al. Identification of the PANoptosome: A molecular platform triggering pyroptosis, apoptosis, and necroptosis (PANoptosis). Frontiers in Cellular and Infection Microbiology. 10, 237 (2020).
- Malireddi, R. K. S., et al. RIPK1 distinctly regulates Yersinia-induced inflammatory cell death, PANoptosis. Immunohorizons. 4 (12), 789-796 (2020).
- Zheng, M., Karki, R., Vogel, P., Kanneganti, T. D. Caspase-6 is a key regulator of innate immunity, inflammasome activation, and host defense. Cell. 181 (3), 674-687 (2020).
- Karki, R., et al. ADAR1 restricts ZBP1-mediated immune response and PANoptosis to promote tumorigenesis. Cell Reports. 37 (3), 109858 (2021).
- Wang, Y., et al. Single cell analysis of PANoptosome cell death complexes through an expansion microscopy method. Cellular and Molecular Life Sciences. 79 (10), 531 (2022).
- Malireddi, R. K. S., et al. Inflammatory cell death, PANoptosis, mediated by cytokines in diverse cancer lineages inhibits tumor growth. Immunohorizons. 5 (7), 568-580 (2021).
- Kesavardhana, S., et al. The Zα2 domain of ZBP1 is a molecular switch regulating influenza-induced PANoptosis and perinatal lethality during development. Journal of Biological Chemistry. 295 (24), 8325-8330 (2020).
- Banoth, B., et al. ZBP1 promotes fungi-induced inflammasome activation and pyroptosis, apoptosis, and necroptosis (PANoptosis). Journal of Biological Chemistry. 295 (52), 18276-18283 (2020).
- Karki, R., et al. Interferon regulatory factor 1 regulates PANoptosis to prevent colorectal cancer. JCI Insight. 5 (12), 136720 (2020).
- Zheng, M., et al. Impaired NLRP3 inflammasome activation/pyroptosis leads to robust inflammatory cell death via caspase-8/RIPK3 during coronavirus infection. Journal of Biological Chemistry. 295 (41), 14040-14052 (2020).
- Karki, R., et al. Synergism of TNF-α and IFN-γ triggers inflammatory cell death, tissue damage, and mortality in SARS-CoV-2 infection and cytokine shock syndromes. Cell. 184 (1), 149-168 (2021).
- Karki, R., et al. ZBP1-dependent inflammatory cell death, PANoptosis, and cytokine storm disrupt IFN therapeutic efficacy during coronavirus infection. Science Immunology. 7 (74), (2022).
- Jiang, W., Deng, Z., Dai, X., Zhao, W. PANoptosis: A new insight into oral infectious diseases. Frontiers in Immunology. 12, 789610 (2021).
- Chi, D., et al. Real-time induction of macrophage apoptosis, pyroptosis, and necroptosis by Enterococcus faecalis OG1RF and two root canal isolated strains. Frontiers in Cellular and Infection Microbiology. 11, 720147 (2021).
- Lin, J. F., et al. Phosphorylated NFS1 weakens oxaliplatin-based chemosensitivity of colorectal cancer by preventing PANoptosis. Signal Transduction and Targeted Therapy. 7 (1), 54 (2022).
- Song, M., et al. Self-assembled polymeric nanocarrier-mediated co-delivery of metformin and doxorubicin for melanoma therapy. Drug Delivery. 28 (1), 594-606 (2021).
- Boucher, D., Chan, A., Ross, C., Schroder, K. Quantifying caspase-1 activity in murine macrophages. Methods in Molecular Biology. 1725, 163-176 (2018).
- Boucher, D., Duclos, C., Denault, J. B. General in vitro caspase assay procedures. Methods in Molecular Biology. 1133, 3-39 (2014).
- Kaushal, V., Herzog, C., Haun, R. S., Kaushal, G. P. Caspase protocols in mice. Methods in Molecular Biology. 1133, 141-154 (2014).
- Swacha, P., Gekara, N. O., Erttmann, S. F. Biochemical and microscopic analysis of inflammasome complex formation. Methods in Enzymology. 625, 287-298 (2019).
- Talley, S., et al. A caspase-1 biosensor to monitor the progression of inflammation in vivo. Journal of Immunology. 203 (9), 2497-2507 (2019).
- Pelegrin, P., Barroso-Gutierrez, C., Surprenant, A. P2X7 receptor differentially couples to distinct release pathways for IL-1beta in mouse macrophage. Journal of Immunology. 180 (11), 7147-7157 (2008).
- Yu, J. W., et al. Cryopyrin and pyrin activate caspase-1, but not NF-kappaB, via ASC oligomerization. Cell Death & Differentiation. 13 (2), 236-249 (2006).
- Henry, C. M., Martin, S. J. Caspase-8 acts in a non-enzymatic role as a scaffold for assembly of a pro-inflammatory "FADDosome" complex upon TRAIL stimulation. Molecular Cell. 65 (4), 715-729 (2017).
- Hoffmann, E., Neumann, G., Kawaoka, Y., Hobom, G., Webster, R. G. A DNA transfection system for generation of influenza A virus from eight plasmids. Proceedings of the National Academy of Sciences of the United States of America. 97 (11), 6108-6113 (2000).
- Simpson, R. J. Homogenization of mammalian tissue. Cold Spring Harbor Protocols. (7), (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon