Method Article
Deep-Learning-basierte Segmentierung von Kryo-Elektronen-Tomogrammen
In diesem Artikel
Zusammenfassung
Hierbei handelt es sich um ein Verfahren zum Trainieren eines Mehrschicht-U-Netzes für die Mehrklassensegmentierung von Kryo-Elektronen-Tomogrammen, wobei ein Teil eines Tomogramms als Trainingseingabe verwendet wird. Wir beschreiben, wie man dieses Netzwerk auf andere Tomogramme ableiten kann und wie man Segmentierungen für weitere Analysen extrahiert, wie z.B. Subtomogramm-Mittelung und Filament-Tracing.
Zusammenfassung
Die Kryo-Elektronentomographie (Kryo-ET) ermöglicht es Forschern, Zellen in ihrem nativen, hydratisierten Zustand mit der höchstmöglichen Auflösung abzubilden. Die Technik hat jedoch mehrere Einschränkungen, die die Analyse der generierten Daten zeitintensiv und schwierig machen. Die manuelle Segmentierung eines einzelnen Tomogramms kann Stunden bis Tage dauern, aber ein Mikroskop kann leicht 50 oder mehr Tomogramme pro Tag erzeugen. Aktuelle Deep-Learning-Segmentierungsprogramme für Kryo-ET existieren zwar, beschränken sich jedoch auf die Segmentierung einer Struktur nach der anderen. Hier werden Multi-Slice-U-Net-Faltungsneuronale Netze trainiert und angewendet, um automatisch mehrere Strukturen gleichzeitig innerhalb von Kryo-Tomogrammen zu segmentieren. Mit der richtigen Vorverarbeitung können diese Netzwerke robust auf viele Tomogramme abgeleitet werden, ohne dass einzelne Netzwerke für jedes Tomogramm trainiert werden müssen. Dieser Arbeitsablauf verbessert die Geschwindigkeit, mit der Kryo-Elektronen-Tomogramme analysiert werden können, erheblich, indem die Segmentierungszeit in den meisten Fällen auf unter 30 Minuten verkürzt wird. Darüber hinaus können Segmentierungen verwendet werden, um die Genauigkeit der Filamentverfolgung innerhalb eines zellulären Kontexts zu verbessern und Koordinaten für die Subtomogrammmittelung schnell zu extrahieren.
Einleitung
Hardware- und Softwareentwicklungen des letzten Jahrzehnts haben zu einer "Auflösungsrevolution" für die Kryo-Elektronenmikroskopie (Kryo-EM) geführt1,2. Mit besseren und schnelleren Detektoren3, Software zur Automatisierung der Datenerfassung 4,5 und Fortschritten bei der Signalverstärkung wie den Phasenplatten6 ist das Sammeln großer Mengen hochauflösender Kryo-EM-Daten relativ einfach.
Cryo-ET liefert einen beispiellosen Einblick in die zelluläre Ultrastruktur in einem nativen, hydratisierten Zustand 7,8,9,10. Die primäre Einschränkung ist die Probendicke, aber mit der Einführung von Methoden wie dem Mahlen mit fokussiertem Ionenstrahl (FIB), bei dem dicke Zell- und Gewebeproben für die Tomographie11 verdünnt werden, erweitert sich der Horizont für das, was mit Kryo-ET abgebildet werden kann, ständig. Die neuesten Mikroskope sind in der Lage, weit über 50 Tomogramme pro Tag zu produzieren, und diese Rate wird aufgrund der Entwicklung schneller Datenerfassungssysteme voraussichtlich nur noch zunehmen12,13. Die Analyse der riesigen Datenmengen, die von Kryo-ET erzeugt werden, bleibt ein Engpass für diese Bildgebungsmodalität.
Die quantitative Analyse von tomographischen Informationen erfordert, dass sie zuerst annotiert werden. Traditionell erfordert dies eine manuelle Segmentierung durch einen Experten, was zeitaufwändig ist. Abhängig von der molekularen Komplexität, die im Kryo-Tomogramm enthalten ist, kann es Stunden bis Tage dauern. Künstliche neuronale Netze sind eine attraktive Lösung für dieses Problem, da sie trainiert werden können, um den Großteil der Segmentierungsarbeit in einem Bruchteil der Zeit zu erledigen. Convolutional Neural Networks (CNNs) eignen sich besonders für Computer-Vision-Aufgaben14 und wurden kürzlich für die Analyse von Kryo-Elektronen-Tomogrammenangepasst 15,16,17.
Herkömmliche CNNs benötigen viele tausend annotierte Trainingsproben, was für biologische Bildanalyseaufgaben oft nicht möglich ist. Daher hat sich die U-Net-Architektur in diesem Bereich18 hervorgetan, da sie auf Datenerweiterung angewiesen ist, um das Netzwerk erfolgreich zu trainieren und die Abhängigkeit von großen Trainingssätzen zu minimieren. Beispielsweise kann eine U-Net-Architektur mit nur wenigen Schichten eines einzelnen Tomogramms (vier oder fünf Schichten) trainiert und ohne erneutes Training robust auf andere Tomogramme abgeleitet werden. Dieses Protokoll bietet eine Schritt-für-Schritt-Anleitung für das Training neuronaler Netzwerkarchitekturen von U-Net zur Segmentierung von Elektronenkryo-Tomogrammen in Dragonfly 2022.119.
Dragonfly ist eine kommerziell entwickelte Software, die für die 3D-Bildsegmentierung und -analyse durch Deep-Learning-Modelle verwendet wird und für den akademischen Gebrauch frei verfügbar ist (es gelten einige geografische Einschränkungen). Es verfügt über eine fortschrittliche grafische Oberfläche, die es einem Nicht-Experten ermöglicht, die Möglichkeiten des Deep Learning sowohl für die semantische Segmentierung als auch für die Rauschunterdrückung von Bildern voll auszuschöpfen. Dieses Protokoll demonstriert, wie Kryo-Elektronen-Tomogramme in Dragonfly vorverarbeitet und annotiert werden, um künstliche neuronale Netze zu trainieren, die dann abgeleitet werden können, um große Datensätze schnell zu segmentieren. Darüber hinaus wird erläutert und kurz demonstriert, wie segmentierte Daten für weitere Analysen verwendet werden können, z. B. für die Filamentverfolgung und die Koordinatenextraktion für die Mittelung von Subtomogrammen.
Protokoll
HINWEIS: Dragonfly 2022.1 erfordert eine leistungsstarke Workstation. Systemempfehlungen sind zusammen mit der Hardware der für dieses Protokoll verwendeten Workstation in der Materialtabelle enthalten. Alle Tomogramme, die in diesem Protokoll verwendet werden, werden 4x von einer Pixelgröße von 3,3 bis 13,2 ang/pix gebündelt. Die in den repräsentativen Ergebnissen verwendeten Proben stammen von einem Unternehmen (siehe Materialtabelle), das Tierpflegerichtlinien befolgt, die sich an den ethischen Standards dieser Institution orientieren. Das in diesem Protokoll verwendete Tomogramm und der Multi-ROI, der als Trainingseingabe generiert wurde, wurden als gebündelter Datensatz in die Ergänzungsdatei 1 (die unter https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct zu finden ist) aufgenommen, so dass der Benutzer auf Wunsch mit denselben Daten nachvollziehen kann. Dragonfly hostet auch eine Open-Access-Datenbank namens Infinite Toolbox, in der Benutzer trainierte Netzwerke gemeinsam nutzen können.
1. Einrichtung
- Ändern des Standardarbeitsbereichs:
- Um den Arbeitsbereich so zu ändern, dass er den in diesem Protokoll verwendeten widerspiegelt, scrollen Sie auf der linken Seite des Hauptfensters nach unten zum Abschnitt Ansichtseigenschaften der Szene und deaktivieren Sie Legenden anzeigen. Scrollen Sie nach unten zum Abschnitt " Layout " und wählen Sie die Ansicht " Einzelszene" und "Vier gleiche Ansichten" aus.
- Um die Standardeinheit zu aktualisieren, gehen Sie zu Datei | Präferenzen. Ändern Sie im sich öffnenden Fenster die Standardeinheit von Millimeter in Nanometer.
- Hilfreiche Standard-Tastenkombinationen:
- Drücken Sie Esc, um das Fadenkreuz in den 2D-Ansichten anzuzeigen und die 3D-Volumendrehung in der 3D-Ansicht zuzulassen. Drücken Sie X, um das Fadenkreuz in den 2D-Ansichten auszublenden und die 2D-Übersetzung und die 3D-Volumenübersetzung in der 3D-Ansicht zuzulassen.
- Bewegen Sie den Mauszeiger über das Fadenkreuz , um kleine Pfeile anzuzeigen, auf die geklickt und gezogen werden kann, um den Winkel der Betrachtungsebene in den anderen 2D-Ansichten zu ändern.
- Drücken Sie Z, um den Zoomstatus in beiden Ansichten aufzurufen, sodass Benutzer auf eine beliebige Stelle klicken und ziehen können, um zu vergrößern und zu verkleinern.
- Doppelklicken Sie auf eine Ansicht in der Vier-Ansichten-Szene , um nur diese Ansicht zu fokussieren. Doppelklicken Sie erneut, um zu allen vier Ansichten zurückzukehren.
- Speichern Sie den Fortschritt in regelmäßigen Abständen, indem Sie alles auf der Registerkarte Eigenschaften als ORS-Objekt exportieren, um den Import zu vereinfachen. Wählen Sie alle Objekte in der Liste aus und klicken Sie mit der rechten Maustaste auf Exportieren | Als ORS-Objekt. Benennen Sie die Datei und speichern Sie sie. Alternativ können Sie auch zu Datei | Sitzung speichern. Um die automatische Speicherfunktion in der Software zu verwenden, aktivieren Sie sie über Datei | Präferenzen | Automatisches Speichern.
2. Bild-Import
- Um Bilder zu importieren, gehen Sie zu Datei | Importieren Sie Bilddateien. Klicken Sie auf Hinzufügen, navigieren Sie zur Bilddatei und klicken Sie auf Öffnen | Weiter | Fertig.
HINWEIS: Die Software erkennt keine .rec-Dateien. Alle Tomogramme müssen das Suffix .mrc haben. Wenn Sie die bereitgestellten Daten verwenden, gehen Sie stattdessen zu Datei | Objekt(e) importieren. Navigieren Sie zur Datei "Training.ORSObject", klicken Sie auf "Öffnen" und dann auf "OK".
3. Vorverarbeitung (Abbildung 1.1)
- Erstellen Sie eine benutzerdefinierte Intensitätsskala (zur Kalibrierung von Bildintensitäten über Datensätze hinweg). Gehen Sie zu Dienstprogramme | Leiter der Dimensionseinheit. Klicken Sie unten links auf + , um eine neue Dimensionseinheit zu erstellen.
- Wählen Sie ein Merkmal mit hoher Intensität (hell) und niedriger Intensität (dunkel), das in allen Tomogrammen von Interesse enthalten ist. Geben Sie der Einheit einen Namen und eine Abkürzung (z. B. stellen Sie für diese Skala die Passerperlen auf 0,0 Standardintensität und den Hintergrund auf 100,0 ein). Speichern Sie die benutzerdefinierte Bemaßungseinheit.
HINWEIS: Eine benutzerdefinierte Intensitätsskala ist eine beliebige Skala, die erstellt und auf die Daten angewendet wird, um sicherzustellen, dass sich alle Daten auf derselben Intensitätsskala befinden, obwohl sie zu unterschiedlichen Zeiten oder auf verschiedenen Geräten erfasst wurden. Wählen Sie helle und dunkle Merkmale, die den Bereich, in den das Signal fällt, am besten darstellen. Wenn die Daten keine Passermarken enthalten, wählen Sie einfach das dunkelste Merkmal aus, das segmentiert werden soll (z. B. die dunkelste Region des Proteins). - Um Bilder auf die benutzerdefinierte Intensitätsskala zu kalibrieren, klicken Sie mit der rechten Maustaste auf das Dataset in der Spalte Eigenschaften auf der rechten Seite des Bildschirms und wählen Sie Intensitätsskala kalibrieren. Scrollen Sie auf der Registerkarte Main auf der linken Seite des Bildschirms nach unten zum Abschnitt Sonde . Klicken Sie mit dem kreisförmigen Sondenwerkzeug mit einem geeigneten Durchmesser auf einige Stellen im Hintergrundbereich des Tomogramms und notieren Sie die durchschnittliche Zahl in der Spalte Rohintensität . Wiederholen Sie diesen Vorgang für Passermarken, und klicken Sie dann auf "Kalibrieren". Passen Sie bei Bedarf den Kontrast an, um Strukturen mit dem Flächenwerkzeug im Abschnitt Fensternivellierung der Registerkarte Haupt wieder sichtbar zu machen.
- Bildfilterung:
HINWEIS: Die Bildfilterung kann das Rauschen reduzieren und das Signal verstärken. Dieses Protokoll verwendet drei Filter, die in die Software integriert sind, da sie für diese Daten am besten funktionieren, aber es gibt viele Filter. Sobald Sie sich für ein Bildfilterprotokoll für die interessierenden Daten entschieden haben, müssen Sie vor der Segmentierung genau dasselbe Protokoll auf alle Tomogramme anwenden.- Scrollen Sie auf der Hauptregisterkarte auf der linken Seite nach unten zum Bildbearbeitungsfenster. Klicken Sie auf Erweitert und warten Sie, bis sich ein neues Fenster öffnet. Wählen Sie im Bedienfeld "Eigenschaften" den zu filternden Datensatz aus und machen Sie ihn sichtbar, indem Sie auf das Augensymbol links neben dem Datensatz klicken.
- Verwenden Sie im Bedienfeld " Operationen " das Dropdown-Menü, um die Option " Histogrammentzerrung " (im Abschnitt "Kontrast ") für den ersten Vorgang auszuwählen. Wählen Sie Vorgang hinzufügen | Gauß (unter dem Abschnitt Glättung ). Ändern Sie die Kernel-Dimension in 3D.
- Fügen Sie einen dritten Vorgang hinzu. Wählen Sie dann Unscharf (im Abschnitt Schärfen ). Lassen Sie die Ausgabe für diese. Wenden Sie sie auf alle Slices an, lassen Sie die Filterung laufen, schließen Sie dann das Bildverarbeitungsfenster, um zur Hauptoberfläche zurückzukehren.
4. Trainingsdaten erstellen (Abbildung 1.2)
- Identifizieren Sie den Trainingsbereich, indem Sie zuerst den ungefilterten Datensatz ausblenden, indem Sie im Bereich "Dateneigenschaften" auf das Augensymbol links daneben klicken. Zeigen Sie dann den neu gefilterten Datensatz an (der automatisch den Namen DataSet-HistEq-Gauss-Unsharp erhält). Identifizieren Sie mithilfe des gefilterten Datasets einen Teilbereich des Tomogramms, der alle interessierenden Merkmale enthält.
- Um ein Feld um den gewünschten Bereich herum zu erstellen, scrollen Sie auf der linken Seite auf der Hauptregisterkarte nach unten zur Kategorie Shapes und wählen Sie Box erstellen aus. Verwenden Sie im Bedienfeld " Vier Ansichten" die verschiedenen 2D-Ebenen , um die Kanten des Felds zu führen/zu ziehen, um nur den gewünschten Bereich in allen Dimensionen einzuschließen. Wählen Sie in der Datenliste den Bereich " Box" aus, und ändern Sie die Farbe des Rahmens, um die Anzeige zu erleichtern, indem Sie auf das graue Quadrat neben dem Augensymbol klicken.
HINWEIS: Die kleinste Patchgröße für ein 2D-U-Net beträgt 32 x 32 Pixel. 400 x 400 x 50 Pixel ist eine vernünftige Boxgröße für den Anfang. - Um einen Multi-ROI zu erstellen, wählen Sie auf der linken Seite die Registerkarte Segmentierung | Neu und aktivieren Sie Als Multi-ROI erstellen. Stellen Sie sicher, dass die Anzahl der Klassen der Anzahl der interessierenden Features + einer Hintergrundklasse entspricht. Benennen Sie die Multi-ROI-Trainingsdaten, und stellen Sie sicher, dass die Geometrie dem Dataset entspricht, bevor Sie auf OK klicken.
- Segmentierung der Trainingsdaten
- Scrollen Sie durch die Daten, bis Sie sich innerhalb der Grenzen des eingerahmten Bereichs befinden. Wählen Sie den Multi-ROI im Eigenschaftenmenü auf der rechten Seite aus. Doppelklicken Sie auf den ersten leeren Klassennamen im Multi-ROI, um ihn zu benennen.
- Malen Sie mit dem 2D-Pinsel. Scrollen Sie auf der Registerkarte Segmentierung auf der linken Seite nach unten zu 2D-Werkzeuge , und wählen Sie einen kreisförmigen Pinsel aus. Wählen Sie dann Adaptive Gauß oder Lokale OTSU aus dem Dropdown-Menü. Halten Sie zum Malen die linke Strg-Taste gedrückt und klicken Sie. Halten Sie zum Löschen die linke Umschalttaste gedrückt und klicken Sie.
HINWEIS: Der Pinsel spiegelt die Farbe der aktuell ausgewählten Klasse wider. - Wiederholen Sie den vorherigen Schritt für jede Objektklasse im Multi-ROI. Stellen Sie sicher, dass alle Strukturen innerhalb des Box-Bereichs vollständig segmentiert sind, da sie sonst vom Netzwerk als Hintergrund betrachtet werden.
- Wenn alle Strukturen beschriftet sind, klicken Sie mit der rechten Maustaste auf die Hintergrundklasse im Multi-ROI , und wählen Sie Alle unbeschrifteten Voxel zur Klasse hinzufügen aus.
- Erstellen Sie einen neuen ROI für eine einzelne Klasse mit dem Namen Mask. Stellen Sie sicher, dass die Geometrie auf das gefilterte Dataset festgelegt ist, und klicken Sie dann auf Übernehmen. Klicken Sie auf der Registerkarte "Eigenschaften" auf der rechten Seite mit der rechten Maustaste auf das Kästchen, und wählen Sie "Zum ROI hinzufügen" aus. Fügen Sie es dem ROI der Maske hinzu.
- Um die Trainingsdaten mithilfe der Maske zu kürzen, wählen Sie auf der Registerkarte Eigenschaften sowohl den Multi-ROI für Trainingsdaten als auch den ROI für die Maske aus, indem Sie die Strg-Taste gedrückt halten und jeweils darauf klicken. Klicken Sie anschließend unter der Liste der Dateneigenschaften im Abschnitt Boolesche Vorgänge auf Überschneiden. Nennen Sie das neue Dataset Gekürzte Trainingseingabe, und stellen Sie sicher, dass die Geometrie dem gefilterten Dataset entspricht, bevor Sie auf OK klicken.
5. Verwenden des Segmentierungsassistenten für iteratives Training (Abbildung 1.3)
- Importieren Sie die Trainingsdaten in den Segmentierungs-Assistenten, indem Sie zuerst mit der rechten Maustaste auf das gefilterte Dataset auf der Registerkarte Eigenschaften klicken und dann die Option Segmentierungs-Assistent auswählen. Wenn sich ein neues Fenster öffnet, suchen Sie auf der rechten Seite nach der Registerkarte Eingabe . Klicken Sie auf Frames aus einem Multi-ROI importieren und wählen Sie die getrimmte Trainingseingabe aus.
- (Fakultativ) Erstellen Sie einen visuellen Feedback-Rahmen, um den Trainingsfortschritt in Echtzeit zu überwachen.
- Wählen Sie einen Frame aus den Daten aus, der nicht segmentiert ist, und klicken Sie auf + , um ihn als neuen Frame hinzuzufügen. Doppelklicken Sie auf die gemischte Beschriftung rechts neben dem Rahmen und ändern Sie sie in Überwachung.
- Um ein neues neuronales Netzwerkmodell zu generieren, klicken Sie auf der rechten Seite auf der Registerkarte Modelle auf die Schaltfläche + , um ein neues Modell zu generieren. Wählen Sie U-Net aus der Liste aus, wählen Sie dann für Eingabedimension 2,5D und 5 Slices aus, und klicken Sie dann auf Generieren.
- Um das Netzwerk zu trainieren, klicken Sie unten rechts im SegWiz-Fenster auf Trainieren.
HINWEIS: Das Training kann vorzeitig abgebrochen werden, ohne dass der Fortschritt verloren geht. - Um das trainierte Netzwerk zum Segmentieren neuer Frames zu verwenden, erstellen Sie nach Abschluss des U-Net-Trainings einen neuen Frame und klicken Sie auf Vorhersagen (unten rechts). Klicken Sie dann auf den Aufwärtspfeil oben rechts im vorhergesagten Frame, um die Segmentierung auf den realen Frame zu übertragen.
- Um die Vorhersage zu korrigieren, klicken Sie bei gedrückter Strg-Taste auf zwei Klassen , um die segmentierten Pixel einer Klasse in die andere zu ändern. Wählen Sie beide Klassen aus und malen Sie mit dem Pinsel , um nur Pixel zu malen, die zu einer der beiden Klassen gehören. Korrigieren Sie die Segmentierung in mindestens fünf neuen Frames.
HINWEIS: Das Malen mit dem Pinselwerkzeug, während beide Klassen ausgewählt sind, bedeutet, dass anstelle des Löschens durch Umschalttaste, wie es normalerweise der Fall ist, Pixel der ersten Klasse in die zweite konvertiert werden. Strg-Klick bewirkt das Gegenteil. - Klicken Sie für ein iteratives Training erneut auf die Schaltfläche Trainieren und lassen Sie das Netzwerk weitere 30-40 Epochen weiter trainieren, beenden Sie dann das Training und wiederholen Sie die Schritte 4.5 und 4.6 für eine weitere Trainingsrunde.
HINWEIS: Auf diese Weise kann ein Modell mit einem einzigen Datensatz iterativ trainiert und verbessert werden. - Um das Netzwerk zu veröffentlichen und mit seiner Leistung zufrieden zu sein, beenden Sie den Segmentierungs-Assistenten. Wählen Sie im Dialogfeld, das automatisch angezeigt wird, in dem Sie gefragt werden, welche Modelle veröffentlicht (gespeichert) werden sollen, das erfolgreiche Netzwerk aus, benennen Sie es und veröffentlichen Sie es, um das Netzwerk für die Verwendung außerhalb des Segmentierungsassistenten verfügbar zu machen.
6. Anwenden des Netzwerks (Abbildung 1.4)
- Wenn Sie das Trainingstomogramm zuerst anwenden möchten, wählen Sie das gefilterte Dataset im Eigenschaftenpanel aus. Scrollen Sie im Bereich "Segmentierung" auf der linken Seite nach unten zum Abschnitt "Segment mit KI". Stellen Sie sicher, dass der richtige Datensatz ausgewählt ist, wählen Sie das Modell aus, das gerade im Dropdown-Menü veröffentlicht wurde, und klicken Sie dann auf Segment | Alle Scheiben. Wählen Sie alternativ Vorschau aus, um eine One-Slice-Vorschau der Segmentierung anzuzeigen.
- Um es auf ein Inferenzdataset anzuwenden, importieren Sie das neue Tomogramm. Vorverarbeitung gemäß Schritt 3 (Abbildung 1.1). Wechseln Sie im Bereich "Segmentierung" zum Abschnitt "Segment mit KI". Stellen Sie sicher, dass das neu gefilterte Tomogramm das ausgewählte Dataset ist, wählen Sie das zuvor trainierte Modell aus, und klicken Sie auf Segment | Alle Scheiben.
7. Manipulation und Bereinigung der Segmentierung
- Bereinigen Sie Rauschen schnell, indem Sie zuerst eine der Klassen auswählen, die das Rauschen und das interessierende Feature segmentiert hat. Rechtsklick | Prozess-Inseln | Entfernen nach Voxelanzahl | Wählen Sie eine Voxelgröße aus. Fangen Sie klein an (~200) und erhöhen Sie die Anzahl schrittweise, um den größten Teil des Rauschens zu entfernen.
- Klicken Sie zur Segmentierungskorrektur bei gedrückter Strg-Taste auf zwei Klassen, um nur Pixel zu zeichnen, die zu diesen Klassen gehören. Klicken Sie bei gedrückter Strg-Taste + ziehen Sie mit den Segmentierungswerkzeugen, um Pixel der zweiten Klasse in die erste zu ändern, und klicken Sie bei gedrückter Umschalttaste + Ziehen, um das Gegenteil zu erreichen. Fahren Sie damit fort, um falsch beschriftete Pixel schnell zu korrigieren.
- Getrennte angeschlossene Komponenten.
- Wählen Sie eine Klasse. Klicken Sie mit der rechten Maustaste auf eine Klasse in Multi-ROI | Trennen Sie verbundene Komponenten , um eine neue Klasse für jede Komponente zu erstellen, die nicht mit einer anderen Komponente derselben Klasse verbunden ist. Verwenden Sie die Schaltflächen unter dem Multi-ROI, um die Klassen einfach zusammenzuführen.
- Exportieren Sie den ROI als Binär/TIFF.
- Wählen Sie eine Klasse im Multi-ROI aus, klicken Sie dann mit der rechten Maustaste und extrahieren Sie die Klasse als ROI. Wählen Sie im Eigenschaftenbereich oben den neuen ROI aus, klicken Sie mit der rechten Maustaste auf | Exportieren | ROI als Binärwert (stellen Sie sicher, dass die Option zum Exportieren aller Bilder in eine Datei ausgewählt ist).
HINWEIS: Benutzer können mit dem IMOD-Programm tif2mrc20 einfach vom tiff- in das mrc-Format konvertieren. Dies ist nützlich für die Filamentverfolgung.
- Wählen Sie eine Klasse im Multi-ROI aus, klicken Sie dann mit der rechten Maustaste und extrahieren Sie die Klasse als ROI. Wählen Sie im Eigenschaftenbereich oben den neuen ROI aus, klicken Sie mit der rechten Maustaste auf | Exportieren | ROI als Binärwert (stellen Sie sicher, dass die Option zum Exportieren aller Bilder in eine Datei ausgewählt ist).
8. Generieren von Koordinaten für die Sub-Tomogramm-Mittelung aus dem ROI
- Extrahieren Sie eine Klasse.
- Klicken Sie mit der rechten Maustaste auf Klasse, die für die Mittelwertbildung verwendet werden soll | Extrahieren Sie die Klasse als ROI. ROI der Rechtsklick-Klasse | Vernetzte Komponenten | Neuer Multi-ROI (26 verbunden).
- Generieren Sie Koordinaten.
- Klicken Sie mit der rechten Maustaste auf den neuen Multi-ROI | Skalarer Generator. Erweitern Sie die Basismessungen mit Datensatz | Überprüfen Sie den gewichteten Schwerpunkt X, Y und Z. Wählen Sie das Dataset aus, und berechnen Sie es. Klicken Sie mit der rechten Maustaste auf Multi-ROI | Exportieren Sie Skalarwerte. Aktivieren Sie das Kontrollkästchen Alle Skalar-Slots auswählen und dann OK , um die Schwerpunkt-Weltkoordinaten für jede Klasse im Multi-ROI als CSV-Datei zu generieren.
HINWEIS: Wenn die Partikel nahe beieinander liegen und sich die Segmentierungen berühren, kann es erforderlich sein, eine Wasserscheidentransformation durchzuführen, um die Komponenten in einen Multi-ROI zu trennen.
- Klicken Sie mit der rechten Maustaste auf den neuen Multi-ROI | Skalarer Generator. Erweitern Sie die Basismessungen mit Datensatz | Überprüfen Sie den gewichteten Schwerpunkt X, Y und Z. Wählen Sie das Dataset aus, und berechnen Sie es. Klicken Sie mit der rechten Maustaste auf Multi-ROI | Exportieren Sie Skalarwerte. Aktivieren Sie das Kontrollkästchen Alle Skalar-Slots auswählen und dann OK , um die Schwerpunkt-Weltkoordinaten für jede Klasse im Multi-ROI als CSV-Datei zu generieren.
9. Transformation der Wasserscheide
- Extrahieren Sie die Klasse, indem Sie mit der rechten Maustaste auf die Klasse in Multi-ROI klicken, um sie für die Mittelwertbildung zu verwenden | Extrahieren Sie die Klasse als ROI. Nennen Sie diese ROI-Wasserscheidenmaske.
- (Fakultativ) Löcher schließen.
- Wenn die segmentierten Partikel Löcher oder Öffnungen aufweisen, schließen Sie diese für die Wasserscheide. Klicken Sie in den Dateneigenschaften auf den ROI. Gehen Sie auf der Registerkarte Segmentierung (auf der linken Seite) zu Morphologische Operationen, und verwenden Sie die erforderliche Kombination aus Erweitern, Erodieren und Schließen, um feste Segmentierungen ohne Löcher zu erzielen.
- Kehren Sie den ROI um, indem Sie auf die Schaltfläche ROI | Ausgewähltes Objekt kopieren (unter Dateneigenschaften). Wählen Sie den kopierten ROI aus, und klicken Sie auf der linken Seite auf der Registerkarte Segmentierung auf Invertieren.
- Erstellen Sie eine Entfernungskarte, indem Sie mit der rechten Maustaste auf den invertierten ROI | Mapping erstellen von | Entfernungskarte. Erstellen Sie zur späteren Verwendung eine Kopie der Entfernungskarte und kehren Sie sie um (Rechtsklick | Modifizieren und transformieren | Werte invertieren | Bewerben). Nennen Sie diese invertierte Karte Landschaft.
- Erstellen Sie Startpunkte.
- Blenden Sie den ROI aus und zeigen Sie die Entfernungskarte an. Klicken Sie auf der Registerkarte Segmentierung auf Bereich definieren und verringern Sie den Bereich , bis nur noch wenige Pixel in der Mitte jedes Punktes hervorgehoben sind und keines mit einem anderen Punkt verbunden ist. Klicken Sie unten im Abschnitt Bereich auf Zu Neu hinzufügen. Nennen Sie diesen neuen ROI Seedpoints.
- Führen Sie eine Transformation des Wassereinzugsgebiets durch.
- Klicken Sie mit der rechten Maustaste auf Seedpoints ROI | Vernetzte Komponenten | Neuer Multi-ROI (26 verbunden). Klicken Sie mit der rechten Maustaste auf den neu generierten Multi-ROI | Wasserscheide-Transformation. Wählen Sie die Entfernungskarte mit dem Namen Landschaft aus und klicken Sie auf OK. Wählen Sie den ROI mit dem Namen Watershed Mask aus, und klicken Sie auf OK, um eine Watershed-Transformation von jedem Startpunkt zu berechnen und einzelne Partikel im Multi-ROI in separate Klassen zu trennen. Generieren Sie Koordinaten wie in Schritt 8.2.
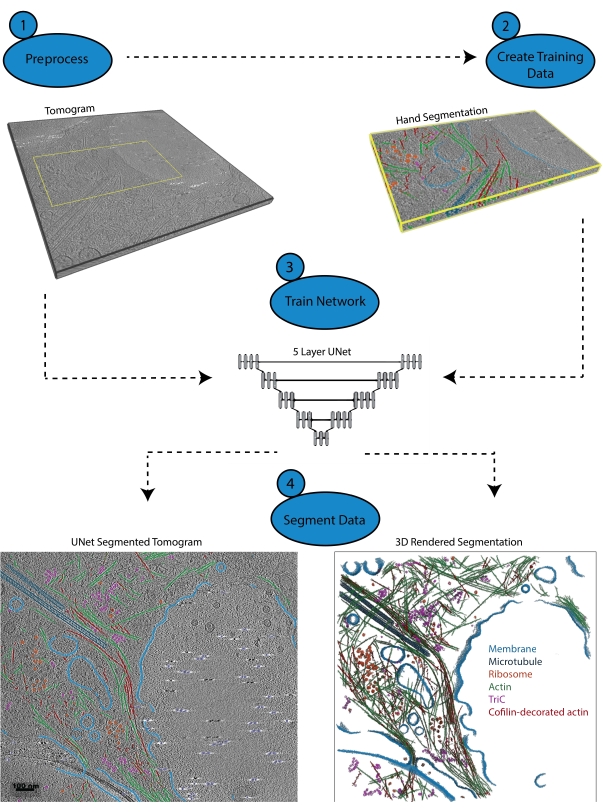
Abbildung 1: Arbeitsablauf. 1) Vorverarbeitung des Trainingstomogramms, indem Sie die Intensitätsskala kalibrieren und den Datensatz filtern. 2) Erstellen Sie die Trainingsdaten, indem Sie einen kleinen Teil eines Tomogramms mit allen geeigneten Beschriftungen, die der Benutzer identifizieren möchte, von Hand segmentieren. 3) Unter Verwendung des gefilterten Tomogramms als Eingabe und der Handsegmentierung als Trainingsausgabe wird ein fünfschichtiges Mehrschicht-U-Net mit mehreren Schichten im Segmentierungsassistenten trainiert. 4) Das trainierte Netzwerk kann auf das vollständige Tomogramm angewendet werden, um es zu kommentieren, und aus jeder segmentierten Klasse kann ein 3D-Rendering generiert werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergebnisse
Im Anschluss an das Protokoll wurde ein Fünf-Schicht-U-Net mit einem einzigen Tomogramm (Abbildung 2A) trainiert, um fünf Klassen zu identifizieren: Membran, Mikrotubuli, Aktin, Passermarker und Hintergrund. Das Netzwerk wurde insgesamt dreimal iterativ trainiert und dann auf das Tomogramm angewendet, um es vollständig zu segmentieren und zu annotieren (Abbildung 2B, C). Die minimale Bereinigung wurde mit den Schritten 7.1 und 7.2 durchgeführt. Die nächsten drei interessierenden Tomogramme (Abbildung 2D, G, J) wurden zur Vorverarbeitung in die Software geladen. Vor dem Bildimport musste eines der Tomogramme (Abbildung 2J) die Pixelgröße von 17,22 Å/px auf 13,3 Å/px anpassen, da es mit einem anderen Mikroskop bei einer etwas anderen Vergrößerung aufgenommen wurde. Das IMOD-Programm squeezevol wurde zum Ändern der Größe mit folgendem Befehl verwendet:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
In diesem Befehl bezieht sich -f auf den Faktor, um den die Pixelgröße geändert werden soll (in diesem Fall: 13,3/17,22). Nach dem Import wurden alle drei Inferenzziele gemäß den Schritten 3.2 und 3.3 vorverarbeitet, und dann wurde das Fünf-Schicht-U-Net angewendet. Es wurden erneut minimale Aufräumarbeiten durchgeführt. Die endgültigen Segmentierungen sind in Abbildung 2 dargestellt.
Mikrotubuli-Segmentierungen aus jedem Tomogramm wurden als binäre (Schritt 7.4) TIF-Dateien exportiert, in MRC (IMOD tif2mrc-Programm ) konvertiert und dann für die Zylinderkorrelation und Filamentverfolgung verwendet. Binäre Segmentierungen von Filamenten führen zu einer viel robusteren Filamentverfolgung als die Verfolgung über Tomogramme. Koordinatenkarten aus der Filamentverfolgung (Abbildung 3) werden für weitere Analysen verwendet, wie z. B. Messungen des nächsten Nachbarn (Filamentpackung) und helikale Subtomogramm-Mittelung entlang einzelner Filamente, um die Orientierung der Mikrotubuli zu bestimmen.
Erfolglose oder unzureichend trainierte Netzwerke sind leicht zu ermitteln. Ein ausgefallenes Netzwerk ist überhaupt nicht in der Lage, Strukturen zu segmentieren, während ein unzureichend trainiertes Netzwerk in der Regel einige Strukturen korrekt segmentiert und eine beträchtliche Anzahl von Fehlalarmen und Fehlalarmen aufweist. Diese Netzwerke können korrigiert und iterativ trainiert werden, um ihre Leistung zu verbessern. Der Segmentierungsassistent berechnet automatisch den Würfelähnlichkeitskoeffizienten eines Modells (im SegWiz als Punktzahl bezeichnet), nachdem es trainiert wurde. Diese Statistik gibt eine Schätzung der Ähnlichkeit zwischen den Trainingsdaten und der U-Net-Segmentierung an. Dragonfly 2022.1 verfügt außerdem über ein integriertes Tool zur Bewertung der Leistung eines Modells, auf das auf der Registerkarte "Künstliche Intelligenz " oben auf der Benutzeroberfläche zugegriffen werden kann (siehe Dokumentation zur Verwendung).
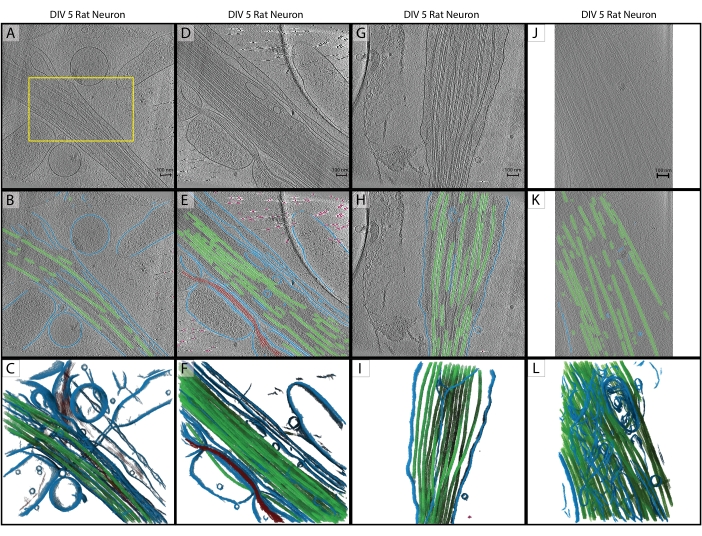
Abbildung 2: Inferenz. (A-C) Originales Trainingstomogramm eines DIV 5 Hippocampus-Rattenneurons, aufgenommen 2019 an einem Titan Krios. Dies ist eine rückprojizierte Rekonstruktion mit CTF-Korrektur in IMOD. (A) Das gelbe Kästchen stellt den Bereich dar, in dem die manuelle Segmentierung für die Trainingseingabe durchgeführt wurde. (B) 2D-Segmentierung aus dem U-Net nach Abschluss des Trainings. (C) 3D-Darstellung der segmentierten Regionen mit Membran (blau), Mikrotubuli (grün) und Aktin (rot). (D-F) DIV 5 Hippocampus-Rattenneuron aus der gleichen Sitzung wie das Trainingstomogramm. (E) 2D-Segmentierung aus dem U-Net ohne zusätzliche Schulung und schnelle Bereinigung. Membran (blau), Mikrotubuli (grün), Aktin (rot), Passermarken (rosa). (F) 3D-Rendering der segmentierten Bereiche. (G-I) DIV 5 Hippocampus-Rattenneuron aus der Sitzung 2019. (H) 2D-Segmentierung aus dem U-Net mit schneller Bereinigung und (I) 3D-Rendering. (J-L) DIV 5 Hippocampus-Rattenneuron, gesammelt im Jahr 2021 auf einem anderen Titan Krios mit einer anderen Vergrößerung. Die Pixelgröße wurde mit dem IMOD-Programm squeezevol an das Trainingstomogramm angepasst. (K) 2D-Segmentierung aus dem U-Net mit schneller Bereinigung, die eine robuste Inferenz über Datensätze hinweg mit ordnungsgemäßer Vorverarbeitung demonstriert, und (L) 3D-Rendering der Segmentierung. Maßstabsbalken = 100 nm. Abkürzungen: DIV = Tage in vitro; CTF = Kontrastübertragungsfunktion. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
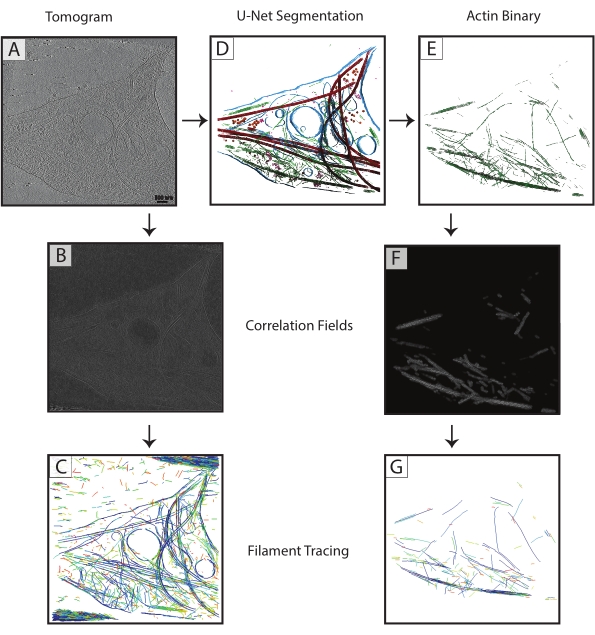
Abbildung 3: Verbesserung der Filamentverfolgung . (A) Tomogramm eines DIV 4 Ratten-Hippocampus-Neurons, gesammelt auf einem Titan Krios. (B) Korrelationskarte, die aus der Zylinderkorrelation über Aktinfilamenten generiert wurde. (C) Filamentverfolgung von Aktin unter Verwendung der Intensitäten der Aktinfilamente in der Korrelationskarte zur Definition von Parametern. Bei der Verfolgung werden die Membran und die Mikrotubuli sowie das Rauschen erfasst, während versucht wird, nur Aktin zu verfolgen. (D) U-Net-Segmentierung des Tomogramms. Membran blau hervorgehoben, Mikrotubuli rot, Ribosomen orange, triC lila und Aktin grün. (E) Aktin-Segmentierung, extrahiert als binäre Maske für die Filamentverfolgung. (F) Korrelationskarte, die aus der Zylinderkorrelation mit den gleichen Parametern aus (B) generiert wurde. (G) Signifikant verbesserte Filamentverfolgung von reinen Aktinfilamenten aus dem Tomogramm. Abkürzung: DIV = Tage in vitro. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Ergänzende Datei 1: Das in diesem Protokoll verwendete Tomogramm und der Multi-ROI, der als Trainingseingabe generiert wurde, sind als gebündelter Datensatz (Training.ORSObject) enthalten. Siehe https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Diskussion
Dieses Protokoll legt ein Verfahren für die Verwendung der Software Dragonfly 2022.1 fest, um ein U-Net mit mehreren Klassen aus einem einzelnen Tomogramm zu trainieren, und wie dieses Netzwerk auf andere Tomogramme abgeleitet werden kann, die nicht aus demselben Datensatz stammen müssen. Das Training ist relativ schnell (kann so schnell wie 3-5 Minuten pro Epoche oder so langsam wie ein paar Stunden sein, abhängig von dem Netzwerk, das trainiert wird, und der verwendeten Hardware), und das Umtrainieren eines Netzwerks zur Verbesserung seines Lernens ist intuitiv. Solange die Vorverarbeitungsschritte für jedes Tomogramm durchgeführt werden, ist die Inferenz in der Regel robust.
Eine konsistente Vorverarbeitung ist der wichtigste Schritt für die Deep-Learning-Inferenz. Es gibt viele Bildfilter in der Software und der Benutzer kann experimentieren, um festzustellen, welche Filter für bestimmte Datensätze am besten geeignet sind. Beachten Sie, dass die Filterung, die für das Trainingstomogramm verwendet wird, auf die gleiche Weise auf die Inferenztomogramme angewendet werden muss. Es muss auch darauf geachtet werden, dass das Netzwerk mit genauen und ausreichenden Schulungsinformationen versorgt wird. Es ist wichtig, dass alle Merkmale, die innerhalb der Trainingsscheiben segmentiert sind, so sorgfältig und präzise wie möglich segmentiert werden.
Die Bildsegmentierung wird durch eine ausgeklügelte Benutzeroberfläche in kommerzieller Qualität erleichtert. Es bietet alle notwendigen Werkzeuge für die Handsegmentierung und ermöglicht die einfache Neuzuweisung von Voxeln von einer Klasse in eine andere vor dem Training und der Umschulung. Der Benutzer kann Voxel innerhalb des gesamten Kontexts des Tomogramms von Hand segmentieren, und er erhält mehrere Ansichten und die Möglichkeit, das Volumen frei zu drehen. Darüber hinaus bietet die Software die Möglichkeit, Multi-Class-Netzwerke zu verwenden, die tendenziell eine bessere Leistungerbringen 16 und schneller sind als die Segmentierung mit mehreren Single-Class-Netzwerken.
Natürlich gibt es Einschränkungen für die Fähigkeiten eines neuronalen Netzes. Kryo-ET-Daten sind von Natur aus sehr verrauscht und in der Winkelabtastung begrenzt, was zu orientierungsspezifischen Verzerrungen bei identischen Objekten führt21. Das Training beruht auf einem Experten, der Strukturen genau von Hand segmentiert, und ein erfolgreiches Netzwerk ist nur so gut (oder so schlecht) wie die Trainingsdaten, die es erhält. Die Bildfilterung zur Signalverstärkung ist für den Trainer hilfreich, aber es gibt immer noch viele Fälle, in denen es schwierig ist, alle Pixel einer bestimmten Struktur genau zu identifizieren. Es ist daher wichtig, dass bei der Erstellung der Trainingssegmentierung große Sorgfalt walten gelassen wird, damit das Netzwerk die bestmöglichen Informationen hat, um während des Trainings zu lernen.
Dieser Workflow kann leicht an die Vorlieben jedes Benutzers angepasst werden. Es ist zwar wichtig, dass alle Tomogramme auf die gleiche Weise vorverarbeitet werden, es ist jedoch nicht erforderlich, die genauen Filter zu verwenden, die im Protokoll verwendet werden. Die Software verfügt über zahlreiche Bildfilteroptionen, und es wird empfohlen, diese für die jeweiligen Daten des Benutzers zu optimieren, bevor Sie sich auf ein großes Segmentierungsprojekt mit vielen Tomogrammen einlassen. Es gibt auch eine ganze Reihe von Netzwerkarchitekturen, die verwendet werden können: Es wurde festgestellt, dass ein Multi-Slice-U-Net für die Daten aus diesem Labor am besten geeignet ist, aber ein anderer Benutzer könnte feststellen, dass eine andere Architektur (z. B. ein 3D-U-Net oder ein Sensor 3D) besser funktioniert. Der Segmentierungsassistent bietet eine komfortable Schnittstelle zum Vergleich der Leistung mehrerer Netzwerke mit denselben Trainingsdaten.
Tools wie die hier vorgestellten werden die manuelle Segmentierung von Volltomogrammen zu einer Aufgabe der Vergangenheit machen. Mit gut trainierten neuronalen Netzen, die robust ableger sind, ist es durchaus möglich, einen Arbeitsablauf zu erstellen, bei dem tomographische Daten so schnell rekonstruiert, verarbeitet und vollständig segmentiert werden, wie das Mikroskop sie erfassen kann.
Offenlegungen
Die Open-Access-Lizenz für dieses Protokoll wurde von Object Research Systems bezahlt.
Danksagungen
Diese Studie wurde vom Penn State College of Medicine und der Abteilung für Biochemie und Molekularbiologie sowie vom Tobacco Settlement Fund (TSF) Grant 4100079742-EXT unterstützt. Die in diesem Projekt verwendeten CryoEM- und CryoET-Core-Dienste und -Instrumente (RRID:SCR_021178) wurden teilweise vom Pennsylvania State University College of Medicine über das Büro des Vizedekans für Forschung und Doktoranden und das Pennsylvania Department of Health mit Tobacco Settlement Funds (CURE) finanziert. Der Inhalt liegt ausschließlich in der Verantwortung der Autoren und spiegelt nicht unbedingt die offiziellen Ansichten der Universität oder des College of Medicine wider. Das Gesundheitsministerium von Pennsylvania lehnt ausdrücklich die Verantwortung für Analysen, Interpretationen oder Schlussfolgerungen ab.
Materialien
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Referenzen
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten