Method Article
Segmentazione basata sul deep learning dei tomogrammi crioelettronici
In questo articolo
Riepilogo
Questo è un metodo per addestrare un U-Net multi-fetta per la segmentazione multi-classe di tomogrammi crioelettronici utilizzando una porzione di un tomogramma come input di allenamento. Descriviamo come dedurre questa rete ad altri tomogrammi e come estrarre segmentazioni per ulteriori analisi, come la media dei sottotomogrammi e il tracciamento dei filamenti.
Abstract
La tomografia crioelettronica (cryo-ET) consente ai ricercatori di visualizzare le cellule nel loro stato nativo e idratato alla massima risoluzione attualmente possibile. La tecnica ha diverse limitazioni, tuttavia, che rendono l'analisi dei dati che genera dispendiosa in termini di tempo e difficile. La segmentazione manuale di un singolo tomogramma può richiedere da ore a giorni, ma un microscopio può facilmente generare 50 o più tomogrammi al giorno. Gli attuali programmi di segmentazione del deep learning per la crio-ET esistono, ma sono limitati alla segmentazione di una struttura alla volta. Qui, le reti neurali convoluzionali multi-slice U-Net vengono addestrate e applicate per segmentare automaticamente più strutture contemporaneamente all'interno dei criotogrammi. Con una corretta pre-elaborazione, queste reti possono essere dedotte in modo robusto per molti tomogrammi senza la necessità di addestrare singole reti per ciascun tomogramma. Questo flusso di lavoro migliora notevolmente la velocità con cui i tomogrammi crioelettronici possono essere analizzati riducendo il tempo di segmentazione a meno di 30 minuti nella maggior parte dei casi. Inoltre, le segmentazioni possono essere utilizzate per migliorare l'accuratezza del tracciamento dei filamenti all'interno di un contesto cellulare e per estrarre rapidamente le coordinate per la media dei sottotomogrammi.
Introduzione
Gli sviluppi hardware e software nell'ultimo decennio hanno portato a una "rivoluzione della risoluzione" per la microscopia crioelettronica (cryo-EM)1,2. Con rivelatori migliori e più veloci3, software per automatizzare la raccolta dei dati4,5 e progressi di potenziamento del segnale come le piastre di fase6, la raccolta di grandi quantità di dati crio-EM ad alta risoluzione è relativamente semplice.
Cryo-ET offre una visione senza precedenti dell'ultrastruttura cellulare in uno stato nativo e idratato 7,8,9,10. Il limite principale è lo spessore del campione, ma con l'adozione di metodi come la fresatura a fascio ionico focalizzato (FIB), in cui i campioni cellulari e tissutali spessi vengono assottigliati per la tomografia11, l'orizzonte per ciò che può essere ripreso con crio-ET è in continua espansione. I microscopi più recenti sono in grado di produrre ben oltre 50 tomogrammi al giorno, e questo tasso è destinato ad aumentare solo a causa dello sviluppo di schemi di raccolta rapida dei dati12,13. L'analisi delle grandi quantità di dati prodotti da cryo-ET rimane un collo di bottiglia per questa modalità di imaging.
L'analisi quantitativa delle informazioni tomografiche richiede che prima siano annotate. Tradizionalmente, ciò richiede la segmentazione manuale da parte di un esperto, che richiede tempo; A seconda della complessità molecolare contenuta all'interno del criotogramma, possono essere necessarie ore o giorni di attenzione dedicata. Le reti neurali artificiali sono una soluzione interessante a questo problema poiché possono essere addestrate a svolgere la maggior parte del lavoro di segmentazione in una frazione del tempo. Le reti neurali convoluzionali (CNN) sono particolarmente adatte ai compiti di visione artificiale14 e sono state recentemente adattate per l'analisi dei tomogrammi crioelettronici15,16,17.
Le CNN tradizionali richiedono molte migliaia di campioni di addestramento annotati, il che spesso non è possibile per le attività di analisi delle immagini biologiche. Quindi, l'architettura U-Net ha eccelso in questo spazio18 perché si basa sull'aumento dei dati per addestrare con successo la rete, riducendo al minimo la dipendenza da grandi set di addestramento. Ad esempio, un'architettura U-Net può essere addestrata solo con poche sezioni di un singolo tomogramma (quattro o cinque fette) e dedotta in modo robusto ad altri tomogrammi senza riaddestramento. Questo protocollo fornisce una guida passo-passo per addestrare le architetture di rete neurale U-Net per segmentare i criotogrammi elettronici all'interno di Dragonfly 2022.119.
Dragonfly è un software sviluppato commercialmente utilizzato per la segmentazione e l'analisi di immagini 3D mediante modelli di deep learning ed è disponibile gratuitamente per uso accademico (si applicano alcune restrizioni geografiche). Ha un'interfaccia grafica avanzata che consente a un non esperto di sfruttare appieno le potenze del deep learning sia per la segmentazione semantica che per il denoising delle immagini. Questo protocollo dimostra come preelaborare e annotare tomogrammi crioelettronici all'interno di Dragonfly per addestrare reti neurali artificiali, che possono quindi essere dedotte per segmentare rapidamente grandi set di dati. Discute ulteriormente e dimostra brevemente come utilizzare i dati segmentati per ulteriori analisi, come il tracciamento dei filamenti e l'estrazione delle coordinate per la media dei sub-tomogrammi.
Protocollo
NOTA: Dragonfly 2022.1 richiede una workstation ad alte prestazioni. Le raccomandazioni di sistema sono incluse nella tabella dei materiali insieme all'hardware della workstation utilizzata per questo protocollo. Tutti i tomogrammi utilizzati in questo protocollo sono binned 4x da una dimensione dei pixel da 3,3 a 13,2 ang / pix. I campioni utilizzati nei risultati rappresentativi sono stati ottenuti da un'azienda (vedere la tabella dei materiali) che segue le linee guida per la cura degli animali che si allineano agli standard etici di questa istituzione. Il tomogramma utilizzato in questo protocollo e il multi-ROI generato come input di formazione sono stati inclusi come set di dati in bundle nel file supplementare 1 (che può essere trovato in https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct) in modo che l'utente possa seguire gli stessi dati se lo desidera. Dragonfly ospita anche un database ad accesso aperto chiamato Infinite Toolbox in cui gli utenti possono condividere reti addestrate.
1. Configurazione
- Modifica dell'area di lavoro predefinita:
- Per modificare l'area di lavoro in modo che rispecchi quella utilizzata in questo protocollo, sul lato sinistro del pannello Principale, scorrere verso il basso fino alla sezione Proprietà vista scena e deselezionare Mostra legende. Scorri verso il basso fino alla sezione Layout e seleziona la vista Scena singola e Quattro viste uguali .
- Per aggiornare l'unità predefinita, vai a File | Preferenze. Nella finestra che si apre, cambia l'unità predefinita da millimetri a nanometri.
- Utili keybinds predefiniti:
- Premere ESC per visualizzare il mirino nelle viste 2D e consentire la rotazione del volume 3D nella vista 3D. Premere X per nascondere il mirino nelle viste 2D e consentire la traduzione 2D e la traduzione del volume 3D nella vista 3D.
- Passa il mouse sopra il mirino per vedere piccole frecce che possono essere cliccate e trascinate per cambiare l'angolo del piano di visualizzazione nelle altre viste 2D.
- Premere Z per immettere lo stato dello zoom in entrambe le viste, consentendo agli utenti di fare clic e trascinare in un punto qualsiasi per ingrandire e rimpicciolire.
- Fate doppio clic su una vista nella scena Four View per mettere a fuoco solo quella vista; Fare di nuovo doppio clic per tornare a tutte e quattro le viste.
- Salvare periodicamente lo stato di avanzamento esportando tutto nella scheda Proprietà come oggetto ORS per una facile importazione. Seleziona tutti gli oggetti nell'elenco e fai clic con il pulsante destro del mouse su Esporta | Come obietta ORS. Assegnare un nome al file e salvare. In alternativa, vai a File | Salva sessione. Per utilizzare la funzione di salvataggio automatico nel software, abilitarla tramite File | Preferenze | Salvataggio automatico.
2. Importazione di immagini
- Per l'importazione di immagini, vai a File | Importare file immagine. Fare clic su Aggiungi, passare al file di immagine e fare clic su Apri | Successivo | Finire.
NOTA: il software non riconosce i file .rec. Tutti i tomogrammi devono avere il suffisso .mrc. Se utilizzi i dati forniti, vai invece su File | Importare oggetti. Individuare il file Training.ORSObject e fare clic su Apri, quindi fare clic su OK.
3. Pre-elaborazione (Figura 1.1)
- Creare una scala di intensità personalizzata (utilizzata per calibrare le intensità delle immagini tra i set di dati). Vai a Utilità | Responsabile unità dimensionali. In basso a sinistra, fate clic su + per creare una nuova unità di quota.
- Scegli una caratteristica ad alta intensità (luminosa) e bassa intensità (scura) che sia in tutti i tomogrammi di interesse. Dare all'unità un nome e un'abbreviazione (ad esempio, per questa scala, impostare le perline fiduciali su 0,0 Intensità standard e lo sfondo su 100,0). Salvate l'unità di quota personalizzata.
NOTA: una scala di intensità personalizzata è una scala arbitraria che viene creata e applicata ai dati per garantire che tutti i dati si trovino sulla stessa scala di intensità nonostante siano raccolti in momenti diversi o su apparecchiature diverse. Scegli le caratteristiche chiare e scure che meglio rappresentano la gamma in cui rientra il segnale. Se non ci sono fiduciali nei dati, scegli semplicemente la caratteristica più scura che verrà segmentata (la regione più scura della proteina, per esempio). - Per calibrare le immagini in base alla scala di intensità personalizzata, fare clic con il pulsante destro del mouse sul set di dati nella colonna Proprietà sul lato destro dello schermo e selezionare Calibra scala di intensità. Nella scheda Principale sul lato sinistro dello schermo, scorrere verso il basso fino alla sezione Sonda . Utilizzando lo strumento sonda circolare con un diametro appropriato, fare clic su alcuni punti nella regione di sfondo del tomogramma e registrare il numero medio nella colonna Intensità grezza ; ripetere l'operazione per i marcatori fiduciali, quindi fare clic su Calibra. Se necessario, regolate il contrasto per rendere nuovamente visibili le strutture con lo strumento Area nella sezione Livellamento finestra della scheda Principale.
- Filtraggio delle immagini:
NOTA: il filtraggio delle immagini può ridurre il rumore e aumentare il segnale. Questo protocollo utilizza tre filtri integrati nel software in quanto funzionano al meglio per questi dati, ma ci sono molti filtri disponibili. Una volta stabilito un protocollo di filtraggio delle immagini per i dati di interesse, sarà necessario applicare esattamente lo stesso protocollo a tutti i tomogrammi prima della segmentazione.- Nella scheda principale sul lato sinistro, scorri verso il basso fino al pannello di elaborazione delle immagini. Fare clic su Avanzate e attendere l'apertura di una nuova finestra. Dal pannello Proprietà selezionare il dataset da filtrare e renderlo visibile facendo clic sull'icona a forma di occhio a sinistra del dataset.
- Dal pannello Operazioni , utilizzate il menu a discesa per selezionare Equalizzazione istogramma (nella sezione Contrasto ) per la prima operazione. Seleziona Aggiungi operazione | Gaussian (nella sezione Smoothing ). Modificare la dimensione del kernel in 3D.
- Aggiungere una terza operazione; quindi, selezionate Contrasto (nella sezione Nitidezza). Lascia l'output per questo. Applicare a tutte le sezioni e lasciare che il filtro venga eseguito, quindi chiudere la finestra Elaborazione immagini per tornare all'interfaccia principale.
4. Creare dati di addestramento (Figura 1.2)
- Identificare l'area di addestramento nascondendo innanzitutto il set di dati non filtrato facendo clic sull'icona a forma di occhio a sinistra nel riquadro Proprietà dati . Quindi, visualizzare il set di dati appena filtrato (che verrà automaticamente denominato DataSet-HistEq-Gauss-Unsharp). Utilizzando il dataset filtrato, identificare una sottoregione del tomogramma che contenga tutte le caratteristiche di interesse.
- Per creare una casella intorno all'area di interesse, sul lato sinistro, nella scheda principale , scorri verso il basso fino alla categoria Forme e seleziona Crea una casella. Nel pannello Quattro viste, usate i diversi piani 2D per guidare/trascinare i bordi della casella in modo da racchiudere solo la regione di interesse in tutte le dimensioni. Nell'elenco dei dati, selezionare l'area Box e modificare il colore del bordo per una visualizzazione più semplice facendo clic sul quadrato grigio accanto al simbolo dell'occhio.
NOTA: la dimensione più piccola della patch per un U-Net 2D è 32 x 32 pixel; 400 x 400 x 50 pixel è una dimensione ragionevole per iniziare. - Per creare un ROI multiplo, nella parte sinistra, seleziona la scheda Segmentazione | Nuovo e seleziona Crea come multi-ROI. Assicurarsi che il numero di classi corrisponda al numero di feature di interesse + una classe di background. Assegnare un nome ai dati di training multi-ROI e assicurarsi che la geometria corrisponda al set di dati prima di fare clic su OK.
- Segmentazione dei dati di allenamento
- Scorrere i dati fino a raggiungere i limiti dell'area riquadrata. Seleziona Multi-ROI nel menu delle proprietà a destra. Fai doppio clic sul primo nome di classe vuota nel multi-ROI per assegnargli un nome.
- Dipingi con il pennello 2D. Nella scheda di segmentazione a sinistra, scorri verso il basso fino a Strumenti 2D e seleziona un pennello circolare. Quindi, seleziona Gaussiana adattiva o OTSU locale dal menu a discesa. Per colorare, tieni premuto il tasto Ctrl sinistro e fai clic. Per cancellare, tieni premuto Maiusc sinistro e fai clic.
NOTA: il pennello rifletterà il colore della classe attualmente selezionata. - Ripetere il passaggio precedente per ogni classe di oggetti nel multi-ROI. Assicurarsi che tutte le strutture all'interno della regione in scatola siano completamente segmentate o saranno considerate in background dalla rete.
- Quando tutte le strutture sono state etichettate, fare clic con il pulsante destro del mouse sulla classe Background nel Multi-ROI e selezionare Aggiungi tutti i voxel senza etichetta alla classe.
- Crea un nuovo ROI a classe singola denominato Mask. Assicuratevi che la geometria sia impostata sul dataset filtrato, quindi fate clic su Applica. Nella scheda delle proprietà a destra, fai clic con il pulsante destro del mouse sulla casella e seleziona Aggiungi a ROI. Aggiungilo al ROI della maschera.
- Per tagliare i dati di allenamento utilizzando la maschera, nella scheda Proprietà selezionare sia il ROI multiplo dei dati di allenamento che il ROI della maschera tenendo premuto Ctrl e facendo clic su ciascuno. Quindi, fare clic su Interseca sotto l'elenco delle proprietà dei dati nella sezione denominata Operazioni booleane. Denominare il nuovo dataset Trimmed Training Input e assicurarsi che la geometria corrisponda al dataset filtrato prima di fare clic su OK.
5. Utilizzo della procedura guidata di segmentazione per la formazione iterativa (Figura 1.3)
- Importare i dati di training nella procedura guidata di segmentazione facendo clic con il pulsante destro del mouse sul set di dati filtrato nella scheda Proprietà e quindi selezionando l'opzione Segmentazione guidata . Quando si apre una nuova finestra, cerca la scheda di input sul lato destro. Fare clic su Importa fotogrammi da un multi-ROI e selezionare l'input di allenamento tagliato.
- (Facoltativo) Crea un Visual Feedback Frame per monitorare i progressi dell'allenamento in tempo reale.
- Selezionate un frame dai dati non segmentati e fate clic su + per aggiungerlo come nuovo frame. Fate doppio clic sull'etichetta mista a destra del fotogramma e impostatela su Monitoraggio.
- Per generare un nuovo modello di rete neurale, sul lato destro della scheda Modelli , fare clic sul pulsante + per generare un nuovo modello. Selezionate U-Net dall'elenco, quindi per la quota di input, selezionate 2.5D e 5 fette, quindi fate clic su Genera (Generate).
- Per addestrare la rete, fare clic su Treno in basso a destra della finestra SegWiz .
NOTA: l'allenamento può essere interrotto in anticipo senza perdere i progressi. - Per utilizzare la rete addestrata per segmentare nuovi frame, al termine dell'addestramento U-Net, creare un nuovo frame e fare clic su Predict (in basso a destra). Quindi, fai clic sulla freccia Su nell'angolo in alto a destra del fotogramma previsto per trasferire la segmentazione al fotogramma reale.
- Per correggere la previsione, fare clic tenendo premuto Ctrl su due classi per modificare i pixel segmentati di una nell'altra. Selezionate entrambe le classi e colorate con il pennello per colorare solo i pixel appartenenti a una delle due classi. Correggete la segmentazione in almeno cinque nuovi fotogrammi.
NOTA: se si disegna con lo strumento pennello mentre entrambe le classi sono selezionate, invece di cancellare tenendo premuto Maiusc, come avviene normalmente, i pixel della prima classe verranno convertiti nella seconda. Ctrl-clic realizzerà il contrario. - Per l'allenamento iterativo, fare nuovamente clic sul pulsante Treno e consentire alla rete di allenarsi ulteriormente per altre 30-40 epoche, a quel punto interrompere l'allenamento e ripetere i passaggi 4.5 e 4.6 per un altro ciclo di allenamento.
NOTA: in questo modo, un modello può essere addestrato e migliorato in modo iterativo utilizzando un singolo set di dati. - Per pubblicare la rete, quando si è soddisfatti delle prestazioni, uscire dalla Segmentazione guidata. Nella finestra di dialogo che viene visualizzata automaticamente chiedendo quali modelli pubblicare (salvare), selezionare la rete riuscita, assegnarle un nome , quindi pubblicarla per rendere la rete disponibile per l'uso al di fuori della procedura guidata di segmentazione.
6. Applicare la rete (Figura 1.4)
- Per applicare prima il tomogramma di addestramento, selezionare il set di dati filtrato nel pannello Proprietà . Nel pannello Segmentazione a sinistra, scorri verso il basso fino alla sezione Segmento con AI . Assicurati che sia selezionato il set di dati corretto, scegli il modello appena pubblicato nel menu a discesa, quindi fai clic su Segmenta | Tutte le fette. In alternativa, selezionate Anteprima per visualizzare un'anteprima di una sezione della segmentazione.
- Per applicare a un set di dati di inferenza, importare il nuovo tomogramma. Pre-elaborazione secondo il passaggio 3 (Figura 1.1). Nel pannello Segmentazione, vai alla sezione Segmento con intelligenza artificiale. Assicurandosi che il tomogramma appena filtrato sia il set di dati selezionato, scegliere il modello sottoposto a training in precedenza e fare clic su Segmenta | Tutte le fette.
7. Manipolazione e pulizia della segmentazione
- Ripulisci rapidamente il rumore scegliendo prima una delle classi che ha segmentato il rumore e la caratteristica di interesse. Fare clic con il pulsante destro del mouse | Isole di processo | Rimuovi da Voxel Count | Seleziona una dimensione del voxel. Inizia in piccolo (~ 200) e aumenta gradualmente il conteggio per rimuovere la maggior parte del rumore.
- Per la correzione della segmentazione, tenete premuto Ctrl e fate clic su due classi per colorare solo i pixel appartenenti a tali classi. Ctrl-clic + trascinamento con gli strumenti di segmentazione per cambiare i pixel della seconda classe nella prima e Maiusc e clic su + trascinamento per ottenere il contrario. Continua a farlo per correggere rapidamente i pixel etichettati in modo errato.
- Separare i componenti collegati.
- Scegli una classe. Fare clic con il pulsante destro del mouse su una classe in Multi-ROI | Separare i componenti connessi per creare una nuova classe per ogni componente non connesso a un altro componente della stessa classe. Usa i pulsanti sotto il Multi-ROI per unire facilmente le classi.
- Esportare il ROI come Binary/TIFF.
- Scegli una classe nel Multi-ROI, quindi fai clic con il pulsante destro del mouse ed estrai classe come ROI. Nel pannello delle proprietà in alto, seleziona il nuovo ROI, fai clic con il pulsante destro del mouse | Esportazione | ROI come binario (assicurarsi che l'opzione per esportare tutte le immagini in un unico file sia selezionata).
NOTA: Gli utenti possono facilmente convertire dal formato tiff al formato mrc utilizzando il programma IMOD tif2mrc20. Questo è utile per il tracciamento dei filamenti.
- Scegli una classe nel Multi-ROI, quindi fai clic con il pulsante destro del mouse ed estrai classe come ROI. Nel pannello delle proprietà in alto, seleziona il nuovo ROI, fai clic con il pulsante destro del mouse | Esportazione | ROI come binario (assicurarsi che l'opzione per esportare tutte le immagini in un unico file sia selezionata).
8. Generazione di coordinate per la media dei sottotomogrammi dal ROI
- Estrarre una classe.
- Fare clic con il pulsante destro del mouse su Classe da utilizzare per la creazione della media | Estrai la classe come ROI. Clic destro sul ROI della classe | Componenti connessi | Nuovo Multi-ROI (26 connessi).
- Generare coordinate.
- Fare clic con il pulsante destro del mouse sul nuovo Multi-ROI | Generatore scalare. Espandi le misure di base con il set di dati | controlla Centro di massa ponderato X, Y e Z. Selezionare il set di dati e il calcolo. Fare clic con il pulsante destro del mouse su Multi-ROI | Esportare valori scalari. Seleziona Seleziona tutti gli slot scalari, quindi OK per generare le coordinate mondiali del centroide per ogni classe nel multi-ROI come file CSV.
NOTA: Se le particelle sono vicine tra loro e le segmentazioni si toccano, potrebbe essere necessario eseguire una trasformazione spartiacque per separare i componenti in un multi-ROI.
- Fare clic con il pulsante destro del mouse sul nuovo Multi-ROI | Generatore scalare. Espandi le misure di base con il set di dati | controlla Centro di massa ponderato X, Y e Z. Selezionare il set di dati e il calcolo. Fare clic con il pulsante destro del mouse su Multi-ROI | Esportare valori scalari. Seleziona Seleziona tutti gli slot scalari, quindi OK per generare le coordinate mondiali del centroide per ogni classe nel multi-ROI come file CSV.
9. Trasformazione spartiacque
- Estrarre la classe facendo clic con il pulsante destro del mouse sulla classe in Multi-ROI da utilizzare per la media | Estrai la classe come ROI. Assegna un nome a questa maschera ROI Watershed.
- (Facoltativo) Chiudere i fori.
- Se le particelle segmentate hanno buchi o aperture, chiuderli per lo spartiacque. Fare clic sul ROI in Proprietà dati. Nella scheda Segmentazione (a sinistra), vai a Operazioni morfologiche e usa qualsiasi combinazione di Dilatare, Erodere e Chiudi necessaria per ottenere segmentazioni solide senza fori.
- Invertire il ROI facendo clic sul ROI | Copiare l'oggetto selezionato (sotto Proprietà dati). Seleziona il ROI copiato e sul lato sinistro della scheda Segmentazione fai clic su Inverti.
- Creare una mappa delle distanze facendo clic con il pulsante destro del mouse sul ROI invertito | Crea mappatura di | Mappa delle distanze. Per un uso successivo, fare una copia della mappa delle distanze e invertirla (fare clic con il pulsante destro del mouse | Modifica e trasformazione | Inverti valori | Applica). Assegna a questa mappa invertita il nome Paesaggio.
- Creare punti iniziali.
- Nascondere il ROI e visualizzare la mappa delle distanze. Nella scheda Segmentazione , fate clic su Definisci intervallo (Define Range ) e riducete l'intervallo fino a evidenziare solo pochi pixel al centro di ciascun punto e nessuno è collegato a un altro punto. Nella parte inferiore della sezione Intervallo , fai clic su Aggiungi a nuovo. Denominare questo nuovo ROI Seedpoints.
- Eseguire la trasformazione spartiacque.
- Fare clic con il pulsante destro del mouse sul ROI di Seedpoints | Componenti connessi | Nuovo Multi-ROI (26 connessi). Fare clic con il pulsante destro del mouse sul multi-ROI appena generato | Trasformazione spartiacque. Selezionare la mappa delle distanze denominata Paesaggio e fare clic su OK; selezionare il ROI denominato Watershed Mask e fare clic su OK per calcolare una trasformazione spartiacque da ciascun punto di inizializzazione e separare le singole particelle in classi separate nel multi-ROI. Generare le coordinate come nel passaggio 8.2.
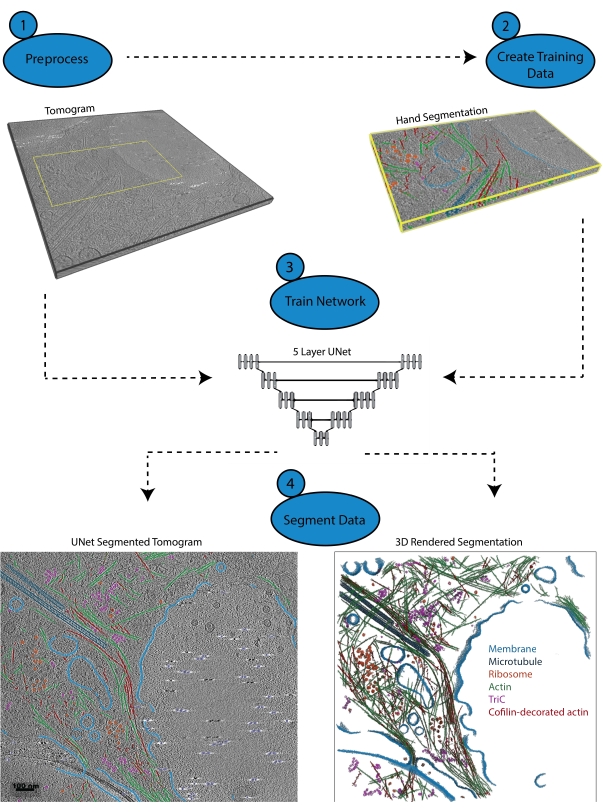
Figura 1: Flusso di lavoro. 1) Pre-elaborare il tomogramma di addestramento calibrando la scala di intensità e filtrando il set di dati. 2) Creare i dati di allenamento segmentando manualmente una piccola porzione di un tomogramma con tutte le etichette appropriate che l'utente desidera identificare. 3) Utilizzando il tomogramma filtrato come input e la segmentazione della mano come output di allenamento, un U-Net multi-fetta a cinque strati viene addestrato nella procedura guidata di segmentazione. 4) La rete addestrata può essere applicata al tomogramma completo per annotarlo e un rendering 3D può essere generato da ogni classe segmentata. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
Seguendo il protocollo, un U-Net a cinque fette è stato addestrato su un singolo tomogramma (Figura 2A) per identificare cinque classi: membrana, microtubuli, actina, marcatori fiduciali e background. La rete è stata addestrata iterativamente per un totale di tre volte, quindi applicata al tomogramma per segmentarla e annotarla completamente (Figura 2B,C). La pulizia minima è stata eseguita utilizzando i passaggi 7.1 e 7.2. I successivi tre tomogrammi di interesse (Figura 2D,G,J) sono stati caricati nel software per la pre-elaborazione. Prima dell'importazione dell'immagine, uno dei tomogrammi (Figura 2J) richiedeva la regolazione della dimensione dei pixel da 17,22 Å / px a 13,3 Å / px poiché veniva raccolto su un microscopio diverso con un ingrandimento leggermente diverso. Il programma IMOD squeezevol è stato utilizzato per il ridimensionamento con il seguente comando:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
In questo comando, -f si riferisce al fattore con cui modificare la dimensione dei pixel (in questo caso: 13.3/17.22). Dopo l'importazione, tutti e tre gli obiettivi di inferenza sono stati pre-elaborati secondo i passaggi 3.2 e 3.3, quindi è stato applicato U-Net a cinque sezioni. È stata nuovamente eseguita una pulizia minima. Le segmentazioni finali sono visualizzate nella Figura 2.
Le segmentazioni dei microtubuli da ciascun tomogramma sono state esportate come file TIF binari (passo 7.4), convertiti in MRC (programma IMOD tif2mrc ) e quindi utilizzati per la correlazione dei cilindri e il tracciamento dei filamenti. Le segmentazioni binarie dei filamenti si traducono in un tracciamento dei filamenti molto più robusto rispetto al tracciamento sui tomogrammi. Le mappe di coordinate dal tracciamento dei filamenti (Figura 3) saranno utilizzate per ulteriori analisi, come le misurazioni del vicino più vicino (impacchettamento del filamento) e la media del subtomogramma elicoidale lungo i singoli filamenti per determinare l'orientamento dei microtubuli.
Le reti non riuscite o non adeguatamente addestrate sono facili da determinare. Una rete guasta non sarà in grado di segmentare alcuna struttura, mentre una rete non adeguatamente addestrata in genere segmenterà correttamente alcune strutture e avrà un numero significativo di falsi positivi e falsi negativi. Queste reti possono essere corrette e addestrate iterativamente per migliorare le loro prestazioni. La procedura guidata di segmentazione calcola automaticamente il coefficiente di somiglianza dei dadi di un modello (chiamato punteggio nella SegWiz) dopo che è stato addestrato. Questa statistica fornisce una stima della somiglianza tra i dati di allenamento e la segmentazione U-Net. Dragonfly 2022.1 ha anche uno strumento integrato per valutare le prestazioni di un modello a cui è possibile accedere nella scheda Intelligenza artificiale nella parte superiore dell'interfaccia (vedere la documentazione per l'utilizzo).
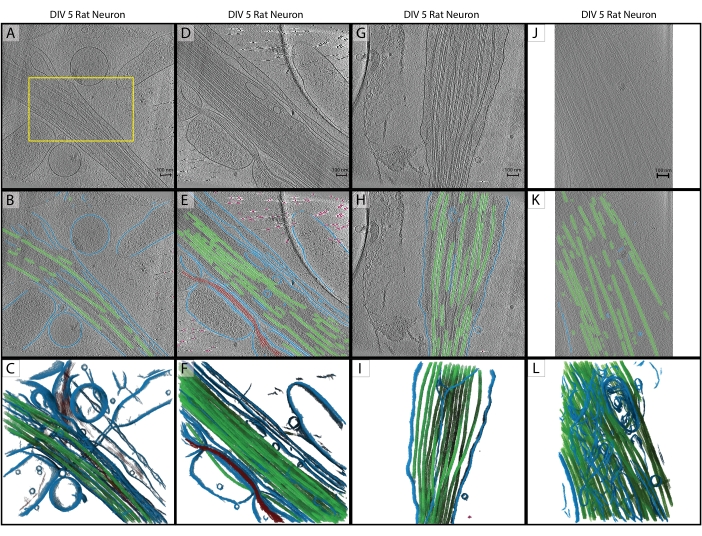
Figura 2: Inferenza. (A-C) Tomogramma di addestramento originale di un neurone di ratto ippocampale DIV 5, raccolto nel 2019 su un Titan Krios. Questa è una ricostruzione retroproiettata con correzione CTF in IMOD. (A) La casella gialla rappresenta la regione in cui è stata eseguita la segmentazione della mano per l'input di formazione. (B) Segmentazione 2D da U-Net al termine dell'addestramento. (C) Rendering 3D delle regioni segmentate che mostrano membrana (blu), microtubuli (verde) e actina (rosso). (D-F) DIV 5 neurone di ratto ippocampale dalla stessa sessione del tomogramma di allenamento. (E) Segmentazione 2D da U-Net senza formazione aggiuntiva e pulizia rapida. Membrana (blu), microtubuli (verde), actina (rosso), fiduciali (rosa). (F) Rendering 3D delle regioni segmentate. (G-I) DIV 5 neurone di ratto ippocampale dalla sessione 2019. (H) segmentazione 2D da U-Net con pulizia rapida e (I) rendering 3D. (J-L) DIV 5 neurone di ratto ippocampale, raccolto nel 2021 su un diverso Titan Krios a un diverso ingrandimento. La dimensione dei pixel è stata modificata con il programma IMOD squeezevol per abbinare il tomogramma di allenamento. (K) Segmentazione 2D dalla rete U-Net con pulizia rapida, dimostrando una solida inferenza tra i set di dati con un'adeguata pre-elaborazione e (L) rendering 3D della segmentazione. Barre di scala = 100 nm. Abbreviazioni: DIV = giorni in vitro; CTF = funzione di trasferimento del contrasto. Fare clic qui per visualizzare una versione ingrandita di questa figura.
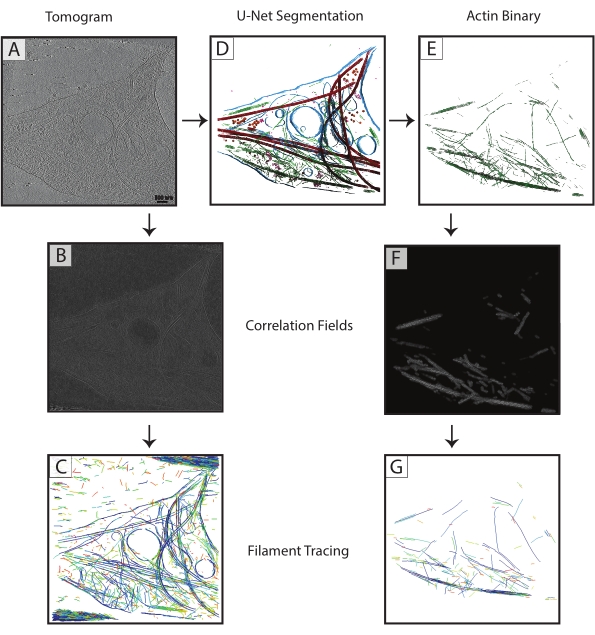
Figura 3: Miglioramento del tracciamento dei filamenti . (A) Tomogramma di un neurone ippocampale di ratto DIV 4, raccolto su un Titan Krios. (B) Mappa di correlazione generata dalla correlazione dei cilindri sui filamenti di actina. (C) Tracciatura dei filamenti di actina utilizzando le intensità dei filamenti di actina nella mappa di correlazione per definire i parametri. Il tracciamento cattura la membrana e i microtubuli, così come il rumore, mentre cerca di tracciare solo l'actina. (D) Segmentazione U-Net del tomogramma. Membrana evidenziata in blu, microtubuli in rosso, ribosomi in arancione, triC in viola e actina in verde. (E) Segmentazione dell'actina estratta come maschera binaria per il tracciamento dei filamenti. (F) Mappa di correlazione generata dalla correlazione del cilindro con gli stessi parametri di (B). (G) Tracciatura dei filamenti significativamente migliorata dei soli filamenti di actina dal tomogramma. Abbreviazione: DIV = giorni in vitro. Fare clic qui per visualizzare una versione ingrandita di questa figura.
File supplementare 1: il tomogramma utilizzato in questo protocollo e il multi-ROI generato come input di training sono inclusi come set di dati in bundle (Training.ORSObject). Vedi https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Discussione
Questo protocollo stabilisce una procedura per l'utilizzo del software Dragonfly 2022.1 per addestrare un U-Net multiclasse da un singolo tomogramma e come dedurre quella rete ad altri tomogrammi che non devono provenire dallo stesso set di dati. La formazione è relativamente veloce (può essere veloce come 3-5 minuti per epoca o lenta come poche ore, a seconda interamente della rete che viene addestrata e dell'hardware utilizzato), e riqualificare una rete per migliorare il suo apprendimento è intuitivo. Finché le fasi di pre-elaborazione vengono eseguite per ogni tomogramma, l'inferenza è in genere robusta.
La pre-elaborazione coerente è il passaggio più critico per l'inferenza del deep learning. Ci sono molti filtri di imaging nel software e l'utente può sperimentare per determinare quali filtri funzionano meglio per particolari set di dati; Si noti che qualsiasi filtro utilizzato sul tomogramma di addestramento deve essere applicato allo stesso modo ai tomogrammi di inferenza. Occorre inoltre prestare attenzione a fornire alla rete informazioni di formazione accurate e sufficienti. È fondamentale che tutte le caratteristiche segmentate all'interno delle sezioni di allenamento siano segmentate nel modo più accurato e preciso possibile.
La segmentazione delle immagini è facilitata da una sofisticata interfaccia utente di livello commerciale. Fornisce tutti gli strumenti necessari per la segmentazione della mano e consente la semplice riassegnazione dei voxel da una classe all'altra prima dell'addestramento e della riqualificazione. L'utente è autorizzato a segmentare a mano i voxel all'interno dell'intero contesto del tomogramma, e gli vengono date più viste e la possibilità di ruotare liberamente il volume. Inoltre, il software offre la possibilità di utilizzare reti multiclasse, che tendono a funzionare meglio16 e sono più veloci rispetto alla segmentazione con più reti a classe singola.
Ci sono, naturalmente, limitazioni alle capacità di una rete neurale. I dati Cryo-ET sono, per natura, molto rumorosi e limitati nel campionamento angolare, il che porta a distorsioni specifiche dell'orientamento in oggetti identici21. La formazione si basa su un esperto per segmentare accuratamente le strutture e una rete di successo è buona (o cattiva) solo quanto i dati di addestramento che viene fornita. Il filtraggio delle immagini per aumentare il segnale è utile per il trainer, ma ci sono ancora molti casi in cui identificare con precisione tutti i pixel di una determinata struttura è difficile. È quindi importante prestare molta attenzione quando si crea la segmentazione della formazione in modo che la rete abbia le migliori informazioni possibili per apprendere durante la formazione.
Questo flusso di lavoro può essere facilmente modificato in base alle preferenze di ciascun utente. Mentre è essenziale che tutti i tomogrammi siano pre-elaborati esattamente nello stesso modo, non è necessario utilizzare i filtri esatti utilizzati nel protocollo. Il software ha numerose opzioni di filtraggio delle immagini e si consiglia di ottimizzarle per i dati particolari dell'utente prima di intraprendere un grande progetto di segmentazione che copre molti tomogrammi. Ci sono anche alcune architetture di rete disponibili per l'uso: un U-Net multi-slice è stato trovato per funzionare meglio per i dati di questo laboratorio, ma un altro utente potrebbe scoprire che un'altra architettura (come un U-Net 3D o un Sensor 3D) funziona meglio. La procedura guidata di segmentazione fornisce una comoda interfaccia per confrontare le prestazioni di più reti utilizzando gli stessi dati di addestramento.
Strumenti come quelli presentati qui renderanno la segmentazione manuale di tomogrammi completi un compito del passato. Con reti neurali ben addestrate che sono solidamente desumibili, è del tutto fattibile creare un flusso di lavoro in cui i dati tomografici vengono ricostruiti, elaborati e completamente segmentati con la stessa rapidità con cui il microscopio può raccoglierli.
Divulgazioni
La licenza ad accesso aperto per questo protocollo è stata pagata da Object Research Systems.
Riconoscimenti
Questo studio è stato sostenuto dal Penn State College of Medicine e dal Dipartimento di Biochimica e Biologia Molecolare, nonché dalla sovvenzione del Tobacco Settlement Fund (TSF) 4100079742-EXT. I servizi e gli strumenti CryoEM e CryoET Core (RRID: SCR_021178) utilizzati in questo progetto sono stati finanziati, in parte, dal Pennsylvania State University College of Medicine attraverso l'Ufficio del Vice Decano della Ricerca e degli Studenti Laureati e il Dipartimento della Salute della Pennsylvania utilizzando Tobacco Settlement Funds (CURE). Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali dell'Università o del College of Medicine. Il Dipartimento della Salute della Pennsylvania declina espressamente la responsabilità per eventuali analisi, interpretazioni o conclusioni.
Materiali
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Riferimenti
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon