Method Article
Сегментация криоэлектронных томограмм на основе глубокого обучения
В этой статье
Резюме
Это метод обучения многосрезовой U-Net для многоклассовой сегментации криоэлектронных томограмм с использованием части одной томограммы в качестве обучающего входа. Мы описываем, как вывести эту сеть из других томограмм и как извлечь сегментации для дальнейшего анализа, такого как усреднение субтомограмм и трассировка нитей.
Аннотация
Криоэлектронная томография (крио-ЭТ) позволяет исследователям визуализировать клетки в их естественном, гидратированном состоянии с максимально возможным в настоящее время разрешением. Однако этот метод имеет несколько ограничений, которые делают анализ данных, которые он генерирует, трудоемким и сложным. Ручная сегментация одной томограммы может занять от нескольких часов до нескольких дней, но микроскоп может легко генерировать 50 или более томограмм в день. Современные программы сегментации глубокого обучения для крио-ЭТ существуют, но ограничены сегментацией одной структуры за раз. Здесь многосрезовые сверточные нейронные сети U-Net обучаются и применяются для автоматической сегментации нескольких структур одновременно в криотомограммах. При надлежащей предварительной обработке эти сети могут быть надежно выведены для многих томограмм без необходимости обучения отдельных сетей для каждой томограммы. Этот рабочий процесс значительно повышает скорость, с которой криоэлектронные томограммы могут быть проанализированы, сокращая время сегментации до 30 минут в большинстве случаев. Кроме того, сегментация может быть использована для повышения точности отслеживания нитей в клеточном контексте и для быстрого извлечения координат для усреднения субтомограммы.
Введение
Аппаратные и программные разработки последнего десятилетия привели к «революции разрешения» для криоэлектронной микроскопии (крио-ЭМ)1,2. Благодаря более совершенным и быстрым детекторам3, программному обеспечению для автоматизации сбора данных4,5 и достижениям в области усиления сигнала, таким как фазовые пластины6, сбор больших объемов крио-ЭМ-данных высокого разрешения является относительно простым.
Cryo-ET обеспечивает беспрецедентное понимание клеточной ультраструктуры в нативном, гидратированном состоянии 7,8,9,10. Основным ограничением является толщина образца, но с принятием таких методов, как фрезерование сфокусированным ионным пучком (FIB), при котором толстые клеточные и тканевые образцы истончаются для томографии11, горизонт того, что можно визуализировать с помощью крио-ET, постоянно расширяется. Новейшие микроскопы способны производить более 50 томограмм в день, и этот показатель, по прогнозам, будет только увеличиваться из-за разработки схем быстрого сбора данных12,13. Анализ огромных объемов данных, полученных с помощью крио-ЭТ, остается узким местом для этого метода визуализации.
Количественный анализ томографической информации требует, чтобы она сначала была аннотирована. Традиционно для этого требуется сегментация рук экспертом, что отнимает много времени; В зависимости от молекулярной сложности, содержащейся в криотомограмме, это может занять от нескольких часов до нескольких дней. Искусственные нейронные сети являются привлекательным решением этой проблемы, поскольку их можно обучить выполнять основную часть работы по сегментации за долю времени. Сверточные нейронные сети (CNN) особенно подходят для задач компьютерного зрения14 и недавно были адаптированы для анализа криоэлектронных томограмм15,16,17.
Традиционные CNN требуют многих тысяч аннотированных обучающих выборок, что не часто возможно для задач анализа биологических изображений. Следовательно, архитектура U-Net преуспела в этом пространстве18 , потому что она полагается на увеличение данных для успешного обучения сети, сводя к минимуму зависимость от больших обучающих наборов. Например, архитектура U-Net может быть обучена с помощью всего нескольких срезов одной томограммы (четыре или пять срезов) и надежно выведена для других томограмм без повторного обучения. Этот протокол предоставляет пошаговое руководство по обучению архитектур нейронных сетей U-Net для сегментации электронных криотомограмм в Dragonfly 2022.119.
Dragonfly — это коммерчески разработанное программное обеспечение, используемое для сегментации и анализа 3D-изображений с помощью моделей глубокого обучения, и оно свободно доступно для академического использования (применяются некоторые географические ограничения). Он имеет расширенный графический интерфейс, который позволяет неспециалисту в полной мере использовать возможности глубокого обучения как для семантической сегментации, так и для шумоподавления изображения. Этот протокол демонстрирует, как предварительно обрабатывать и аннотировать криоэлектронные томограммы в Dragonfly для обучения искусственных нейронных сетей, которые затем могут быть выведены для быстрого сегментирования больших наборов данных. Далее обсуждается и кратко демонстрируется, как использовать сегментированные данные для дальнейшего анализа, такого как трассировка нитей и выделение координат для усреднения субтомограммы.
протокол
ПРИМЕЧАНИЕ: Для Dragonfly 2022.1 требуется высокопроизводительная рабочая станция. Системные рекомендации включены в Таблицу материалов вместе с аппаратным обеспечением рабочей станции, используемой для этого протокола. Все томограммы, используемые в этом протоколе, объединяются в 4 раза от размера пикселя от 3,3 до 13,2 ang/pix. Образцы, использованные в репрезентативных результатах, были получены от компании (см. Таблицу материалов), которая следует рекомендациям по уходу за животными, которые соответствуют этическим стандартам этого учреждения. Томограмма, используемая в этом протоколе, и мульти-ROI, которая была сгенерирована в качестве обучающих входных данных, были включены в виде объединенного набора данных в дополнительный файл 1 (который можно найти по адресу https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct), чтобы пользователь мог следить за теми же данными, если захочет. Dragonfly также имеет базу данных открытого доступа под названием Infinite Toolbox, где пользователи могут совместно использовать обученные сети.
1. Настройка
- Изменение рабочей области по умолчанию:
- Чтобы изменить рабочую область так, чтобы она отражала рабочую область, используемую в этом протоколе, в левой части главной панели прокрутите вниз до раздела « Свойства вида сцены» и снимите флажок « Показать легенды». Прокрутите вниз до раздела « Макет » и выберите представление « Одна сцена» и «Четыре равных вида ».
- Чтобы обновить единицу измерения по умолчанию, перейдите в меню Файл | Предпочтения. В открывшемся окне измените единицу измерения по умолчанию с миллиметров на нанометры.
- Полезные привязки клавиш по умолчанию:
- Нажмите клавишу Esc, чтобы отобразить перекрестие в 2D-видах и разрешить поворот 3D-объема в 3D-виде. Нажмите X, чтобы скрыть перекрестие в 2D-видах и разрешить 2D-перевод и 3D-перевод объема в 3D-виде.
- Наведите курсор на перекрестие , чтобы увидеть маленькие стрелки , которые можно щелкнуть и перетащить, чтобы изменить угол плоскости обзора в других 2D-видах.
- Нажмите Z, чтобы войти в состояние масштабирования в обоих видах, позволяя пользователям щелкать и перетаскивать в любое место для увеличения и уменьшения масштаба .
- Дважды щелкните вид в сцене Four View , чтобы сфокусироваться только на этом виде; Дважды щелкните еще раз, чтобы вернуться ко всем четырем представлениям.
- Периодически сохраняйте ход выполнения, экспортируя все, что находится на вкладке "Свойства ", в виде объекта ORS для удобного импорта. Выберите все объекты в списке и щелкните правой кнопкой мыши Экспорт | Как возражает ОРС. Назовите файл и сохраните. Кроме того, перейдите в раздел Файл | Сохранить сессию. Чтобы использовать функцию автосохранения в программном обеспечении, включите ее через Файл | Предпочтения | Автосохранение.
2. Импорт изображений
- Для импорта изображений перейдите в раздел Файл | Импорт файлов изображений. Нажмите кнопку Добавить, перейдите к файлу изображения и нажмите кнопку Открыть | Далее | Финиш.
ПРИМЕЧАНИЕ: Программное обеспечение не распознает файлы .rec. Все томограммы должны иметь суффикс .mrc. Если вы используете предоставленные данные, вместо этого перейдите в Файл | Импорт объектов. Перейдите к файлу Training.ORSObject и нажмите кнопку « Открыть», а затем нажмите кнопку «ОК».
3. Предварительная обработка (рис. 1.1)
- Создайте пользовательскую шкалу интенсивности (используется для калибровки интенсивности изображения в наборах данных). Перейти в раздел «Коммунальные услуги» | Менеджер измерения единицы. В левом нижнем углу нажмите + , чтобы создать новую единицу измерения.
- Выберите признак высокой интенсивности (яркий) и низкой интенсивности (темный), который присутствует во всех интересующих вас томограммах. Дайте устройству название и аббревиатуру (например, для этой шкалы установите фидуциальные бусины на 0,0 стандартной интенсивности , а фон на 100,0). Сохраните единицу измерения пользовательского размера.
ПРИМЕЧАНИЕ: Пользовательская шкала интенсивности - это произвольная шкала, которая создается и применяется к данным, чтобы гарантировать, что все данные находятся на одной и той же шкале интенсивности, несмотря на то, что они собраны в разное время или на разном оборудовании. Выбирайте светлые и темные объекты, которые лучше всего представляют диапазон, в который попадает сигнал. Если в данных нет фидуциалов, просто выберите самый темный объект, который будет сегментирован (например, самую темную область белка). - Чтобы откалибровать изображения в соответствии с пользовательской шкалой интенсивности, щелкните правой кнопкой мыши набор данных в столбце «Свойства» в правой части экрана и выберите «Калибровка шкалы интенсивности». На вкладке «Основные» в левой части экрана прокрутите вниз до раздела «Зонд». Используя инструмент кругового зонда соответствующего диаметра, щелкните несколько мест в фоновой области томограммы и запишите среднее число в столбце «Необработанная интенсивность»; повторите эти действия для реперных маркеров, затем нажмите «Калибровка». При необходимости отрегулируйте контрастность, чтобы снова сделать структуры видимыми с помощью инструмента «Область» в разделе «Выравнивание окна» вкладки «Основные».
- Фильтрация изображений:
ПРИМЕЧАНИЕ: Фильтрация изображений может уменьшить шум и усилить сигнал. Этот протокол использует три фильтра, встроенных в программное обеспечение, поскольку они лучше всего работают с этими данными, но доступно множество фильтров. После того, как мы определились с протоколом фильтрации изображений для интересующих данных, необходимо будет применить один и тот же протокол ко всем томограммам перед сегментацией.- На главной вкладке слева прокрутите вниз до панели обработки изображений. Нажмите «Дополнительно » и дождитесь открытия нового окна. На панели «Свойства » выберите набор данных, который нужно отфильтровать, и сделайте его видимым, щелкнув значок глаза слева от набора данных.
- На панели « Операции » в раскрывающемся меню выберите « Выравнивание гистограммы » (в разделе «Контрастность ») для первой операции. Выберите Добавить операцию | Гауссов (в разделе «Сглаживание »). Измените размер ядра на 3D.
- Добавьте третью операцию; затем выберите «Нерезкий » (в разделе « Резкость »). Оставьте вывод для этого. Примените ко всем фрагментам и дайте фильтрации запуститься, затем закройте окно обработки изображений , чтобы вернуться к основному интерфейсу.
4. Создание обучающих данных (рис. 1.2)
- Определите область обучения, сначала скрыв нефильтрованный набор данных, щелкнув значок глаза слева от него на панели « Свойства данных ». Затем покажите только что отфильтрованный набор данных (который будет автоматически называться DataSet-HistEq-Gauss-Unsharp). Используя отфильтрованный набор данных, определите подобласть томограммы, содержащую все интересующие объекты.
- Чтобы создать рамку вокруг интересующей области, слева на главной вкладке прокрутите вниз до категории « Фигуры » и выберите « Создать поле». Находясь на панели «Четыре вида », используйте различные 2D-плоскости , чтобы направлять/перетаскивать края прямоугольника, чтобы охватить только интересующую область во всех измерениях. В списке данных выберите область «Поле » и измените цвет границы для более удобного просмотра, щелкнув серый квадрат рядом с символом глаза.
ПРИМЕЧАНИЕ: Наименьший размер патча для 2D U-Net составляет 32 x 32 пикселя; 400 x 400 x 50 пикселей — разумный размер коробки для начала. - Чтобы создать мульти-ROI, в левой части выберите вкладку Сегментация | Создать и установить флажок Создать как Multi-ROI. Убедитесь, что количество классов соответствует количеству интересующих объектов + фоновый класс. Назовите обучающие данные с несколькими ROI и убедитесь, что геометрия соответствует набору данных, прежде чем нажимать кнопку ОК.
- Сегментация обучающих данных
- Прокрутите данные до тех пор, пока они не окажутся в пределах области в рамке. Выберите Multi-ROI в меню свойств справа. Дважды щелкните первое пустое имя класса в мульти-ROI, чтобы присвоить ему имя.
- Рисуйте 2D-кистью. На вкладке сегментации слева прокрутите вниз до 2D-инструментов и выберите круглую кисть. Затем выберите «Адаптивный гауссов» или « Локальный OTSU » в раскрывающемся меню. Чтобы раскрасить, удерживайте левую клавишу ctrl и щелкните. Чтобы стереть, удерживайте левый сдвиг и щелкните.
ПРИМЕЧАНИЕ: Кисть будет отражать цвет выбранного в данный момент класса. - Повторите предыдущий шаг для каждого класса объектов в мульти-ROI. Убедитесь, что все структуры в коробочном регионе полностью сегментированы, иначе они будут рассматриваться сетью как фоновые.
- Когда все структуры будут помечены, щелкните правой кнопкой мыши класс Background в Multi-ROI и выберите Add All Unlabeled Voxels to Class (Добавить все непомеченные воксели в класс).
- Создайте новый одноклассный ROI с именем Mask. Убедитесь, что геометрия соответствует отфильтрованному набору данных, затем нажмите кнопку «Применить». На вкладке свойств справа щелкните правой кнопкой мыши поле и выберите «Добавить к рентабельности инвестиций». Добавьте его в ROI маски.
- Чтобы обрезать обучающие данные с помощью маски, на вкладке « Свойства » выберите «Мультирентабельность инвестиций в обучающие данные » и « Рентабельность инвестиций в маску », удерживая клавишу Ctrl и щелкнув каждый из них. Затем нажмите кнопку « Пересечь» под списком свойств данных в разделе « Логические операции». Назовите новый набор данных Trimped Training Input и убедитесь, что геометрия соответствует отфильтрованному набору данных, прежде чем нажимать кнопку ОК.
5. Использование мастера сегментации для итеративного обучения (рис. 1.3)
- Импортируйте обучающие данные в мастер сегментации, сначала щелкнув правой кнопкой мыши отфильтрованный набор данных на вкладке Свойства , а затем выбрав параметр Мастер сегментации . Когда откроется новое окно, найдите вкладку ввода справа. Нажмите «Импортировать кадры из Multi-ROI» и выберите « Обрезанные входные данные обучения».
- (Необязательно) Создайте визуальную рамку обратной связи, чтобы отслеживать прогресс обучения в режиме реального времени.
- Выберите кадр из данных, который не сегментирован, и нажмите + , чтобы добавить его в качестве нового кадра. Дважды щелкните смешанную метку справа от фрейма и измените ее на «Мониторинг».
- Чтобы создать новую модель нейронной сети, справа на вкладке «Модели » нажмите кнопку + , чтобы создать новую модель. Выберите U-Net из списка, а затем в качестве входного параметра выберите 2.5D и 5 срезов, а затем нажмите кнопку Создать.
- Чтобы обучить сеть, нажмите «Обучить » в правом нижнем углу окна SegWiz .
ПРИМЕЧАНИЕ: Тренировки можно прекратить досрочно без потери прогресса. - Чтобы использовать обученную сеть для сегментации новых кадров, после завершения обучения U-Net создайте новый кадр и нажмите «Предсказать » (внизу справа). Затем нажмите стрелку вверх в правом верхнем углу прогнозируемого кадра, чтобы перенести сегментацию в реальный кадр.
- Чтобы исправить прогноз, щелкните два класса, удерживая нажатой клавишу Ctrl, чтобы изменить сегментированные пикселы одного на другой. Выберите оба класса и рисуйте кистью , чтобы рисовать только пикселы, принадлежащие к одному из классов. Исправьте сегментацию как минимум в пяти новых кадрах.
ПРИМЕЧАНИЕ: Рисование с помощью инструмента «Кисть», когда выбраны оба класса, означает, что вместо стирания при нажатой клавише «Shift», как это обычно происходит, он преобразует пиксели первого класса во второй. Щелчок с нажатой клавишей Ctrl сделает обратное. - Для итеративного обучения снова нажмите кнопку «Обучить » и позвольте сети продолжить обучение еще 30-40 эпох, после чего остановите обучение и повторите шаги 4.5 и 4.6 для следующего раунда обучения.
ПРИМЕЧАНИЕ: Таким образом, модель может быть итеративно обучена и улучшена с использованием одного набора данных. - Чтобы опубликовать сеть, когда она будет удовлетворена ее производительностью, выйдите из мастера сегментации. В автоматически всплывающем диалоговом окне с вопросом о том, какие модели следует опубликовать (сохранить), выберите успешную сеть, назовите ее, а затем опубликуйте , чтобы сделать сеть доступной для использования вне мастера сегментации.
6. Примените сеть (рис. 1.4)
- Чтобы сначала применить к обучающей томограмме, выберите отфильтрованный набор данных на панели «Свойства ». На панели «Сегментация » слева прокрутите вниз до раздела «Сегмент с искусственным интеллектом ». Убедитесь, что выбран правильный набор данных, выберите только что опубликованную модель в раскрывающемся меню, затем нажмите «Сегмент» | Все ломтики. Кроме того, можно выбрать «Предварительный просмотр », чтобы просмотреть сегментацию в виде одного фрагмента.
- Чтобы применить его к набору данных вывода, импортируйте новую томограмму. Предварительная обработка в соответствии с Шагом 3 (рис. 1.1). На панели «Сегментация» перейдите в раздел «Сегмент с ИИ». Убедившись, что только что отфильтрованная томограмма является выбранным набором данных, выберите ранее обученную модель и нажмите кнопку Сегмент | Все ломтики.
7. Манипуляции с сегментацией и очистка
- Быстро очистите шум, сначала выбрав один из классов с сегментированным шумом и интересующей особенностью. Щелчок правой кнопкой мыши | Технологические острова | Удалить с помощью Voxel Count | Выберите размер вокселей. Начните с малого (~ 200) и постепенно увеличивайте количество, чтобы удалить большую часть шума.
- Для коррекции сегментации щелкните два класса, удерживая клавишу Ctrl , чтобы закрасить только пикселы, принадлежащие этим классам. Щелкните, удерживая нажатой клавишу Ctrl + перетащить , с помощью инструментов сегментации , чтобы изменить пиксели второго класса на первый, и щелкните, удерживая нажатой клавишу Shift, + перетаскивание, чтобы добиться обратного. Продолжайте делать это, чтобы быстро исправить неправильно помеченные пиксели.
- Отдельные подключенные компоненты.
- Выберите класс. Щелкните правой кнопкой мыши класс в Multi-ROI | Разделите подключенные компоненты , чтобы создать новый класс для каждого компонента, который не подключен к другому компоненту того же класса. Используйте кнопки под Multi-ROI, чтобы легко объединить классы.
- Экспортируйте ROI в виде двоичного файла / TIFF.
- Выберите класс в Multi-ROI, затем щелкните правой кнопкой мыши и извлеките класс как ROI. На панели свойств выше выберите новый ROI, щелкните правой кнопкой мыши | Экспорт | ROI как двоичный (убедитесь, что выбрана опция экспорта всех изображений в один файл ).
ПРИМЕЧАНИЕ: Пользователи могут легко конвертировать из tiff в формат mrc с помощью программы IMOD tif2mrc20. Это полезно для трассировки нити.
- Выберите класс в Multi-ROI, затем щелкните правой кнопкой мыши и извлеките класс как ROI. На панели свойств выше выберите новый ROI, щелкните правой кнопкой мыши | Экспорт | ROI как двоичный (убедитесь, что выбрана опция экспорта всех изображений в один файл ).
8. Генерация координат для усреднения субтомограммы от ROI
- Извлеките класс.
- Щелкните правой кнопкой мыши Class, который будет использоваться для усреднения | Извлеките класс как ROI. Окупаемость инвестиций в класс по щелчку правой кнопкой мыши | Подключенные компоненты | Новый Multi-ROI (26 подключенных).
- Сгенерируйте координаты.
- Щелкните правой кнопкой мыши новый Multi-ROI | Скалярный генератор. Разверните основные измерения с помощью набора данных | проверьте взвешенный центр масс X, Y и Z. Выберите набор данных и вычислите. Щелкните правой кнопкой мыши Multi-ROI | Экспорт скалярных значений. Установите флажок «Выбрать все скалярные слоты», затем «ОК», чтобы сгенерировать координаты мира центроида для каждого класса в мульти-ROI в виде CSV-файла.
ПРИМЕЧАНИЕ: Если частицы расположены близко друг к другу и сегментации соприкасаются, может потребоваться выполнить преобразование водораздела, чтобы разделить компоненты в мульти-ROI.
- Щелкните правой кнопкой мыши новый Multi-ROI | Скалярный генератор. Разверните основные измерения с помощью набора данных | проверьте взвешенный центр масс X, Y и Z. Выберите набор данных и вычислите. Щелкните правой кнопкой мыши Multi-ROI | Экспорт скалярных значений. Установите флажок «Выбрать все скалярные слоты», затем «ОК», чтобы сгенерировать координаты мира центроида для каждого класса в мульти-ROI в виде CSV-файла.
9. Трансформация водораздела
- Извлеките класс, щелкнув правой кнопкой мыши класс в Multi-ROI, который будет использоваться для усреднения | Извлеките класс как ROI. Назовите эту маску водораздела ROI.
- (Необязательно) Закройте отверстия.
- Если сегментированные частицы имеют отверстия или отверстия, закройте их для водораздела. Щелкните ROI в свойствах данных. На вкладке «Сегментация» (слева) перейдите в раздел «Морфологические операции» и используйте любую комбинацию «Расширение», «Эрозию» и «Закрытие», необходимую для достижения сплошной сегментации без отверстий.
- Инвертируйте рентабельность инвестиций, нажав кнопку ROI | Скопируйте выбранный объект (в разделе «Свойства данных»). Выберите скопированный ROI и слева на вкладке «Сегментация » нажмите «Инвертировать».
- Создайте карту расстояний, щелкнув правой кнопкой мыши инвертированную рентабельность инвестиций | Создать сопоставление | Карта расстояний. Для последующего использования сделайте копию карты расстояний и инвертируйте ее (щелкните правой кнопкой мыши | Модифицировать и трансформировать | Инвертировать значения | Подать заявку). Назовите эту перевернутую карту Пейзаж.
- Создание начальных точек.
- Скройте ROI и отобразите карту расстояний. На вкладке « Сегментация » нажмите кнопку «Определить диапазон» и уменьшайте диапазон до тех пор, пока не будет выделено всего несколько пикселов в центре каждой точки и ни один из них не будет соединен с другой точкой. В нижней части раздела «Диапазон » нажмите кнопку «Добавить в новый». Назовите это новым начальным моментом ROI.
- Выполните преобразование водораздела.
- Окупаемость инвестиций в Seedpoints | Подключенные компоненты | Новый Multi-ROI (26 подключенных). Щелкните правой кнопкой мыши только что сгенерированный Multi-ROI | Преображение водораздела. Выберите карту расстояний с именем «Пейзаж» и нажмите «ОК»; выберите ROI с именем Watershed Mask и нажмите OK, чтобы рассчитать преобразование водораздела из каждой точки посева и разделить отдельные частицы на отдельные классы в мульти-ROI. Сгенерируйте координаты, как показано на шаге 8.2.
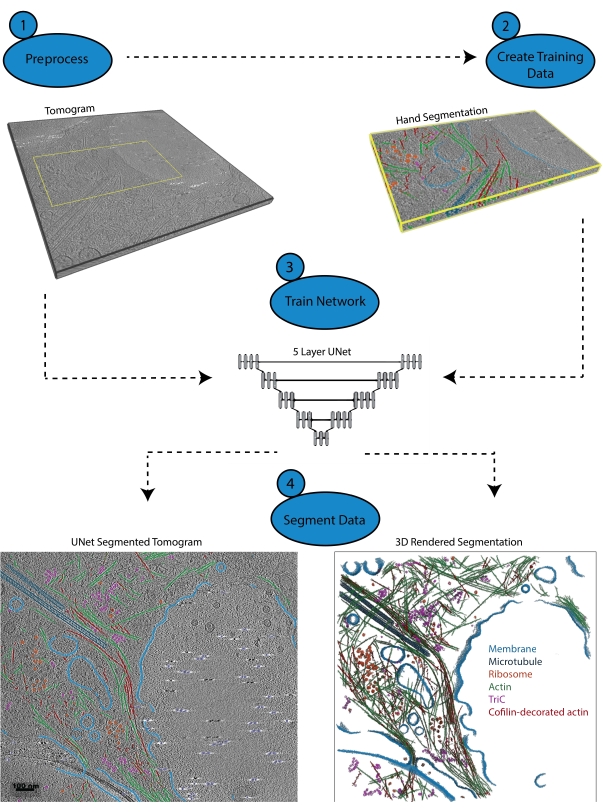
Рисунок 1: Рабочий процесс. 1) Предварительно обработайте тренировочную томограмму, откалибровав шкалу интенсивности и отфильтровав набор данных. 2) Создайте обучающие данные, вручную сегментировав небольшую часть томограммы со всеми соответствующими метками, которые пользователь хочет идентифицировать. 3) Используя отфильтрованную томограмму в качестве входных данных и сегментацию рук в качестве обучающего выхода, пятислойная многосрезовая U-Net обучается в мастере сегментации. 4) Обученная сеть может быть применена к полной томограмме, чтобы аннотировать ее, и 3D-рендеринг может быть сгенерирован из каждого сегментированного класса. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Результаты
В соответствии с протоколом пятисрезовая U-Net была обучена на одной томограмме (рис. 2A) для идентификации пяти классов: мембрана, микротрубочки, актин, фидуциальные маркеры и фон. Сеть была итеративно обучена в общей сложности три раза, а затем применена к томограмме, чтобы полностью сегментировать и аннотировать ее (рис. 2B, C). Минимальная очистка была выполнена с помощью шагов 7.1 и 7.2. Следующие три томограммы, представляющие интерес (рис. 2D, G, J), были загружены в программное обеспечение для предварительной обработки. Перед импортом изображения одна из томограмм (рис. 2J) требовала корректировки размера пикселя с 17,22 Å/px до 13,3 Å/px, поскольку она была собрана на другом микроскопе с немного другим увеличением. Программа IMOD squeezevol использовалась для изменения размера с помощью следующей команды:
'squeezevol -f 0.772 inputfile.mrc outputfile.mrc'
В этой команде -f относится к коэффициенту, с помощью которого изменяется размер пикселя (в данном случае: 13,3/17,22). После импорта все три цели вывода были предварительно обработаны в соответствии с шагами 3.2 и 3.3, а затем была применена пятисрезовая U-Net. Снова была проведена минимальная очистка. Окончательные сегментации показаны на рисунке 2.
Сегментации микротрубочек из каждой томограммы экспортировались в виде двоичных (шаг 7.4) файлов TIF, преобразовывались в MRC (программа IMOD tif2mrc ), а затем использовались для корреляции цилиндров и трассировки нитей. Двоичная сегментация филаментов приводит к гораздо более надежной трассировке филаментов, чем трассировка по томограммам. Координатные карты, полученные на основе трассировки нитей (рис. 3), будут использоваться для дальнейшего анализа, такого как измерения ближайших соседей (упаковка нити) и усреднение спиральной субтомограммы вдоль отдельных нитей для определения ориентации микротрубочек.
Неудачные или недостаточно обученные сети легко определить. Неисправная сеть вообще не сможет сегментировать какие-либо структуры, в то время как недостаточно обученная сеть обычно правильно сегментирует некоторые структуры и имеет значительное количество ложноположительных и ложноотрицательных результатов. Эти сети могут быть исправлены и итеративно обучены для повышения их производительности. Мастер сегментации автоматически вычисляет коэффициент подобия Dice модели (называемый оценкой в SegWiz) после ее обучения. Эта статистика дает оценку сходства между данными обучения и сегментацией U-Net. Dragonfly 2022.1 также имеет встроенный инструмент для оценки производительности модели, доступ к которому можно получить на вкладке « Искусственный интеллект » в верхней части интерфейса (см. документацию по использованию).
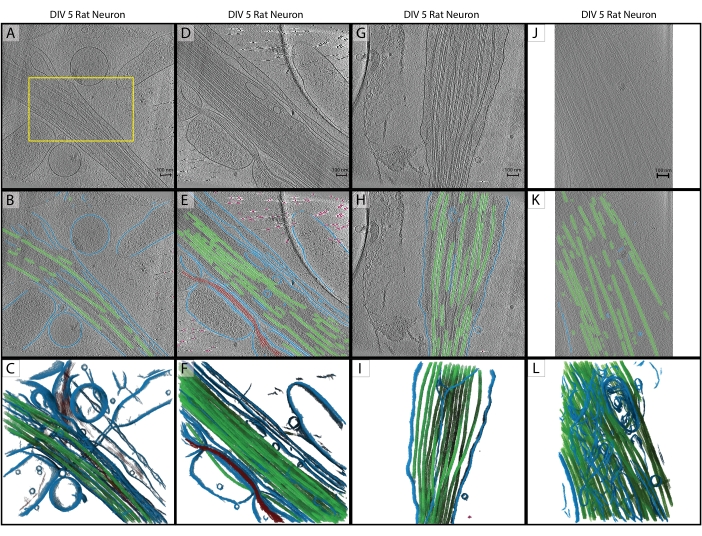
Рисунок 2: Вывод. (A-C) Оригинальная тренировочная томограмма нейрона гиппокампа крысы DIV 5, собранная в 2019 году на Titan Krios. Это реконструкция, спроецированная назад с коррекцией CTF в IMOD. (A) Желтая рамка представляет область, в которой была выполнена сегментация рук для обучающих входных данных. (B) 2D-сегментация из U-Net после завершения обучения. (C) 3D-рендеринг сегментированных областей, показывающих мембрану (синий), микротрубочки (зеленый) и актин (красный). (Д-Ж) DIV 5 нейрон гиппокампа крысы из того же сеанса, что и тренировочная томограмма. (E) 2D-сегментация из U-Net без дополнительной подготовки и быстрой очистки. Мембрана (синяя), микротрубочки (зеленая), актин (красная), фидуциальные (розовая). (F) 3D-рендеринг сегментированных областей. (Г-И) DIV 5 нейрон гиппокампа крысы с сессии 2019 года. (H) 2D-сегментация из U-Net с быстрой очисткой и (I) 3D-рендерингом. (Ж-Л) Нейрон гиппокампа крысы DIV 5, собранный в 2021 году на другом Titan Krios с другим увеличением. Размер пикселя был изменен с помощью программы IMOD squeezevol в соответствии с тренировочной томограммой. (K) 2D-сегментация из U-Net с быстрой очисткой, демонстрирующая надежный вывод по наборам данных с надлежащей предварительной обработкой и (L) 3D-рендерингом сегментации. Масштабные линейки = 100 нм. Сокращения: DIV = дни in vitro; CTF = функция переноса контраста. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
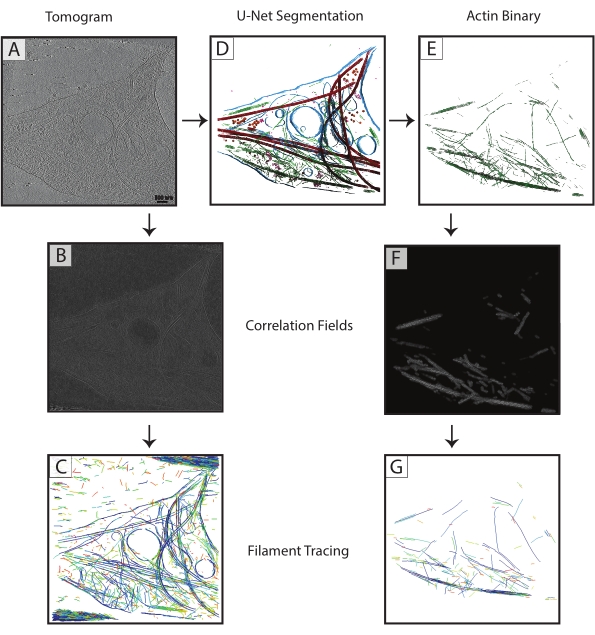
Рисунок 3: Улучшение трассировки нити . (A) Томограмма нейронов гиппокампа крысы DIV 4, собранных на Titan Krios. (B) Корреляционная карта, полученная на основе корреляции цилиндров над актиновыми филаментами. (C) Нитевое отслеживание актина с использованием интенсивности актиновых филаментов в корреляционной карте для определения параметров. Трассировка захватывает мембрану и микротрубочки, а также шум, пытаясь отследить только актин. (D) U-нет-сегментация томограммы. Мембрана выделена синим цветом, микротрубочки - красным, рибосомы - оранжевым, triC - фиолетовым, актин - зеленым. (E) Сегментация актина, извлеченная в виде бинарной маски для трассировки филаментов. (F) Корреляционная карта, полученная на основе корреляции цилиндра с теми же параметрами, что и (B). (G) Значительно улучшено отслеживание филаментов только актиновых филаментов по томограмме. Аббревиатура: DIV = дни in vitro. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этого рисунка.
Дополнительный файл 1: Томограмма, используемая в этом протоколе, и мульти-ROI, который был сгенерирован в качестве входных данных для обучения, включены в виде связанного набора данных (Training.ORSObject). См. https://datadryad.org/stash/dataset/doi:10.5061/dryad.rxwdbrvct.
Обсуждение
В этом протоколе изложена процедура использования программного обеспечения Dragonfly 2022.1 для обучения многоклассовой U-Net по одной томограмме и как вывести эту сеть из других томограмм, которые не обязательно должны быть из того же набора данных. Обучение происходит относительно быстро (может длиться от 3 до 5 минут за эпоху или от нескольких часов, в зависимости от обучаемой сети и используемого оборудования), а переобучение сети для улучшения ее обучения интуитивно понятно. До тех пор, пока этапы предварительной обработки выполняются для каждой томограммы, вывод, как правило, является надежным.
Согласованная предварительная обработка является наиболее важным шагом для вывода глубокого обучения. В программном обеспечении есть много фильтров изображений, и пользователь может экспериментировать, чтобы определить, какие фильтры лучше всего подходят для определенных наборов данных; Обратите внимание, что любая фильтрация, используемая на обучающей томограмме, должна применяться таким же образом к томограммам вывода. Необходимо также позаботиться о том, чтобы предоставить сети точную и достаточную информацию об обучении. Очень важно, чтобы все функции, сегментированные в тренировочных срезах, были сегментированы как можно тщательнее и точнее.
Сегментация изображений облегчается сложным пользовательским интерфейсом коммерческого уровня. Он предоставляет все необходимые инструменты для сегментации рук и позволяет легко переназначать воксели из любого класса в другой перед обучением и переподготовкой. Пользователю разрешается вручную сегментировать воксели в рамках всего контекста томограммы, и им предоставляется несколько представлений и возможность свободно вращать громкость. Кроме того, программное обеспечение предоставляет возможность использовать многоклассовые сети, которые, как правило, работают лучше16 и быстрее, чем сегментация с несколькими одноклассовыми сетями.
Конечно, существуют ограничения возможностей нейронной сети. Данные Cryo-ET по своей природе очень зашумлены и ограничены в угловой выборке, что приводит к искажениям, специфичным для ориентации, в идентичных объектах21. Обучение опирается на эксперта, который точно сегментирует структуры, и успешная сеть хороша настолько, насколько хороши (или плохи) данные обучения, которые ей предоставляются. Фильтрация изображений для усиления сигнала полезна для тренера, но есть еще много случаев, когда точная идентификация всех пикселей данной структуры затруднена. Поэтому важно, чтобы при создании сегментации обучения уделялось большое внимание, чтобы сеть имела наилучшую информацию, возможную для изучения во время обучения.
Этот рабочий процесс можно легко изменить в соответствии с предпочтениями каждого пользователя. Хотя важно, чтобы все томограммы были предварительно обработаны точно таким же образом, нет необходимости использовать точные фильтры, используемые в протоколе. Программное обеспечение имеет множество опций фильтрации изображений, и рекомендуется оптимизировать их для конкретных данных пользователя, прежде чем приступать к большому проекту сегментации, охватывающему множество томограмм. Существует также довольно много сетевых архитектур, доступных для использования: было обнаружено, что многосрезовая U-Net лучше всего подходит для данных из этой лаборатории, но другой пользователь может обнаружить, что другая архитектура (например, 3D U-Net или Sensor 3D) работает лучше. Мастер сегментации предоставляет удобный интерфейс для сравнения производительности нескольких сетей с использованием одних и тех же обучающих данных.
Инструменты, подобные представленным здесь, сделают ручную сегментацию полных томограмм задачей прошлого. С помощью хорошо обученных нейронных сетей, которые надежно выводимы, вполне возможно создать рабочий процесс, в котором томографические данные реконструируются, обрабатываются и полностью сегментируются так быстро, как микроскоп может их собрать.
Раскрытие информации
Лицензия открытого доступа для этого протокола была оплачена компанией Object Research Systems.
Благодарности
Это исследование было поддержано Медицинским колледжем штата Пенсильвания и Департаментом биохимии и молекулярной биологии, а также грантом Фонда урегулирования табака (TSF) 4100079742-EXT. Услуги и инструменты CryoEM и CryoET Core (RRID:SCR_021178), используемые в этом проекте, частично финансировались Медицинским колледжем Университета штата Пенсильвания через Управление заместителя декана по исследованиям и аспирантам и Департамент здравоохранения Пенсильвании с использованием фондов урегулирования табака (CURE). Содержание является исключительной ответственностью авторов и не обязательно отражает официальную точку зрения Университета или Медицинского колледжа. Департамент здравоохранения Пенсильвании специально снимает с себя ответственность за любые анализы, интерпретации или выводы.
Материалы
| Name | Company | Catalog Number | Comments |
| Dragonfly 2022.1 | Object Research Systems | https://www.theobjects.com/dragonfly/index.html | |
| E18 Rat Dissociated Hippocampus | Transnetyx Tissue | KTSDEDHP | https://tissue.transnetyx.com/faqs |
| IMOD | University of Colorado | https://bio3d.colorado.edu/imod/ | |
| Intel® Xeon® Gold 6124 CPU 3.2GHz | Intel | https://www.intel.com/content/www/us/en/products/sku/120493/intel-xeon-gold-6134-processor-24-75m-cache-3-20-ghz/specifications.html | |
| NVIDIA Quadro P4000 | NVIDIA | https://www.nvidia.com/content/dam/en-zz/Solutions/design-visualization/productspage/quadro/quadro-desktop/quadro-pascal-p4000-data-sheet-a4-nvidia-704358-r2-web.pdf | |
| Windows 10 Enterprise 2016 | Microsoft | https://www.microsoft.com/en-us/evalcenter/evaluate-windows-10-enterprise | |
| Workstation Minimum Requirements | https://theobjects.com/dragonfly/system-requirements.html |
Ссылки
- Bai, X. -C., Mcmullan, G., Scheres, S. H. W. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- de Oliveira, T. M., van Beek, L., Shilliday, F., Debreczeni, J., Phillips, C. Cryo-EM: The resolution revolution and drug discovery. SLAS Discovery. 26 (1), 17-31 (2021).
- Danev, R., Yanagisawa, H., Kikkawa, M. Cryo-EM performance testing of hardware and data acquisition strategies. Microscopy. 70 (6), 487-497 (2021).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Tomography 5 and Tomo Live Software User-friendly batch acquisition for and on-the-fly reconstruction for cryo-electron tomography Datasheet. , Available from: https://assets.thermofisher.com/TFS-Assets/MSD/Datasheets/tomography-5-software-ds0362.pdf (2022).
- Danev, R., Baumeister, W. Expanding the boundaries of cryo-EM with phase plates. Current Opinion in Structural Biology. 46, 87-94 (2017).
- Hylton, R. K., Swulius, M. T. Challenges and triumphs in cryo-electron tomography. iScience. 24 (9), (2021).
- Turk, M., Baumeister, W. The promise and the challenges of cryo-electron tomography. FEBS Letters. 594 (20), 3243-3261 (2020).
- Oikonomou, C. M., Jensen, G. J. Cellular electron cryotomography: Toward structural biology in situ. Annual Review of Biochemistry. 86, 873-896 (2017).
- Wagner, J., Schaffer, M., Fernández-Busnadiego, R. Cryo-electron tomography-the cell biology that came in from the cold. FEBS Letters. 591 (17), 2520-2533 (2017).
- Lam, V., Villa, E. Practical approaches for Cryo-FIB milling and applications for cellular cryo-electron tomography. Methods in Molecular Biology. 2215, 49-82 (2021).
- Chreifi, G., Chen, S., Metskas, L. A., Kaplan, M., Jensen, G. J. Rapid tilt-series acquisition for electron cryotomography. Journal of Structural Biology. 205 (2), 163-169 (2019).
- Eisenstein, F., Danev, R., Pilhofer, M. Improved applicability and robustness of fast cryo-electron tomography data acquisition. Journal of Structural Biology. 208 (2), 107-114 (2019).
- Esteva, A., et al. Deep learning-enabled medical computer vision. npj Digital Medicine. 4 (1), (2021).
- Liu, Y. -T., et al. Isotropic reconstruction of electron tomograms with deep learning. bioRxiv. , (2021).
- Moebel, E., et al. Deep learning improves macromolecule identification in 3D cellular cryo-electron tomograms. Nature Methods. 18 (11), 1386-1394 (2021).
- Chen, M., et al. Convolutional neural networks for automated annotation of cellular cryo-electron tomograms. Nature Methods. 14 (10), 983-985 (2017).
- Ronneberger, O., Fischer, P., Brox, T. U-net: Convolutional networks for biomedical image segmentation. Lecture Notes in Computer Science (including subseries Lecture Notes in Artificial Intelligence and Lecture Notes in Bioinformatics). 9351, 234-241 (2015).
- Dragonfly 2021.3 (Computer Software). , Available from: http://www.theobjects.com/dragonfly (2021).
- Kremer, J. R., Mastronarde, D. N., McIntosh, J. R. Computer visualization of three-dimensional image data using IMOD. Journal of Structural Biology. 116 (1), 71-76 (1996).
- Iancu, C. V., et al. A "flip-flop" rotation stage for routine dual-axis electron cryotomography. Journal of Structural Biology. 151 (3), 288-297 (2005).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены