Method Article
Robuster Nachweis der Genamplifikation in formalinfixierten, in Paraffin eingebetteten Proben durch Fluoreszenz-In-situ-Hybridisierung
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet eine reproduzierbare Methode zur Visualisierung der Genamplifikation in formalinfixierten, paraffineingebetteten (FFPE) Gewebeproben.
Zusammenfassung
Die fokale Genamplifikation, wie z. B. die extrachromosomale DNA (ecDNA), spielt eine wichtige Rolle bei der Krebsentstehung und Therapieresistenz. Während sequenzierungsbasierte Methoden eine unvoreingenommene Identifizierung von ecDNA ermöglichen, bleiben zytogenetische Techniken, wie z. B. die Fluoreszenz-in-situ-Hybridisierung (FISH), zeit- und kosteneffizient für die Identifizierung von ecDNA in klinischen Proben. Die Anwendung von FISH in formalinfixierten, paraffineingebetteten (FFPE) Gewebeproben bietet einen einzigartigen Weg zum Nachweis amplifizierter Gene, insbesondere wenn keine lebensfähigen Proben für die Karyotypuntersuchung zur Verfügung stehen. Allerdings fehlt es an Konsensverfahren für diese Technik. Dieses Protokoll bietet umfassende, vollständig optimierte Schritt-für-Schritt-Anweisungen für die Durchführung von FISH zum Nachweis der Genamplifikation, einschließlich ecDNA, in FFPE-Gewebeproben, die einzigartige Herausforderungen mit sich bringen, die mit diesem Protokoll überwunden und standardisiert werden sollen. Durch die Befolgung dieses Protokolls können Forscher reproduzierbar hochwertige Bildgebungsdaten erfassen, um die Genamplifikation zu beurteilen.
Einleitung
Die Untersuchung der fokalen Onkogenamplifikation ist von entscheidender Bedeutung, da sie die Entstehung, das Fortschreiten und die Therapieresistenz von Krebs fördert1. Wichtig ist, dass Onkogene und immunregulatorische Gene als extrachromosomale DNAs (ecDNA) amplifiziert werden können, deren asymmetrische Vererbung die genetische Heterogenität bei Krebs fördert 2,3. ecDNA wurde mit Therapieresistenz und ungünstigen klinischen Ergebnissen in Verbindung gebracht 4,5,6.
Formalinfixierte, in Paraffin eingebettete (FFPE) Gewebeproben stellen eine umfangreiche Archivressource in pathologischen Laboratorien dar und bieten reichlich Informationen für retrospektive Studien. Die Extraktion molekularer Daten aus FFPE-Proben durch PCR oder Sequenzierung stellt jedoch aufgrund der Fragmentierung, des Abbaus und der Vernetzung von Nukleinsäuren während der Fixierung eine Herausforderungdar 7. Unter den verfügbaren Techniken für die molekulare Analyse von FFPE-Geweben hat sich die Fluoreszenz-in-situ-Hybridisierung (FISH) bei der Visualisierung spezifischer DNA-Sequenzen als wirksam erwiesen8.
Trotz der Weiterentwicklung moderner molekulardiagnostischer Verfahren bietet die Fähigkeit von FISH, die Genamplifikation auf Einzelzellebene zu visualisieren und zu quantifizieren, wertvolle Einblicke in die molekularen Mechanismen, die der Tumorgenese und den klinischen Ergebnissen zugrunde liegen. Durch die Verwendung von fluoreszenzmarkierten Sonden, die komplementär zum Zielgen von Interesse sind, kann FISH die Lokalisierung eines Onkogens bequem aufklären und auf die Form der Onkogen-Amplifikation (wie ecDNA) innerhalb einzelner Zellen schließen, die sonst mit anderen Technologien unmöglich oder teuer wäre. Daher bietet FISH eine kostengünstige Möglichkeit, die Heterogenität von Tumoren und die klonale Evolution zu beurteilen9. Darüber hinaus haben Fortschritte in der Automatisierung, Bildgebung und computergestützten Analyse die Hochdurchsatzanalyse von FISH-Daten erleichtert und ermöglichen so eine robuste Quantifizierung der Genamplifikation über große Gewebekohortenhinweg 10.
Die Anwendung von FISH auf FFPE-Gewebe birgt jedoch inhärente Herausforderungen, einschließlich der Vernetzung von Artefakten und der Hintergrundautofluoreszenz. Die Überwindung dieser Hindernisse erfordert eine sorgfältige Optimierung jedes Verfahrens, um genaue und reproduzierbare Ergebnisse zu gewährleisten. Dieses Papier bietet ein Schritt-für-Schritt-Protokoll, das vollständig optimiert ist, für die Anwendung von FISH zur Untersuchung der Genamplifikation in FFPE-Gewebeproben. Mit Hilfe einer Sonde, die auf den ERBB2 (HER2)-Genlocus abzielt, zeigen wir, dass FISH den ERBB2-Amplifikationsstatus in FFPE-Proben von Brustkrebspatientinnen robust nachweisen kann. Es ist sogar möglich abzuschätzen, ob ERBB2 als ecDNAs amplifiziert wird. Durch die Synthese der vorhandenen Literatur und unserer experimentellen Ergebnisse verdeutlichen wir die methodischen Überlegungen, technischen Herausforderungen und potenziellen Fallstricke der FISH-basierten Analyse. Wir diskutieren auch die klinische Relevanz von Genamplifikationsprofilen bei verschiedenen Krebsarten und unterstreichen ihre prognostische Bedeutung und ihr Potenzial für personalisierte therapeutische Strategien.
Zusammenfassend unterstreicht diese Arbeit die Bedeutung von FISH als wertvolles Werkzeug für die Untersuchung der Genamplifikation in FFPE-Gewebeproben, das beispiellose Einblicke in die Tumorbiologie bietet und die klinische Entscheidungsfindung in der Onkologie leitet. Durch die kontinuierliche Verfeinerung und Integration mit komplementären molekularen Assays ist die FISH-basierte Analyse in der Lage, unser Verständnis der Krebspathogenese weiter zu verbessern und die Patientenergebnisse im Zeitalter der Präzisionsmedizin zu verbessern.
Protokoll
Dieses Forschungsprotokoll wurde vom Institutional Review Board (IRB) des Southwestern Medical Center der University of Texas genehmigt. Vor der Operation wurde von allen Patienten eine Einverständniserklärung eingeholt.
1. Vorbereitung von Reagenzien und Materialien
- Bereiten Sie die 0,2 N Natriumchlorid (HCl)-Lösung in einem Abzug vor, indem Sie langsam 8,212 ml HCl (37 % w/w oder 12,1 N) zu 491,788 mL ddH2O hinzufügen. Bei Raumtemperatur (RT) lagern.
ACHTUNG: Geben Sie langsam Säure in das Wasser. Fügen Sie der Säure kein Wasser hinzu. - Bereiten Sie 10 mM Zitronensäurelösung (pH 6,0) vor, indem Sie 1,47 g Trinatriumcitrat (Dihydrat) in 400 ml ddH2O lösen. Verwenden Sie HCl, um den pH-Wert 6,0 einzustellen, und bringen Sie dann das endgültige Volumen mit ddH2O auf 500 mL. Lagern Sie den Puffer bei RT.
- Bereiten Sie die 10%ige Tween-20-Lösung vor, indem Sie 100 μl Tween-20 bis 900 μl ddH2O hinzufügen. Lagern Sie sie bei RT.
- Bereiten Sie die 10%ige IGEPAL-Lösung vor, indem Sie 5 mL IGEPAL CA-630 zu 45 mL ddH2O hinzufügen. Lagern Sie es bei RT.
- Bereiten Sie die 20x SSC-Lösung (pH 7,0, 3 M NaCl, 0,3 M Natriumcitrat) vor, indem Sie 44,1 g Trinatriumcitrat (Dihydrat) und 87,65 g Natriumchlorid (NaCl) in 900 ml ddH2O lösen. Verwenden Sie HCl, um es auf pH 7,0 einzustellen, und bringen Sie dann das endgültige Volumen mit ddH2O auf 1000 ml. Lagern Sie den Puffer bei RT.
- Bereiten Sie die 2x SSC-Lösung vor, indem Sie 100 mL 20x SSC zu 900 mL ddH2O hinzufügen. Falls erforderlich, fügen Sie 0,5 ml Konservierungsmittel hinzu (Materialtabelle). Lagern Sie es bei RT.
- Bereiten Sie den Sondenhybridisierungspuffer vor, indem Sie 910 μl ddH2O, 500 μl 20x SSC, 50 μl 10 % Tween-20, 40 μl RNase A, 1 ml 50 % Dextransulfat und 2,5 ml Formamid mischen. Aliquotieren Sie in 1 mL und lagern Sie sie bei -20 °C.
- Bereiten Sie die 0,4x SSC mit 0,3% iger IGEPAL-Lösung vor, indem Sie 100 mL 2x SSC, 15 mL 10% IGEPAL und 385 mL ddH2O mischen. Lagern Sie es bei RT.
- Bereiten Sie die 2x SSC mit 0,1% IGEPAL-Lösung vor, indem Sie 5 mL 10% IGEPAL zu 495 mL 2x SSC-Lösung hinzufügen. Lagern Sie es bei RT.
- Bereiten Sie den Proteinase-K-Verdauungspuffer vor der Verwendung frisch vor, indem Sie 1 μl Proteinase K zu 99 μl Tris-EDTA-Puffer hinzufügen.
- Bereiten Sie 1 mg/ml DAPI-Lagerbestand vor, indem Sie 1 mg DAPI in 1 mL ddH2O auflösen. Lagern Sie es bei -20 °C und vor Licht geschützt. Bereiten Sie die DAPI-Arbeitslösung vor, indem Sie 1 μl des DAPI-Lagerbestands zu 999 μl 2x SSC-Lösung hinzufügen. Bis zum Gebrauch von Licht fernhalten.
2. Vorbehandlung der Proben
HINWEIS: Der hier verwendete Objektträger enthält die Probe.
- Lassen Sie den Objektträger 20 Minuten lang oder über Nacht bei 60-90 °C reifen (Abbildung 1).
HINWEIS: Dieser Schritt erleichtert das Schmelzen von Paraffin. In der Regel sind 20 min Aufheizen ausreichend. Es kann auf eine Nacht verlängert werden, um dem Zeitplan gerecht zu werden. - Entparaffinieren Sie den Objektträger, indem Sie ihn 10 Minuten lang in Xylol oder seine Ersatzstoffe in einem Coplin-Glas eintauchen. Wiederholen Sie diesen Schritt mit frischem Xylol oder seinen Ersatzstoffen. Führen Sie alle folgenden Vorbehandlungs- und Waschschritte in einem Coplin-Tiegel durch.
HINWEIS: Xylol-Ersatzstoffe sind sichere und umweltfreundliche Alternativen zu Xylol. Einer der Ersatzstoffe (siehe Materialtabelle) schneidet genauso gut ab wie Xylol, wenn nicht sogar besser. Obwohl Xylolersatzstoffe weniger Geruch erzeugen als Xylol, wird empfohlen, sie in einem Abzug zu verwenden. Wenn Xylolersatzstoffe nicht zugänglich sind, sind alle Verfahren unverändert mit der Xylol-basierten Entparaffinisierung kompatibel. - Den Xylol-Ersatz mit 100%igem Ethanol 5 min abwaschen.
- Rehydrieren Sie den Objektträger 5 Minuten lang mit 70% Ethanol-Immersion.
- Tauchen Sie den Objektträger 20 Minuten lang in 0,2 N Salzsäure (HCl) bei RT.
HINWEIS: HCl extrahiert effektiv säurelösliche Proteine, wie z. B. basische Kernproteine, um die Zugänglichkeit der DNA zu FISH-Sonden zu verbessern11. - Tauchen Sie den Objektträger in 10 mM heiße Zitronensäurelösung und inkubieren Sie ihn 20 Minuten lang bei 90-95 °C.
HINWEIS: Bei der Zitronensäurebehandlung bei hohen Temperaturen werden ebenfalls säurelösliche Proteine extrahiert. Es wird angenommen, dass beide Säurebehandlungen extrazelluläre Matrixproteine extrahieren, um die Autofluoreszenz zu verringern12. Es wird empfohlen, die Zitronenlösung auf den gewünschten Temperaturbereich vorzuwärmen, bevor sie auf den Objektträger aufgetragen wird. Die Mikrowelle kann eine bequeme Möglichkeit sein, dies zu tun. Ein Wasserbad, wie z. B. bei einem Sous-Vide-Garer, ist die effektivste und wirtschaftlichste Lösung für die Hochtemperatur-Inkubation. - Spülen Sie den Objektträger kurz in 2x SSC aus, um den pH-Wert zu neutralisieren.
- Verdauen Sie das Gewebe durch Zugabe von 100-200 μl (genug, um das Gewebe je nach Größe des Schnitts vollständig zu bedecken) Proteinase K Verdauungspuffer und inkubieren Sie 1 Minute lang bei RT.
HINWEIS: Der Proteinase-K-Verdau erhöht die Zugänglichkeit für FISH-Sonden weiter und reduziert die Autofluoreszenz. Der Zeitpunkt der Verdauung sollte je nach Gewebetyp optimiert werden. In den meisten Fällen ist 1 min Verdauung ausreichend. Eine Überverdauung führt zu einer haloförmigen Kernmorphologie, und die Verdauungszeit sollte verkürzt werden. - Stoppen Sie sofort den Proteinase-K-Verdau und dehydrieren Sie den Objektträger, indem Sie ihn 2 Minuten lang in 70 % Ethanol eintauchen, gefolgt von einer 85 %igen und 100 % Ethanolbehandlung für jeweils 2 Minuten.
3. FISH und Bildgebung
- Bereiten Sie die FISH-Hybridisierungsmischung vor, indem Sie 2 μl FISH-Sondenschaft mit 8 μl Hybridisierungspuffer verdünnen und dann auf den Objektträger auftragen. Decken Sie die Probe mit einem Deckglas ab.
HINWEIS: Das insgesamt verwendete FISH-Sondenmaterial liegt je nach Bildqualität zwischen 0,5 und 4 μl. Wenn das Signal zu niedrig ist, erhöhen Sie den Sondeneingang. Reduzieren Sie den Eingang der FISH-Sonde, wenn der Hintergrund zu hoch ist, insbesondere wenn fluoreszierende Ablagerungen außerhalb der Zellkerne beobachtet werden. Der Hybridisierungspuffer kann entweder derjenige sein, der mit den kommerziell erworbenen Sonden geliefert wird, oder wie in Abschnitt 1 hergestellt. - Legen Sie die Objektträger auf eine heiße Platte, z. B. ein Hybridisierungssystem für Objektträger, um die DNA 2-5 Minuten lang bei 75 °C zu denaturieren. Übertragen Sie dann den Objektträger auf eine andere auf 37 °C eingestellte Heizplatte, um über Nacht zu hybridisieren.
HINWEIS: Wenn die Heizplatte über eine Wasserwanne oder ein Reservoir verfügt, um die Feuchtigkeit während der Hybridisierung aufrechtzuerhalten, ist es nicht erforderlich, das Deckglas mit Gummizement abzudichten. - Tauchen Sie den Objektträger nach der Hybridisierung in 40-60 °C erwärmtes 0,4x SSC mit 0,3 % IGEPAL CA-630 Waschpuffer und entfernen Sie dann vorsichtig das Deckglas. Setzen Sie die Wäsche zweimal für jeweils 5 min im Dunkeln fort, unter Rühren für die ersten 10-15 s.
HINWEIS: Das Eintauchen des Objektträgers in den Waschpuffer hilft, das Deckglas vorsichtig zu lösen. - Waschen Sie die Folie in SSC mit 0,1% IGEPAL CA-630 für 5 min bei RT im Dunkeln, unter Rühren für die ersten 10-15 s.
- Um die Autofluoreszenz zu löschen, behandeln Sie den Objektträger mit dem Autofluoreszenz-Quenchkit (siehe Materialtabelle), indem Sie 150 μl Reagenz (50 μl + 50 μl + 50 μl Reagenzien A, B, C) für 2-5 min auftragen und dann 5 min lang mit 2x SSC waschen.
HINWEIS: Dies ist ein optionaler Schritt. Die Autofluoreszenz des Gewebes stammt hauptsächlich von extrazellulären Matrixbestandteilen wie Kollagen und Elastin. Es wird auch signifikant von Lysosomen und Mitochondrien beeinflusst, da sie Lipofuszin-, NADPH- und Flavin-Coenzym-Gehalt enthalten. Die Aldehydfixierung und das Vorhandensein von Blutzellen können die Autofluoreszenz ebenfalls gut erhöhen 13,14. - Färben Sie die Folie 10 Minuten lang mit DAPI. Spülen Sie den Objektträger 5 min lang mit 2x SSC-Puffer aus.
HINWEIS: Wenn der Objektträger nicht mit dem Autofluoreszenz-Quenching-Kit behandelt wird, kann die DAPI-Färbung auf 2 min reduziert werden. - Tauchen Sie die Rutsche schnell für nicht länger als 1 s in entionisiertes Wasser und trocknen Sie sie dann schnell, indem Sie zusätzliche Feuchtigkeit mit einem Papiertuch aufnehmen.
HINWEIS: Dies ist ein optionaler Schritt. Die Behandlung mit deionisiertem Wasser verhindert effektiv die Ablagerung von SSC-Puffer durch Salzkristalle auf dem Objektträger und verbessert die Bildqualität. Unter solch ionenarmen Bedingungen werden jedoch die Wasserstoffbrückenbindungen zwischen der FISH-Sonde und der Ziel-DNA geschwächt, was zu Dissoziation und Signalverlust der Sonde führt. Daher ist es entscheidend, den Wasseraufbereitungsschritt für eine sehr kurze Zeit aufrechtzuerhalten. - Trocknen Sie die Folie und montieren Sie sie dann mit Antifading-Montagemedien. Versiegeln Sie das Deckglas vor der Bildgebung mit Nagellack.
HINWEIS: Wenn der Objektträger mit einem Autofluoreszenz-Quenching-Reagenz behandelt wird, montieren Sie den Objektträger mit einem Antifading-Einbettmedium gemäß den Anweisungen des Herstellers. Darüber hinaus muss die Probe je nach Art des Einbettmediums, härtend oder nicht aushärtend, 1-24 h lang ausgehärtet werden, bevor sie versiegelt und abgebildet wird. Es wird empfohlen, die Probe mindestens über Nacht auszuhärten, um den besten Brechungsindex für die Bildgebung zu erzielen. - Verwenden Sie eine 60×Öllinse, um das Fluoreszenzsignal zu erfassen. Verwenden Sie den DAPI-Kanal, um den Fokus anzupassen. Stellen Sie sicher, dass Sie mehrere Z-Stacks erhalten. In der Regel sind 5-10 Z-Stapel mit einem Abstand von 1 μm ausreichend. Führen Sie eine maximale 3D-Projektion durch, um die beste Auflösung zu erzielen. Wenden Sie Dekonvolution oder andere Algorithmen zur Hintergrundbereinigung an, um die Bildqualität weiter zu verbessern.
Ergebnisse
Wir verwendeten FFPE-Proben von HER2-positivem und -negativem Brustkrebs, um das Ergebnis der FISH-Bildgebung zu demonstrieren. Die Amplifikation von HER2 (kodiert durch das ERBB2-Gen ) ist aufgrund der Verfügbarkeit und Wirksamkeit von molekularen HER2-Targeting-Therapien ein günstiger Marker. Im Gegensatz dazu sehen sich Patientinnen mit dreifach negativem Brustkrebs, bei denen die Expression von HER2, Östrogenrezeptor (ER) und Progesteronrezeptor (PR) fehlt, aufgrund begrenzter therapeutischer Optionen mit schlechten Ergebnissen konfrontiert. Daher ist die Bestimmung des HER2-Status für die Erforschung und Behandlung von Brustkrebs von entscheidenderBedeutung 15.
In der dreifach negativen Brustkrebsprobe zeigen die meisten Zellkerne zwei unterschiedliche Punkte, die HER2/ERBB2-FISH-Signale darstellen. Einige Kerne haben aufgrund der Schnittverzerrung möglicherweise nur einen Punkt (Abbildung 2, links). Im Gegensatz dazu weisen HER2-positive Proben reichlich FISH-Signale mit zwei unterschiedlichen Mustern auf. Ein Muster zeigt verstreute Punkte im gesamten Zellkern (Abbildung 2, Mitte). Dieses Muster ist ein Merkmal der ecDNA-Morphologie, da ecDNAs möglicherweise kein einzigartiges und organisiertes Kerngebiet besetzen16. Darüber hinaus führt die asymmetrische Segregation von ecDNAs während der Mitose zur Variation der Kopienzahl, was zu einer Signalheterogenität zwischen den Zellkernenführt 17. Einige Zellkerne können gelegentlich Cluster aufweisen, was auf ecDNA-Hubshinweist 18 (Abbildung 2, rechts). Die andere Art der HER2-Amplifikation zeigt hauptsächlich stäbchenförmige, kondensierte Aggregate. Diese Morphologie deutet wahrscheinlich auf eine chromosomenbasierte Amplifikation hin, wie z. B. homogen färbende Regionen (HSR)19 oder durch den Breakage-Fusion-Bridge-Zyklus (BFB)20. Bemerkenswert ist, dass ecDNA-, HSR- und BFB-Amplifikation im selben Zellkern koexistieren können. Daher wird empfohlen, mehrere Kerne zu untersuchen, um auf die Form der fokalen Amplifikation zu schließen.
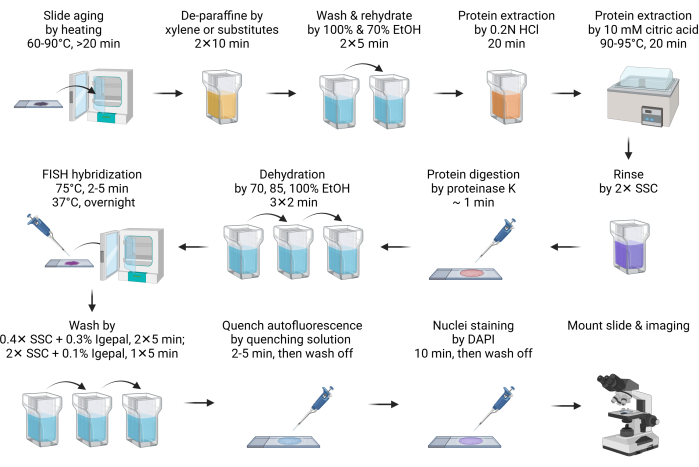
Abbildung 1: Schematische Darstellung von FISH in FFPE-Proben. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
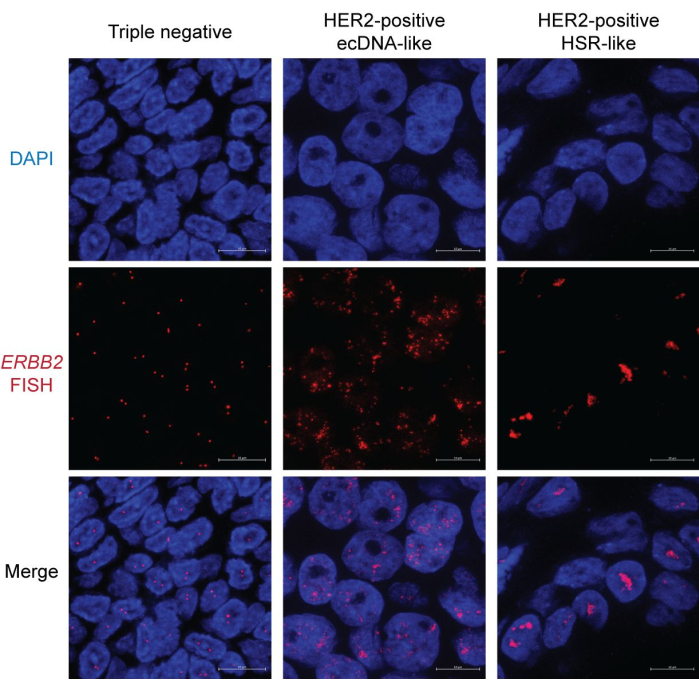
Abbildung 2: Repräsentatives FISH-Bild in FFPE-Proben von Brustkrebs. Vergrößerung: 600x; Maßstab: 10 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
FISH ist eine schnelle und kostengünstige Option für die zytogenetische Diagnose. Insbesondere bei der Bestimmung, ob ecDNA bei Krebs vorhanden ist, bleibt die FISH-Evidenz der Goldstandard1. FISH in FFPE-Gewebe ermöglicht eine schnelle Bestimmung des Genstatus in den Biopsieproben eines Patienten, was eine schnellere Diagnose und die Verfolgung von Veränderungen während des gesamten Krankheitsverlaufs ermöglicht. Diese Technik ist besonders wertvoll für die Untersuchung klinischer Proben, die bereits für die Pathologie entnommen wurden.
Dieses Protokoll umfasst mehrere kritische Schritte. Der erste Schritt ist die gründliche Entparaffinierung. Restparaffin kann die FISH-Hybridisierung stören. Wenn die Probe nach Schritt 2 immer noch wachsartig erscheint, sollte sie erneut mit frischem Xylol oder dessen Ersatz behandelt werden.
Zweitens sind die Proteinextraktion und -verdauung von entscheidender Bedeutung. Diese Prozesse verbessern nicht nur die Zugänglichkeit der DNA für die FISH-Sonde, sondern reduzieren auch die Autofluoreszenz erheblich. Dieses Protokoll umfasst drei Deproteinisierungsschritte. Während die Behandlung mit 0,2 N HCl und 10 mM Zitronensäure unkompliziert ist, kann der Proteinase-K-Verdau optimiert werden müssen. Eine Überverdauung ist der häufigste Fehler bei der Verwendung von Proteinase K, der zu haloförmigen Zellkernen führt. Durch die Verkürzung der Verdauungszeit wird die Morphologie der Zellkerne verbessert. Darüber hinaus wird empfohlen, nicht mehr als vier Proben gleichzeitig zu verdauen, um den Zeitunterschied zwischen der ersten und der letzten Probe zu minimieren. Es ist wichtig zu beachten, dass selbst ein intakter Zellkern unter hoher Vergrößerung und hochauflösender Mikroskopie als Halo erscheinen kann. Das liegt daran, dass sich der Zellkern nicht auf der gleichen Fokusebene befindet. Daher wird empfohlen, mehrere Z-Stapel zu verwenden und eine maximale Projektion durchzuführen, um die Kernmorphologie zu untersuchen.
Schließlich wird eine Quench-Autofluoreszenz empfohlen. Obwohl die Säureextraktion und der Proteinase-K-Verdau den proteinabgeleiteten Hintergrund signifikant reduzieren können, können fluoreszierende Metaboliten die Bildgebungsqualität beeinträchtigen.
Obwohl FISH eine beispiellose räumliche Auflösung bei der Identifizierung der fokalen Genamplifikation bietet, hat es Einschränkungen. Erstens sind der Inhalt und der Durchsatz im Vergleich zu PCR- oder Next-Generation-Sequencing-basierten (NGS)-basierten Ansätzen gering. In der Regel können ein bis drei FISH-Sonden in verschiedenen Farben ohne spezielle Ausrüstung auf einen einzigen Objektträger aufgebracht werden. Nichtsdestotrotz haben Fortschritte in der Automatisierungstechnologie FISH mit hohem Gehalt und hohem Durchsatz, wie z. B. die In-situ-Sequenzierung 21, möglich gemacht. Zweitens erfordert das Design der FISH-Sonde vorherige Informationen. Die laufenden Bemühungen zur Identifizierung wiederkehrender fokaler Amplifikationsereignisse bei Krebs haben die Entwicklung von vorgefertigten FISH-Panels für Labor- und klinische Anwendungen ermöglicht. Zum Beispiel werden Onkogene der MYC-Familie häufig als ecDNA bei kleinzelligem Lungenkrebs amplifiziert, um Chemotherapieresistenz zu vermitteln. Daher kann ein FISH-Panel, das auf die MYC-, MYCL- und MYCN-Gene abzielt, die Bestimmung des Behandlungsansprechens in Biopsien beschleunigen. Im Vergleich dazu ermöglicht NGS ein unvoreingenommeneres Screening von Genen von Interesse. Von den NGS-basierten Technologien kann ecDNA jedoch nur durch die Ganzgenomsequenzierung mit rechenintensiver Analyse22 charakterisiert werden.
Zusammenfassend stellen wir robuste und umfassende Anweisungen zur Untersuchung der fokalen Genamplifikation in FFPE-Proben vor. Durch die Untersuchung des FISH-Signalmusters wird eindeutig klar, ob und wie ein Genlocus amplifiziert wird. Wir erwarten die Integration von maschinellem Lernen in die Bildanalyse23 von Interphase-Zellkernen, um zytogenetische Informationen bezüglich der Kopienzahl und der Form der Amplifikation (Chromosom oder ecDNA) zu extrahieren, wodurch der molekulare Diagnoseprozess rationalisiert und unser Verständnis der pathogenetischen Mechanismen bei Krebs verbessert wird.
Offenlegungen
S.W. ist Mitglied des wissenschaftlichen Beirats von Dimension Genomics Inc.
Danksagungen
S.W. ist Stipendiat des Cancer Prevention and Research Institute of Texas (RR210034) und wird von diesem unterstützt
Materialien
| Name | Company | Catalog Number | Comments |
| DAPI | Tocris Bioscience | 5748 | Nucleus staining |
| Dextran sulfate 50% solution | EMD Millipore Sigma | S4030 | Probe hybridization buffer |
| ERBB2 (HER2) FISH Probe | Empire Genomics | ERBB2-20-RE | FISH probe |
| Ethanol | Decon Labs | 2716 | Dehydrating and hydrating tissue |
| Formamide | Thermo Scientific Chemicals | 205821000 | Probe hybridization buffer |
| Formula 83 (Xylene substitute) | CBG Biotech | CH0104A | Removing paraffin |
| Hydrochloric acid | Fisher Chemical | A144-500 | Sample pretreatment |
| IGEPAL CA-630 | Thermo Scientific Chemicals | J19628K2 | Slide washing |
| Proclin 300 | Sigma-Aldrich | 48914-U | Preservative for SSC buffer (optional) |
| Proteinase K (800 units/mL) | New England Biolabs | P8107S | Protein digestion |
| RNase A (20 mg/mL) | New England Biolabs | T3018L | Probe hybridization buffer |
| Slide Moat Hybridization System | Boekel Scientific | 280001 | Sample denature and hybridization. Alternative hot plates are acceptable. |
| Sodium chloride | Fisher Chemical | S2713 | SSC buffer |
| Sodium citrate dihydrate | Fisher BioReagents | FLBP3271 | SSC buffer and sample pretreatment |
| Tris-EDTA (TE) buffer | Fisher BioReagents | BP2473500 | Proteinase K digestion buffer |
| Tween-20 | Fisher BioReagents | BP337-500 | Probe hybridization buffer |
| Vectashield antifade mounting media | Vector Laboratories | H190010 | Slide mounting |
| Vector TrueVIEW | Vector Laboratories | SP8400 | Autofluorescence quenching kit |
Referenzen
- ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Nature. 578 (7793), 82-93 (2020).
- Wu, S., Bafna, V., Chang, H. Y., Mischel, P. S. Extrachromosomal DNA: An emerging hallmark in human cancer. Annu Rev Pathol. 17, 367-386 (2022).
- Luebeck, J., et al. Extrachromosomal DNA in the cancerous transformation of Barrett's oesophagus. Nature. 616 (7958), 798-805 (2023).
- Nathanson, D. A., et al. Targeted therapy resistance mediated by dynamic regulation of extrachromosomal mutant EGFR DNA. Science. 343 (6166), 72-76 (2014).
- Kim, H., et al. Extrachromosomal DNA is associated with oncogene amplification and poor outcome across multiple cancers. Nat Genet. 52 (9), 891-897 (2020).
- Pal Choudhuri, S., et al. Acquired cross-resistance in small cell lung cancer due to extrachromosomal DNA amplification of MYC paralogs. Cancer Discov. 14 (5), 804-827 (2024).
- Greytak, S. R., Engel, K. B., Bass, B. P., Moore, H. M. Accuracy of molecular data generated with FFPE biospecimens: Lessons from the literature. Cancer Res. 75 (8), 1541-1547 (2015).
- Chrzanowska, N. M., Kowalewski, J., Lewandowska, M. A. Use of fluorescence in situ hybridization (FISH) in diagnosis and tailored therapies in solid tumors. Molecules. 25 (8), 1864 (2020).
- Cui, C., Shu, W., Li, P. Fluorescence in situ hybridization: Cell-based genetic diagnostic and research applications. Front Cell Dev Biol. 4, 89 (2016).
- Finn, E. H., Misteli, T. A high-throughput DNA FISH protocol to visualize genome regions in human cells. STAR Protoc. 2 (3), 100741 (2021).
- Watters, A. D., Bartlett, J. M. S. Fluorescence in situ hybridization in paraffin tissue sections. Mol Biotechnol. 21 (3), 217-220 (2002).
- Richardson, S. O., et al. One-fits-all pretreatment protocol facilitating fluorescence in situ hybridization on formalin-fixed paraffin-embedded, fresh frozen and cytological slides. Mol Cytogenet. 12, 27 (2019).
- Monici, M. Cell and tissue autofluorescence research and diagnostic applications. Biotechnol Annu Rev. 11, 227-256 (2005).
- Davis, A. S., et al. Characterizing and diminishing autofluorescence in formalin-fixed paraffin-embedded human respiratory tissue. J Histochem Cytochem. 62 (6), 405-423 (2014).
- Cancer Genome Atlas Network. Comprehensive molecular portraits of human breast tumours. Nature. 490 (7418), 61-70 (2012).
- Liang, Z., et al. Chromatin-associated RNA dictates the ecDNA interactome in the nucleus. bioRxiv. , (2023).
- Lange, J. T., et al. The evolutionary dynamics of extrachromosomal DNA in human cancers. Nat Genet. 54 (10), 1527-1533 (2022).
- Hung, K. L., et al. ecDNA hubs drive cooperative intermolecular oncogene expression. Nature. 600 (7890), 731-736 (2021).
- Storlazzi, C. T., et al. Gene amplification as double minutes or homogeneously staining regions in solid tumors: origin and structure. Genome Res. 20 (9), 1198-1206 (2010).
- Guerin, T. M., Marcand, S. Breakage in breakage-fusion-bridge cycle: an 80-year-old mystery. Trends Genet. 38 (7), 641-645 (2022).
- Nguyen, H. Q., et al. 3D mapping and accelerated super-resolution imaging of the human genome using in situ sequencing. Nat Methods. 17 (8), 822-832 (2020).
- Deshpande, V., et al. Exploring the landscape of focal amplifications in cancer using AmpliconArchitect. Nat Commun. 10 (1), 392 (2019).
- Rajkumar, U., et al. EcSeg: Semantic segmentation of metaphase images containing extrachromosomal DNA. iScience. 21, 428-435 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten