Method Article
Rilevamento robusto dell'amplificazione genica in campioni inclusi in paraffina fissati in formalina mediante ibridazione in situ a fluorescenza
In questo articolo
Riepilogo
Questo protocollo fornisce un metodo riproducibile per visualizzare l'amplificazione genica in campioni di tessuto inclusi in paraffina (FFPE) fissati in formalina.
Abstract
L'amplificazione genica focale, come il DNA extracromosomico (ecDNA), svolge un ruolo importante nello sviluppo del cancro e nella resistenza alla terapia. Mentre le metodologie basate sul sequenziamento consentono un'identificazione imparziale dell'ecDNA, le tecniche basate sulla citogenetica, come l'ibridazione in situ a fluorescenza (FISH), rimangono efficienti in termini di tempo e costi per l'identificazione dell'ecDNA nei campioni clinici. L'applicazione della FISH in campioni di tessuto inclusi in formalina fissata in paraffina (FFPE) offre una via unica per rilevare geni amplificati, in particolare quando non sono disponibili campioni vitali per l'esame del cariotipo. Tuttavia, mancano procedure di consenso per questa tecnica. Questo protocollo fornisce istruzioni dettagliate, complete e completamente ottimizzate per l'esecuzione di FISH per rilevare l'amplificazione genica, incluso l'ecDNA, in campioni di tessuto FFPE che presentano sfide uniche che questo protocollo mira a superare e standardizzare. Seguendo questo protocollo, i ricercatori possono acquisire in modo riproducibile dati di imaging di alta qualità per valutare l'amplificazione genica.
Introduzione
Lo studio dell'amplificazione focale dell'oncogene è fondamentale in quanto guida la formazione, la progressione e la resistenza alla terapiadel cancro 1. È importante sottolineare che gli oncogeni e i geni immunoregolatori possono amplificarsi come DNA extracromosomici (ecDNA), la cui eredità asimmetrica promuove l'eterogeneità genetica nel cancro 2,3. L'ecDNA è stato collegato alla resistenza alla terapia e agli esiti clinici sfavorevoli 4,5,6.
I campioni di tessuto FFPE (FFPE) fissati in formalina rappresentano una vasta risorsa archivistica nei laboratori di patologia, offrendo informazioni abbondanti per studi retrospettivi. Tuttavia, l'estrazione di dati molecolari da campioni FFPE attraverso la PCR o il sequenziamento è difficile a causa della frammentazione, della degradazione e della reticolazione degli acidi nucleici durante la fissazione7. Tra le tecniche disponibili per l'analisi molecolare dei tessuti FFPE, l'ibridazione fluorescente in situ (FISH) si è dimostrata efficace per la visualizzazione di specifiche sequenze di DNA8.
Nonostante il progresso delle moderne tecniche diagnostiche molecolari, la capacità della FISH di visualizzare e quantificare l'amplificazione genica a livello di singola cellula fornisce preziose informazioni sui meccanismi molecolari alla base della tumorigenesi e sugli esiti clinici. Utilizzando sonde marcate in fluorescenza complementari al gene bersaglio di interesse, FISH può risolvere convenientemente la localizzazione di un oncogene e può dedurre la forma di amplificazione dell'oncogene (come l'ecDNA) all'interno delle singole cellule, cosa altrimenti impossibile o costosa con altre tecnologie. Pertanto, la FISH offre un modo economico per valutare l'eterogeneità del tumore e l'evoluzione clonale9. Inoltre, i progressi nell'automazione, nell'imaging e nell'analisi computazionale hanno facilitato l'analisi ad alto rendimento dei dati FISH, consentendo una solida quantificazione dell'amplificazione genica in grandi coorti di tessuti10.
Tuttavia, l'applicazione di FISH al tessuto FFPE presenta sfide intrinseche, tra cui artefatti di reticolazione e autofluorescenza di fondo. Il superamento di questi ostacoli richiede un'attenta ottimizzazione di ogni procedura per garantire risultati accurati e riproducibili. Questo articolo fornisce un protocollo passo-passo e completamente ottimizzato per l'applicazione della FISH per studiare l'amplificazione genica nei campioni di tessuto FFPE. Utilizzando una sonda che ha come bersaglio il locus del gene ERBB2 (HER2), dimostriamo che la FISH è in grado di rilevare in modo robusto lo stato di amplificazione di ERBB2 nei campioni di FFPE di pazienti con carcinoma mammario. È anche possibile stimare se ERBB2 è amplificato come ecDNA. Sintetizzando la letteratura esistente e i nostri risultati sperimentali, chiariamo le considerazioni metodologiche, le sfide tecniche e le potenziali insidie dell'analisi basata sulla FISH. Discutiamo anche la rilevanza clinica del profilo di amplificazione genica in vari tipi di cancro, evidenziandone il significato prognostico e il potenziale per strategie terapeutiche personalizzate.
In sintesi, questo articolo sottolinea l'importanza della FISH come strumento prezioso per lo studio dell'amplificazione genica in campioni di tessuto FFPE, offrendo intuizioni senza precedenti sulla biologia dei tumori e guidando il processo decisionale clinico in oncologia. Grazie al continuo perfezionamento e all'integrazione con saggi molecolari complementari, l'analisi basata sulla FISH è pronta a migliorare ulteriormente la nostra comprensione della patogenesi del cancro e a migliorare gli esiti dei pazienti nell'era della medicina di precisione.
Protocollo
Questo protocollo di ricerca è stato approvato dall'Institutional Review Board (IRB) del Southwestern Medical Center dell'Università del Texas. Il consenso informato è stato ottenuto da tutti i pazienti prima dell'intervento chirurgico.
1. Preparazione dei reagenti e dei materiali
- Preparare la soluzione di cloruro di sodio (HCl) 0,2 N in una cappa aspirante aggiungendo lentamente 8,212 mL di HCl (37% p/p o 12,1 N) a 491,788 mL di ddH2O. Conservare a temperatura ambiente (RT).
ATTENZIONE: Aggiungere lentamente acido all'acqua. Non aggiungere acqua all'acido. - Preparare 10 mM di soluzione di acido citrico (pH 6,0) sciogliendo 1,47 g di citrato trisodico (diidrato) in 400 mL di ddH2O. Utilizzare HCl per regolare a pH 6,0, quindi portare il volume finale a 500 mL con ddH2O. Conservare il tampone a RT.
- Preparare la soluzione di Tween-20 al 10% aggiungendo 100 μl di Tween-20 a 900 μl di ddH2O. Conservarla in RT.
- Preparare la soluzione di IGEPAL al 10% aggiungendo 5 mL di IGEPAL CA-630 a 45 mL di ddH2O. Conservarla in RT.
- Preparare la soluzione 20x SSC (pH 7,0, 3 M NaCl, 0,3 M citrato di sodio) sciogliendo 44,1 g di citrato trisodico (diidrato) e 87,65 g di cloruro di sodio (NaCl) in 900 mL di ddH2O. Utilizzare HCl per regolare a pH 7,0, quindi portare il volume finale a 1000 mL con ddH2O. Conservare il tampone a RT.
- Preparare la soluzione 2x SSC aggiungendo 100 mL di 20x SSC a 900 mL di ddH2O. Se necessario, aggiungere 0,5 ml di conservante (Tabella dei materiali). Conservalo su RT.
- Preparare il tampone di ibridazione della sonda miscelando 910 μL di ddH2O, 500 μL di SSC 20x, 50 μL di Tween-20 al 10%, 40 μL di RNasi A, 1 mL di destrano solfato al 50% e 2,5 mL di formammide. Aliquotare in 1 mL e conservarli a -20 °C.
- Preparare l'SSC 0,4x con la soluzione di IGEPAL allo 0,3% mescolando 100 mL di SSC 2x, 15 mL di IGEPAL al 10% e 385 mL di ddH2O. Conservarlo in RT.
- Preparare il 2x SSC con soluzione di IGEPAL allo 0,1% aggiungendo 5 mL di IGEPAL al 10% a 495 mL di soluzione 2x SSC. Conservalo su RT.
- Preparare nuovamente il tampone di digestione della proteinasi K prima dell'uso aggiungendo 1 μl di proteinasi K a 99 μl di tampone Tris-EDTA.
- Preparare 1 mg/mL di riserva DAPI sciogliendo 1 mg di DAPI in 1 mL di ddH2O. Conservarlo a -20 °C e al riparo dalla luce. Preparare la soluzione di lavoro DAPI aggiungendo 1 μl del materiale di stoccaggio DAPI a 999 μl di soluzione SSC 2x. Tenere lontano dalla luce fino all'uso.
2. Pretrattamento del campione
NOTA: Il vetrino utilizzato qui contiene il campione.
- Invecchiare il vetrino a 60-90°C per 20 minuti o durante la notte (Figura 1).
NOTA: Questo passaggio facilita la fusione della paraffina. Di solito, 20 minuti di riscaldamento sono sufficienti. Può essere esteso a notte per adattarsi al programma. - Deparaffinare il vetrino immergendolo nello xilene o nei suoi sostituti in un barattolo di Coplin per 10 minuti. Ripeti questo passaggio con xilene fresco o suoi sostituti. Eseguire tutte le seguenti fasi di pretrattamento e lavaggio in un barattolo Coplin.
NOTA: I sostituti dello xilene sono alternative sicure ed ecologiche allo xilene. Uno dei sostituti (vedi Tabella dei materiali) si comporta bene come lo xilene, se non meglio. Sebbene i sostituti dello xilene producano meno odore dello xilene, si consiglia di utilizzarlo all'interno di una cappa aspirante. Se i sostituti dello xilene non sono accessibili, tutte le procedure sono compatibili con la deparaffinazione a base di xilene senza alcuna modifica. - Lavare via il sostituto dello xilene con etanolo al 100% per 5 minuti.
- Reidratare il vetrino con un'immersione di etanolo al 70% per 5 minuti.
- Immergere il vetrino in acido cloridrico 0,2 N (HCl) a RT per 20 minuti.
NOTA: L'HCl estrae efficacemente le proteine solubili in acido, come le proteine nucleari basiche, per migliorare l'accessibilità del DNA alle sonde FISH11. - Immergere il vetrino in 10 mM di soluzione calda di acido citrico e incubare a 90-95 °C per 20 minuti.
NOTA: Il trattamento con acido citrico ad alte temperature estrae in modo simile le proteine solubili in acido. Si ritiene che entrambi i trattamenti acidi estraggono le proteine della matrice extracellulare per ridurre l'autofluorescenza12. Si consiglia di preriscaldare la soluzione citrica all'intervallo di temperatura desiderato prima di essere applicata al vetrino. Il microonde può essere un modo conveniente per farlo. Un bagno d'acqua, come con un fornello sous vide, è la soluzione più efficace ed economica per l'incubazione ad alta temperatura. - Sciacquare brevemente il vetrino in 2x SSC per neutralizzare il pH.
- Digerire il tessuto aggiungendo 100-200 μL (sufficienti a coprire completamente il tessuto a seconda delle dimensioni della sezione) di tampone per digestione Proteinasi K e incubare a RT per 1 minuto.
NOTA: La digestione della proteinasi K aumenta ulteriormente l'accessibilità per le sonde FISH e riduce l'autofluorescenza. Il tempo di digestione deve essere ottimizzato in base ai tipi di tessuto. Nella maggior parte dei casi, 1 minuto di digestione è sufficiente. L'eccessiva digestione porta a una morfologia dei nuclei a forma di alone e il tempo di digestione dovrebbe essere ridotto. - Interrompere immediatamente la digestione della proteinasi K e disidratare il vetrino immergendolo in etanolo al 70% per 2 minuti, seguito da un trattamento con etanolo all'85% e al 100% per 2 minuti ciascuno.
3. FISH e imaging
- Preparare la miscela di ibridazione FISH diluendo 2 μL di stock di sonda FISH con 8 μL di tampone di ibridazione, quindi applicarla al vetrino. Coprire il campione con un vetrino coprioggetti.
NOTA: Lo stock totale di sonde FISH utilizzate varia da 0,5 a 4 μL, a seconda della qualità dell'immagine. Se il segnale è troppo basso, aumentare l'ingresso della sonda. Ridurre l'ingresso della sonda FISH se lo sfondo è troppo alto, soprattutto quando si osservano detriti fluorescenti all'esterno dei nuclei. Il tampone di ibridazione può essere quello fornito con le sonde acquistate in commercio o preparato come nella sezione 1. - Posizionare i vetrini su una piastra calda, come un sistema di ibridazione con fossato per vetrini, per denaturare il DNA a 75 °C per 2-5 minuti. Quindi, trasferire il vetrino su un'altra piastra riscaldante impostata a 37 °C per ibridare durante la notte.
NOTA: Se la piastra riscaldante è dotata di una vaschetta per l'acqua o di un serbatoio per mantenere l'umidità durante l'ibridazione, non è necessario sigillare il vetrino coprioggetti con cemento di gomma. - Dopo l'ibridazione, immergere il vetrino in SSC riscaldato 0,4x a 40-60 °C con tampone di lavaggio IGEPAL CA-630 allo 0,3%, quindi rimuovere con cautela il vetrino coprioggetti. Continuare il lavaggio due volte per 5 minuti ciascuno al buio, agitando per i primi 10-15 s.
NOTA: Immergere il vetrino nel tampone di lavaggio aiuta a rilasciare delicatamente il vetrino coprioggetti. - Lavare il vetrino in SSC con IGEPAL CA-630 allo 0,1% per 5 minuti a RT al buio, agitando per i primi 10-15 s.
- Per spegnere l'autofluorescenza, trattare il vetrino con il kit di quenching in autofluorescenza (vedere la tabella dei materiali) applicando 150 μL di reagente (50 μL + 50 μL + 50 μL di reagenti A, B, C) per 2-5 minuti, quindi lavarlo con 2x SSC per 5 minuti.
NOTA: Questo è un passaggio facoltativo. L'autofluorescenza tissutale ha origine principalmente da componenti della matrice extracellulare, come il collagene e l'elastina. È anche significativamente influenzato dai lisosomi e dai mitocondri a causa del loro contenuto di lipofuscina, NADPH e coenzima flavina. La fissazione dell'aldeide e la presenza di cellule del sangue possono anche aumentare l'autofluorescenza13,14. - Colorare il vetrino con DAPI per 10 min. Sciacquare il vetrino con 2 tamponi SSC per 5 minuti.
NOTA: Se il vetrino non viene trattato con il kit di quenching in autofluorescenza, la colorazione DAPI può essere ridotta a 2 minuti. - Immergere rapidamente il vetrino in acqua deionizzata per non più di 1 s, quindi asciugarlo rapidamente assorbendo l'umidità in eccesso con un tovagliolo di carta.
NOTA: Questo è un passaggio facoltativo. Il trattamento con acqua deionizzata previene efficacemente la deposizione di cristalli di sale del tampone SSC sul vetrino e migliora la qualità dell'imaging. Tuttavia, in condizioni di ioni così bassi, i legami idrogeno tra la sonda FISH e il DNA bersaglio si indeboliscono, portando alla dissociazione della sonda e alla perdita del segnale. Pertanto, è fondamentale mantenere la fase di trattamento dell'acqua per un tempo molto breve. - Asciugare la diapositiva, quindi montarla con un supporto di montaggio antisbiadimento. Sigillare il vetrino coprioggetti con smalto per unghie prima dell'imaging.
NOTA: Se il vetrino viene trattato con un reagente di spegnimento in autofluorescenza, montare il vetrino con un mezzo di montaggio antisbiadimento secondo le istruzioni del produttore. Inoltre, a seconda del tipo di mezzo di montaggio, indurimento o non indurimento, il campione deve essere indurito per 1-24 ore prima della sigillatura e dell'imaging. Si consiglia di polimerizzare il campione almeno durante la notte per ottenere il miglior indice di rifrazione per l'imaging. - Utilizzare una lente a olio da 60× per catturare il segnale di fluorescenza. Utilizzare il canale DAPI per regolare la messa a fuoco. Assicurati di ottenere più Z-stack. In genere, sono sufficienti 5-10 z-stack con un intervallo di 1 μm. Esegui una proiezione 3D massima per ottenere la migliore risoluzione. Applica la deconvoluzione o altri algoritmi di cancellazione dello sfondo per migliorare ulteriormente la qualità dell'immagine.
Risultati
Abbiamo utilizzato campioni FFPE di entrambi i tumori mammari HER2-positivi e negativi per dimostrare il risultato dell'imaging FISH. L'amplificazione di HER2 (codificata dal gene ERBB2 ) è un marcatore favorevole a causa della disponibilità e dell'efficacia delle terapie di targeting molecolare di HER2. Al contrario, le pazienti con carcinoma mammario triplo negativo, che mancano di espressione di HER2, del recettore degli estrogeni (ER) e del recettore del progesterone (PR), affrontano esiti scarsi a causa delle limitate opzioni terapeutiche. Pertanto, determinare lo stato di HER2 è fondamentale nella ricerca e nel trattamento del cancro al seno15.
Nel campione di carcinoma mammario triplo negativo, la maggior parte dei nuclei mostra due punti distinti che rappresentano i segnali HER2/ERBB2 FISH. Alcuni nuclei possono avere un solo punto a causa della distorsione di sezionamento (Figura 2, a sinistra). Al contrario, i campioni HER2-positivi presentano abbondanti segnali FISH con due diversi pattern. Un modello mostra punti sparsi in tutto il nucleo (Figura 2, al centro). Questo modello è una caratteristica della morfologia dell'ecDNA, poiché gli ecDNA potrebbero non occupare un territorio nucleare unico e organizzato16. Inoltre, la segregazione asimmetrica degli ecDNA durante la mitosi guida la variazione del numero di copie, portando all'eterogeneità del segnale tra i nuclei17. Alcuni nuclei possono mostrare cluster occasionali, indicativi di hub di ecDNA18 (Figura 2, a destra). L'altro tipo di amplificazione HER2 presenta principalmente aggregati condensati a forma di bastoncino. Questa morfologia probabilmente indica un'amplificazione basata sui cromosomi, come le regioni di colorazione omogenea (HSR)19 o attraverso il ciclo di rottura-fusione-ponte (BFB)20. In particolare, l'amplificazione di ecDNA, HSR e BFB può coesistere nello stesso nucleo. Pertanto, si raccomanda di esaminare più nuclei per dedurre la forma di amplificazione focale.
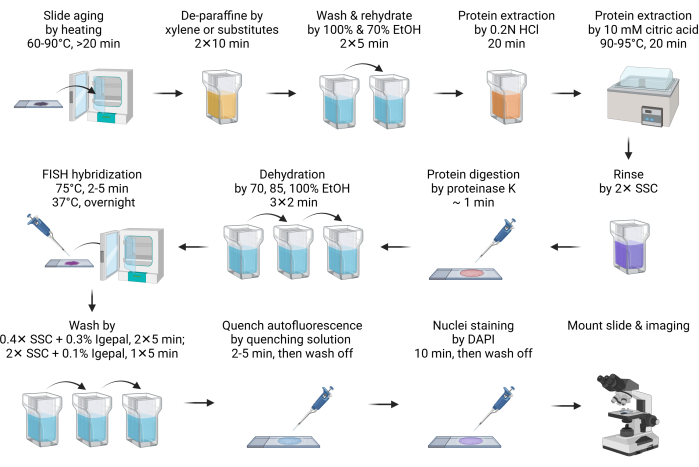
Figura 1: Schema per FISH in campioni FFPE. Fare clic qui per visualizzare una versione più grande di questa figura.
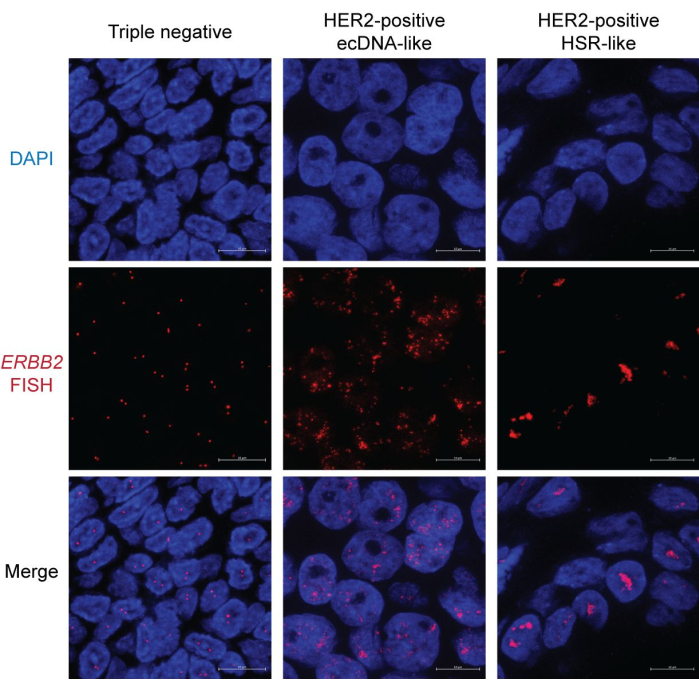
Figura 2: Immagine rappresentativa della FISH in campioni di FFPE per il cancro al seno. Ingrandimento: 600x; Barra della scala: 10 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
La FISH è un'opzione rapida ed economica per la diagnosi citogenetica. Soprattutto nel determinare se l'ecDNA è presente nel cancro, l'evidenza FISH rimane il gold standard1. La FISH nel tessuto FFPE consente una rapida determinazione dello stato genico nei campioni bioptici di un paziente, consentendo una diagnosi più rapida e il monitoraggio dei cambiamenti durante il progresso della malattia. Questa tecnica è particolarmente utile per testare campioni clinici che sono già stati raccolti per la patologia.
Questo protocollo prevede diversi passaggi critici. Il primo passo è la deparaffinazione completa. La paraffina residua può interrompere l'ibridazione della FISH. Se il campione appare ancora ceroso dopo il passaggio 2, deve essere trattato nuovamente con xilene fresco o suoi sostituti.
In secondo luogo, l'estrazione e la digestione delle proteine sono fondamentali. Questi processi non solo migliorano l'accessibilità del DNA alla sonda FISH, ma riducono anche significativamente l'autofluorescenza. Questo protocollo include tre fasi di deproteinizzazione. Mentre il trattamento con 0,2 N di HCl e 10 mM di acido citrico è semplice, la digestione della proteinasi K può richiedere un'ottimizzazione. La sovradigestione è l'errore più comune quando si utilizza la proteinasi K, con conseguente nuclei a forma di alone. Accorciare il tempo di digestione migliorerà la morfologia dei nuclei. Inoltre, si raccomanda di non digerire più di quattro campioni contemporaneamente per ridurre al minimo la differenza di tempo tra il primo e l'ultimo campione. È importante notare che anche un nucleo intatto può apparire come un alone al microscopio ad alto ingrandimento e ad alta risoluzione. Questo perché il nucleo non si trova sullo stesso piano focale. Pertanto, si suggerisce di prendere più Z-stack ed eseguire una proiezione massima per ispezionare la morfologia nucleare.
Infine, si consiglia l'autofluorescenza estinta. Sebbene l'estrazione acida e la digestione della proteinasi K possano ridurre significativamente il background di origine proteica, i metaboliti fluorescenti possono comunque influenzare la qualità dell'imaging.
Sebbene la FISH offra una risoluzione spaziale senza precedenti nell'identificazione dell'amplificazione genica focale, presenta dei limiti. In primo luogo, il contenuto e la produttività sono bassi rispetto agli approcci basati sulla PCR o sul sequenziamento di nuova generazione (NGS). In genere, da una a tre sonde FISH di colori diversi possono essere applicate a un singolo vetrino senza attrezzature specializzate. Ciononostante, i progressi nelle tecnologie di automazione hanno reso fattibile la FISH ad alto contenuto e ad alto rendimento, come il sequenziamento in situ 21. In secondo luogo, il progetto della sonda FISH richiede informazioni preliminari. Gli sforzi in corso per identificare gli eventi di amplificazione focale ricorrenti nel cancro hanno permesso la creazione di pannelli FISH pre-progettati per applicazioni di laboratorio e cliniche. Ad esempio, gli oncogeni della famiglia MYC sono spesso amplificati come ecDNA nel carcinoma polmonare a piccole cellule per mediare la resistenza alla chemioterapia. Pertanto, un pannello FISH mirato ai geni MYC, MYCL e MYCN può accelerare la determinazione delle risposte al trattamento nelle biopsie. In confronto, l'NGS consente uno screening più imparziale dei geni di interesse. Tuttavia, tra le tecnologie basate su NGS, solo il sequenziamento dell'intero genoma con analisi computazionale22 può caratterizzare l'ecDNA.
In sintesi, presentiamo istruzioni solide e complete per studiare l'amplificazione genica focale nei campioni FFPE. Esaminando il modello di segnale FISH, diventa inequivocabilmente chiaro se e come un locus genico viene amplificato. Prevediamo l'integrazione dell'apprendimento automatico nell'analisi delle immagini23 dei nuclei interfasici per estrarre informazioni citogenetiche riguardanti il numero di copie e la forma di amplificazione (cromosoma o ecDNA), semplificando così il processo di diagnosi molecolare e migliorando la nostra comprensione dei meccanismi patogenetici nel cancro.
Divulgazioni
S.W. è membro del comitato consultivo scientifico di Dimension Genomics Inc.
Riconoscimenti
S.W. è uno studioso ed è supportato dal Cancer Prevention and Research Institute of Texas (RR210034)
Materiali
| Name | Company | Catalog Number | Comments |
| DAPI | Tocris Bioscience | 5748 | Nucleus staining |
| Dextran sulfate 50% solution | EMD Millipore Sigma | S4030 | Probe hybridization buffer |
| ERBB2 (HER2) FISH Probe | Empire Genomics | ERBB2-20-RE | FISH probe |
| Ethanol | Decon Labs | 2716 | Dehydrating and hydrating tissue |
| Formamide | Thermo Scientific Chemicals | 205821000 | Probe hybridization buffer |
| Formula 83 (Xylene substitute) | CBG Biotech | CH0104A | Removing paraffin |
| Hydrochloric acid | Fisher Chemical | A144-500 | Sample pretreatment |
| IGEPAL CA-630 | Thermo Scientific Chemicals | J19628K2 | Slide washing |
| Proclin 300 | Sigma-Aldrich | 48914-U | Preservative for SSC buffer (optional) |
| Proteinase K (800 units/mL) | New England Biolabs | P8107S | Protein digestion |
| RNase A (20 mg/mL) | New England Biolabs | T3018L | Probe hybridization buffer |
| Slide Moat Hybridization System | Boekel Scientific | 280001 | Sample denature and hybridization. Alternative hot plates are acceptable. |
| Sodium chloride | Fisher Chemical | S2713 | SSC buffer |
| Sodium citrate dihydrate | Fisher BioReagents | FLBP3271 | SSC buffer and sample pretreatment |
| Tris-EDTA (TE) buffer | Fisher BioReagents | BP2473500 | Proteinase K digestion buffer |
| Tween-20 | Fisher BioReagents | BP337-500 | Probe hybridization buffer |
| Vectashield antifade mounting media | Vector Laboratories | H190010 | Slide mounting |
| Vector TrueVIEW | Vector Laboratories | SP8400 | Autofluorescence quenching kit |
Riferimenti
- ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium. Pan-cancer analysis of whole genomes. Nature. 578 (7793), 82-93 (2020).
- Wu, S., Bafna, V., Chang, H. Y., Mischel, P. S. Extrachromosomal DNA: An emerging hallmark in human cancer. Annu Rev Pathol. 17, 367-386 (2022).
- Luebeck, J., et al. Extrachromosomal DNA in the cancerous transformation of Barrett's oesophagus. Nature. 616 (7958), 798-805 (2023).
- Nathanson, D. A., et al. Targeted therapy resistance mediated by dynamic regulation of extrachromosomal mutant EGFR DNA. Science. 343 (6166), 72-76 (2014).
- Kim, H., et al. Extrachromosomal DNA is associated with oncogene amplification and poor outcome across multiple cancers. Nat Genet. 52 (9), 891-897 (2020).
- Pal Choudhuri, S., et al. Acquired cross-resistance in small cell lung cancer due to extrachromosomal DNA amplification of MYC paralogs. Cancer Discov. 14 (5), 804-827 (2024).
- Greytak, S. R., Engel, K. B., Bass, B. P., Moore, H. M. Accuracy of molecular data generated with FFPE biospecimens: Lessons from the literature. Cancer Res. 75 (8), 1541-1547 (2015).
- Chrzanowska, N. M., Kowalewski, J., Lewandowska, M. A. Use of fluorescence in situ hybridization (FISH) in diagnosis and tailored therapies in solid tumors. Molecules. 25 (8), 1864 (2020).
- Cui, C., Shu, W., Li, P. Fluorescence in situ hybridization: Cell-based genetic diagnostic and research applications. Front Cell Dev Biol. 4, 89 (2016).
- Finn, E. H., Misteli, T. A high-throughput DNA FISH protocol to visualize genome regions in human cells. STAR Protoc. 2 (3), 100741 (2021).
- Watters, A. D., Bartlett, J. M. S. Fluorescence in situ hybridization in paraffin tissue sections. Mol Biotechnol. 21 (3), 217-220 (2002).
- Richardson, S. O., et al. One-fits-all pretreatment protocol facilitating fluorescence in situ hybridization on formalin-fixed paraffin-embedded, fresh frozen and cytological slides. Mol Cytogenet. 12, 27 (2019).
- Monici, M. Cell and tissue autofluorescence research and diagnostic applications. Biotechnol Annu Rev. 11, 227-256 (2005).
- Davis, A. S., et al. Characterizing and diminishing autofluorescence in formalin-fixed paraffin-embedded human respiratory tissue. J Histochem Cytochem. 62 (6), 405-423 (2014).
- Cancer Genome Atlas Network. Comprehensive molecular portraits of human breast tumours. Nature. 490 (7418), 61-70 (2012).
- Liang, Z., et al. Chromatin-associated RNA dictates the ecDNA interactome in the nucleus. bioRxiv. , (2023).
- Lange, J. T., et al. The evolutionary dynamics of extrachromosomal DNA in human cancers. Nat Genet. 54 (10), 1527-1533 (2022).
- Hung, K. L., et al. ecDNA hubs drive cooperative intermolecular oncogene expression. Nature. 600 (7890), 731-736 (2021).
- Storlazzi, C. T., et al. Gene amplification as double minutes or homogeneously staining regions in solid tumors: origin and structure. Genome Res. 20 (9), 1198-1206 (2010).
- Guerin, T. M., Marcand, S. Breakage in breakage-fusion-bridge cycle: an 80-year-old mystery. Trends Genet. 38 (7), 641-645 (2022).
- Nguyen, H. Q., et al. 3D mapping and accelerated super-resolution imaging of the human genome using in situ sequencing. Nat Methods. 17 (8), 822-832 (2020).
- Deshpande, V., et al. Exploring the landscape of focal amplifications in cancer using AmpliconArchitect. Nat Commun. 10 (1), 392 (2019).
- Rajkumar, U., et al. EcSeg: Semantic segmentation of metaphase images containing extrachromosomal DNA. iScience. 21, 428-435 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon