Method Article
Charakterisierung des vermittelten extrazellulären Elektronentransfers in Milchsäurebakterien mit einem bioelektrochemischen System mit drei Elektroden und zwei Kammern
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Protokoll zur Charakterisierung des vermittelten extrazellulären Elektronentransfers (EET) in Milchsäurebakterien unter Verwendung eines bioelektrochemischen Systems mit drei Elektroden und zwei Kammern vor. Wir veranschaulichen diese Methode mit Lactiplantibacillus plantarum und dem Redoxmediator 1,4-Dihydroxy-2-naphthoinsäure und geben eine ausführliche Beschreibung der elektrochemischen Techniken, die zur Evaluierung der vermittelten EET verwendet werden.
Zusammenfassung
Viele Bakterien führen einen extrazellulären Elektronentransfer (EET) durch, bei dem Elektronen von der Zelle zu einem extrazellulären terminalen Elektronenakzeptor übertragen werden. Dieser Elektronenakzeptor kann eine Elektrode sein, und Elektronen können indirekt über ein redoxaktives Mediatormolekül abgegeben werden. Hier stellen wir ein Protokoll zur Untersuchung der vermittelten EET bei Lactiplantibacillus plantarum, einem probiotischen Milchsäurebakterium, das in der Lebensmittelindustrie weit verbreitet ist, unter Verwendung eines bioelektrochemischen Systems vor. Wir beschreiben, wie ein bioelektrochemisches System mit drei Elektroden und zwei Kammern zusammengebaut wird, und geben eine Anleitung zur Charakterisierung von EET in Gegenwart eines löslichen Mediators unter Verwendung von Chronoamperometrie und zyklischer Voltammetrie. Wir verwenden repräsentative Daten aus 1,4-Dihydroxy-2-naphthosäure (DHNA)-vermittelten EET-Experimenten mit L. plantarum , um die Datenanalyse und -interpretation zu demonstrieren. Die in diesem Protokoll beschriebenen Techniken können neue Möglichkeiten für die Elektrofermentation und Bioelektrokatalyse eröffnen. Jüngste Anwendungen dieser elektrochemischen Technik mit L. plantarum zeigten eine Beschleunigung des Stoffwechselflusses hin zur Herstellung von Fermentationsendprodukten, die wichtige Geschmackskomponenten bei der Lebensmittelfermentation sind. Als solches hat dieses System das Potenzial, weiterentwickelt zu werden, um Aromen in der Lebensmittelproduktion zu verändern oder wertvolle Chemikalien herzustellen.
Einleitung
Bioelektrochemische Systeme verbinden Mikroben mit Elektroden, was die Untersuchung von Mechanismen des extrazellulären Elektronentransfers (EET) ermöglicht und erneuerbare Ansätze für die Bioelektrokatalyse bietet 1,2,3. Mikroben, die auf natürliche Weise EET durchführen, werden als Exoelektronen bezeichnet, die Elektronen aus dem Stoffwechsel auf extrazelluläre terminale Elektronenakzeptoren übertragen, z. B. Eisen(hydr)oxide und Elektroden1. EET-Signalwege wurden erstmals bei Geobacter und Shewanella Spezies 4,5 charakterisiert und seitdem in vielen Bakterien identifiziert. Diese Exoelektronengene spielen eine zentrale Rolle in mehreren mikrobiellen elektrochemischen Technologien, wie z. B. der Erzeugung elektrischer Energie aus Abfallströmen, der Bindung von CO2 und der Herstellung wertvoller Chemikalien durch Elektrosynthese 1,6,7,8,9,10,11,12.
Eines dieser Exoelektronen ist Lactiplantibacillus plantarum, ein grampositives Milchsäurebakterium13. L. plantarum ist ein nomadisches, probiotisches Bakterium, das in einer Vielzahl von Umgebungen vorkommt, einschließlich des Darms von Menschen und anderen Wirbeltieren sowie vieler Arten von Lebensmitteln wie Fleisch, Getreide, Gemüse und fermentierten Lebensmitteln und Getränken 14,15,16,17. Sein Genom kodiert für einen flexiblen, heterofermentativen Stoffwechsel, der eine erfolgreiche Anpassung an diese verschiedenen Umgebungen ermöglicht. Es ist gut untersucht, in der Lebensmittel- und Gesundheitsindustrie weit verbreitet und von der Food and Drug Administration allgemein als sicher anerkannt18,19. Als solches hat L. plantarum das Potenzial, als nützliche Plattform für EET-basierte Technologien zu dienen.
Neuere Forschungen an L. plantarum haben ein Multi-Gen-Operon identifiziert, das für einen komplexen EET-Signalweg kodiert, der ursprünglich in Listeria monocytogenes charakterisiert wurde 13,20. In L. plantarum erleichtern die aus diesem Operon synthetisierten Proteine die EET in einem bioelektrochemischen System (BES), wenn ihnen das Chinon 1,4-Dihydroxy-2-naphthosäure (DHNA) als Elektronenmediator13 zur Verfügung gestellt wird. Das erste essentielle Protein in diesem Signalweg ist eine membrangebundene NADH-Chinon-Oxidoreduktase (Ndh2), die NADH oxidiert und DHNA reduziert. DHNA liefert Elektronen entweder direkt an eine Elektrode oder indirekt über das akzessorische Protein PplA (Abbildung 1)13,21,22. Neuere Forschungen deuten darauf hin, dass L. plantarum auch andere Chinonen, die strukturell ähnlich zu DHNA sind, als Elektronenmediatoren verwenden kann; L. plantarum ist jedoch nicht in der Lage, DHNA oder diese alternativen Chinone zu produzieren, so dass Mediatoren exogen in der Umwelt vorhanden sein müssen, damit EET auftretenkann 13,22,23.
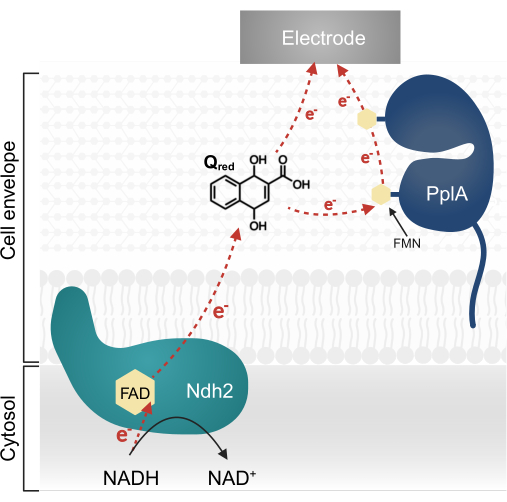
Abbildung 1: Elektronenfluss in Lactiplantibacillus plantarum EET. Ndh2 leitet Elektronen von NADH an das Chinon DHNA weiter. Elektronen werden zur Elektrode transportiert, um Strom zu erzeugen, entweder direkt durch reduziertes Chinon oder indirekt durch das akzessorische Protein PplA. Abkürzungen: FAD = Flavinadenindinukleotid; FMN = Flavin-Mononukleotid; EET = extrazellulärer Elektronentransfer; NADH = reduziertes Nicotinamid-Adenin-Dinukleotid; Ndh2 = NADH-Chinon-Oxidoreduktase; DHNA = 1,4-Dihydroxy-2-naphthosäure; PplA = Phospholipase A. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
In diesem Artikel stellen wir ein umfassendes Protokoll für die Verwendung einer BES-basierten Methode zur Charakterisierung der DHNA-vermittelten EET in L. plantarum vor. Ein System mit drei Elektroden und zwei Kammern schließt Bakterien an die Arbeitselektrode an und ermöglicht eine präzise Steuerung des an die Bakterien angelegten Potentials bei gleichzeitiger Verhinderung eines Übersprechens zwischen Arbeits- und Gegenelektrode. Wir präsentieren ein umfassendes Protokoll über 5 Tage, das die Vorbereitung vor dem Experiment, die BES-Assemblierung, die EET-Analyse mittels Chronoamperometrie (CA) und zyklischer Voltammetrie (CV) sowie die Probenanalyse nach dem Experiment umfasst. Dieses Protokoll kann angewendet werden, um die Mechanismen der EET-Signalwege zu entschlüsseln und Systeme für die Elektrofermentation und Elektrokatalyse zu bauen.
Protokoll
HINWEIS: BES-Baugruppen mit zwei Kammern werden im folgenden Protokoll als "Reaktoren" bezeichnet.
1. Vorbereitung der Medien
- Bereiten Sie das Kulturmedium L. plantarum vor.
- Bereiten Sie kommerzielle MRS-Medien (de Man Rogosa Sharpe) wie angewiesen und mMRS-Medien24 wie in Tabelle 1 beschrieben vor. Stellen Sie den pH-Wert von mMRS auf 6,5 ein. Beide Medien werden durch einen 0,22-μm-Filter filtersterilisiert und bis zur Verwendung bei 4 °C gelagert.
- Bereiten Sie ein chemisch definiertes Medium mit Mannitol (mCDM)13,25 vor, wie in Tabelle 2 beschrieben, und stellen Sie den pH-Wert auf 6,5 ein. Bereiten Sie genügend Medium vor, um die anodische Kammer jedes Reaktors mit 110 ml Medium zu füllen. Filtersterilisieren Sie mCDM durch einen 0,22-μm-Filter.
HINWEIS: mCDM sollte am Tag der bestimmungsgemäßen Verwendung frisch zubereitet werden. Die Komponentenlösungen können im Voraus vorbereitet werden. Alle Komponentenlösungen werden durch einen 0,22-μm-Filter filtriert und bei 4 °C gelagert. Wenn Sie Wolfe's Vitamine zubereiten, stellen Sie den pH-Wert vor dem Sterilisieren auf 11 ein und lagern Sie es dunkel oder in Folie eingewickelt. Wenn Sie Wolfe's Mineralien zubereiten, stellen Sie den pH-Wert auf 8 ein, nachdem Sie Nitrilotriacetinsäure (NTA) hinzugefügt haben. Fügen Sie dann die restlichen Komponenten hinzu, sterilisieren Sie sie und lagern Sie sie im Dunkeln oder in Folie eingewickelt.
- 1x PBS vorbereiten und zum Sterilisieren autoklavieren. Für die Verwendung in diesem Protokoll bei 4 °C lagern.
- Bereiten Sie handelsübliche M9-Medien wie angegeben vor (Tabelle 3) und autoklavieren Sie sie zum Sterilisieren. Bereiten Sie genügend Medien vor, um die kathodische Kammer jedes Reaktors mit 110 ml zu füllen. Bei Raumtemperatur lagern.
2. Tag 1: BES-Reaktormontage und ersteKultivierungvon L. plantarum
HINWEIS: In Abbildung 2 finden Sie einen Schaltplan eines BES-Reaktors und ein Diagramm, in dem die im Protokoll angegebenen Montageteile aufgeführt sind.
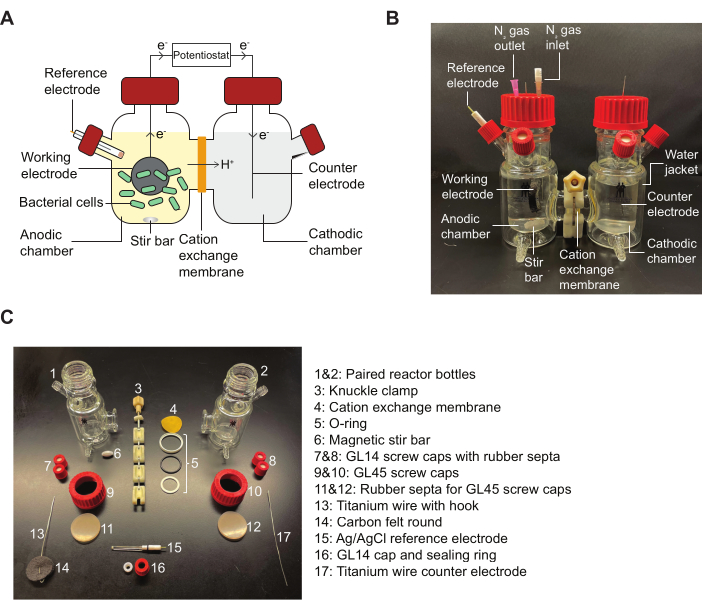
Abbildung 2: BES-Komponenten und Diagramm für die Montage. (A) Schematische Darstellung eines Zweikammer-BES-Reaktors. Bakterien (grün) in der anodischen Kammer übertragen Elektronen auf eine Arbeitselektrode (schwarzer Kreis) in Gegenwart eines Chinonmediators. Durch den Schaltkreis fließen Elektronen in die kathodische Kammer, so dass mit einem Potentiostaten Strommessungen zwischen Anode und Kathode durchgeführt werden können. (B) Ein Bild, das einen vollständig zusammengebauten BES-Reaktor darstellt, einschließlich N2-Einlass - und -Auslassnadeln in der anodischen Kammer. (C) Ein Bild, das alle Teile eines zerlegten Reaktors darstellt. Abkürzung: BES = bioelectrochemical system. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Reaktoren zusammenbauen und sterilisieren.
- Bereiten Sie die Arbeits- und Gegenelektroden vor. Vorgeschnittene Titandrähte mit einem Durchmesser von 1,0 mm für die Arbeits- und Gegenelektroden mit Aluminiumoxid-Schleifpapier gleichmäßig auf Hochglanz schleifen. Biegen Sie mit einer Zange ein Ende jedes Arbeitselektrodendrahtes zu einem kleinen Haken. Schieben Sie eine 16 cmlange 2 Carbonfilzrunde auf jeden Arbeitselektrodendraht, weben Sie den Draht einmal in die Carbonfilzrunde hinein und wieder heraus und ziehen Sie die Runde über den Draht nach unten, bis sie am Haken befestigt ist. Befestigen Sie die Arbeits- und Gegenelektroden in GL45-Kappen, indem Sie das Gummiseptum mit dem Draht durchstechen und einige Zentimeter durch die Kappe ziehen.
- Um gepaarte Reaktoren zusammenzubauen, montieren Sie zuerst den O-Ring und setzen Sie eine vorgeschnittene Kationenaustauschmembran, die in Wasser vorgetränkt wurde, in den zusammengebauten O-Ring ein. Platzieren Sie den O-Ring mit der Membran zwischen den großen Bodenöffnungen zweier gepaarter Reaktorflaschen. Befestigen Sie die Reaktorpaarflaschen und den O-Ring mit der Membran mit einer Knöchelklemme, um sie fingerfest zu halten. Lassen Sie einen magnetischen Rührstab in jede anodische Kammer fallen und verschließen Sie dann alle kleinen Öffnungen an der Oberseite jeder Flasche mit GL14-Kappen, die mit Gummisepten versehen sind.
HINWEIS: Ziehen Sie die Fingerknöchelklemmen oder Kappen nicht zu fest an, da dies zu Rissen oder Brüchen in den Flaschen führen kann. - Füllen Sie jede Reaktorflasche mit 110 ml deionisiertem (DI) Wasser und verschließen Sie dann jede Flasche mit der entsprechenden elektrodenmontierten GL45-Kappe. Setzen Sie Kappen ein, die mit einer runden Arbeitselektrode aus Carbonfilz versehen sind, indem Sie die Oberseite der Filzrunde vorsichtig nach unten drücken, damit die Patrone auf dem Haken bleibt.
HINWEIS: Frischer Kohlenstofffilz ist hydrophob und weist Wasser im Reaktor in der Erstmontage ab. Durch das Autoklavieren wird der Kohlenstofffilz für Experimente entsprechend hydrophil. - Sammeln Sie GL14-Elektrodenkappen, die mit Silikon-Dichtungsringen ausgestattet sind.
- Lösen Sie vor dem Autoklavieren vorsichtig alle GL45-Kappen. Autoklavieren Sie wassergefüllte Reaktoren und Elektrodenkappen zum Sterilisieren. Lassen Sie die Reaktoren nach dem Autoklavieren auf Raumtemperatur abkühlen.
- Kultur L. plantarum NCIMB8826. Kratzen Sie unter sterilen Bedingungen einige Bakterien von der Oberseite eines Glycerinvorrats ab und inokulieren Sie sie in 3 ml handelsübliches MRS-Medium. Lassen Sie die Kultur über Nacht bei 37 °C züchten, ohne sie zu schütteln.
HINWEIS: L. plantarum verwendet keinen Sauerstoff als terminalen Elektronenakzeptor; Daher ist es nicht erforderlich, das Medium durch Schütteln mit Sauerstoff anzureichern26. Die Kultivierungsbedingungen variieren jedoch, wenn andere Mikroben verwendet werden.
3. Tag 2: Vorbereitung der Referenzelektroden, Vorbereitung der Reaktoren für den Versuchsstart und Subkultivierung von L. plantarum
- Bereiten Sie Ag/AgCl-Referenzelektroden vor.
HINWEIS: Diese Schritte beschreiben die Vorbereitung der in der Materialtabelle angegebenen Ag/AgCl-Referenzelektroden. Benutzer sollten die Anweisungen des Herstellers befolgen, wenn sie eine andere Referenzelektrode verwenden.- Demontieren Sie die Ag/AgCl-Referenzelektroden und schleifen Sie die Drähte. Ziehen Sie die Elektrode vorsichtig aus dem Glasgehäuse und schnippen Sie mit dem Glasgehäuse, um die alte KCl-Lösung zu entleeren. Schleifen Sie die Elektrodendrähte vorsichtig mit Aluminiumoxid-Schleifpapier ab, bis sie gleichmäßig glänzen, um oxidiertes Material zu entfernen.
- Sammeln Sie alle geschliffenen Referenzelektrodendrähte in einem kleinen Becherglas mit einem kleinen Rührstab und füllen Sie das Becherglas mit 100% Bleichmittel, bis die Drähte vollständig eingetaucht sind. Chloridieren Sie die Drähte, indem Sie die Drähte auf einer Rührplattform 30 Minuten lang bleichen lassen, bis die Elektroden dunkler grau werden. Spülen Sie die Elektrodendrähte nach dem Bleichen gründlich mit DI-Wasser ab.
- Um eine Referenzelektrode wieder zusammenzubauen, füllen Sie das Glasgehäuse mit einer Spritze vollständig mit 3 M KCl-Lösung, die mit Silberchlorid gesättigt ist, und klopfen Sie vorsichtig auf die Seite, um alle Luftblasen zu entfernen. Füllen Sie die Elektrodendrahtkappe mit derselben Spritze mit KCl-Lösung und führen Sie den Draht in das Gehäuse ein. Legen Sie die Unterseite des Glasgehäuses gegen ein Papiertuch auf der Tischplatte, führen Sie die Elektrode in das Glasgehäuse ein und drücken Sie dann fest auf die Kappe, um die Elektrode zu schließen. Lagern Sie die Referenzelektroden bis zur Verwendung in einem flach mit KCl-Lösung gefüllten Becherglas und wiederholen Sie den Vorgang mit allen verbleibenden Referenzelektroden.
- Verwenden Sie ein Digitalmultimeter, um die Spannung von selbstgemachten Ag/AgCl-Referenzelektroden zu messen.
- Tauchen Sie die Enden der Ag/AgCl-Referenzelektroden (geschätzt auf 197 mV im Vergleich zur Standard-Wasserstoffelektrode, SHE) flach in ein Becherglas, das mit 3 M KCl gefüllt ist. Verbinden Sie das Multimeter sicher mit einer im Handel erhältlichen gesättigten Kalomelelektrode (SCE, 241 mV gegenüber SHE), die ebenfalls in dieselben KCl-Elektrolyte eingetaucht ist.
- Messen Sie die Potentialdifferenz zwischen den einzelnen Referenzelektroden und der SCE. Vergewissern Sie sich, dass sich die Referenzelektroden um 44 ± 10 mV von der SCE unterscheiden. Zerlegen Sie alle Referenzelektroden, die außerhalb dieses Bereichs liegen, und bauen Sie sie wieder zusammen.
- Versiegeln Sie die Naht an der Stelle, an der die Kappe auf das Glasgehäuse trifft, mit Parafilm.
- Bereiten Sie die Reaktoren für Experimente vor.
- Tauschen Sie in einer sterilen Biosicherheitswerkbank das autoklavierte Wasser in den Reaktoren gegen die entsprechenden Medien aus. Gießen Sie das autoklavierte Wasser aus. Füllen Sie die kathodischen Kammern mit 110 mL autoklaviertem M9-Medium. Füllen Sie die anodischen Kammern mit 110 mL frisch zubereitetem mCDM.
- Installieren Sie die Referenzelektroden. Entfernen Sie aus jeder anodischen Kammer eine GL14-Kappe und ersetzen Sie sie durch eine autoklavierte Elektrodenkappe (GL14-Kappe mit Silikon-Dichtungsring). Besprühen Sie die Referenzelektroden zum Sterilisieren mit 70 % Ethanol und platzieren Sie dann eine Referenzelektrode durch die Elektrodenkappe in jede anodische Kammer.
HINWEIS: Stellen Sie sicher, dass die Referenzelektrode keinen direkten Kontakt mit dem Carbonfilzrund hat. - Bevor Sie die Reaktoren aus der Biosicherheitswerkbank entfernen, ziehen Sie alle Kappen und Klemmen fingerfest an, um ein Auslaufen zu vermeiden.
- Befestigen Sie die Reaktoren an das Wasserpumpensystem. Stellen Sie jeden Reaktor auf die entsprechende Rührstab-Plattform. Verbinden Sie die Wassermantelstutzen jedes Reaktors mit einem Gummischlauch mit dem nächsten und verbinden Sie die Endreaktoren mit den Zu- und Abflussrohren der Wasserpumpe.
HINWEIS: Stellen Sie sicher, dass alle Verbindungen dicht und undicht sind, und verwenden Sie bei Bedarf Kabelbinder, um die Schläuche zu sichern. - Füllen Sie die Pumpe mit Wasser und fügen Sie 4-6 Tropfen Wasseraufbereiter hinzu. Schalten Sie das Pumpensystem ein und stellen Sie die Temperatur auf 30 °C ein. Starten Sie die Pumpe und beobachten Sie, wie das Wasser durch alle Wassermäntel des Reaktors fließt, um sicherzustellen, dass an den Anschlüssen keine Lecks vorhanden sind.
- Schalten Sie die Rührplattformen ein und stellen Sie sie auf Dauerrühren bei 220 U/min ein.
- Befestigen Sie die Reaktoren an den stickstoffreichen Gasleitungen. Befestigen Sie einen Luftfilter an einer 4-Zoll-Nadel mit 22 G und führen Sie die Nadel durch das obere Septum einer anodischen Kammer des Reaktors in das Medium ein, das als Stickstoffeinlass dient. Führen Sie eine weitere 1-Zoll-Nadel mit 18 G in das obere Septum der anodischen Kammer ein, die als Stickstoffauslass dient. Schließen Sie die Gasleitungen einer Stickstoffquelle an den Luftfilter an und öffnen Sie das Ventil, damit das Gas sanft durch den Reaktor sprudeln kann. Stellen Sie sicher, dass Stickstoff während des gesamten Experiments kontinuierlich durch alle anodischen Kammern sprudelt, um anaerobe Bedingungen aufrechtzuerhalten.
HINWEIS: Stellen Sie sicher, dass die Einlaufnadel von der Carbonfilzrunde entfernt ist. Der Blasenstrom sollte weder mit dem Kohlenstofffilz noch mit den Referenzelektroden in Berührung kommen. - Befestigen Sie die Bioreaktoren an den Potentiostatenleitungen. Verbinden Sie die Arbeits-, Zähler- und Referenzelektroden-Krokodilklemmenleitungen vom Potentiostaten mit den entsprechenden Elektroden.
HINWEIS: Verwenden Sie ein Multimeter, um den Widerstand zwischen dem Draht-/Stromabnehmer und den Elektroden zu überprüfen, um ordnungsgemäße elektrische Verbindungen sicherzustellen und potenzielle Messfehler zu minimieren.
- Eingabe der Potentiostatenparameter für den Vorlauf.
HINWEIS: Kritische Technikeinstellungen sind unten aufgeführt. In Tabelle 4 finden Sie eine erweiterte Liste der Softwareeinstellungen für jede Technik.- Schalten Sie den Potentiostaten ein und initialisieren Sie die EC-lab Software auf dem Rechner. Schließen Sie den Potentiostaten an den Computer an, indem Sie auf die Schaltfläche mit dem Potentiostatensymbol oben links unter Geräte klicken. Sobald die Verbindung hergestellt ist, wird der Gerätename mit einem grünen Kreis im Textfeld unten angezeigt.
- Synchronisieren Sie alle mit den Bioreaktoren verbundenen Kanäle in einer Gruppe, indem Sie auf die Registerkarte Bearbeiten klicken und dann Synchronisieren auswählen. Klicken Sie auf die entsprechenden Felder für die Kanalnummer | OK.
- Fügen Sie dem Potentiostaten die Technik Leerlaufspannung (OCV) hinzu, indem Sie auf die Schaltfläche + im Bereich Parametereinstellungen auf der linken Seite klicken. Im blauen Einstellungsfeld; Stellen Sie es so ein, dass das Arbeitselektrodenpotential (EWE) gegen die Referenzelektrode (RE) gemessen wird, wobei die Zeit im Intervall dt alle 36 s für insgesamt 3 h aufgezeichnet wird.
- Als nächstes fügen Sie dem Potentiostaten die Technik zyklische Voltammetrie (CV) hinzu. Stellen Sie das Anfangspotential des EWE auf Ei von 0 V gegenüber RE mit einer Abtastrate von 2 mV/s ein. Sweep auf ein Vertex-Potential (E1) von 0,4 V im Vergleich zu RE und Umkehrung auf ein Vertex-Potential (E2) von -0,7 V im Vergleich zu RE. Wiederholen Sie den Scan für insgesamt zwei Sweeps.
HINWEIS: In mikrobiellen elektrochemischen Systemen induzieren die mikrobielle Inokulation und die Biofilmbildung im Vergleich zu Metallmaterialien oder anorganischen Molekülen hohe Kapazitäten. Bei der zyklischen Voltammetrie-Abtastung variiert das Potential, und ein Ladestrom fungiert als Hintergrund. Um ein hohes Signal-Rausch-Verhältnis zu erreichen, sind niedrigere Scanraten notwendig, was jedoch die Dauer des Scans erhöht. Da wir zwei Zyklen abtasten, die ein breites elektrochemisches Fenster von 1,1 V abdecken, führt eine Abtastrate von 2 mV/s zu einem Halbzyklus, der bis zu 9,1 min dauert. Folglich dauern zwei Zyklen insgesamt 36,4 min. Eine weitere Reduzierung der Scanrate wäre übermäßig zeitaufwändig. - Hinzu kommt die Technik Chronoamperometrie (CA). Es wird ein konstantes Potential EWE von 0,2 V gegenüber RE für eine Zeit von t = 200 h angelegt und die Zeit alle 25-40 s in den Intervallen dt aufgezeichnet. Passen Sie das Zeitintervall basierend auf der Potentiostat-Software an, um den gewünschten Detaillierungsgrad zu erhalten.
HINWEIS: Das Mittelpunktspotenzial von DHNA beträgt ca. -0,093 V gegenüber Ag/AgCl; daher ist ein angelegtes Potential von 0,2 V gegenüber RE an die Arbeitselektrode ausreichend, um einen Elektronentransfer von DHNA zur Elektrode zu ermöglichen. - Nachdem Sie alle Parameter eingegeben haben, starten Sie den Lauf, indem Sie das grüne Startdreieck drücken. Speichern Sie die Datei wie gewünscht gemäß den Anweisungen der Software und klicken Sie dann auf Speichern. Die Software beginnt mit Technik 1, "OCV". Beobachten Sie die OCV-Spuren einige Minuten lang, um sicherzustellen, dass alle Reaktoren positiv ablesen und mit einem stetigen Signal dicht beieinander schließen. Lassen Sie das Experiment über Nacht laufen, um OCV und anfängliche CV abzuschließen (siehe Ergänzende Abbildung S1 für die Mediensteuerungs-CV), und lassen Sie CA laufen, bis sie stabilisiert ist.
- Unter sterilen Bedingungen subkulturieren Sie die MRS-Kultur von L. plantarum 1:200 in 50 mL mMRS. Züchten Sie die Zellen über Nacht bei 37 °C ohne Schütteln.
HINWEIS: Eine 50-ml-Übernachtkultur produziert in der Regel mehr als genug Zellen für sechs Reaktoren, wenn man von einem endgültigen OD600 von 0,2 im Reaktor ausgeht. Passen Sie das Kulturvolumen für größere oder kleinere Experimente entsprechend an.
4. Tag 3: Injektion von Zellen und DHNA/DMSO
- Waschen Sie die Zellen und injizieren Sie sie in Reaktoren.
- Nehmen Sie die mMRS L. plantarum-Kultur morgens aus dem Inkubator. Übertragen Sie die Kultur unter sterilen Bedingungen in ein konisches 50-ml-Röhrchen und legen Sie die Kultur auf Eis.
- Waschen Sie die Zellen 2x in sterilem, kaltem 1x PBS. Zentrifugieren Sie dazu die Kultur bei 4.000 × g für 5 min in einer 4 °C Zentrifuge, um die Zellen zu pelletieren. Unter sterilen Bedingungen werden die Zellen in 50 ml PBS vorsichtig, aber gründlich resuspendiert und dann wie zuvor erneut zentrifugiert. Wiederholen Sie den Vorgang für eine zweite Wäsche. Nach der abschließenden Zentrifuge resuspendieren Sie die Zellen in kaltem PBS auf OD600 = 11.
- Unter sterilen Bedingungen werden 2 ml resuspendierter Zellen in eine 3-ml-Spritze geladen, die mit einer Nadel für jeden Reaktor versehen ist.
- Entschließen Sie an der Reaktorstation eine Zellspritze und führen Sie die Nadel in den oberen Teil einer anodischen Kammer des Reaktors ein. Drücken Sie zu diesem Zeitpunkt nicht auf den Spritzenkolben. Wiederholen Sie dies für alle Reaktoren. Sobald alle Spritzen an Ort und Stelle sind, drücken Sie die Kolben, um die Zellen in die Reaktoren zu injizieren, und notieren Sie den Zeitpunkt der Injektion anhand der CA-Spur. Dieses Zellvolumen erzeugt in den Reaktoren einen End-OD600 von 0,2. Entsorgen Sie alle Spritzen in Behältern für biologische Gefahrstoffe und Nadeln in den dafür vorgesehenen Behälter für biologische Gefahrstoffe für scharfe und scharfe Gegenstände. Lassen Sie den Strom auf der CA-Leiterbahn für 2-4 Stunden auf flach stabilisieren.
HINWEIS: Nach der Injektion können Stromschwankungen auf der CA-Leiterbahn beobachtet werden. Nach 2-4 h stabilisieren sich diese Schwankungen auf einen flachen Strom (<2 μA Stromänderung im Laufe einer Stunde), woraufhin DHNA injiziert werden kann.
- Messen Sie den anfänglichen pH-Wert und injizieren Sie DHNA.
- Bereiten Sie eine DHNA-Lösung vor. Bereiten Sie in einem 1,5-ml-Röhrchen 500 μl einer 20 mg/ml-DHNA-Lösung vor, indem Sie pulverförmiges DHNA in 100 % DMSO auflösen. Füllen Sie 3 Insulinspritzen mit 110 μl DHNA-Lösung und 3 Insulinspritzen nur mit 110 μl DMSO.
HINWEIS: Während DHNA in Wasser leicht löslich ist, ist DMSO in dieser Konzentration ein besseres Lösungsmittel für DHNA-Aktien. Das Lösungsmittel des Mediators kann variieren, wenn andere Mediatoren eingesetzt werden. - Beschriften Sie in der BES-Reaktorstation die Versuchsreaktoren als + DHNA und die Lösungsmittelkontrollreaktoren als - DHNA. Entschließen Sie eine DHNA-Spritze und führen Sie die Spritze oben in jede anodische Kammer ein, die als + DHNA gekennzeichnet ist. Führen Sie eine reine DMSO-Spritze in jede anodische Kammer ein, die als - DHNA gekennzeichnet ist. Drücken Sie zu diesem Zeitpunkt nicht auf die Kolben der Spritze.
- Entnahme von Proben zu einem Zeitpunkt von 0 h für die Probenanalyse. Entnehmen Sie mit einer 3-ml-Spritze, die mit einer 2-Zoll-21-G-Nadel ausgestattet ist, eine 2-ml-Medienprobe aus jeder anodischen Kammer durch das unbenutzte Septum mit kleiner Kappe und übertragen Sie die Proben auf eine 24-Liter-Deep-Well-Platte, um pH-Messungen für den 0-Stunden-Zeitpunkt (DHNA-Injektionspunkt) durchzuführen. Falls gewünscht, entnehmen Sie 1 ml verbrauchtes Medium aus jeder anodischen Kammer und filtrieren Sie es durch einen 0,2-μm-Filter in sauber markierte Röhrchen zur Quantifizierung von Metaboliten mittels HPLC oder anderen Assays. Lagern Sie verbrauchte Medienproben bei -80 °C.
- Drücken Sie die Kolben aller DHNA- und DMSO-Spritzen, um in die Reaktoren zu injizieren. Zeichnen Sie den Zeitpunkt der Injektion aus dem CA-Trace auf. Entsorgen Sie alle Spritzen und Nadeln ordnungsgemäß.
- Messen und protokollieren Sie die 0-Stunden-pH-Proben für jeden Reaktor.
- Bereiten Sie eine DHNA-Lösung vor. Bereiten Sie in einem 1,5-ml-Röhrchen 500 μl einer 20 mg/ml-DHNA-Lösung vor, indem Sie pulverförmiges DHNA in 100 % DMSO auflösen. Füllen Sie 3 Insulinspritzen mit 110 μl DHNA-Lösung und 3 Insulinspritzen nur mit 110 μl DMSO.
5. Tag 4: Abschluss des Experiments und Probenentnahme
- Führen Sie 24 Stunden nach der DHNA-Injektion eine elektrochemische Analyse durch und entnehmen Sie die Endproben.
- Beenden Sie den CA-Lauf 24 Stunden nach der DHNA-Injektion.
- Entnahme von 24 h Zeitpunktproben für die Probenanalyse gemäß Schritt 4.2.3.
- Führen Sie CV erneut für den 24-Stunden-Zeitpunkt gemäß den in Schritt 3.3.4 beschriebenen Parametern aus.
- Messen und notieren Sie den pH-Wert für die 24-Stunden-Proben aus jedem Reaktor.
- Zerlegen und reinigen Sie die Reaktoren.
- Schalten Sie den Potentiostaten aus. Trennen Sie dann die Arbeits-, Zähler- und Referenzkabel von jedem Reaktor. Wischen Sie jegliche Feuchtigkeit von den Potentiostaten Blei-Krokodilklemmen ab.
- Schalten Sie den Stickstoffgasfluss aus. Trennen Sie die Gasleitungen und entfernen Sie dann die Zu- und Abflussnadeln. Entsorgen Sie alle Nadeln ordnungsgemäß in einem Behälter für scharfe Gegenstände.
- Schalten Sie die Wasserpumpe aus. Trennen Sie die Zu- und Abflussschläuche von der Pumpe und lassen Sie das Wasser in einen Eimer ablaufen. Halten Sie beim Trennen die Schlauchenden über der Wasserleitung in der Pumpe erhöht, um zu verhindern, dass Wasser auf den Boden tropft. Trennen Sie nacheinander jeden Reaktor von den Wassermantelschläuchen und arbeiten Sie vom endgültigen Auslaufreaktor zum ersten Zuflussreaktor.
- Entleeren Sie alle Medien aus den Reaktoren in einen großen Behälter für biologische Gefahren. Befolgen Sie zur Entsorgung die üblichen Bleichmethoden für Kulturen.
- Nach der Entleerung alle Reaktorteile zerlegen und reinigen. Entsorgen Sie Kationenaustauschermembranen und Carbonfilzkugeln in den richtigen Biomüllabfall. Reinigen Sie Referenzelektroden und Titandrähte mit 70% Ethanol und lagern Sie die gereinigten Referenzelektroden in einem flach mit Wasser gefüllten Becherglas. Reinigen Sie Reaktorflaschen, Kappen, O-Ringe und Klemmen vorsichtig in warmem Wasser mit Laborreiniger, spülen Sie sie gründlich mit DI-Wasser ab und trocknen Sie alle Teile an der Luft, bevor Sie sie lagern.
6. Tag 5: Elektrochemische Analyse
HINWEIS: Nachfolgend finden Sie eine allgemeine Beschreibung der Datendarstellung für dieses Protokoll. Detailliertere Beschreibungen zur Analyse und Dateninterpretation finden Sie im Abschnitt "Repräsentative Ergebnisse".
- Für die CA-Analyse: Legen Sie den Zeitpunkt der DHNA-Injektion auf den Zeitpunkt 0 h fest. Der Durchschnitt und die Standardabweichung der gemessenen Stromdichte (j in μA/cm2) alle 36 s für alle Replikate ab dem Zeitpunkt 0 h werden als Funktion der Zeit (h) aufgetragen. Berechnen Sie die Stromdichte in Abhängigkeit von der Arbeitselektrodenfläche (16 cm2).
- Für die CV-Analyse: Erstellen Sie für jede experimentelle Bedingung (nur Medien, DMSO oder DHNA) eine repräsentative CV-Spur, die die Stromdichte (j in μA/cm2) als Funktion des Arbeitselektrodenpotentials (EWE in V) darstellt. Plotzyklus zwei des ausgewählten CV-Laufs.
Ergebnisse
Chronoamperometrie-Analyse
Die EET von L. plantarum kann anhand der in Abbildung 3 dargestellten Chronoamperometrie (CA)-Daten beobachtet werden, in denen die Stromdichtekurve den Elektronentransfer von L. plantarum zur Arbeitselektrode visualisiert. Wir überwachten die Stromdichte (j) über die Zeit unter Beibehaltung eines konstanten Potentials von +200 mV gegenüber Ag/AgCl für 24 h. Bei der Injektion von 20 μg/ml DHNA in die rührende Elektrolytlösung wurde ein abiotischer DHNA-Oxidationsanstieg beobachtet, gefolgt von einem raschen Anstieg der biotischen Stromdichte, der etwa nach 8 h bei 132,0 ± 2,47 μA/cm2 seinen Höhepunkt erreichte. Umgekehrt führte die Injektion von DMSO zu einer vernachlässigbaren Stromdichte. Diese Ergebnisse unterstreichen die Bedeutung von DHNA als notwendiger und effizienter Mediator, um den Elektronentransfer zwischen L. plantarum und der Elektrode zu erleichtern. Der Benutzer kann die Stromausgabe anpassen, indem er die Konzentration von DHNA im BES anpasst. Frühere Forschungen deuten auch darauf hin, dass L. plantarum dosisabhängig über einen weiten Bereich von DHNA-Konzentrationen auf DHNA reagiert und in Gegenwart von DHNA-Konzentrationen von nur 0,01 μg/ml einen signifikanten Strom erzeugt13,22.
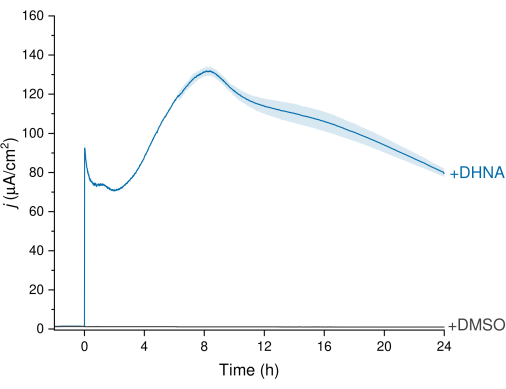
Abbildung 3: Chronoamperometrische Analyse von Lactiplantibacillus plantarum EET vermittelt durch DHNA. DHNA (20 μg/ml) oder DMSO wurde in mCDM-Elektrolyte (pH ~ 6,5) injiziert, wobei die Injektionszeit als t = 0 angegeben wurde. j steht für die Stromdichte in Abhängigkeit von der Arbeitselektrodenfläche. Die Experimente wurden bei 200 mV im Vergleich zu Ag/AgCl mit einer Kohlenstofffilzelektrode (16 cm2) und Rühren durchgeführt. Die Werte werden als Mittelwert ± sd aufgetragen, der in dreifachen BES-Reaktoren erhalten wurde. Abkürzungen: EET = extrazellulärer Elektronentransfer; DHNA = 1,4-Dihydroxy-2-naphthosäure; DMSO = Dimethylsulfoxid; mCDM = Chemisch definiertes Medium mit Mannitol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Zyklische Voltammetrie-Analyse
Um die DHNA-vermittelte EET bei L. plantarum weiter zu untersuchen, führten wir 24 Stunden nach der DHNA-Injektion eine zyklische Voltammetrie durch. Hier zeigen wir CV-Spuren für drei Erkrankungen: L. plantarum mit 20 μg/mL DHNA, L. plantarum mit DMSO und Medien mit 20 μg/mL DHNA. Wie in Abbildung 4A gezeigt, führte das Vorhandensein von 20 μg/ml DHNA in Reaktoren, die L. plantarum enthielten, zu einem deutlichen Anstieg des oxidativen Stroms bei 50 mV, der in Gegenwart von DMSO allein nicht auftrat. Diese Daten bestätigen, dass die Zugabe des Redoxmediators DHNA notwendig ist, um den Elektronentransfer zwischen L. plantarum und der Anode zu erleichtern. Während wir verschiedene kleinere Redox-Peaks in der L. plantarum + DMSO-Spur beobachteten, waren diese Peaks ähnlich wie in der Medienkontrollspur und werden wahrscheinlich auf redoxaktive Komponenten in mCDM zurückgeführt (Ergänzende Abbildung S1). In Abbildung 4B verglichen wir Spuren von DHNA unter biotischen Bedingungen (L. plantarum + DHNA) mit DHNA unter abiotischen Bedingungen (Media + DHNA). Während beide Spuren einen ausgeprägten oxidativen DHNA-Peak um 50 mV aufwiesen, beobachteten wir einen anhaltenden Anstieg des Stroms über 50 mV hinaus nur unter biotischen Bedingungen. Der katalytische Peak erreichte eine Stromdichte von 129 μA/cm2 bei 300 mV, was einer Steigerung von 256% im Vergleich zur abiotischen Spur entspricht. Dieses Umsatz-CV-Profil ist charakteristisch für mikrobielles EET27 und deutet auf eine Rereduktion von DHNA durch L. plantarum-Zellen in Gegenwart einer Elektronenquelle (Mannitol) nach Oxidation von DHNA an der Anode hin. Darüber hinaus wies die abiotische Spur neue oxidative Peaks um -240 mV und -180 mV auf. Frühere Forschungen deuten darauf hin, dass das Auftreten dieser Peaks auf den Abbau von DHNA in ACNQ (2-Amino-3-carboxy-1,4-naphthochinon) zurückzuführen sein könnte21,28. Wir haben diese Peaks in der biotischen Spur nicht beobachtet, was darauf hindeutet, dass die Interaktion von L. plantarum-Zellen mit DHNA DHNA stabilisieren und den Abbau verhindern könnte. Zu beachten ist, dass die 24-Stunden-Spur für Medien mit 20 μg/ml DHNA separat nach diesem Protokoll ohne Zugabe von Zellen durchgeführt wurde.
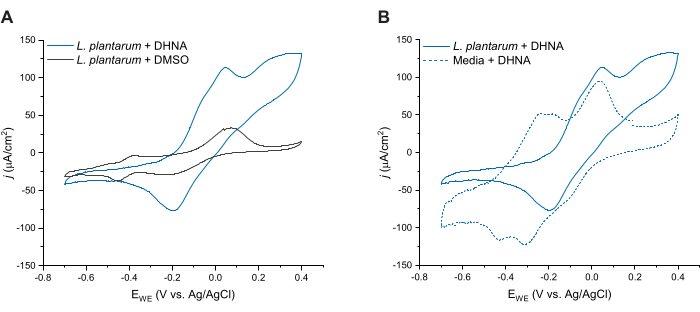
Abbildung 4: Repräsentative zyklische Voltammetrie-Kurven. Alle CV-Experimente wurden in mCDM unter Verwendung von Kohlenstofffilz (16 cm2) als Arbeitselektrode bei einer Abtastrate von 2 mV/s unter Rühren der Lösung durchgeführt. (A) kardiovaskuläre Spuren von Lactiplantibacillus plantarum mit entweder DHNA (20 μg/ml) oder DMSO bei t = 24 h. (B) kardiovaskuläre Spuren von 20 μg/ml DHNA in L. plantarum (biotische Bedingungen) oder nur mCDM (abiotische Bedingungen) bei t = 24 h. Abkürzungen: CV = zyklische Voltammetrie; mCDM = Chemisch definiertes Medium mit Mannitol; DHNA = 1,4-Dihydroxy-2-naphthosäure; DMSO = Dimethylsulfoxid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
pH-Analyse
Die EET-Aktivität in L. plantarum führte zu einem deutlichen Abfall des pH-Werts über 24 h. Wie in Abbildung 5 gezeigt, sank der durchschnittliche pH-Wert der Probe von L. plantarum, die DHNA ausgesetzt war, auf 3,33 ± 0,01 (p = 6,85 × 10-6, n = 3), während der durchschnittliche pH-Wert der Probe von L. plantarum, die DMSO ausgesetzt war, auf 6,50 ± 0,06 sank (p = 0,0409, n = 3). Wie in früheren Forschungen gezeigt, wird dieser Rückgang auf eine Zunahme des fermentativen Stoffwechsels zurückgeführt, die auftritt, wenn L. plantarum EET13 durchführt. L. plantarum metabolisiert Mannitol normalerweise durch Glykolyse und fermentative Wege, die Acetat, Laktat und Ethanol als Endfermentationsprodukte produzieren und ATP durch Phosphorylierung auf Substratebene erzeugen29. Unter EET-Bedingungen nimmt der metabolische Fluss durch Fermentation zu, wodurch die Produktion von Endfermentationsprodukten in den BES-Medien13 erhöht wird. Diese metabolische Verschiebung führt dazu, dass der pH-Wert des Mediums in den Reaktoren mit DHNA im Vergleich zu den DMSO-Regelreaktoren schneller abfällt.
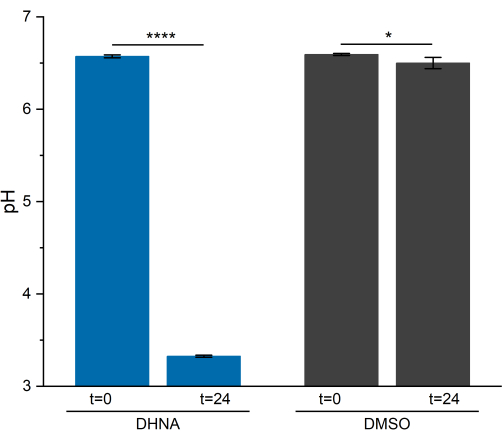
Abbildung 5: pH-Analyse des bioelektrochemischen Systems Lactiplantibacillus plantarum . Die Proben wurden bei t = 0 und t = 24 h während der Chronoamperometrie entnommen. Die Werte werden als Mittelwert ± sd aufgetragen, der in dreifachen BES-Reaktoren erhalten wurde. Die Signifikanz wurde durch einen einseitigen t-Test bestimmt. DHNA: P-Wert = 6,85 × 10-6. DMSO: P-Wert = 0,0409. Abkürzungen: DHNA = 1,4-Dihydroxy-2-naphthoinsäure; DMSO = Dimethylsulfoxid. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Zutaten für die Zubereitung von mMRS-Medien24. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: Zutaten für die Herstellung von mCDM-Medien. Diese Tabelle stammt von Tejedor-Sanz et al.13 und Aumiller et al.25. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 3: Zutaten für die Zubereitung von M9-Medien. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 4: EC-Lab-Parametereinstellungen für OCV-, CA- und CV-Techniken. Abkürzungen: OCV = Leerlaufspannung; CA = Chronoamperometrie; CV = zyklische Voltammetrie. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Abbildung S1: Repräsentative zyklische Voltammetrie-Spuren von Lactiplantibacillus plantarum allein mit DMSO und mCDM. CV-Traces für L. plantarum mit DMSO bei t = 24 h und mCDM allein bei t = 0 h. Alle CV-Experimente wurden mit Kohlenstofffilz (16 cm2) als Arbeitselektrode bei einer Abtastrate von 2 mV/s unter Rühren der Lösung durchgeführt. Abkürzungen: CV = zyklische Voltammetrie; mCDM = Chemisch definiertes Medium mit Mannitol; DHNA = 1,4-Dihydroxy-2-naphthosäure; DMSO = Dimethylsulfoxid. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Mit dem hier beschriebenen bioelektrochemischen System mit drei Elektroden und zwei Kammern konnten wir die Messung der Stromerzeugung durch DHNA-vermittelte EET in L. plantarum zeigen. Diese BES-Experimente generieren qualitativ hochwertige Daten; BES sind jedoch sensibel. Daher hängt der Erfolg des Protokolls von der Präzision des Benutzers ab, insbesondere bei der Montage von Reaktor- und Referenzelektroden, der Positionierung von Nadeln und Elektroden in der anodischen Kammer und dem Austausch der Kationenaustauschmembran. Es ist wichtig, die Reaktoren sorgfältig zusammenzubauen, um sicherzustellen, dass während des Autoklavierens oder Experimentierens kein Wasser/Medien austritt. Wasserleckagen können behoben werden, indem sichergestellt wird, dass die Kationenaustauschmembranen so geschnitten werden, dass sie genau auf den O-Ring passen, und indem die Achsschenkelklemme festgezogen wird. Es ist auch wichtig, den rund filzierten Kohlenstoff während des Autoklavierens vollständig in Wasser einzutauchen, damit er für Experimente hydrophil werden kann. Wir empfehlen neuen Benutzern, neu zusammengebaute mit Wasser gefüllte Reaktoren vor dem Autoklavieren 2 Stunden lang stehen zu lassen und auf Anzeichen von langsamen Lecks unter den Hauptflaschenanschlüssen zu prüfen. Darüber hinaus garantiert die Sicherstellung einer geeigneten Referenzelektrodenanordnung eine konsistente Datenreplikation über Reaktoren hinweg. Verfärbt sich die Teflonfritte im Inneren des Glasgehäuses, reißt oder trocknet, kann dies zu einem hohen Widerstand der Referenzelektrode führen. Benutzer können das Glasgehäuse austauschen, um die Leistung der Referenzelektrode wiederherzustellen.
Die richtige Ausrichtung aller Nadeln und Elektroden in der anodischen Kammer während des Experiments ist entscheidend für den Erfolg des Experiments. Die Referenzelektrode darf keinen Teil der Arbeitselektrode aus Kohlenstofffilz direkt berühren. Der Benutzer kann die Position des Carbonfilzes einstellen, indem er den Titandraht der Arbeitselektrode sanft von oberhalb des Reaktors dreht. Darüber hinaus sollte die Platzierung der Nadel für das Stickstoff-Sparging keinen direkten Kontakt mit Elektroden innerhalb der Kammer oder Elektroden-/Potentiostatenverbindungen über der Kammer haben. Der Stickstoffstrom sollte so eingestellt werden, dass er nicht in eine der Elektroden fließt. Schließlich sollte der Benutzer sicherstellen, dass der Rührstab die Arbeitselektrode nicht berührt, indem er die Arbeitselektrode 1-2 cm über dem Rührstab positioniert. Wenn ein unregelmäßiges Signal in OCV beobachtet wird, kann dies in der Regel behoben werden, indem die richtige Platzierung der Elektroden und des Stickstoffstroms im Reaktor sichergestellt wird und überprüft wird, ob die Verbindungen zwischen den Potentiostatenleitungen und den Reaktorelektroden korrekt und sicher sind. Schließlich zeigt unsere Erfahrung, dass Elektronenmediatoren wie DHNA in der Kationenaustauschmembran zurückgehalten werden können und bei zu vielen Wiederverwendungen einen hohen Hintergrundstrom verursachen. Wir empfehlen, die Kationenaustauschmembran nach zwei bis drei Anwendungen auszutauschen, insbesondere bei der Untersuchung der vermittelten EET, um zuverlässige experimentelle Ergebnisse zu gewährleisten.
Im Gegensatz zur direkten EET, bei der die direkte mikrobielle Bindung an die Elektrode den Elektronentransfer erleichtert, erfordert die vermittelte EET eine konsistente Diffusion von Elektronenshuttles über die Zellmembran und die Elektrode, was zu den hier beschriebenen einzigartigen BES-Einstellungen führt. Zunächst wählten wir in unserem Protokoll ein Doppelkammer-BES gegenüber dem Einkammer-Gegenstück, um anodische und kathodische Reaktionen mit einer Kationenaustauschmembran zu trennen. Diese Trennung verhindert, dass die frei diffundierenden Elektronenmediatoren (DHNA) und Mikroben mit der Kathode wechselwirken, wodurch sichergestellt wird, dass die mikrobielle EET die wichtigste Elektronenquelle ist, um die Elektronenmediatoren und die Anode zu reduzieren. Die Trennung ermöglicht auch eine präzise Kontrolle über Parameter wie die Konzentration/Verteilung des Mediators und das auf die Anode ausgerichtete Potenzial. Darüber hinaus haben wir uns für Carbonfilz als Anodenmaterial entschieden, neben anderen Optionen wie Graphitstäben, Metallelektroden, glasigem Kohlenstoff oder Indiumzinnoxid (ITO). Dies liegt daran, dass die poröse 3D-Struktur des Kohlenstofffilzes eine viel größere Oberfläche als die Elektroden30 bietet, was eine effiziente Nutzung von Mediatoren auch bei hohen Konzentrationen ermöglicht. Unsere BES-Einstellungen mit drei Elektroden und zwei Kammern bieten eine zuverlässige und reproduzierbare Auslesung der vermittelten EET auch bei Langzeitüberwachung. Dieser Prozess hat jedoch einen relativ geringen Durchsatz. Dieses Protokoll eignet sich für das Verständnis von EET-Mechanismen im Labormaßstab oder zum Testen von Prototypen von EET-Anwendungen. Alternative BES-Architekturen, wie z. B. tragbare oder gedruckte BESs31, 32, komplementäre Metalloxid-Halbleiter-Arrays (CMOS)33 oder hochskalierte BESs34, können von Forschern für verschiedene grundlegende oder Anwendungszwecke in Betracht gezogen werden.
In diesem Protokoll finden Sie detaillierte Anweisungen für die am häufigsten verwendeten elektrochemischen Techniken: Chronoamperometrie (CA) und zyklische Voltammetrie (CV). Es ist erwähnenswert, dass andere elektrochemische Techniken, wie z. B. die elektrochemische Impedanzspektroskopie (EIS) und die Differentialpulsvoltammetrie (DPV), durch die Analyse des Ladungsübertragungswiderstands und der Doppelschichtkapazität 35,36,37 tiefere Einblicke in das BES bieten können. Während dieses BES-Protokoll EET-Messungen ermöglicht, kann die Ergänzung elektrochemischer Daten durch Messungen der Stoffwechselaktivität und der Zellbiomasse für eine umfassende Analyse unerlässlich sein. Mikroben wie L. plantarum nutzen EET als eine der Elektronensenken neben anderen Fermentationsnebenprodukten wie Laktat und Ethanol. Darüber hinaus ist es bemerkenswert, dass das Wachstum der Zellbiomasse auch als Elektronensenke dient13. Daher bieten die Quantifizierung verbrauchter Elektronendonatoren (z. B. Mannitol), die Bewertung des Wachstums der Zellbiomasse und die Überwachung von Fermentationsnebenprodukten tiefere Einblicke in die Effizienz und physiologischen Auswirkungen von EET. Zelluläre Metaboliten werden üblicherweise mit Hilfe von Chromatographie und enzymatischen Assays quantifiziert, während die Lebensfähigkeit und das Wachstum der Zellen durch Zählen koloniebildender Einheiten und Messen der optischen Dichte verbrauchter Medien bei 600 nm bzw.13 bewertet werden. Es ist auch wichtig zu beachten, dass EET-Messungen empfindlich auf kleine Störungen unter experimentellen Bedingungen reagieren. Dazu gehören unter anderem der pH-Wert, die Temperatur, die Rührgeschwindigkeit und die Stickstoffgas-Begasungsrate38. Daher fungiert die Normalisierung der gemessenen EET-Spiegel mit bioanalytischen Messungen als interne Kontrolle, die eine konsistente Bewertung über Experimente hinweg ermöglicht, die an verschiedenen Tagen durchgeführt werden.
Durch die Kombination elektrochemischer Techniken mit anderen bioanalytischen Messungen schafft die vermittelte EET neue Möglichkeiten für die Elektrofermentation und Bioelektrokatalyse. Der konventionelle Einsatz von organischen, anorganischen oder enzymatischen Elektrokatalysatoren stellt aufgrund ihrer hohen Kosten eine Herausforderung dar und ist anfällig für Degradation. Alternativ bietet die Verwendung von Mikroben als lebende Elektrokatalysatoren aufgrund der Selbstreparatur- und Selbstreplikationsfähigkeiten der Mikroben eine kostengünstigere und skalierbarere Lösung39. L. plantarum, allgemein als sicheres Milchsäurebakterium anerkannt, ist ein besonders faszinierendes Chassis. Unter Verwendung identischer elektrochemischer Aufbauten, die in diesem Protokoll beschrieben werden, haben wir zuvor gezeigt, dass L. plantarum Grünkohlsaft unter EET-Bedingungen fermentieren und den Stoffwechselfluss hin zur Produktion von mehr Fermentationsendprodukten wie Laktat, Acetat und Succinat beschleunigenkann 13; Diese organischen Säuren sind essentielle Aromastoffe bei der Lebensmittelfermentation. Dies impliziert, dass durch den Einsatz elektrochemischer Techniken die vermittelte EET in L. plantarum möglicherweise gekapert werden kann, um den Stoffwechselfluss zu manipulieren, den Geschmack von Lebensmitteln zu verändern oder wertvolle Chemikalien zu produzieren. Es ist erwähnenswert, dass die in diesem Protokoll vorgestellten elektrochemischen Techniken nicht nur auf L. plantarum angewendet werden können, sondern auch generisch auf andere native oder gentechnisch veränderte Mikroben, die eine vermittelte EETdurchführen 40,41. Verschiedene Elektronenmediatoren, wie Flavin, Ferrocen, Neutralrot, Ferricyanid, Lawson und Menadion, können basierend auf dem Elektronentransfermechanismus der spezifischen verwendeten Mikrobe ausgewählt werden22,42. Darüber hinaus kann das in dieser Arbeit etablierte BES-Protokoll auf Exoelektronengene erweitert werden, die eine mediatorlose EET durchführen, wie zuvor an Shewanella- und Geobacter-Spezies gezeigt wurde 43,44. Ein optimiertes Wachstumsmedium sollte verwendet werden, um die zelluläre Aktivität der jeweiligen Mikrobe zu unterstützen und ihre EET-Leistung zu erleichtern. Dieses Protokoll stimmt die Parameter für die DHNA-vermittelte EET in L. plantarum feinab, aber Änderungen sind zu erwarten, wenn eine andere Mikrobe und Elektronenmediatoren angewendet werden.
Offenlegungen
Die Autoren haben keine konkurrierenden Interessen zu erklären.
Danksagungen
Wir danken den Mitgliedern des Ajo-Franklin-Labors für die aufschlussreichen Diskussionen über die BES-Montage, Wartung, kritische Schritte und Fehlerbehebung. Die Forschung wurde vom Army Research Office gesponsert und unter der Fördernummer W911NF-22-1-0239 (an C. M. A-F, zur Unterstützung von R. A.) und vom Cancer Prevention and Research Institute of Texas, Zuschuss # RR190063 (an C. M. A-F, zur Unterstützung von R. C., S. L. und B. B. K.) durchgeführt. Abbildung 1 wurde mit BioRender.com erstellt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1,4-Dihydroxy-2-naphthoic acid (DHNA) | Sigma-Aldrich | 281255-25G | |
| 1.0 mm diameter titanium wire | Thermo Fisher Scientific | 045485.BY | Cut to size for working and counter electrodes |
| 120-C Aluminum Oxide Sheets 9" x 11" | Johnson Abrasives | 10108-15 | |
| 3 mL plastic syringes | Thermo Fisher Scientific | 14955457 | |
| 3M KCl solution saturated with silver chloride | Millipore Sigma | 60137-250ML | |
| 6.35-mm-thick carbon felt | Thermo Fisher Scientific | 043200.RF | Cut into 16 cm2 rounds |
| Ag/AgCl reference electrode | CH Instruments | CH111 | |
| Air-Tite Premium Hypodermic Needles | Thermo Fisher Scientific | 14-817-102 | |
| AlK(SO)4 * 12H2O | Sigma-Aldrich | 237086-100G | |
| Ammonium citrate tribasic | Millipore Sigma | A1332 | |
| Avanti J-15R Centrifuge | Beckman Coulter | B99517 | |
| BD Precision Glide Needle, 18 G x 1 inch | Thermo Fisher Scientific | 14-826-5G | |
| BD Precision Glide Needle, 21 G x 2 inch | Thermo Fisher Scientific | 14-821-13N | |
| Bel-Art SP Scienceware Cleanware Aqua-Clear Water Condtioner | Thermo Fisher Scientific | 23-278339 | |
| Biotin | Millipore Sigma | B4639 | |
| CaCl2 | Millipore Sigma | C4901 | |
| Calcium D-(+)-pantothenate | Millipore Sigma | 1087009 | |
| Casamino acids | Millipore Sigma | 2240-OP | |
| cation exchange membrane | Membranes International | CMI-7000 | Cut into rounds fit to the BES O-ring |
| CoCl2 * 6H2O | Millipore Sigma | C8661 | |
| CuSO4 * 5H2O | Millipore Sigma | C8027 | |
| Cysteine-HCl * H2O | Millipore Sigma | 30129 | |
| DMSO | Millipore Sigma | 5439001000 | |
| DS-11+ Spectrophotometer | Denovix | N/A | |
| EC-Lab Software | BioLogic | N/A | |
| ECO E 4 S heating circulator | Lauda-Brinkmann | Cat. No. 115 V; 60 Hz : L001191 | |
| FeSO4 * 7H2O | Millipore Sigma | 215422 | |
| Folic acid | Millipore Sigma | F8758 | |
| H3BO3 | Millipore Sigma | B6768 | |
| Insulin syringes with BD Micro-Fine IV Needle | Thermo Fisher Scientific | 14-829-1A | |
| Lactiplantibacillus plantarum NCIMB8826 | N/A | N/A | Reference: Tejedor-Sanz et al., 2022 |
| Lactobacillus MRS Broth | HiMedia | M369 | |
| M9 Broth | Milliport Sigma | 63011 | |
| Magnesium sulfate anhydrous | Millipore Sigma | 208094 | |
| Manganese sulfate monohydrate | Millipore Sigma | 221287 | |
| mannitol | Millipore Sigma | M1902-1KG | |
| Mettler Toledo FiveEasy Benchtop pH Meter | Hogentogler | F20-KIT | |
| MgCl2 * 6H2O | Millipore Sigma | M9272 | |
| MgSO4 * 7H2O | Millipore Sigma | M2773 | |
| Millex - GV 0.22 µm PVDF Membrane Filter Unit | Millipore Sigma | SLGV004SL | |
| MnCl2 * 4H2O | Millipore Sigma | 203734 | |
| MnSO4 * H2O | Millipore Sigma | 221287 | |
| MOPS | Millipore Sigma | M1442 | |
| N2 gas | Airgas | NI UHP300 | Filter before use |
| Na2MoO4 * 2H2O | Millipore Sigma | 331058 | |
| Na2SO4 | Millipore Sigma | 238597 | |
| NaCl | Millipore Sigma | S9888 | |
| NH4Cl | Millipore Sigma | A9434 | |
| Nicotinic acid | Millipore Sigma | N-0761 | |
| Nitrilotriacetic acid (NTA) | Millipore Sigma | 72560 | |
| p-Aminobenzoic acid | Millipore Sigma | P9879 | |
| Phosphate buffered saline, 10x solution | Thermo Fisher Scientific | BP399-1 | |
| Potassium phosphate dibasic | Millipore Sigma | P8281 | |
| potentiostat | BioLogic | VMP-300 | |
| Protease peptone #3 | Bacto | 211693 | |
| Pyridoxine HCl | Millipore Sigma | P6280 | |
| Riboflavin | Millipore Sigma | 555682 | |
| RO10 magnetic stir bar platform | IKA | 3691000 | |
| Sodium acetate trihydrate | Millipore Sigma | 935700 | |
| Stir bar, egg-shaped | Thermo Fisher Scientific | 14-512-121 | Place in anodic chamber of BES |
| Thiamine HCl | Millipore Sigma | V-014 | |
| Thioctic acid (α-Lipoic acid) | Millipore Sigma | T-1395 | |
| Tryptophan | Millipore Sigma | 9136 | |
| Tween80 | Millipore Sigma | P4780 | |
| Vitamin B12 | Millipore Sigma | V6629 | |
| Jacketed MCF set, 100 ml, NW25, 2 x GL14 port | Adams & Chittenden Scientific Glass | NA | Customized |
| Yeast extract | Millipore Sigma | Y1625 | |
| ZnSO4 * 7H2O | Millipore Sigma | Z0251 |
Referenzen
- TerAvest, M. A., Ajo-Franklin, C. M. Transforming exoelectrogens for biotechnology using synthetic biology. Biotechnol Bioeng. 113 (4), 687-697 (2016).
- Chen, H., Dong, F., Minteer, S. D. The progress and outlook of bioelectrocatalysis for the production of chemicals, fuels and materials. Nat Catal. 3 (3), 225-244 (2020).
- Chen, H., et al. Fundamentals, applications, and future directions of bioelectrocatalysis. Chem Rev. 120 (23), 12903-12993 (2020).
- Lovley, D. R., Phillips, E. J. P. Novel mode of microbial energy metabolism: organic carbon oxidation coupled to dissimilatory reduction of iron or manganese. Appl Environ Microbiol. 54 (6), 1472-1480 (1988).
- Myers, C. R., Nealson, K. H. Bacterial manganese reduction and growth with manganese oxide as the sole electron acceptor. Science. 240 (4857), 1319-1321 (1988).
- Koch, C., Harnisch, F. Is there a specific ecological niche for electroactive Microorganisms. ChemElectroChem. 3 (9), 1282-1295 (2016).
- Zhao, J., et al. Microbial extracellular electron transfer and strategies for engineering electroactive microorganisms. Biotechnol Adv. 53, 107682 (2021).
- Zhang, J., Li, F., Liu, D., Liu, Q., Song, H. Engineering extracellular electron transfer pathways of electroactive microorganisms by synthetic biology for energy and chemicals production. Chem Soc Rev. 53 (3), 1375-1446 (2024).
- Logan, B. E. Exoelectrogenic bacteria that power microbial fuel cells. Nat Rev Microbiol. 7 (5), 375-381 (2009).
- Logan, B. E., Rabaey, K. Conversion of wastes into bioelectricity and chemicals by using microbial electrochemical technologies. Science. 337 (6095), 686-690 (2012).
- Gu, L., Xiao, X., Yup Lee, S., Lai, B., Solem, C. Superior anodic electro-fermentation by enhancing capacity for extracellular electron transfer. Bioresour Technol. 389, 129813 (2023).
- Boucher, D. G., et al. Bioelectrocatalytic synthesis: concepts and applications. Angew Chem Int Ed. 62 (46), e202307780 (2023).
- Tejedor-Sanz, S., et al. Extracellular electron transfer increases fermentation in lactic acid bacteria via a hybrid metabolism. eLife. 11, e70684 (2022).
- Martino, M. E., et al. Nomadic lifestyle of Lactobacillus plantarum revealed by comparative genomics of 54 strains isolated from different habitats. Environ Microbiol. 18 (12), 4974-4989 (2016).
- Fidanza, M., Panigrahi, P., Kollmann, T. R. Lactiplantibacillus plantarum-nomad and ideal probiotic. Front Microbiol. 12, 712236 (2021).
- Duar, R. M., et al. Lifestyles in transition: evolution and natural history of the genus Lactobacillus. FEMS Microbiol Rev. 41 (Suppl_1), S27-S48 (2017).
- Kaushik, J. K., et al. Functional and probiotic attributes of an indigenous isolate of Lactobacillus plantarum. PLOS ONE. 4 (12), e8099 (2009).
- Seddik, H. A., et al. Lactobacillus plantarum and its probiotic and food potentialities. Probiotics Antimicrob Proteins. 9 (2), 111-122 (2017).
- Siezen, R. J., et al. Phenotypic and genomic diversity of Lactobacillus plantarum strains isolated from various environmental niches. Environ Microbiol. 12 (3), 758-773 (2010).
- Light, S. H., et al. A flavin-based extracellular electron transfer mechanism in diverse Gram-positive bacteria. Nature. 562 (7725), 140-144 (2018).
- Tolar, J. G., Li, S., Ajo-Franklin, C. M. The differing roles of flavins and quinones in extracellular electron transfer in Lactiplantibacillus plantarum. Appl Environ Microbiol. 89 (1), e0131322 (2023).
- Li, S., De Groote Tavares, C., Tolar, J. G., Ajo-Franklin, C. M. Selective bioelectronic sensing of pharmacologically relevant quinones using extracellular electron transfer in Lactiplantibacillus plantarum. Biosens Bioelectron. 243, 115762 (2024).
- Blackburn, B., Alba, R., Hatch, A., Ajo-Franklin, C. M., Mevers, E. Understanding the Chemical Properties that Drive Extracellular Electron Shuttles. ChemRxiv. , (2024).
- De Man, J. C., Rogosa, M., Sharpe, M. E. A medium for the cultivation of Lactobacilli. J Appl Bacteriol. 23 (1), 130-135 (1960).
- Aumiller, K., et al. A chemically-defined growth medium to support Lactobacillus-Acetobacter sp. community analysis. PLOS ONE. 18 (10), e0292585 (2023).
- Brooijmans, R. J. W., de Vos, W. M., Hugenholtz, J. Lactobacillus plantarum WCFS1 electron transport chains. Appl Environ Microbiol. 75 (11), 3580-3585 (2009).
- LaBelle, E., Bond, D. R. Cyclic voltammetry for the study of microbial electron transfer at electrodes. Bio-electrochemical systems: from extracellular electron transfer to biotechnological application. , (2009).
- Mevers, E., et al. An elusive electron shuttle from a facultative anaerobe. eLife. 8, e48054 (2019).
- Dirar, H., Collins, E. B. End-products, Fermentation balances and molar growth yields of homofermentative Lactobacilli. Microbiology. 73 (2), 233-238 (1972).
- Huong Le, T. X., Bechelany, M., Cretin, M. Carbon felt based-electrodes for energy and environmental applications: A review. Carbon. 122, 564-591 (2017).
- Zhou, A. Y., Baruch, M., Ajo-Franklin, C. M., Maharbiz, M. M. A portable bioelectronic sensing system (BESSY) for environmental deployment incorporating differential microbial sensing in miniaturized reactors. PLOS ONE. 12 (9), e0184994 (2017).
- Benjamin, S. R., de Lima, F., Nascimento, V. A. d. o., de Andrade, G. M., Oriá, R. B. Advancement in paper-based electrochemical biosensing and emerging diagnostic methods. Biosensors. 13 (7), 689 (2023).
- Kumashi, S. R., et al. A CMOS multi-modal electrochemical and impedance cellular sensing array for massively paralleled exoelectrogen screening. IEEE Trans Biomed Circuits Syst. 15 (2), 221-234 (2021).
- Jadhav, D. A., et al. Scale-up of the bioelectrochemical system: Strategic perspectives and normalization of performance indices. Bioresour Technol. 363, 127935 (2022).
- Kim, J., Cestellos-Blanco, S., Shen, Y., Cai, R., Yang, P. Enhancing biohybrid CO2 to multicarbon reduction via adapted whole-cell catalysts. Nano Lett. 22 (13), 5503-5509 (2022).
- He, Z., Mansfeld, F. Exploring the use of electrochemical impedance spectroscopy (EIS) in microbial fuel cell studies. Energy Environ Sci. 2 (2), 215-219 (2009).
- Okamoto, A., Hashimoto, K., Nealson, K. H., Nakamura, R. Rate enhancement of bacterial extracellular electron transport involves bound flavin semiquinones. Proc Natl Acad Sci U S A. 110 (19), 7856-7861 (2013).
- Zhang, X., Li, X., Zhao, X., Li, Y. Factors affecting the efficiency of a bioelectrochemical system: a review. RSC Adv. 9 (34), 19748-19761 (2019).
- Kornienko, N., Zhang, J. Z., Sakimoto, K. K., Yang, P., Reisner, E. Interfacing nature's catalytic machinery with synthetic materials for semi-artificial photosynthesis. Nat Nanotechnol. 13 (10), 890-899 (2018).
- Glasser, N. R., Saunders, S. H., Newman, D. K. The colorful world of extracellular electron shuttles. Annu Rev Microbiol. 71 (1), 731-751 (2017).
- Gemünde, A., Lai, B., Pause, L., Krömer, J., Holtmann, D. Redox mediators in microbial electrochemical systems. ChemElectroChem. 9 (13), e202200216 (2022).
- Kundu, B. B., et al. Extracellular respiration is a latent energy metabolism in Escherichia coli. bioRxiv. , (2024).
- O'Brien, J. P., Malvankar, N. S. A simple and low-cost procedure for growing Geobacter sulfurreducens cell cultures and biofilms in bioelectrochemical systems. Curr Protoc Microbiol. 43 (1), A.4K.1-A.4K.27 (2016).
- Su, L., Fukushima, T., Ajo-Franklin, C. M. A hybrid cyt c maturation system enhances the bioelectrical performance of engineered Escherichia coli by improving the rate-limiting step. Biosens Bioelectron. 165, 112312 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten