Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Cuantificar cronológico levadura Duración de la vida por consecuencia de células envejecidas
En este artículo
Resumen
Envejecimiento cronológico en la levadura se refiere a la pérdida de la viabilidad celular asociado con el tiempo en la fase estacionaria. Aquí se describe un método de alto rendimiento para determinar cuantitativamente la levadura abarcan la vida cronológica.
Resumen
La levadura en ciernes
Protocolo
Parte 1: Preparación de los cultivos de envejecimiento
- Cepas racha de interés de las existencias congeladas en placas de agar YEPD (1% extracto de levadura, 2% Bacto-peptona, 2% de agar, 2% de glucosa).
- Se incuban las células a 30 ˚ C durante 48 horas o hasta que las colonias solo aparecen.
- Elige colonias aisladas e inocular en 5 ml de medio líquido YEPD (1% extracto de levadura, 2% Bacto-peptona, 2% de glucosa) en tubos de ensayo.
- Crecer cultivos de una noche a 30 ˚ C, manteniendo agitación constante usando un agitador o un tambor de rodillos.
- Inocular 5 ml de sintético completo (SC) medio (Tabla 1) con 50 l de la cultura durante la noche. En general las tres culturas SC se preparan para cada cepa para proporcionar la replicación triplicado biológica de los análisis de ciclo de vida de cada cepa se está examinando.
- Mantener los cultivos a 30 ˚ C con agitación constante en un tambor durante todo el experimento (por lo general dos o más semanas).
Parte 2: Tomar una viabilidad edad de punto
Después de dos días de la cultura en SC medios de comunicación, las células deben estar en la fase estacionaria y la primera edad de punto está listo para ser tomado. Con posterioridad la edad puntos se deben tomar cada 2-3 días durante un mínimo de dos semanas. Para cada punto de la edad:
- Preparar el Bioscreen 100 y placas de nido de abeja para la inoculación, llenando cada pozo con 145 l de YEPD. Asegúrese de dejar al menos un pozo lleno de sólo YEPD y no las células para el análisis de los datos más tarde.
- Eliminar los cultivos de envejecimiento de la incubadora.
- Agitar brevemente la primera cultura que se inocula en la placa de nido de abeja, teniendo cuidado de no derramar de la cultura.
- Tomar 5 l de la cultura mestiza y la pipeta que en el primer pocillo de la placa de panal. Llama la boca de cada tubo de ensayo antes y después de la eliminación de la alícuota de 5 mL.
- Repita este procedimiento para cada cultura del envejecimiento estar seguro de tener en cuenta la posición, así que corresponde a cada cultura. Idénticas posiciones y se debe utilizar para cada edad subsiguiente puntos durante todo el experimento.
- Vuelva a colocar la cultura en la incubadora de 30 ˚ C cuando haya terminado con las inoculaciones.
Parte 3: Carga de la máquina Bioscreen C MBR
- Exponer el compartimiento de la incubadora por levantar la tapa y retire la tapa de la bandeja de muestras.
- Inserte la placa recién inoculado de nido de abeja en la bandeja de la muestra (el uso de la ranura de la izquierda si sólo estás leyendo una placa).
- Vuelva a colocar la tapa de la bandeja de muestras y bajar la tapa del compartimiento de la incubadora.
- Asegúrese de que el fluido de transferencia de calor está por encima del nivel mínimo de llenado. Si está bajo, agregue más con una pipeta 1.000 l con el fluido de transferencia de calor siempre.
- Usando el software Bioscreen "EZExperiment", establecer los parámetros siguientes para obtener curvas de crecimiento adecuadas para Saccharomyces cerevisiae:
- Número de muestras: Escriba el número de pozos con los medios de comunicación o 200
- Filtro: 420-580nm, de banda ancha
- Temperatura: 30 ˚ C
- Duración experimento: un día, 0 horas, 0 segundos
- Intervalo de medición: 30 minutos
- Temblando: On, agitación continua, de alta
- Haga clic en "Inicio" para comenzar las lecturas.
Parte 4: Análisis de Datos
Por lo general, de seis años a los puntos se toman en el transcurso de dos semanas. Los puntos de la edad se toman en los días 2, 4, 6, 9, 11 y 13. Dependiendo del diseño experimental y tensiones a que está probado, puede ser conveniente tener la edad puntos más o menos frecuencia o por más de 2 semanas. Es importante que la carga de la placa de nido de abeja en el mismo orden para todas las edades-tal punto que cada cultura se corresponde con el envejecimiento de la misma posición y en todas las edades-punto, ya que esto hará que el análisis de datos mucho más fácil.
- Obtener los archivos de salida de la máquina Bioscreen C MBR. El software "EZExperiment" la salida de los datos Bioscreen como un archivo delimitado por tabuladores que es compatible con Microsoft Excel, así como otros programas. La primera columna muestra a qué hora de la medición de OD fue tomada durante el experimento, con columnas posteriores en representación de cada bien en el Bioscreen placas alveolares (Figura 1).
- Eliminar la primera lectura de DO de cada columna. Esta lectura es "ruido".
- Normalizar los datos al restar el valor de la DO del pozo con YEPD solo de todos los valores de DO de cada columna. Esto elimina la absorbancia de fondo por los medios de comunicación.
- Trazar las curvas consecuencia. Las curvas se desplazarán en función de la edad (Figura 2). Por ejemplo, mediante el trazado de las curvas de extensión de la columna 1 (y uno de la placa de nido de abeja) durante los seis puntos de momento, no hay un cambio claro hacia la derecha de las curvas en el tiempo.
- Calcular el tiempo de duplicación (δ) para cada bien sobre la base de la cinética de crecimiento de la edad de 2 días-punto. La ecuación utilizada para calcular el tiempo de duplicación is:

donde OD 1 y 2 representan OD sucesivas mediciones de OD y t 1 y t 2 es el tiempo entre las mediciones. Calcular tiempos de duplicación sólo entre los valores de DO de 0,2 a 0,5. El promedio de estos valores es el tiempo de duplicación para ese bien. Calcular tiempos de duplicación sólo entre los valores de DO de 0,2 a 0,5. El promedio de estos valores es el tiempo de duplicación para ese bien. La mayoría de las cepas salvajes de levadura debe dar un valor duplicada en un tiempo entre 85 a 90 minutos. - Para cada edad puntos, calcular el cambio de tiempo (T) en las curvas de extensión en relación con el punto inicial de la edad (día 2). Una manera fácil de hacer esto es determinar la diferencia en la cantidad de tiempo que le tomó a cada pocillo para llegar a un diámetro exterior de 0,3 entre el primer punto de la edad, y en cada edad de punto. El tiempo que un determinado pozo alcanzó una DO de 0,3 se puede calcular a partir de la ecuación de regresión lineal correspondiente a ln (DO) en función del tiempo que transcurre entre la puesta entre paréntesis OD dos puntos de tiempo = 0,3.
- Calcular la fracción superviviente en cada punto de la edad con el fin de generar una curva de supervivencia (Figura 3). Definir el punto inicial de la edad (día 2) al 100% de viabilidad. Para cada edad sucesivos puntos de calcular el porcentaje de supervivencia mediante la ecuación:
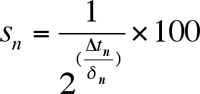
s, donde n es el porcentaje de supervivencia, Dt n es el cambio de hora, y δ n es el tiempo de duplicación. - Generar las curvas de supervivencia (Figura 3B) como se desee para cada bien (o pozos de replicar) por el trazado de la fracción (o porcentaje) de células viables en función de la edad.
- Calcular la supervivencia integral (SI) para cada pozo. SI se define como el área bajo la curva de supervivencia y se puede estimar mediante la fórmula:

donde la edad es la edad n puntos (por ejemplo, 2, 4, 6, 9, 11 y 13) y s n es el valor de supervivencia a esa edad-punto. - Determinar los parámetros estadísticos de los pozos de replicar los datos de la SI y la supervivencia. Común los parámetros estadísticos de interés, como media, mediana y la varianza para cada conjunto de réplicas biológicas. Un t-test o análisis similar puede ser utilizado para la comparación de pares de SI con fines experimentales y grupos control. También puede ser deseable para generar las curvas de supervivencia, como se describe en 4.8 de promedio réplicas biológicas.
Parte 5: Los resultados representativos
Al finalizar el experimento, se han trazado la curva de supervivencia y el análisis realizado de datos suficientes para determinar el potencial de envejecimiento cronológico de diferentes cepas o condiciones. Si se realiza correctamente, las curvas de crecimiento obtenidas de la máquina Bioscreen C MBR debe ser similar a los que se muestran en la Figura 2 y las curvas de supervivencia resultante debe ser similar a los que se muestran en la Figura 3. Por lo general, de tipo salvaje células cultivadas en las condiciones descritas aquí, tendrá un período cronológico la vida media del orden de 7 días. Variación sustancial en la supervivencia se observa en las diferentes cepas y bajo ciertas condiciones, tales como el crecimiento en los medios de la glucosa al 0,05%, la mediana de supervivencia puede exceder de 30 días.

Figura 1. Salida de datos desde el programa Bioscreen "EZExperiment". Una columna muestra la hora en que se tomó una lectura de absorbancia. Sucesivas columnas representan los pozos de la placa de nido de abeja inoculados con células obtenidas de cultivos de envejecimiento.
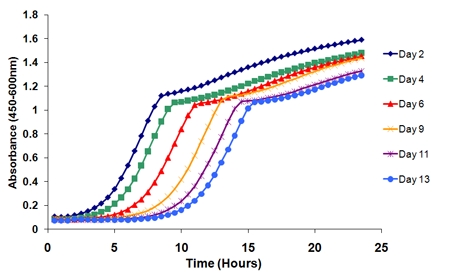
Figura 2. Curvas consecuencia de una réplica biológica única en el transcurso de un experimento. Hay un cambio notable en las curvas con el tiempo, las células en la cultura del envejecimiento pierden su viabilidad. El tiempo transcurrido entre el momento inicial (día 2) y un punto de tiempo sucesivos que determina la viabilidad a esa edad en particular.
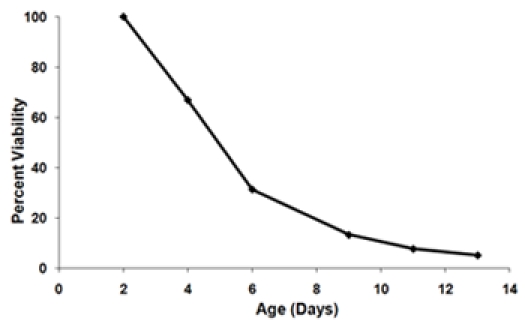
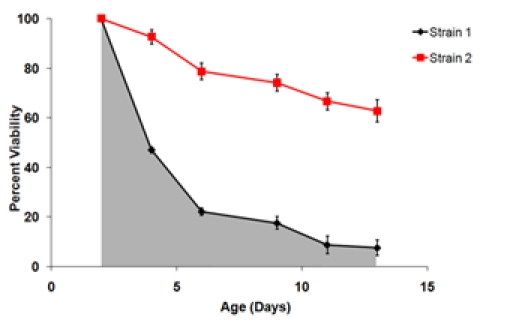
Figura 3. A) una curva de supervivencia generado con los datos fruto de la Figura 1. El día 2 punto de tiempo se establece como el punto de viabilidad del 100%. B) Curvas de supervivencia final de las dos cepas analizadas en el mismo experimento. Estas curvas de supervivencia representan las viabilidades promedio de tres réplicas biológicas de cada cepa. Las barras de error representan la desviación estándar dentro de réplicas biológicas. El área sombreada bajo la curva de supervivencia representa la supervivencia integral (SI) para la cepa 1.
| Tabla 1. Medio sintético definidos que se utilizan para estudios de envejecimiento cronológico (cepa backgrouª BY4743) | |
| Componente | Concentración (g / L) |
| D-glucosa | 20 |
| Levadura base de nitrógeno (-AA/AS) | 1.7 |
| (NH 4) 2 SO 4 | 5.0 |
| Adenina | 0.04 |
| L-Arginina | 0.02 |
| Ácido L-aspártico | 0.1 |
| L-ácido glutámico | 0.1 |
| L-histidina | 0.1 |
| L-leucina | 0.3 |
| L-lisina | 0.03 |
| L-Metionina | 0.02 |
| L-fenilalanina | 0.05 |
| L-serina | 0.375 |
| L-Treonina | 0.2 |
| L-triptófano | 0.04 |
| L-tirosina | 0.03 |
| L-valina | 0.15 |
| Uracilo | 0.1 |
Nota: Esta receta explica auxotrophies en BY4743 cepa diploide. Auxotrophies aminoácidos deben ser compensados mediante la adición de una concentración final de 5 veces el medio completo sintético.
Discusión
El alto rendimiento de la vida cronológica ensayo de duración se describe aquí es un método eficaz para cuantificar el potencial de envejecimiento de un gran número de cepas con alta exactitud y precisión. El principal avance de este método sobre los métodos clásicos para determinar la supervivencia de contar unidades formadoras de colonias (véase, por ejemplo 3) es el uso de un dispositivo de lectura agitador / incubadora / placa como la máquina Bioscreen C MBR para obtener alta resolución de las...
Agradecimientos
Este trabajo fue apoyado por el NIH subvención 1R21AG031965-01A1. MK es una Ellison Medical Foundation de Nueva Académico en el envejecimiento.
Materiales
| Name | Company | Catalog Number | Comments | |
| Bacto Peptone | Reagent | BD Biosciences | 211677 | |
| Bacto Yeast Extract | Reagent | BD Biosciences | 288620 | |
| Difco Agar | Reagent | BD Biosciences | 214530 | |
| Yeast Nitrogen Base w/o A.A. and A.S. | Reagent | MidSci | J630-500G | |
| Amino Acids | Reagent | Sigma-Aldrich | ||
| Ammonium Sulfate | Reagent | Spectrum | AM185 | |
| Dextrose | Reagent | Fisher Scientific | D16-10 | |
| Bioscreen C MBR machine | Tool | Growth Curves USA | 5101370 | |
| Bioscreen 100-well Honeycomb plate | Tool | Growth Curves USA | 9502550 |
Referencias
- Steinkraus, K. A., Kaeberlein, M., Kennedy, B. K. Replicative aging in yeast: the means to the end. Annu Rev Cell Dev Biol. 24, 29 (2008).
- Kaeberlein, M., Conn, P. M. . Handbook of models for human aging. , 109 (2006).
- Fabrizio, P., Longo, V. D. The chronological life span of Saccharomyces cerevisiae. Aging Cell. 2 (2), 73 (2003).
- Murakami, C. J., Burtner, C. R., Kennedy, B. K., Kaeberlein, M. A method for high-throughput quantitative analysis of yeast chronological life span. J Gerontol A Biol Sci Med Sci. 63 (2), 113 (2008).
- Piper, P. W., Harris, N. L., MacLean, M. Preadaptation to efficient respiratory maintenance is essential both for maximal longevity and the retention of replicative potential in chronologically ageing yeast. Mech Ageing Dev. 127 (9), 733 (2006).
- Fabrizio, P., et al. Superoxide is a mediator of an altruistic aging program in Saccharomyces cerevisiae. J Cell Biol. 166 (7), 1055 (2004).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados