Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Quantifier Life Span levure chronologique par excroissance de cellules âgées
Dans cet article
Résumé
Vieillissement chronologique dans la levure se réfère à la perte de la viabilité cellulaire associée avec le temps dans la phase stationnaire. Nous décrivons ici une méthode à haut débit pour déterminer quantitativement la durée de vie de la levure chronologique.
Résumé
La levure bourgeonnante
Protocole
Partie 1: Préparation des cultures de vieillissement
- Streak souches d'intérêt des stocks congelés sur des plaques de gélose YEPD (extrait de levure 1%, 2% de bacto-peptone, 2% d'agar, 2% de glucose).
- Incuber les cellules à 30 ˚ C pendant 48 heures ou jusqu'à ce que des colonies isolées apparaissent.
- Prélever des colonies unique et inoculer dans 5 ml de milieu liquide YEPD (extrait de levure 1%, 2% de bacto-peptone, 2% de glucose) dans des tubes à essai.
- Cultivez des cultures nuit à 30 ˚ C, tout en maintenant une agitation constante en utilisant soit un shaker ou à tambour à rouleaux.
- Inoculer 5 ml de synthèse complet (SC) moyen (Tableau 1) avec 50 uL de la culture de la nuit. Généralement trois cultures SC sont préparés pour chaque souche de fournir réplication triple biologiques de l'analyse de la durée de vie pour chaque souche à l'étude.
- Maintenir les cultures à 30 ˚ C avec agitation constante sur un tambour à rouleaux pour toute l'expérience (en général 2 semaines ou plus).
Partie 2: Prendre une viabilité à l'âge points
Après deux jours de culture dans les médias SC, les cellules doivent être en phase stationnaire et le premier point de l'âge est prêt à prendre. Après l'âge de points doivent être pris tous les 2-3 jours pour un minimum de deux semaines. Pour chaque point de l'âge:
- Préparer le Bioscreen 100 des plaques en nid d'abeille pour l'inoculation en remplissant chaque puits avec 145 ul de YEPD. Soyez sûr de laisser au moins un bien rempli avec seulement YEPD et non des cellules pour une analyse ultérieure des données.
- Retirez les cultures du vieillissement de l'incubateur.
- Brièvement au vortex la première culture à être inoculés dans la plaque en nid d'abeille, tout en faisant attention à ne pas répandre de la culture.
- Retirer 5 pi de la culture mixte et il pipette dans le premier puits de la plaque en nid d'abeille. Flamme de l'embouchure de chaque tube à essai, avant et après le retrait du aliquote de 5 ul.
- Répétez cette procédure pour chaque culture de vieillissement en étant sûr de noter la position bien correspondant à chaque culture. Identique positions bien doit être utilisé pour chaque période d'âge tout au long point de toute l'expérience.
- Remplacer les cultures dans l'incubateur à 30 ˚ C lorsque vous avez terminé avec les inoculations.
Partie 3: Chargement du Bioscreen C MBR machines
- Exposer le compartiment incubateur en soulevant le couvercle et retirez le couvercle sur le plateau de l'échantillon.
- Insérez la plaque nouvellement ensemencé Honeycomb dans le bac de l'échantillon (utiliser l'emplacement de gauche si vous êtes la seule lecture une plaque).
- Remettre le couvercle sur le plateau de l'échantillon et abaisser le couvercle du compartiment incubateur.
- Assurez-vous que le fluide caloporteur est au-dessus du niveau de remplissage minimum. Si elle est basse, ajouter plus de 1000 en utilisant une pipette uL avec le fluide caloporteur fourni.
- Utilisation du logiciel Bioscreen "EZExperiment", définir les paramètres suivants pour obtenir des courbes de croissance approprié pour Saccharomyces cerevisiae:
- Nombre d'échantillons: Entrez le nombre de puits avec des médias ou 200
- Filtre: 420-580nm, Wideband
- Température: 30 ˚ C
- Longueur Expérience: 1 Jour, 0 heures, 0 secondes
- Intervalle de mesure: 30 minutes
- Secouer: Sur, agitation continue, haute
- Cliquez sur "Démarrer" pour commencer les lectures.
Partie 4: Analyse des données
Typiquement, six ans-points sont pris au cours de deux semaines. Les points à l'âge sont prises au jour 2, 4, 6, 9, 11 et 13. Selon la conception expérimentale et les souches testées, il peut être souhaitable de prendre des points à l'âge plus ou moins fréquemment ou pendant plus de deux semaines. Il est important de charger la plaque en nid d'abeilles dans le même ordre pour chaque âge un tel point que chaque culture le vieillissement correspond à la même position et à tout âge-là, car cela fera l'analyse de données beaucoup plus facile.
- Obtenir les fichiers de sortie de la machine Bioscreen C MBR. Le logiciel "EZExperiment" va afficher les données dans un fichier Bioscreen délimité par des tabulations qui est compatible avec Microsoft Excel ainsi que d'autres logiciels. La première colonne indique à quel moment la mesure OD a été prise pendant l'expérience, avec des colonnes suivantes représentent chaque puits dans les plaques en nid d'abeille Bioscreen (figure 1).
- Supprimer la première lecture OD de chaque colonne. Cette lecture est un «bruit».
- Normaliser les données en soustrayant la valeur de DO du puits avec YEPD seul de toutes les valeurs OD dans chaque colonne. Cela supprime l'absorbance de fond par les médias.
- Tracer les courbes excroissance. Les courbes se déplacera en fonction de l'âge (figure 2). Par exemple, en traçant les courbes excroissance de la colonne 1 (puits 1 de la plaque en nid d'abeille) sur les six points du temps, il ya un déplacement vers la droite distinctes des courbes au cours du temps.
- Calculer le temps de doublement (δ) pour chaque puits basée sur la cinétique de croissance de l'âge de 2 jours-point. L'équation utilisée pour calculer le temps de doublement is:

où OD 1 et OD 2 mesures successives représentent DO et T 1 et T 2 étant le temps entre les mesures. Calculer des temps de doublement seulement entre les valeurs de DO de 0,2 à 0,5. La moyenne de ces valeurs est le temps de doublement de ce bien. Calculer des temps de doublement seulement entre les valeurs de DO de 0,2 à 0,5. La moyenne de ces valeurs est le temps de doublement de ce bien. La plupart des souches de levures de type sauvage doit donner une valeur temps de doublement compris entre 85 à 90 minutes. - Pour chaque âge-point, calculer le décalage de temps (At) dans les courbes excroissance par rapport à l'âge de départ-point (jour 2). Un moyen facile de le faire est de déterminer la différence dans la longueur du temps qu'il a fallu pour chaque puits pour atteindre une DO de 0,3 entre les initiales d'âge et chaque point de l'âge subséquentes points. Le temps qu'une particulière ainsi atteint une DO de 0,3 peut être calculée à partir de l'équation de régression linéaire correspondant à ln (DO) en fonction du temps entre les deux points de bracketing temps DO = 0,3.
- Calculer la fraction survivante à chaque âge-point afin de générer une courbe de survie (figure 3). Définir le point de départ à l'âge (jour 2) à la viabilité de 100%. Pour chaque âge successifs point de calculer la survie pour cent en utilisant l'équation:
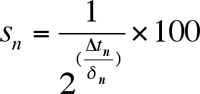
où s n est le pourcentage de survie, At n est le décalage dans le temps, et δ n est le temps de doublement. - Générer les courbes de survie (figure 3B) comme souhaité pour chaque bien (ou puits répliquer) en traçant la fraction (ou pour cent) de cellules viables en fonction de l'âge.
- Calculer la survie (SI) solidaire de chaque puits. SI est définie comme l'aire sous la courbe de survie et peut être estimée par la formule:

l'âge où n est le point de l'âge (par exemple 2, 4, 6, 9, 11 et 13) et s n est la valeur de survie à cet âge-point. - Déterminer des paramètres statistiques des puits de reproduire à partir des données du SI et de survie. Commune paramètres statistiques d'intérêt comprennent moyenne, médiane, et la variance pour chaque ensemble de biologiques répétitions. Un t-test ou une analyse similaire peut être utilisée pour la comparaison par paires des SI à des fins expérimentales et différents groupes de contrôle. Il peut être également souhaitable de générer des courbes de survie, comme décrit dans 4.8 de moyenne biologiques répétitions.
Partie 5: Les résultats représentatifs
À la fin de l'expérience, vous aurez tracé la courbe de survie et effectué une analyse de données suffisantes pour déterminer le potentiel de vieillissement chronologique des différentes souches ou des conditions. Si effectué correctement, les courbes de croissance obtenus à partir de la machine Bioscreen C MBR devrait ressembler à ceux de la figure 2 et les courbes de survie résultant devrait ressembler à ceux de la figure 3. Généralement, les cellules de type sauvage cultivées dans les conditions décrites ici aura une durée médiane de la vie chronologique de l'ordre de 7 jours. Variation importante du taux de survie est observée dans différentes souches et sous certaines conditions, telles que la croissance dans les médias de glucose 0,05%, la médiane de survie peut dépasser 30 jours.

Figure 1. Sortie des données du programme "EZExperiment" Bioscreen. Colonne A affiche l'heure à laquelle une lecture de l'absorbance a été prise. Colonnes successives représentent les puits de la plaque en nid d'abeille inoculées avec des cellules prélevées à partir des cultures de vieillissement.
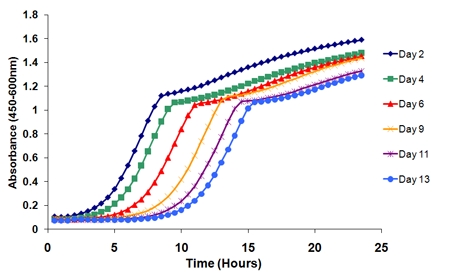
Figure 2. Courbes Excroissance d'une réplique biologique unique au cours d'une expérience. Il ya une nette évolution dans les courbes au fil du temps que les cellules dans la culture du vieillissement perdent leur viabilité. La longueur de temps entre le point de temps initial (jour 2) et un point de temps successifs détermine la viabilité à cet âge en particulier.
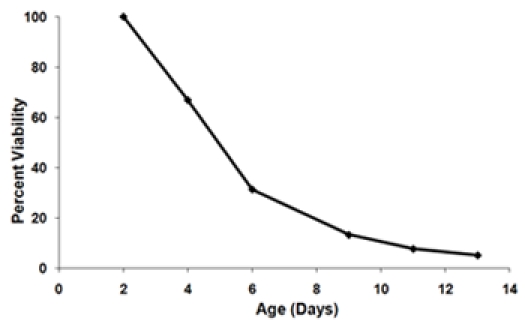
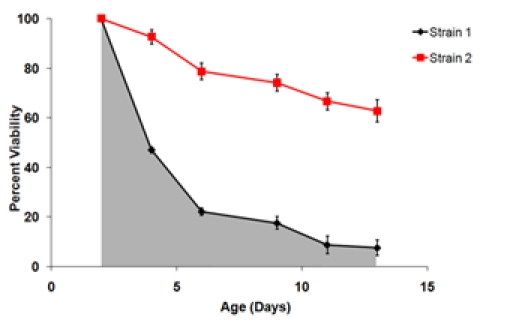
Figure 3. A) Une courbe de survie en utilisant les données générées excroissance de la figure 1. Le point 2 est temps de jour défini comme le point de la viabilité de 100%. B) Les courbes de survie final de deux souches testées dans l'expérience même. Ces courbes de survie représentent les viabilités moyenne de trois répétitions pour chaque biologiques souche. Les barres d'erreur représentent l'écart-type au sein biologiques répétitions. La zone hachurée sous la courbe de survie représente la survie (SI) pour la souche intégrante 1.
| Tableau 1. Synthétique milieu défini Utilisé pour des études de vieillissement chronologique (souche backgrouND BY4743) | |
| Composante | Concentration (g / L) |
| D-glucose | 20 |
| Base azotée de levure (-AA/AS) | 1.7 |
| (NH 4) 2 SO 4 | 5,0 |
| Adénine | 0,04 |
| L-arginine | 0,02 |
| Acide L-aspartique | 0.1 |
| L-acide glutamique | 0.1 |
| L-histidine | 0.1 |
| L-Leucine | 0.3 |
| L-Lysine | 0,03 |
| L-Méthionine | 0,02 |
| L-phénylalanine | 0,05 |
| L-sérine | 0,375 |
| L-Thréonine | 0.2 |
| L-tryptophane | 0,04 |
| La L-Tyrosine | 0,03 |
| L-Valine | 0,15 |
| Uracile | 0.1 |
Note: Cette recette représente auxotrophies dans diploïde BY4743 souche. Auxotrophies acides aminés doit être compensée par l'ajout d'une concentration 5X final au milieu synthétique complet.
Discussion
Le test à haut débit chronologique la durée de vie décrit ici est une méthode efficace pour quantifier le potentiel de vieillissement d'un grand nombre de souches avec une grande précision et de précision. L'avance principal de cette méthode par rapport aux méthodes classiques pour déterminer la survie en comptant unités formant colonie (voir par exemple 3) est l'utilisation d'un dispositif de lecture secoueur / incubateur / plaque telles que la machine Bioscreen C MBR afin d'ob...
Remerciements
Ce travail a été soutenu par des subventions du NIH 1R21AG031965-01A1. MK est une Ellison Medical Foundation New Scholar liés au vieillissement.
matériels
| Name | Company | Catalog Number | Comments | |
| Bacto Peptone | Reagent | BD Biosciences | 211677 | |
| Bacto Yeast Extract | Reagent | BD Biosciences | 288620 | |
| Difco Agar | Reagent | BD Biosciences | 214530 | |
| Yeast Nitrogen Base w/o A.A. and A.S. | Reagent | MidSci | J630-500G | |
| Amino Acids | Reagent | Sigma-Aldrich | ||
| Ammonium Sulfate | Reagent | Spectrum | AM185 | |
| Dextrose | Reagent | Fisher Scientific | D16-10 | |
| Bioscreen C MBR machine | Tool | Growth Curves USA | 5101370 | |
| Bioscreen 100-well Honeycomb plate | Tool | Growth Curves USA | 9502550 |
Références
- Steinkraus, K. A., Kaeberlein, M., Kennedy, B. K. Replicative aging in yeast: the means to the end. Annu Rev Cell Dev Biol. 24, 29 (2008).
- Kaeberlein, M., Conn, P. M. . Handbook of models for human aging. , 109 (2006).
- Fabrizio, P., Longo, V. D. The chronological life span of Saccharomyces cerevisiae. Aging Cell. 2 (2), 73 (2003).
- Murakami, C. J., Burtner, C. R., Kennedy, B. K., Kaeberlein, M. A method for high-throughput quantitative analysis of yeast chronological life span. J Gerontol A Biol Sci Med Sci. 63 (2), 113 (2008).
- Piper, P. W., Harris, N. L., MacLean, M. Preadaptation to efficient respiratory maintenance is essential both for maximal longevity and the retention of replicative potential in chronologically ageing yeast. Mech Ageing Dev. 127 (9), 733 (2006).
- Fabrizio, P., et al. Superoxide is a mediator of an altruistic aging program in Saccharomyces cerevisiae. J Cell Biol. 166 (7), 1055 (2004).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon