Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
El examen de los telómeros G-Estructura en voladizo Trypanosoma brucei
En este artículo
Resumen
Los telómeros son esenciales para la estabilidad de los cromosomas y los telómeros G-voladizo estructura es esencial para la telomerasa mediada mantenimiento de los telómeros. Recientemente hemos aprobado dos métodos para la detección de los telómeros G-estructura en voladizo Trypanosoma brucei, Que son nativos en gel de hibridación y la ligadura mediada por extensión del cebador, que se describe.
Resumen
Los telómeros G-voladizo estructura ha sido identificada en muchos eucariotas como levaduras, los vertebrados, y Trypanosoma brucei. Que sirve como sustrato para la telomerasa para la síntesis de novo del ADN de los telómeros y por lo tanto, es importante para el mantenimiento de los telómeros. T. brucei es un parásito protozoario que causa la enfermedad del sueño en los seres humanos y nagana en el ganado. De acogida, una vez infectados los mamíferos, T. celular brucei regularmente cambia su antígeno de superficie para evadir el ataque inmunológico del huésped. Recientemente hemos demostrado que el T. brucei estructura de los telómeros desempeñan un papel esencial en la regulación de la expresión del gen del antígeno de superficie, lo cual es fundamental para la T. brucei patogénesis. Sin embargo, T. brucei estructura de los telómeros no ha sido ampliamente estudiado debido a la limitación de los métodos para el análisis de esta estructura especializada. Ahora hemos adoptado con éxito los nativos en gel de la hibridación y la ligadura mediada por los métodos de extensión del cebador para el examen de los telómeros G-voladizo estructura y un método de ligadura de adaptador para la determinación de la terminal de los telómeros de nucleótidos en T. brucei células. Aquí, vamos a describir en detalle los protocolos y comparar sus ventajas y limitaciones.
Protocolo
1. Detectar T. brucei telómeros G-pendiente con los nativos en gel hibridación 3
Principio (Figura 1): En condiciones nativas, una final de la etiqueta (CCCTAA) 4 (TELC) oligo sonda sólo puede hibridar con el telómero de cadena simple G-pendiente región. La intensidad de la hibridación es proporcional a la longitud del voladizo. Después de la desnaturalización y neutralización, la misma sonda será capaz de hibridar con la región de los telómeros conjunto. La intensidad de la hibridación representa la cantidad total de ADN y los telómeros se puede utilizar como control de carga. Dos controles estándar para este experimento es la hibridación con final de la etiqueta (TTAGGG) 4 (TELG) oligo sonda, que no debe producir ninguna señal como T. celular brucei no tiene los telómeros C-voladizo estructura, y para tratar el ADN genómico con 3'-específica de cadena simple exonucleasas como Exo I o T Exo antes de la hibridación, que debe eliminar la señal nativa de hibridación TELC.
Paso 1. Prepare los tapones de ADN para la electroforesis en campo pulsado (PFGE)
Cromosomas intactos serán separados por PFGE. Por lo tanto, el ADN genómico se preparó como los tapones de ADN.
- Comience con 100 ml de sangre se forman células (en 1,5 -2 millones de células / ml) o 20 ml de células procíclico (en 10 millones de células / ml).
- Disolver 1,6% de bajo punto de fusión (LMP) de agarosa en tampón L (0,01 M Tris • CL pH 7,6, 0,02 M NaCl, 0,1 M EDTA pH 8.0) y mantenerlo caliente a 50 ° C.
- Las células por centrifugación de la cosecha de 1,5 krpm, 4 ° C durante 10 min. Aspirar el sobrenadante y resuspender el sedimento celular con tampón L a una concentración final de 5 millones de células / ml.
- Incubar la suspensión de células a 42-50 ° C durante 10 min.
- Añadir un volumen de LMP agarosa a 1 volumen de las células. Mezclar bien, pero rápidamente.
- Alícuota de 85 l de la suspensión celular a cada pocillo de un molde de plug desechables (BioRad).
- Después de 5-10 minutos o hasta que los tapones se solidifican, se conecta a un tubo de transferencia cónico de 15 ml con 5 ml de tampón L / 1% Sarkosyl. Añadir 100 ml de 250 mg / ml de proteinasa K o una pizca de proteinasa K en polvo, mezclar bien e incubar a 50 ° C durante 48 horas.
- Deseche el tampón, lavar los tapones con 5 ml de solución tampón L dos veces y repetir la digestión de proteinasa K durante 48 horas. Lave los tapones con tampón L fresco otra vez. Tienda de tapones a 4 ° C en tampón L. Estos tapones se debe utilizar dentro de 2 semanas.
Paso 2. Separados intacta T. brucei cromosomas mediante electroforesis en campo pulsado
- Preparar un gel de agarosa
- Preparar 3.2 L de 0,5 x TBE buffer y la transferencia a la cámara de ejecución de un gel CHEF DRII (BioRad) la unidad. Ajuste la temperatura de funcionamiento, en marcha la bomba, a continuación, iniciar la unidad de refrigeración.
- Preparar 100 ml de un 1,2% en gel de agarosa en 0,5 x TBE. Enfriar la agarosa, mientras que la creación de la bandeja del gel.
- Decidir el orden de la muestra. Recuerde que debe incluir el nivel de ADN. Seque el peine y lo pondré sobre una superficie plana. Colocar cada enchufe de ADN en el diente correspondiente. Quitar exceso de buffer alrededor del tapón de ADN por la aspiración de modo que el enchufe se adhieren a los dientes.
- Verter el gel de agarosa sin insertar el peine. Dejar enfriar a 40-50 ° C (unos minutos), introduzca lentamente el peine con los enchufes conectados en el gel. Asegúrese de que los enchufes no resbale el peine. Deje que el gel se solidifique. Quitar peine de gel lentamente.
- Campo pulsado electroforesis en gel
Corra el gel en una unidad CHEF DRII: 700 s-1500 s legumbres, 2,5 V / cm, a los 12 ° C durante 120 horas.
Paso 3. Mancha y destain el gel de agarosa
Tinción del gel con 1 g / ml de bromuro de etidio en 0,5 x TBE a temperatura ambiente durante 1 hora después en 0,5 x TBE sin bromuro de etidio durante 1 hora con suave balanceo. Tome una fotografía del gel.
Paso 4. Secar el gel de agarosa
El gel de agarosa en dos capas de 3 mm de papel Whatman y cubrir el gel con un envoltorio plástico. Secar el gel a temperatura ambiente (el gel no debe ser calentado a más de 50 ° C para evitar la desnaturalización de las muestras de ADN). Vuelva a colocar el mojado papeles Whatman con los secos después de 2 y 4 horas de secado, respectivamente. Para secar hasta que el gel está completamente seca. Esto puede tomar durante la noche dependiendo de la secadora.
Paso 5. Etiqueta de las sondas oligo
Mezcle 1 l de 50 ng / mL TELC u oligo TELG con 1 L de 10 x polinucleótido quinasa (PNK) de amortiguación, 5 l de γ [32P] ATP, 2 l de ddH2O, y 1 l de T4 PNK. Se incuba a 37 ° C durante 1 hora. Añadir 90 l de las empresas transnacionales buffer (10 mM Tris Cl, pH 8,0, 100 mM NaCl, 10 mM EDTA, pH 8,0, SDS al 1%) a la mezcla de reacción.
Mientras que el etiquetado del oligoes, preparar una columna G-25: poner un poco de lana de vidrio en una jeringa de 3 cc y cosas que hacia abajo con fuerza, con una punta de la pipeta. Llene la jeringa con Sephadex bien G-25 (autoclAVED en TE) para la marca de 3 ml. Dejar reposar en peso. Para purificar la sonda marcada: la carga de la sonda en la parte superior de la columna. Lavar con 700 l de buffer de las empresas transnacionales, y eluir con 600 l de buffer de las empresas transnacionales. Por otra parte, purifican la sonda marcada con un paseo rápido mini ™ columna (Roche Applied Science).
Paso 6: La hibridación
- En pocas palabras húmedo del gel seco en agua destilada y lavar cualquier tipo de papel Whatman atrapado en el gel
- Colocar el gel en una bolsa de hibridación y agregar 20 ml de solución tampón hybridiztion (0,25 M Na 2 HPO 4 pH 7.2, 1 mM EDTA, SDS al 7%, 1% BSA, filtrada a través de 0,22 micras filtro). Incubar en un baño de agua a 50 ° C con suave balanceo por lo menos 1 hora.
- Retire el buffer de pre-hibridación. Hierva la sonda marcada durante 5 minutos y añadir a 25 ml de tampón de hibridación fresca, filtro de esterilizar la mezcla con un filtro de 0,22 micras jeringa y agregue la mezcla de hibridación directamente a la bolsa de hibridación. Selle la bolsa y colóquela en un recipiente poco profundo con agua tibia. Coloque el recipiente entero en el baño de agua. Se incuba a 50 ° C durante la noche con suave balanceo.
- Un pequeño corte en la bolsa de hibridación. Vierta la medida de lo posible la mezcla de hibridación y lo guarda en un tubo cónico de 50 ml. Mantenga la sonda congeladas a -20 ° C. Quitar el gel de la bolsa de hibridación y lo puso en un recipiente.
En este paso todo va a estar caliente y necesita una limpieza extensiva (BANCO, PANTALLA, tijeras, etc. Asegúrese de usar capas dobles de GUANTES! - Lavar el gel en 4 x SSC durante 30 minutos a 50 ° C. Vuelva a colocar con tampón de lavado fresco y repetir dos veces.
- Lave la gel en 4 x SSC/0.1% SDS a 50 ° C.
- Poner el gel en un pedazo de papel Whatman de 3 mm y ligeramente seco el gel.
- Envuelva el gel con papel plástico y exponer a PhosphorImager de ~ 3 días
- Busque en la PhosphorImager
- Incubar el gel en tampón de desnaturalización (1,5 M NaCl, 0,5 M NaOH) a temperatura ambiente durante 30 minutos
- Lavar el gel en tampón de neutralización (3 M NaCl, 0,5 M Tris • / Cl pH 7,0) a temperatura ambiente durante 30 minutos
- Enjuague con agua destilada durante 3 minutos
- Pre-hibridación a 55 ° C durante 1 hora
- Repita la hibridación con la sonda que contienen misma mezcla de hibridación a 55 ° C durante la noche.
- Lavar como se describe arriba, excepto a 55 ° C.
- Envuelva el gel y exponerlo a PhosphorImager de ~ 2 hrs.
- Busque en la phosphorimager. Normalizar la señal de hibridación obtenida antes de la desnaturalización de la que se obtiene después de la desnaturalización.
2. Determinar el telómero terminal de nucleótidos de la ligadura de adaptador de 6
Principio (Figura 2A y 2B): A 5 'final de la etiqueta de oligonucleótidos único se liga a los 3' finales del G-alero. Esto se logra mediante el recocido de oligo específico guía complementaria. Sólo la única guía / oligo secuencias teniendo adaptador compatible con las secuencias de telómero terminal G-alero se ligan. Por T. brucei, los telómeros puede terminar en uno de los seis nucleótidos diferentes de las repeticiones TTAGGG. Por lo tanto, seis oligoes guía se utilizan diferentes (TG1-TG6, Figura 2A). Después de la ligadura, el ADN es digerido con endonucleasas de restricción y se resolvieron en un gel de agarosa.
- Quinasa tratamiento de los oligo único.
Mix 6 l de 10 pmol / l oligo única con 3 l de tampón PNK 10x, 5 l de γ [32 P] ATP, 14 l de ddH2O, y 2 l (10 U) de la T4 PNK. Incubar la mezcla a 37 ° C durante 1 hora. - Quitar incorporadas caliente nucleótidos.
Utilice el equipo de Qiaquick eliminación de nucleótidos para eliminar el calor no incorporadas ATP y eluir los oligo final de la etiqueta con 60 l de tampón de elución (concentración final sería de aproximadamente 1 pmol / l). - Recocido oligo única guía de oligo (para crear el adaptador).
Añadir 10 l de agua purificada oligo única de 2 l de cada oligo guía y un buffer de l recocido (200 mM NaCl, 100 mM Tris-HCl pH 8,0). Poner los tubos en un bloque de 85 ° C de calor y apagar el bloque de calor. Deje que los tubos y el bloque de calor frío a temperatura ambiente. Esto tomará un par de horas. Si en un tubo de transferencia de prisa, después de 5 minutos cada uno a 65 ° C, entonces de 37 ° C y luego dejar enfriar a temperatura ambiente. - Adaptadores de ligar a un total de ADN genómico.
Y 2,5 l de ADN genómico intacto, añadir 4 l de 5 x tampón ligasa, 10 l de oligos recocido, 2,5 l de ddH2O, y 1 l de ADN ligasa de T4. Se incuba a 16 ° C durante la noche. - Restricción de la digestión con endonucleasa
A 20 L de producto de ligación añadir 3 L de 10 x tampón NEB 4, 1,5 l de AluI y Mboi cada uno, y 4 l de ddH 2 O. Incubar la mezcla a 37 ° C durante la noche. - Electroforesis.
Añadir 3 L de 10 x Orange G colorante (50% de glicerol, 0,5% de Orange G) para cada muestra. Cargar las muestras de ADN en un 20X20 cm 0,7% en gel de agarosa en 0,5 x TBE con 0,2 mg / ml de bromuro de etidio. Funcionar a 30V durante 30 minutos, entonces continue con un voltaje más alto, pero menos de 120 de un total de 1000 v hr. Tome una fotografía del gel con una regla al lado del marcador de ADN. - Gel seco y exponer.
Poner el gel de agarosa en una capa de papel de filtro DE81 y dos capas de 3 mm de papel Whatman y cubrir el gel con un envoltorio plástico. Gel seco a temperatura ambiente. Exponer el gel seco para PhosphorImager noche a la mañana.
3. Mediada la ligadura de extensión del cebador 6
Principio (Figura 2A y 2C): Un único oligonucleótido se liga a los 3 'G-pendiente con la secuencia terminal específico, determinado por sus secuencias complementarias en el adaptador (guía) oligo. Después de la purificación, la guía de oligo marcado que sigue siendo templado a la oligo G-overhang/unique imprimación es extendido a la unión ss-ds con T4 ADN polimerasa, que carece de desplazamiento de cadena y 5'-3 'exonucleasa actividades. Los productos de extensión del cebador por lo tanto, dar la longitud de la G-alero.
- Quinasa tratamiento de la oligoes guía y único
- Mix 20 L de 10 pmol / l oligo única con 10 l de buffer de 10 x PNK, 20 l 5 x tampón de ligasa (que contiene 10 mM ATP), 3 l (30 U) de la T4 PNK, y 47 L de ddH2O . Incubar la mezcla a 37 ° C durante 60 minutos.
- Mezcle 1 l de 10 pmol / l con una guía de oligo L de 10 x tampón PNK, 2 l de γ [32 P] ATP, 1 l (10 U) de la T4 PNK, y 5 l de ddH 2 O. Incubar la mezcla a 37 ° C durante 60 minutos.
- Recocido guía oligo y oligo única
Añadir 10 l (20 pmoles) de los oligo fosforilada única y 10 l (10 pmoles) de la final de la etiqueta oligo guía en un tubo Eppendorf de 1,5 ml (la proporción de 2:1 de guía única de oligo asegura que todos los oligo guía es recocido ). Siga el mismo procedimiento descrito en el protocolo II de recocido oligoes. - Ligar la guía de recocido / adaptador oligo única a los fines del telómero
Siga el mismo procedimiento descrito en el Protocolo II de la ligadura de ADN. - Precipitado de ADN con un volumen de 1 / 10 de acetato de sodio y 2,5 volumen de helado de etanol a -80 ° C durante 30 min. Girar el ADN precipitado en 12 krpm, 4 ° C durante 15 min. Lavar el ADN con el 70% de etanol y se disuelven en la paleta de 10 l ddH 2 O.
- Primer extensión
Para cada muestra de ADN 10 l agregar 2 L de 10 x tampón T4 ADN polimerasa, 1 l de 1 mg / ml de BSA, 1 mM dATP L de 25, dCTP, dTTP y cada uno, 1 l (3 U) de la ADN polimerasa T4, y 3 l de ddH 2 O.
Haga una mezcla maestra con la polimerasa, buffer, etc y la cantidad alícuota de la mezcla maestra para cada muestra de ADN. Se incuba a 30 ° C durante 30 min. A continuación, añadir 1,2 l de EDTA 0,5 M de inmediato a la reacción. - Electroforesis
Ejecutar los productos de extensión en un 10% de acrilamida / 7 gel M urea/1xTBE. Carga de 4 l de cada muestra. Hervir las muestras durante 10 minutos antes de la carga. Funcionar a 800V en 1x TBE hasta que el colorante azul llega a la parte inferior del gel. Secar el gel a 65 ° C durante 30 minutos después a temperatura ambiente durante 30 minutos y exponer a PhosphorImager durante 2 horas.
Para preparar 100 ml de gel de poliacrilamida al 10%, se disuelven 41 g de urea y 21 ml de solución de acrilamida al 40% (acrilamida / bis-acrilamida 29:1) en 30 ml de agua destilada, agregar 10 ml de TBE 10x. Montar las placas de cristal. Justo antes de verter el gel, agregar 1 ml de 10% de APS y 100 l de TEMED. Verter el gel y tratar de evitar las burbujas.
4. Los resultados representativos:
Detectar T. brucei telómeros G-alero con estructura nativa en gel de hibridación
Intacta T. brucei cromosomas separados por PFGE se muestran en la Figura 3A y 3B (paneles de la izquierda). T. brucei células normalmente contienen aproximadamente 100 copias de minichromosomes, todos ellos con un tamaño similar (50 a 150 kb) y migrar a la misma posición en el gel (MC). Debido a este hecho, después de la hibridación con sondas oligo TELC, el telómero G-pendiente de la señal es más prominente en minichromosomes (Figura 3A, panel central). Hibridación con sondas oligo TELG normalmente no da ninguna señal de hibridación (Figura 3B, panel central), ya que T. brucei células no tienen los telómeros C-voladizo estructura. Después de la desnaturalización y neutralización, la hibridación, ya sea con o TELC TELG oligo sonda debe revelar señales de los telómeros en todos los extremos de los cromosomas (Figura 3A y 3B paneles a la derecha).
Determinar el telómero terminal de nucleótidos de la ligadura de adaptador
Carga la misma cantidad de ADN en cada carril es fundamental para este ensayo, y esto se demuestra por tinción con bromuro de etidio del gel. Un ejemplo se muestra en la figura 4A, panel de la izquierda.
En este protocolo, el final de la etiqueta única oligo y guía de adaptadores oligo sólo ligado a extremo del cromosoma, cuando el telómero G-voladizos que termina con las secuencias que son compatibles con el oligo guía. Si na vez que el oligo único es final de la etiqueta, el producto de ligación se radiactivos y dar una señal fuerte. Como se muestra en la figura 4A, panel de la derecha, carril 1 da una fuerte señal, que indica que una gran cantidad de unique/TG1 adaptador ha sido ligada a la final del telómero. TG1 tiene una secuencia de final de 5'CCCTAA3. Por lo tanto, el telómero G-salientes ligados a este extremo del adaptador en 5'TTAGGG3. Ninguna cantidad significativa de productos de la ligadura se observa con otros oligoes guía, lo que indica que los telómeros que termina en TTAGGG son predominantes en T. brucei células.
El tratamiento de la ADN genómico con Exo I o T Exo, que son las nucleasas específicas voladizo 3 ', resultó en la pérdida de la ligadura de los productos (Figura 4, panel de la derecha, las calles 2 y 3), lo que indica que la ligadura de hecho el resultado de los telómeros G- voladizo estructura
Mediada la ligadura de extensión del cebador
Sin ligadura, el final de la etiqueta guía oligo es de 22 nt de longitud. Carga final de la etiqueta oligo guía servirá como un control negativo y un marcador de tamaño. (Figura 4, calle 11, el asterisco indica el final de la etiqueta guía oligo). Por lo general también se observa una banda de ~ 44 nt en este control negativo (Figura 4, calle 11, triángulo abierto), que tienen más probabilidades de residuos no desnaturalizado adaptadores. Después de la ligadura de el extremo del cromosoma, el tamaño del producto ampliada refleja la longitud de la estructura de G-alero. La mayoría de T. brucei telómeros G-voladizos fin en 5'TTAGGG3 "(Figura 4). Sin embargo, estos G-salientes parecen ser muy corta (sólo ~ 10 nt de longitud), ya que los productos se extendía desde la liga oligo TG1 guía no son mucho más larga que la guía se oligo (Figura 4, carril 10), pero permiten que la ligadura de TG1 adaptador (Figura 4, panel de la derecha, carril 1). Aunque sólo una pequeña cantidad de TG4 adaptador se ligó a los extremos de los telómeros (Figura 4, panel de la derecha, carril 6), los productos se extendió desde TG4 liga son mucho más largos (Figura 4, calle 7, puntas de flecha), con el mayor producto que se ~ 55 nt. Por lo tanto, observamos dos tipos de los telómeros en voladizo G-T. brucei células. El predominio del telómero G-voladizos fin en 5'TTAGGG3 ', pero son sólo ~ 10 nt de longitud. Pocos telómeros G-voladizos fin en 5'GGGTTA3 ', pero puede ser hasta 40 nt de longitud. Ambos tipos de G-saliente son sensibles a Exo T (o Exo I) el tratamiento (Figura 4, panel de la derecha, el carril 2, 3 y 7; la Figura 4B, carril 1, flechas).

Figura 1. Principio de nativos en gel de hibridación. Izquierda, bajo la condición de nativo, el final de la etiqueta TELC oligo sonda sólo se pueden hibridar con la sola hebra G-ricos voladizo telómeros. La intensidad de la hibridación refleja la longitud de los telómeros G-alero. Derecho, después de la desnaturalización, la TELC oligo sonda se hibridan con todos los ADN de los telómeros. La intensidad de esta hibridación representa la cantidad total de ADN de los telómeros y se utiliza como control de carga, y el final del G-alero nivel se calcula dividiendo la señal de hibridación nativos por la que se obtiene después de la desnaturalización.
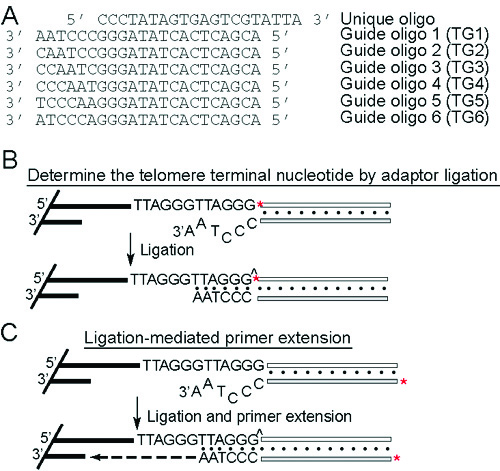
Figura 2. Principio de la ligadura de conexión y extensión de la ligadura de mediados de imprimación. (A) Secuencias de la oligoes guía única y seis. (B) Determinar el telómero terminal de nucleótidos de la ligadura de adaptador. El oligo único es final de la etiqueta (marcados con un asterisco rojo), recocido a la oligoes guía y se ligó con el final del telómero. Sólo cuando el único adaptador / guía tiene pendiente un 3 'que es compatible con la secuencia del telómero terminal del adaptador de ser ligado. (C) En primer extensión ligadura mediada, el oligo guía final de la etiqueta (marcados con un asterisco rojo) y templado a la oligo única antes ligado al cromosoma termina. Sólo el único adaptador / cojinete guía saliente compatible con el G-saliente 3 'se ligan. ^ Indica el vínculo phophordiester liga. Después de la eliminación de los pares de oligo no ligado, primer extensión se llevará a cabo con la ADN polimerasa T4, que carece de desplazamiento de cadena y 5'-3 'exonucleasa actividades. La longitud final de la guía extendida oligo tanto, de la longitud de la G-alero.
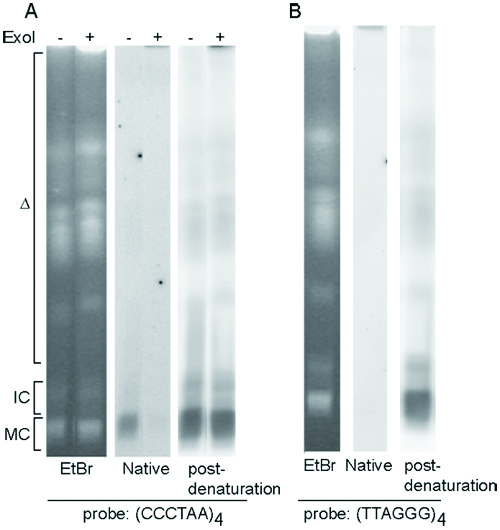
Figura 3. T. brucei telómeros G-voladizo estructura analizada por los nativos en gel de hibridación. (A) En-gel de la hibridación con sondas oligo TELC. (B) En-gel de la hibridación con sondas oligo TELG. En ambos (A) y (B) PFG izquierda, bromuro de Ethedium manchado. Media, resultado de hibridación nativos. Derecho, después de la desnaturalización resultado de la hibridación. De tipo salvaje T. Las células se utilizaron brucei. Intacto o Exo he tratado de ADN genómico fueron separados por PFGE. Triángulo abierto representa los cromosomas megabase, IC, intermedio cromosomas de tamaño, MC, minichromosomes.
"/>
Figura 4. T. brucei telómeros G-voladizo estructura analizada por la ligadura mediada por extensión del cebador. (A) La mayoría de T. brucei telómeros G-salientes terminan en 5'TTAGGG3. Izquierda, el bromuro de Ethedium gel de ADN manchado. Cantidad aproximadamente igual de ADN está cargado en cada carril. Derecho, resultado de la exposición del mismo gel, después del secado. Intacto, Exo I-tratados, o Exo T tratados con ADN se ligó a seis diferentes finales etiquetados única guía / adaptadores oligo como se indica. (B) T. brucei telómeros se corta G-salientes. Intacto o Exo T tratados con ADN genómico se ligó a seis diferentes única guía / adaptadores oligo seguido por extensión del cebador utilizando ADN polimerasa T4. Final de la etiqueta oligo TG4 se ha cargado como un control negativo (calle 11). El propio oligo funciona a 22 nt (*) sino que también dio una señal más débil en el ~ 44 nt (Δ). Sólo unique/TG4 adaptador producido productos de la extensión significativamente mayor que el oligo guía a sí mismo (puntas de flecha, carril 7).
Discusión
Tripanosoma brucei causa la enfermedad del sueño en los seres humanos. Esta enfermedad, si no se trata, es inevitablemente fatal. T. células brucei en huéspedes mamíferos sufren variación antigénica con regularidad con el fin de evadir el ataque inmune del huésped 7. Por lo tanto, es muy difícil de eliminar T. brucei células una vez que la infección se ha establecido. Recientemente hemos demostrado que los telómeros desempeñan un papel importante en la regulaci?...
Divulgaciones
Agradecimientos
Nos gustaría agradecer a la Dra. Carolyn Precio de los debates científicos y sugerencias perspicaces. Este trabajo es apoyado por el NIH subvención AI066095 (PI: Bibo Li).
Materiales
| Name | Company | Catalog Number | Comments |
| SeaKem LE Agarose | Lonza Inc. | 50004 | |
| Agarose Type VII (low melting agarose) | Sigma-Aldrich | A4018 | |
| Proteinase K, recombinant, PCR grade | Roche Group | 03 115801001 | |
| Exo I | New England Biolabs | M0293 | |
| Exo T | New England Biolabs | M0265 | |
| T7 exonuclease | New England Biolabs | M0263 | |
| T4 DNA polymerase | New England Biolabs | M0203 | |
| QIAquick nucleotide removal kit | Qiagen | 28304 |
Referencias
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados