É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Exame da estrutura G-Telomere saliência na Trypanosoma brucei
Neste Artigo
Resumo
Os telômeros são essenciais para a estabilidade cromossômica ea estrutura G-saliência dos telômeros é essencial para a telomerase mediada manutenção dos telômeros. Recentemente, aprovou dois métodos para detectar a estrutura G-saliência dos telômeros em Trypanosoma brucei, Que são nativas em gel-hibridação e ligadura mediada por extensão de primer, que serão descritas.
Resumo
O telômero estrutura G-saliência tem sido identificada em eucariotos, incluindo muitos fungos, vertebrados, e Trypanosoma brucei. Serve como substrato para telomerase para a síntese de DNA de novo do telômero e, portanto, importante para a manutenção dos telômeros. T. brucei é um parasita protozoário que causa a doença do sono em humanos e nagana em bovinos. Uma vez infectado hospedeiro mamífero, T. célula brucei regularmente alterna a sua antígeno de superfície para escapar ataque do sistema imunológico do hospedeiro. Temos recentemente demonstraram que o T. brucei estrutura dos telômeros desempenha um papel essencial na regulação da expressão gênica do antígeno de superfície, que é crítico para T. patogênese brucei. No entanto, T. brucei estrutura dos telômeros não tem sido extensivamente estudada devido à limitação de métodos de análise dessa estrutura especializada. Temos agora adotada com sucesso o nativo em gel hibridação e mediada por ligadura métodos cartilha de extensão para exame da estrutura G-saliência dos telômeros e um método de ligadura adaptador para determinação do terminal de nucleotídeos dos telômeros em T. células brucei. Aqui, vamos descrever os protocolos em detalhe e comparar suas vantagens e limitações.
Protocolo
1. Detectar T. brucei Telomere G saliência usando Native Hibridização In-gel 3
Princípio (Figura 1): Em condições nativas, um fim-rotulados (CCCTAA) 4 (TELC) oligo sonda só pode hibridizar o telômero single-stranded G-saliência região. A intensidade de hibridização é proporcional ao comprimento do balanço. Após desnaturação e neutralização, a mesma sonda será capaz de hibridizar para a região dos telômeros todo. A intensidade de hibridização representa a quantidade total de DNA do telômero e pode ser usado como um controle de carga. Dois controles padrão para este experimento são hibridação com final marcado (TTAGGG) 4 (TELG) oligo sonda, que não deve produzir qualquer sinal como T. célula brucei não tem estrutura C-saliência dos telômeros, e para o tratamento do DNA genômico com 3'-específicas single-stranded exonucleases como Exo I ou T Exo antes da hibridação, que deve eliminar o sinal de hibridização nativa TELC.
Etapa 1. Prepare fichas de DNA Eletroforese em campo pulsado Gel (PFGE)
Cromossomos intactos serão separados por PFGE. Portanto, o DNA genômico é preparado como plugs DNA.
- Comece com 100 mL de células formam corrente sangüínea (menos 1,5 -2000000 células / mL) ou 20 mL de células procíclicos (menos 10 milhões de células / mL).
- Dissolver 1,6% de baixo ponto de fusão (LMP) agarose em tampão L (0,01 M Tris • CL pH 7,6, 0,02 M NaCl, 0,1 M EDTA pH 8.0) e mantê-lo aquecido a 50 ° C.
- Células colheita por centrifugação a 1,5 krpm, 4 ° C por 10 min. Remover o sobrenadante e ressuspender pellet celular com L buffer para uma concentração final de 5 milhões de células / mL.
- Incubar a suspensão celular em 42-50 ° C por 10 min.
- Adicionar um volume de agarose LMP para 1 volume de células. Misture bem, mas rapidamente.
- ML alíquota 85 de suspensão de células em cada poço de um molde ficha descartáveis (BioRad).
- Após 5-10 minutos ou até que as fichas estão solidificadas, fichas de transferência em um tubo cônico de 15 mL com 5 mL de buffer L / Sarkosyl 1%. Adicionar 100 mL de 250 mg / mL proteinase K ou uma pitada de proteinase K em pó, misture bem e incubar a 50 ° C por 48 horas.
- Descarte o tampão, lave plugs com 5 mL de tampão L duas vezes e repita proteinase K digestão por mais 48 hrs. Lavar com tampão plugs L fresca novamente. Loja plugs a 4 ° C em tampão L. Estas fichas devem ser usadas dentro de 2 semanas.
Etapa 2. Separe T. intacta cromossomos brucei usando Eletroforese em Gel de Campo Pulsado
- Prepare gel de agarose
- Prepare 2-3 L de 0,5 x TBE e transferência para a câmara de execução de um gel DRII CHEF unidade (BioRad). Ajustar a temperatura em execução, inicie a bomba, então comece a unidade de refrigeração.
- Prepare 100 mL de um gel de agarose 1,2% em tampão TBE 0,5 x. Arrefecer a agarose durante a configuração do tabuleiro de gel.
- Decidir a ordem da amostra. Lembre-se de incluir o padrão de DNA. Secar o pente e colocá-la sobre uma superfície plana. Coloque cada vela de DNA para o dente apropriado. Remover tampão excessiva em torno do plugue DNA por aspiração para a ficha vai ficar com o dente.
- Despeje o gel agarose sem inserir o pente. Deixe esfriar até 40-50 ° C (alguns minutos), introduza lentamente o pente com as fichas em anexo para o gel. Certifique-se que se conecta não deslizar o pente. Deixe o gel solidificar. Remover pente de gel lentamente.
- Eletroforese em Gel de Campo Pulsado
Executar gel em uma unidade DRII CHEF: 700 s-1500 s pulsos de 2,5 V / cm, a 12 ° C por 120 horas.
Etapa 3. Mancha e destain o gel agarose
Manchar o gel com 1μg / mL de brometo de etídio em 0,5 x TBE na RT para 1 hr depois em 0,5 x TBE sem Brometo de etídio por 1 h com balanço delicado. Tire uma foto do gel.
Etapa 4. Secar o gel agarose
Coloque o gel agarose em duas camadas de 3 milímetros de papel Whatman e cobrir o gel com filme plástico. Secar o gel em temperatura ambiente (o gel não deve ser aquecida mais de 50 ° C para evitar a desnaturação das amostras de DNA). Substituir os papéis molhados Whatman com os secar depois de 2 e 4 horas de secagem, respectivamente. Mantenha a secagem até que o gel é completamente seco. Isso pode levar a noite dependendo do secador.
O passo 5. Rótulo as sondas oligo
Misture 1 mL de 50 TELC ng / mL ou oligo TELG com 1 mL de 10 Polinucleotídeo tampão quinase x (PNK), 5 mL de γ [32P] ATP, 2 mL de DDH 2 O, e 1 mL de PNK T4. Incubar a 37 ° C por 1 hora. Adicionar 90 mL de ETNs buffer (10 mM Tris Cl, pH 8,0, 100 mM NaCl, 10 mM EDTA, pH 8,0, 1% SDS) para a mistura de reação.
Enquanto a rotulagem oligoes, prepare um G-25 coluna: colocar um pouco de lã de vidro para uma seringa cc 3 e enchê-lo para baixo firmemente com uma ponteira. Encha a seringa com Sephadex G-25 fine (autoclAVED em TE) para a 3 mL marca. Deixe repousar por peso. Para purificar a sonda marcada: carga a sonda em cima da coluna. Lavar com 700 mL de tampão ETNs, e eluir com 600 mL de tampão ETNs. Alternativamente, purificar a sonda marcada usando um mini-rotação rápida ™ coluna (Roche Applied Science).
Passo 6: Hibridização
- Brevemente molhado do gel seco em água destilada e lavar qualquer papel Whatman presos no gel
- Coloque o gel dentro de um saco de hibridização e adicionar 20 mL de tampão hybridiztion (0.25m Na 2 HPO 4 pH7.2; 1 mM EDTA, 7% SDS; 1% BSA, 0,22 mM filtrada através de filtro). Incubar em banho-maria a 50 ° C com balanço suave durante pelo menos 1 hr.
- Remova o buffer de pré-hibridação. Ferva a sonda marcada por 5 min e adicioná-lo a 25 mL de tampão de hibridação fresco, filtro de esterilizar a mistura com um filtro de seringa de 0,22 mm e adicione a mistura de hibridização diretamente para o saco de hibridação. Feche o saco e coloque em um recipiente raso com água morna. Coloque o recipiente inteiro no banho-maria. Incubar a 50 ° C durante a noite com balanço delicado.
- Cortar uma pequena abertura no saco de hibridação. Derramarei o tanto quanto possível a mistura de hibridização e salve-o em um tubo de 50 ml. Manter a sonda congeladas a -20 ° C. Remova o gel do saco de hibridização e colocá-lo em um recipiente.
Nesta etapa TUDO será quente e precisa de limpeza EXTENSIVO (BENCH, SCREEN, tesouras, ETC. Certificar-se desgastar duas camadas de luvas! - Lave o gel em 4 x SSC por 30 min a 50 ° C. Substituir com tampão de lavagem novo e repita duas vezes.
- Lave o gel em 4 x SDS SSC/0.1% a 50 ° C.
- Coloque o gel em um pedaço de papel Whatman 3 mm e levemente seca o gel.
- Embrulhe o gel com filme plástico e expor a phosphorimager para ~ 3 dias
- Digitalizar o phosphorimager
- Incubar o gel em tampão de desnaturação (1,5 M NaCl, 0,5 M NaOH) em temperatura ambiente por 30 min
- Lave o gel em tampão de neutralização (3 M NaCl, 0,5 M Tris • / Cl pH 7,0) em temperatura ambiente por 30 min
- Enxágüe em água destilada por 3 min
- Pré-hibridizar a 55 ° C por 1 hr
- Repita a hibridização utilizando a sonda contendo mesmo mix de hibridação a 55 ° C durante a noite.
- Lave como descrito acima, exceto a 55 ° C.
- Embrulhe o gel e exponha ao phosphorimager por aproximadamente 2 horas.
- Digitalizar o phosphorimager. Normalizar o sinal de hibridização obtidos antes desnaturação com o obtido após desnaturação.
2. Determinar o Terminal Telomere Nucleotide por Ligadura Adaptor 6
Princípio (Figura 2A e 2B): A 5 'end-rotulados oligonucleotídeo única é ligada à extremidade 3' do G-beiral. Isto é conseguido através de recozimento para oligo guia específico complementares. Apenas os únicos / guia oligo seqüências adaptador tendo compatível com o telômero seqüências G saliência-terminal vai ser ligada. Para T. brucei, os telômeros pode terminar em uma das seis nucleotídeos diferentes do TTAGGG repete. Daí seis oligoes guia diferentes são usados (TG1-TG6, Figura 2A). Após a ligadura, o DNA é digerido com endonucleases de restrição e resolvidos em gel de agarose.
- Tratamento quinase do oligo único.
Mix 6 mL de 10 pmole / mL oligo única com 3 mL de tampão PNK 10x, 5 mL de γ [32 P] ATP, 14 mL de DDH 2 O e 2 mL (10 U) do PNK T4. Incubar a mistura a 37 ° C por 1 hora. - Remover unincorporated nucleotídeo quente.
Use QIAquick kit remoção de nucleotídeos para remover o unincorporated quente ATP e eluir os oligo-end rotulados com 60 mL de tampão de eluição (concentração final seria pmole ~ 1 / mL). - Recozer oligo única para guiar oligo (para criar o adaptador).
Adicionar 10 mL de purificada oligo única de 2 mL de cada oligo guia e um tampão mL de recozimento (200 mM NaCl, 100 mM Tris-HCl pH 8,0). Coloque os tubos em um bloco de aquecimento 85 ° C e desligue o bloco de aquecimento. Deixe os tubos e bloco de aquecimento legal RT. Isso levará algumas horas. Se em um tubo de transferência de pressa, depois de 5 minutos cada a 65 ° C, então 37 ° C e, em seguida, deixar arrefecer à RT. - Adaptadores de ligadura ao total de DNA genômico.
Para 2,5 mL de DNA genômico intacto, adicionar 4 mL de 5 x Ligase tampão, 10 mL de oligos recozido, 2,5 mL de DDH 2 O, e 1 mL de DNA Ligase T4. Incubar a 16 ° C durante a noite. - Digestão endonuclease de restrição
A 20 mL do produto ligadura adicionar 3 mL de tampão NEB 10 x 4, 1,5 mL de AluI e MboI cada, e 4 mL de DDH 2 O. Incubar a mistura a 37 ° C durante a noite. - Eletroforese.
Adicionar 3 mL de 10 x Laranja G corante (glicerol 50%, 0,5% Laranja G) para cada amostra. Carregar a amostras de DNA em uma 20x20 cm 0,7% agarose gel em 0,5 x TBE com 0,2 mg / mL de brometo de etídio. Executar a 30V por 30 min, em seguida, continue com tensão mais alta, mas menos de 120 para um total de 1000 v hr. Tire uma foto do gel com uma régua ao lado do marcador de DNA. - Gel seco e expor.
Coloque o gel agarose sobre uma camada de papel de filtro DE81 e duas camadas de papel Whatman 3 mm e cobrir o gel com filme plástico. Gel seco a RT. Expõe o gel seco para phosphorimager durante a noite.
3. Ligadura Extensão Primer Mediated 6
Princípio (Figura 2A e 2C): A única oligonucleotídeo é ligada aos 3 'G-saliência com seqüência de terminal específico, determinado por suas seqüências complementares no adaptador (guia) oligo. Após purificação, o oligo guia rotulada que permanece recozido para a oligo G-overhang/unique cartilha é estendido para a junção ss-ds usando DNA polimerase T4, que carece de deslocamento vertente e atividades 5'-3 'exonuclease. Os produtos de extensão de primer, portanto, dar o comprimento do G-beiral.
- Tratamento quinase do oligoes guia e único
- Misture 20 mL de 10 pmole / oligo única mL com 10 mL de 10 x PNK tampão, 20 mL 5 x tampão ligase (que contém 10 mM ATP), 3 mL (30 U) do PNK T4, e 47 mL de DDH 2 O . Incubar a mistura a 37 ° C por 60 minutos.
- Misture 1 mL de 10 pmole / mL guia oligo com 1 mL de 10 x PNK buffer, 2 mL de γ [32 P] ATP, 1 mL (10 U) de PNK T4, e 5 mL de DDH 2 O. Incubar a mistura a 37 ° C por 60 minutos.
- Recozer guia oligo e único oligo
Adicionar 10 mL (20 pmole) do oligo fosforilada único e 10 L (10 pmole) do fim-rotulados guia oligo em um tubo Eppendorf 1,5 mL (a proporção de 2:1 de única para guiar oligo garante que todos os oligo guia é recozido ). Siga o mesmo procedimento descrito no protocolo II para o recozimento oligoes. - Ligadura o guia recozido / adaptador oligo única para fins telomere
Siga o mesmo procedimento descrito no protocolo II para ligadura DNA. - DNA precipitado com volume de 1 / 10 de acetato de sódio e 2,5 volume de etanol gelado a -80 ° C por 30 min. Spin down DNA pellet em 12 krpm, 4 ° C por 15 min. Lavar DNA com 70% de etanol e dissolver em 10 mL de paletes de DDH 2 O.
- Extensão de primer
Para cada amostra de DNA de 10 L, adicionar 2 mL de buffer de 10 x T4 DNA polimerase, 1 mL de 1 mg / mL BSA, 1 mL de 25 mM dATP, dCTP, dTTP e cada um, 1 mL (3 U) da Polimerase DNA T4, e 3 mL de DDH 2 O.
Faça uma mistura com o mestre da polimerase, tampão, etc e quantidade alíquota apropriada do master mix a cada amostra de DNA. Incubar a 30 ° C por 30 min. Em seguida, adicione 1,2 mL de EDTA 0,5 M imediatamente para a reação. - Eletroforese
Executar produtos de extensão em uma acrilamida 10% / 7 M gel urea/1xTBE. Carga 4 mL de cada amostra. Ferva as amostras por 10 minutos antes do carregamento. Executado em 800V em 1x TBE até o corante azul atinge o fundo do gel. Secar o gel a 65 ° C por 30 min, em seguida, à temperatura ambiente por 30 min e expor a phosphorimager durante 2 horas.
Para preparar 100 ml de gel de poliacrilamida 10%, dissolver 41 g de uréia e 21 mL de solução de acrilamida 40% (acrilamida / bis-acrilamida 29:1) em 30 mL de água destilada, adicionar 10 mL de TBE 10x. Montar placas de vidro. Direito antes de derramar o gel, adicionar 1 mL da APS 10% e 100 mL de Temed. Despeje o gel e tentar evitar as bolhas.
4. Resultados representativos:
Detectar T. brucei estrutura dos telômeros G saliência usando nativa em gel de hibridação
T. intacta cromossomos brucei separados por PFGE são mostrados na Figura 3A e 3B (painéis à esquerda). T. células brucei normalmente contêm ~ 100 cópias de minichromosomes, todos com tamanho similar (50-150 kb) e migram para a mesma posição no gel (MC). Devido a este fato, após hibridização com sonda oligo TELC, o telômero sinal G saliência é mais proeminente em minichromosomes (Figura 3A, painel do meio). Hibridação com sonda oligo TELG normalmente não deu qualquer sinal de hibridização (Figura 3B, painel do meio), pois T. células brucei não têm estrutura de C-saliência dos telômeros. Após desnaturação e hibridização de neutralização, com qualquer TELC ou oligo TELG sonda deve revelar sinais dos telômeros em todas as extremidades dos cromossomas (Figura 3A e 3B, painéis à direita).
Determinar o terminal de nucleotídeos dos telômeros pela ligadura adaptador
Carregando igual quantidade de DNA em cada lane é essencial para este ensaio, e isso é mostrado pela coloração com brometo de etídio do gel. Um exemplo é mostrado na figura 4A, painel da esquerda.
Neste protocolo, o fim marcado única adaptadores oligo e oligo guia só são ligados ao fim quando o cromossomo telomere G-saliências que terminam com seqüências que são compatíveis com o oligo guia. Si nce a oligo-end único é rotulado, o produto será ligadura radioativos e dar sinal forte. Conforme mostrado na Figura 4A, painel da direita, pista 1 dá um forte sinal, indicando que uma grande quantidade de unique/TG1 adaptador tem sido ligada ao fim dos telômeros. TG1 tem uma seqüência final de 5'CCCTAA3 '. Daí o telômero G-overhangs ligada a este adaptador no final 5'TTAGGG3 '. Nenhuma quantidade significativa de produtos ligadura é observado usando oligoes outro guia, indicando que os telômeros terminando em TTAGGG são predominantes em T. células brucei.
Tratamento do DNA genômico com Exo I ou T Exo, que são nucleases 3 'saliência específicos, resultou na perda dos produtos ligadura (Figura 4A, painel da direita, faixas 2 e 3), indicando que a ligadura de fato resultou do telômero G- estrutura de balanço
Ligadura Extensão Primer Mediated
Sem ligadura, o fim marcado oligo guia é de 22 nt de comprimento. Carregando final marcado guia oligo serviria como um controle negativo e um marcador de tamanho. (Figura 4B, pista 11, asterisco indica o fim marcado guia oligo). Que normalmente também observar uma banda de ~ 44 nt neste controle negativo (Figura 4B, pista 11, triângulo aberto), que são mais provável resíduo não-desnaturado adaptadores. Após a ligadura para o fim de cromossomos, o tamanho do produto estendida reflete o comprimento do balanço G-estrutura. A maioria T. brucei telomere fim G-saliências em 5'TTAGGG3 '(Figura 4A). No entanto, estes G-overhangs parecem ser muito curto (apenas ~ 10 nt de comprimento), como os produtos se estendia desde o ligada TG1 oligo guia não são muito mais do que o oligo guia própria (Figura 4B, pista 10), mas eles permitem que a ligadura do TG1 adaptador (Figura 4A, painel da direita, pista 1). Embora apenas uma pequena quantidade de TG4 adaptador era ligada às extremidades dos telômeros (Figura 4A, painel da direita, pista 6), os produtos se estendia desde TG4 ligados são muito mais longos (Figura 4B, pista 7, cabeças de seta), com o maior produto que está sendo ~ 55 nt. Assim, observamos dois tipos de saliência dos telômeros G-in T. células brucei. Predominante dos telômeros fim G-saliências em 5'TTAGGG3 ", mas são apenas ~ 10 nt de comprimento. Alguns dos telômeros fim G-saliências em 5'GGGTTA3 ", mas pode ser até 40 nt de comprimento. Ambos os tipos de saliência G-são sensíveis a Exo T (ou Exo I) tratamento (Figura 4A, painel da direita, pista 2, 3, e 7, Figura 4B, pista 1, setas).

Figura 1. Princípio da hibridização em gel nativo. Esquerda, sob a condição de natal, o fim marcado TELC oligo sonda só pode hibridizar com o single-stranded saliência G-ricos dos telômeros. A intensidade da hibridação reflete o comprimento do balanço G-telômero. Direito, após a desnaturação, o TELC oligo sonda irá hibridizar com todo o DNA do telômero. A intensidade desta hibridação representa a quantidade total de DNA do telômero e é usado como um controle de carga, eo nível G saliência-final é calculado dividindo o sinal de hibridização nativa por que a obtida após a desnaturação.
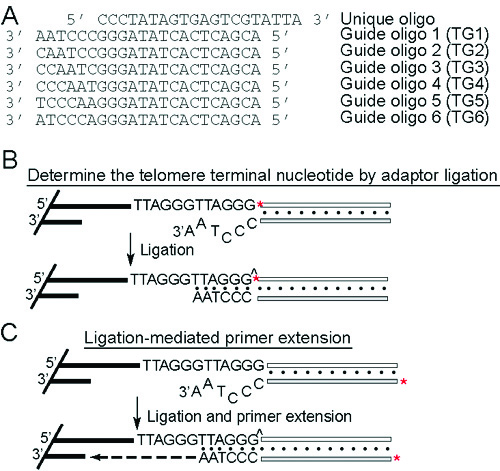
Figura 2. Princípio da ligadura Adaptor e extensão ligadura cartilha mediada. (A) Seqüências do oligoes guia única e seis. (B) Determine o terminal de nucleotídeos dos telômeros pela ligadura adaptador. O único é oligo-end rotulados (marcados com um asterisco vermelho), recozidas ao oligoes guia e ligada ao fim dos telômeros. Apenas quando o adaptador exclusivo / guia traz uma saliência 3 'que é compatível com a seqüência do telômero terminal vai ser ligada ao adaptador. (C) Em ligadura de extensão de primer mediada, o guia é oligo-end rotulados (marcados com um asterisco vermelho) e recozida ao oligo exclusivo antes ligados às extremidades do cromossomo. Somente o adaptador exclusivo / guia tendo saliência compatível com o G-a saliência 3 'vai ser ligada. ^ Indica o vínculo phophordiester ligada. Após a remoção dos pares oligo sem ligadura, extensão cartilha será realizado com T4 DNA polimerase, que carece de deslocamento vertente e atividades 5'-3 'exonuclease. O comprimento final da estendido guia oligo, portanto, reflete o comprimento do G-saliência.
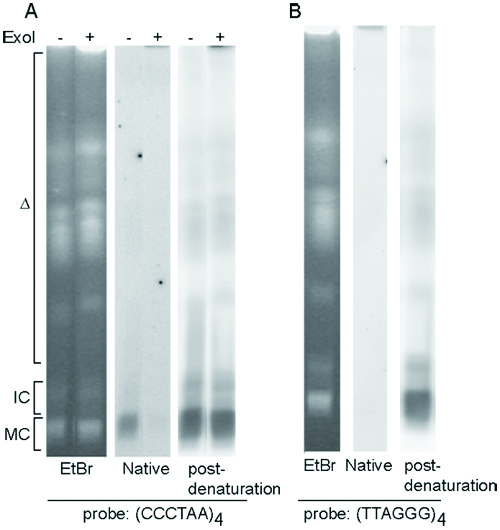
Figura 3. T. brucei estrutura dos telômeros G-saliência analisados por nativa em gel de hibridização. (A) hibridação Em gel com TELC oligo sonda. (B) de hibridização em gel com TELG oligo sonda. Em ambos (A) e (B) Esquerda, Ethedium PFG manchada Brometo. Meio, resultado de hibridização nativa. Direito, resultado de hibridização pós-desnaturação. Do tipo selvagem T. células brucei foram utilizados. Intacto ou Exo tratei DNA genômico foram separados por PFGE. Triângulo aberto representa os cromossomos megabase; IC, intermediário cromossomos empresas; MC, minichromosomes.
"/>
Figura 4. T. brucei estrutura dos telômeros G-saliência analisados por ligadura mediada extensão primer. (A) A maioria T. brucei telomere G-saliências terminam em 5'TTAGGG3 '. Esquerda, Ethedium brometo de gel DNA manchado. Quantidade aproximadamente igual de DNA é carregado em cada pista. Direito, resultado da exposição do mesmo gel após a secagem. Intacto, Exo I-tratada, ou Exo DNA T-tratados foi ligada a seis diferentes finais rotulados adaptadores únicos / guia oligo conforme indicado. (B) T. telômeros brucei têm curto G-overhangs. DNA genômico intacto ou Exo T-tratados foi ligada a seis diferentes adaptadores únicos / guia oligo seguido por extensão de primer usando DNA polimerase T4. Fim-rotulados TG4 oligo foi carregado como um controle negativo (faixa 11). Os oligo-se corre a 22 nt (*), mas também deu um sinal mais fraco em ~ 44 nt (Δ). Apenas unique/TG4 adaptador rendeu produtos de extensão significativamente maior que o oligo guia própria (seta cabeças, pista 7).
Discussão
Trypanosoma brucei causa a doença do sono em humanos. Esta doença, se não tratada, é inevitavelmente fatal. T. células brucei em hospedeiros mamíferos sofrer variação antigênica regularmente, de modo a evitar o ataque imune do hospedeiro 7. Por isso, é muito difícil eliminar a T. células brucei uma vez que uma infecção é estabelecida. Recentemente, demonstrou que os telômeros desempenhar um papel importante na regulação da expressão de T. bru...
Divulgações
Agradecimentos
Gostaríamos de agradecer ao Dr. Carolyn Preço para discussões científicas e sugestões perspicaz. Este trabalho é suportado pelo NIH conceder AI066095 (PI: Bibo Li).
Materiais
| Name | Company | Catalog Number | Comments |
| SeaKem LE Agarose | Lonza Inc. | 50004 | |
| Agarose Type VII (low melting agarose) | Sigma-Aldrich | A4018 | |
| Proteinase K, recombinant, PCR grade | Roche Group | 03 115801001 | |
| Exo I | New England Biolabs | M0293 | |
| Exo T | New England Biolabs | M0265 | |
| T7 exonuclease | New England Biolabs | M0263 | |
| T4 DNA polymerase | New England Biolabs | M0203 | |
| QIAquick nucleotide removal kit | Qiagen | 28304 |
Referências
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados