È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
L'esame dei telomeri G-Struttura in aggetto Trypanosoma brucei
In questo articolo
Riepilogo
I telomeri sono essenziali per la stabilità dei telomeri dei cromosomi e la G-sbalzo struttura è essenziale per la telomerasi mediato conservazione dei telomeri. Abbiamo recentemente adottato due metodi per individuare i telomeri G-sbalzo struttura Trypanosoma brucei, Che sono nativi in gel di ibridazione e ligation-mediata primer extension, che saranno descritti.
Abstract
Il G-sbalzo struttura dei telomeri è stato identificato negli eucarioti tra cui lievito, vertebrati, e Trypanosoma brucei. Esso funge da substrato per la telomerasi per de novo sintesi del DNA dei telomeri e quindi è importante per il mantenimento dei telomeri. T. brucei è un protozoo parassita che causa la malattia del sonno negli esseri umani e nagana nei bovini. Una volta infettato dei mammiferi, T. cellula brucei interruttori regolarmente il suo antigene di superficie di eludere attacco immunitario dell'ospite. Abbiamo recentemente dimostrato che il T. brucei struttura dei telomeri svolge un ruolo essenziale nella regolazione dell'espressione genica antigene di superficie, che è critica per T. brucei patogenesi. Tuttavia, T. brucei struttura dei telomeri non è stata ampiamente studiata a causa delle limitazioni dei metodi per l'analisi di questa struttura specializzata. Ora abbiamo adottato con successo il nativo in gel ibridazione e ligation-mediata metodi fondo estensione per l'esame del telomero G-sbalzo struttura e un metodo di legatura adattatore per la determinazione del terminale dei telomeri nucleotide in T. brucei cellule. Qui, verranno descritti i protocolli in dettaglio e confrontare i loro diversi vantaggi e limitazioni.
Protocollo
1. Rileva T. brucei telomeri G-sbalzo utilizzando nativi In-gel ibridazione 3
Principio (Figura 1): In condizioni native, una soluzione end-etichetta (CCCTAA) 4 (TELC) oligo sonda può ibridare solo al singolo filamento telomeri G-sbalzo regione. L'intensità di ibridazione è proporzionale alla lunghezza dello sbalzo. Dopo la denaturazione e neutralizzazione, la sonda stessa sarà in grado di ibridare la regione dei telomeri intera. L'intensità di ibridazione rappresenta la quantità totale del DNA dei telomeri e può essere utilizzato come controllo di caricamento. Due controlli standard per questo esperimento sono ibridazione con end-etichetta (TTAGGG) 4 (TELG) oligo sonda, che non dovrebbe dare alcun segnale di T. cellula brucei non ha telomeri C-sbalzo struttura, e di trattare il DNA genomico con 3'-specifici a singolo filamento esonuleasi come Exo I o T Exo prima ibridazione, che dovrebbe eliminare il segnale nativo di ibridazione TELC.
Fase 1. Preparare le spine del DNA per elettroforesi su gel a campo pulsato (PFGE)
Cromosomi intatti saranno separati da PFGE. Pertanto, il DNA genomico è preparato come spine DNA.
- Inizia con 100 ml di cellule formano sangue (1,5 -2.000 mila cellule / ml) o 20 ml di cellule procyclic (a 10 milioni di cellule / ml).
- Sciogliere 1,6% a basso punto di fusione (LMP) agarosio in tampone L (0,01 M Tris • CL pH 7,6, 0,02 M di NaCl, 0,1 M EDTA pH 8,0) e tenere in caldo a 50 ° C.
- Celle di raccolta per centrifugazione a 1,5 krpm, 4 ° C per 10 min. Rimuovere il surnatante e risospendere il pellet con tampone L ad una concentrazione finale di 5 milioni di cellule / ml.
- Incubare sospensione cellulare a 42-50 ° C per 10 min.
- Aggiungere 1 volume di agarosio LMP a 1 volume delle cellule. Mescolate bene, ma in fretta.
- Aliquota 85 ml di sospensione cellulare in ogni pozzetto di uno stampo spina usa e getta (BioRad).
- Dopo 5-10 minuti o fino a quando le spine sono solidificate, tappi trasferimento in un tubo da 15 ml con 5 ml di buffer L / 1% Sarkosyl. Aggiungere 100 ml di 250 mg / ml di proteinasi K o un pizzico di proteinasi K in polvere, mescolare bene e incubare a 50 ° C per 48 ore.
- Eliminare il tampone, lavare spine con 5 ml di tampone L ripetere due volte e la digestione proteinasi K per altre 48 ore. Lavare le spine con tampone L fresca di nuovo. Conservare le spine a 4 ° C in tampone L. Questi tappi deve essere utilizzato entro 2 settimane.
Fase 2. Separare intatto T. cromosomi brucei tramite elettroforesi su gel a campo pulsato
- Preparare gel
- Preparare 2-3 L di 0,5 x tampone TBE e trasferimento in camera di gel in esecuzione di un DRII CHEF (BioRad) unità. Impostare la temperatura in esecuzione, avviare la pompa, poi avviare l'unità di raffreddamento.
- Preparare 100 mL di un gel di agarosio 1,2% in 0,5 x tampone TBE. Raffreddare agarosio durante l'impostazione del vassoio gel.
- Decidere l'ordine del campione. Ricordati di inserire lo standard del DNA. Asciugare il pettine e adagiarlo su una superficie piana. Luogo ogni spina del DNA sul dente appropriato. Eliminare la soluzione tampone eccessivo intorno al tappo del DNA per aspirazione in modo che il tappo si attacca al dente.
- Versare il gel senza inserire il pettine. Lasciate raffreddare a 40-50 ° C (pochi minuti), inserire lentamente il pettine con il maschio nella gel. Assicurarsi che spine non scivolare il pettine. Lasciate che il gel solidifichi. Rimuovere pettine da gel lentamente.
- Pulsata campo elettroforesi su gel
Esegui gel in una unità CHEF DRII: 700 s-1500 s legumi, 2,5 V / cm, a 12 ° C per 120 ore.
Fase 3. Macchia e Decolorare il gel di agarosio
Macchia il gel con 1μg / ml di etidio bromuro in 0,5 x TBE a temperatura ambiente per 1 ora in poi 0,5 x TBE senza bromuro di etidio per 1 ora con il dolce dondolio. Scatta una foto del gel.
Fase 4. Asciugare il gel
Posizionare il gel su due strati di carta Whatman mm 3 e coprire il gel con involucro di plastica. Asciugare il gel a temperatura ambiente (il gel non deve essere riscaldata oltre 50 ° C per evitare qualsiasi denaturazione dei campioni di DNA). Sostituire le carte Whatman bagnato con quelle secche dopo 2 e 4 ore di essiccazione, rispettivamente. Mantenere l'essiccazione fino a quando il gel è completamente asciutto. Questa operazione potrebbe richiedere una notte in funzione l'essiccatore.
Fase 5. Etichetta le sonde oligo
Miscelare 1 ml di 50 ng / mL TELC o TELG oligo con 1 ml di 10 x polinucleotide chinasi (PNK) tampone, 5 ml di γ [32P] ATP, 2 l di DDH 2 O e 1 ml di PNK T4. Incubare a 37 ° C per 1 ora. Aggiungere 90 ml di TNES tampone (10 mM Tris Cl, pH 8.0, 100 mM NaCl, 10 mM EDTA, pH 8.0, 1% SDS) alla miscela di reazione.
Mentre l'etichettatura oligoes, preparare una colonna G-25: mettere un po 'di lana di vetro in una siringa da 3 cc e roba del genere è saldamente con la punta della pipetta. Riempire la siringa con Sephadex multa G-25 (autoclAVED in TE) al 3 ml marchio. Lasciate riposare in peso. Per purificare la sonda marcata: caricare la sonda in cima alla colonna. Lavare con 700 l di tampone TNES, ed eluire con 600 ml di tampone TNES. In alternativa, purificare la sonda marcata con un mini-Spin ™ rapida colonna (Roche Applied Science).
Punto 6: ibridazione
- Brevemente bagnato il gel secchi in acqua distillata e lavare via la carta Whatman bloccato sul gel
- Mettere il gel in un sacchetto di ibridazione e aggiungere 20 ml di tampone hybridiztion (0.25M Na 2 HPO 4 pH7.2, 1 mM EDTA, 7% SDS, 1% di BSA, filtrata attraverso filtro 0,22 micron). Incubare in un bagno d'acqua a 50 ° C con dondolo dolce per almeno 1 ora.
- Rimuovere la pre-ibridazione buffer. Lessate le sonda marcata per 5 minuti e aggiungerlo a 25 mL di tampone di ibridazione fresca, filtro-sterilizzare la miscela con un filtro 0,22 micron siringa e aggiungete la miscela di ibridazione direttamente alla borsa di ibridazione. Sigillare il sacchetto e porlo in un contenitore poco profonda con acqua calda. Mettere il recipiente tutto nella vasca d'acqua. Incubare a 50 ° C durante la notte con dolce dondolio.
- Tagliare una piccola apertura nella borsa ibridazione. Riversa il più possibile il mix di ibridazione e salvarlo in una provetta da 50 ml. Tenere la sonda congelato a -20 ° C. Rimuovere il gel dal sacchetto ibridazione e metterlo in un contenitore.
IN QUESTO PASSO TUTTO SARA 'CALDO E BISOGNI una pulizia accurata (BANCO, SCHERMO, forbici, ECC. Assicuratevi di indossare doppi strati di guanti! - Lavare il gel in 4 x SSC per 30 minuti a 50 ° C. Sostituire con tampone di lavaggio fresco e ripetere due volte.
- Lavare il gel in 4 x SSC/0.1% SDS a 50 ° C.
- Mettere il gel su un pezzo di 3 mm Carta Whatman e leggermente asciugare il gel.
- Avvolgere il gel con pellicola trasparente e esporre a phosphorimager per ~ 3 giorni
- Eseguire la scansione del phosphorimager
- Incubare il gel nel tampone denaturante (1,5 M di NaCl, 0,5 M di NaOH) a temperatura ambiente per 30 min
- Lavare il gel nel tampone di neutralizzazione (3 M di NaCl, 0,5 M Tris • / Cl pH 7,0) a temperatura ambiente per 30 min
- Lavare in acqua distillata per 3 minuti
- Pre-ibridazione a 55 ° C per 1 ora
- Ripetere l'ibridazione con la stessa sonda contenenti mix di ibridazione a 55 ° C durante la notte.
- Lavare come sopra descritto, tranne a 55 ° C.
- Avvolgere il gel e esporre a phosphorimager per ~ 2 ore.
- Eseguire la scansione del phosphorimager. Normalizzare il segnale di ibridazione ottenuto prima denaturazione con quella ottenuta dopo denaturazione.
2. Determinare il terminale dei telomeri Nucleotide da Legatura adattatore 6
Principio (Figura 2A e 2B): A 5 'marcata con oligonucleotide unico è legatura al 3' del G-sbalzo. Ciò si ottiene ricottura a specifiche oligo guida complementare. Solo l'unico / guida oligo sequenze cuscinetto adattatore compatibile al telomero G-sbalzo sequenze terminale sarà legato. Per T. brucei, i telomeri può terminare in una delle sei nucleotidi differenti delle ripetizioni TTAGGG. Quindi sei oligoes guida vengono utilizzati diversi (TG1-TG6, Figura 2A). Dopo la legatura, il DNA viene digerito con enzimi di restrizione e risolti su un gel di agarosio.
- Trattamento chinasi della oligo unico.
Mix 6 ml di 10 pmole / mL oligo unico con 3 ml di tampone PNK 10x, 5 ml di γ [32 P] ATP, 14 ml di DDH 2 O, e 2 microlitri (10 U) di PNK T4. Incubare la miscela a 37 ° C per 1 ora. - Rimuovere senza personalità giuridica nucleotide caldo.
Utilizzare il kit di rimozione nucleotide QIAquick per rimuovere la personalità giuridica caldo ATP ed eluire la fine marcata oligo con 60 ml di tampone di eluizione (concentrazione finale sarebbe pmole ~ 1 / mL). - Temprare oligo unica per guidare oligo (per creare l'adattatore).
Aggiungere 10 ml di purificato oligo unici a 2 l di ogni oligo guida e buffer di 1 microlitri di ricottura (200 mM NaCl, 100 mM Tris-HCl pH 8,0). Mettere i tubi in un blocco di calore ° 85 C e spegnere il blocco di calore. Lasciate che i tubi e sul blocco termico freddo per RT. Questo richiederà un paio d'ore. Se in un tubo di fretta, il trasferimento dopo 5 minuti ciascuno a 65 ° C, 37 ° C e poi lasciate raffreddare per RT. - Adattatori legare al totale del DNA genomico.
A 2,5 ml di DNA genomico intatto, aggiungere 4 ml di buffer di 5 x ligasi, 10 ml di oligo ricotto, 2,5 l di DDH 2 O e 1 ml di DNA ligasi T4. Incubare a 16 ° C durante la notte. - Endonucleasi di restrizione digestione
A 20 ml di prodotto legatura aggiungere 3 ml di 10 x ONA buffer da 4, 1,5 l di ALUI e MboI ciascuno, e 4 ml di DDH 2 O. Incubare la miscela a 37 ° C durante la notte. - Elettroforesi.
Aggiungere 3 ml di 10 x Arancio G colorante (50% glicerolo, 0.5% Arancio G) per ogni campione. Caricare i campioni di DNA su un 20x20 cm 0,7% gel in TBE 0,5 x con 0,2 mg / ml di bromuro di etidio. Funzionare a 30V per 30 minuti, poi continue con una maggiore tensione, ma a meno di 120 V per un totale di 1000 ore v. Scatta una foto del gel con un righello vicino al marcatore del DNA. - Gel secco ed esporre.
Mettere il gel su uno strato di DE81 carta da filtro e da due strati di 3 millimetri di carta Whatman e coprire il gel con involucro di plastica. Gel secco a temperatura ambiente. Esporre il gel essiccato al phosphorimager durante la notte.
3. Legatura Primer Extension mediata 6
Principio (Figura 2A e 2C): un oligonucleotide unico è legatura al 3 'G-sbalzo con la sequenza terminale specifico, determinato dal suo sequenze complementari l'adattatore (guida) oligo. Dopo la purificazione, la guida oligo etichetta che rimane ricotta al oligo G-overhang/unique fondo è estesa alla ss ds-giunzione con T4 DNA polimerasi, che manca di spostamento filo e 5'-3 'attività esonucleasi. I prodotti di fondo estensione quindi dare la lunghezza del G-sbalzo.
- Trattamento chinasi del oligoes guida e unico
- Mix 20 l di 10 pmole / mL oligo unico con 10 ml di 10 x PNK tampone, 20 l 5 x tampone ligasi (che contiene 10 mM ATP), 3 ml (30 U) di PNK T4, e 47 ml di DDH 2 O . Incubare la miscela a 37 ° C per 60 minuti.
- Miscelare 1 ml di 10 pmole / mL guida oligo con 1 l di 10 x PNK tampone, 2 l di γ [32 P] ATP, 1 ml (10 U) di PNK T4, e 5 ml di DDH 2 O. Incubare la miscela a 37 ° C per 60 minuti.
- Temprare guida oligo e unico oligo
Aggiungere 10 L (20 pmole) della oligo fosforilato unico e 10 L (10 pmole) della fine marcato guida oligo in una provetta 1,5 ml Eppendorf (il rapporto 2:1 di unico per guidare oligo assicura che tutti i oligo guida è ricotto ). Seguire la stessa procedura descritta nel protocollo II per ricottura oligoes. - Legare la guida di ricotta / adattatore oligo unica a fini telomero
Seguire la stessa procedura descritta nel protocollo II per la legatura del DNA. - DNA precipitato con il volume 1 / 10 di acetato di sodio e 2,5 volumi di etanolo ghiacciato a -80 ° C per 30 min. Spin down DNA pellet a 12 krpm, 4 ° C per 15 min. Lavare il DNA con il 70% di etanolo e si dissolvono pallet in 10 ml di DDH 2 O.
- Primer estensione
Per ogni campione di 10 microlitri di DNA aggiungere 2 ml di buffer di 10 T4 DNA polimerasi x, 1 l di 1 mg / ml BSA, 1 l di mM dATP 25, dCTP e dTTP ciascuno, 1 ml (3 U) di DNA polimerasi T4, e 3 ml di DDH 2 O.
Fare un master mix con la polimerasi, buffer, ecc e la quantità appropriata aliquota del master mix per ogni campione di DNA. Incubare a 30 ° C per 30 min. Quindi aggiungere 1,2 ml di EDTA 0.5M immediatamente la reazione. - Elettroforesi
Esegui prodotti di estensione su un acrilamide 10% / 7 M gel urea/1xTBE. Carico di 4 ml di ogni campione. Bollire i campioni per 10 minuti prima di caricare. Funzionare a 800V in TBE 1x fino a quando il colorante blu raggiunge il fondo del gel. Asciugare il gel a 65 ° C per 30 minuti poi a temperatura ambiente per 30 min e esporre a phosphorimager per 2 ore.
Per preparare 100 ml di gel di poliacrilammide al 10%, sciogliere 41 g di urea e 21 ml di soluzione di acrilamide al 40% (acrilamide / bis-acrilamide 29:1) in 30 ml di acqua distillata, aggiungere 10 ml di TBE 10x. Montare lastre di vetro. A destra prima di versare il gel, aggiungere 1 mL di 10% di APS e 100 ml di TEMED. Versare il gel e cercare di evitare eventuali bolle.
4. Rappresentante dei risultati:
Rileva T. brucei telomeri G-sbalzo con struttura nativa in-gel ibridazione
Intatta T. cromosomi brucei separati da PFGE sono mostrati in figura 3A e 3B (pannelli a sinistra). T. cellule brucei normalmente contengono circa 100 copie di minichromosomes, il tutto con dimensioni simili (50-150 kb) e migrare verso la stessa posizione sul gel (MC). A causa di questo fatto, dopo ibridazione con TELC oligo sonda, il G-sbalzo telomeri segnale è più evidente su minichromosomes (Figura 3A, pannello centrale). Ibridazione con TELG oligo sonda normalmente non produce alcun segnale di ibridazione (figura 3B, pannello centrale) a causa T. cellule brucei non hanno telomeri C-sbalzo struttura. Dopo la denaturazione e neutralizzazione, l'ibridazione sia con TELC o TELG oligo sonda dovrebbe rivelare i segnali su tutte le estremità dei telomeri dei cromosomi (figura 3A e 3B, pannelli di destra).
Determinare il terminale dei telomeri nucleotide mediante legatura adattatore
Caricamento quantità uguale di DNA in ogni corsia è fondamentale per questa analisi, e questo è dimostrato dalla colorazione con etidio bromuro del gel. Un esempio è mostrato nella Figura 4A, pannello a sinistra.
In questo protocollo, la fine marcati unico oligo e guida adattatori oligo sono solo legatura alla fine dei cromosomi, quando il telomero G-aggetti che terminano con sequenze che sono compatibili con il oligo guida. Si Una volta che l'oligo unico fine è marcato, il prodotto sarà legatura radioattivi e dare segnale forte. Come mostrato nella Figura 4A, pannello di destra, corsia 1 fornisce un segnale forte, che indica che una grande quantità di unique/TG1 adattatore è stato legatura alla fine dei telomeri. TG1 ha una sequenza finale di 5'CCCTAA3 '. Di qui il telomero G-sbalzi legatura a tal fine adattatore in 5'TTAGGG3 '. Nessuna quantità significativa di prodotti legatura si osserva utilizzando oligoes altra guida, indicando che i telomeri che terminano in TTAGGG sono predominanti in T. brucei cellule.
Trattare il DNA genomico con Exo I o Exo T, che sono 3 'nucleasi sbalzo specifici, ha determinato la perdita dei prodotti legatura (4A, pannello di destra, corsie 2 e 3), che indica che la legatura infatti il risultato del telomero G- sbalzo struttura
Legatura mediata Estensione Primer
Senza legatura, la fine marcata oligo guida è di 22 nt lungo. Caricamento fine marcato guida oligo servirebbe come controllo negativo e un marker dimensioni. (Figura 4B, corsia 11, asterisco indica la fine marcato guida oligo). Di solito anche osservare una banda di ~ 44 nt in questo controllo negativo (Figura 4B, corsia 11, triangolo aperto), che sono più probabili residui non denaturato adattatori. Dopo la legatura alla fine dei cromosomi, la dimensione del prodotto esteso riflette la lunghezza del G-sbalzo struttura. La maggior parte T. brucei telomeri G-sbalzi fine a 5'TTAGGG3 '(Figura 4A). Tuttavia, questi sbalzi G-sembrano essere molto breve (solo ~ 10 nt di lunghezza), i prodotti si estendeva dalla legatura TG1 guida oligo non sono molto più lungo del oligo guida stessa (figura 4B, corsia 10), ma permettono di legatura TG1 adattatore (4A, pannello di destra, corsia 1). Anche se solo una piccola quantità di TG4 adattatore è stato legatura alle estremità dei telomeri (4A, pannello di destra, corsia 6), i prodotti si estendeva dal TG4 legatura sono molto più lunghi (Figura 4B, corsia 7, punte di frecce), con la più lunga prodotto da ~ 55 nt. Quindi, abbiamo osservato due tipi di telomeri G-sbalzo in T. brucei cellule. Il predominante telomeri G-sbalzi fine a 5'TTAGGG3 ', ma sono solo ~ 10 nt lungo. Pochi telomeri G-sbalzi fine a 5'GGGTTA3 ', ma può essere lungo fino a 40 nt. Entrambi i tipi di G-sbalzo sono sensibili a Exo T (o Exo I) trattamento (4A, pannello di destra, corsia 2, 3 e 7; Figura 4B, corsia 1, frecce).

Figura 1. Principio di nativi-gel ibridazione. Sinistra, con la condizione nativa, la fine marcata TELC oligo sonda può ibridare con il singolo filamento G-ricchi sbalzo dei telomeri. L'intensità della ibridazione riflette la lunghezza del telomero G-sbalzo. Destra, dopo denaturazione, la TELC oligo sonda ibridare con tutto il DNA dei telomeri. L'intensità di questa ibridazione rappresenta la quantità totale di DNA dei telomeri e viene utilizzato come controllo di caricamento, e la finale del G-sbalzo livello è calcolato dividendo il segnale nativo di ibridazione con quella ottenuta dopo denaturazione.
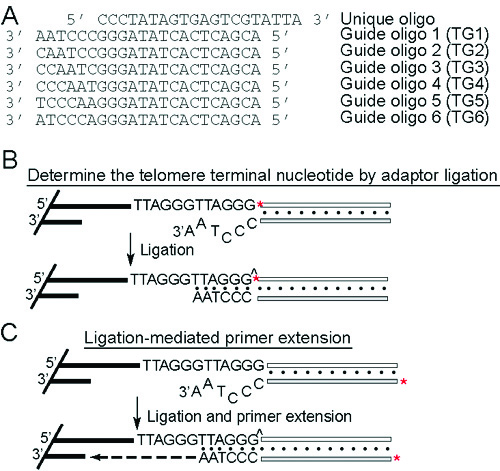
Figura 2. Principio di legatura adattatore e mediato estensione legatura primer. (A) sequenze del oligoes guida unica e sei. (B) Determinare il terminale dei telomeri nucleotide con legatura adattatore. L'oligo unico fine è marcato (contrassegnati con un asterisco rosso), ricotta al oligoes guida e legato fino alla fine dei telomeri. Solo quando l'adattatore unico / guida sbalzo porta a 3 'che è compatibile con la sequenza dei telomeri terminale l'adattatore essere legata. (C) Per l'estensione di fondo legatura mediata, la guida è oligo fine marcato (contrassegnati con un asterisco rosso) e ricotta al oligo univoco prima legatura alle estremità dei cromosomi. Solo l'adattatore unico / guida cuscinetto sbalzo compatibile con il G-sbalzo di 3 'sarà legato. ^ Indica il legame legatura phophordiester. Dopo la rimozione delle coppie di oligo unligated, primer extension sarà effettuato con la DNA polimerasi T4, che manca di spostamento filo e 5'-3 'attività esonucleasi. La lunghezza finale del estesa guida oligo riflette quindi la lunghezza del G-sbalzo.
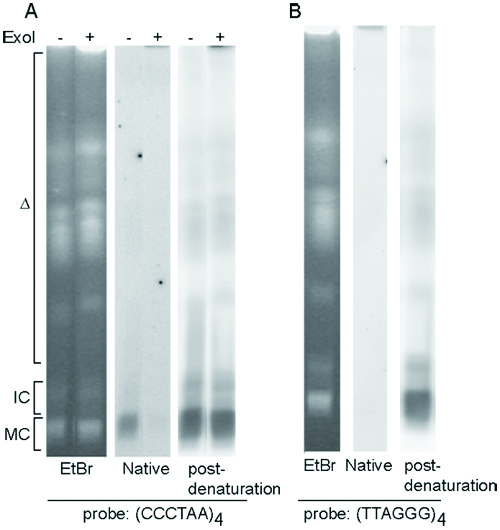
Figura 3. T. brucei telomeri G-sbalzo struttura analizzata da nativi in gel-ibridazione. (A) In-gel ibridazione con TELC oligo sonda. (B) In-gel ibridazione con TELG oligo sonda. In entrambi (A) e (B) Sinistra, PFG Bromuro Ethedium macchiato. Mezzo, risultato dell'ibridazione nativi. A destra, dopo denaturazione risultato ibridazione. Wild-type T. cellule brucei sono stati utilizzati. Intatti o Exo ho trattato DNA genomico erano separati da PFGE. Triangolo sta per aprire i cromosomi megabase, IC, intermedio cromosomi imprese; MC, minichromosomes.
"/>
Figura 4. T. brucei telomeri G-sbalzo struttura analizzata da ligation-mediata primer extension. (A) La maggior parte T. brucei telomeri G-sbalzi 5'TTAGGG3 terminano in '. Sinistra, Ethedium DNA gel colorato bromuro. Importo pari del DNA è caricata in ogni corsia. A destra, risultato esposizione del gel stesso dopo l'essiccazione. Intatta, Exo I-trattati, o Exo T-DNA è stata trattata legatura a sei diversi end-etichettati unica guida / adattatori oligo come indicato. (B) T. brucei telomeri sono brevi G-strapiombi. Intatti o Exo T trattati con DNA genomico è stato legatura a sei diverse unici / guida adattatori oligo seguita da primer extension con T4 DNA polimerasi. End-etichettati TG4 oligo è stato caricato come un controllo negativo (corsia 11). La stessa oligo corre a 22 nt (*), ma ha anche dato un segnale più debole a ~ 44 nt (Δ). Solo unique/TG4 adattatore prodotto prodotti estensione significativamente più lungo rispetto alla oligo guida stessa (punte di freccia, corsia 7).
Discussione
Trypanosoma brucei causa la malattia del sonno negli esseri umani. Questa malattia, se non trattata, è inevitabilmente fatale. T. cellule brucei a ospiti mammiferi subiscono variazione antigenica regolarmente in modo da eludere l'attacco immunitario dell'ospite 7. Quindi è molto difficile da eliminare T. cellule brucei una volta l'infezione è stabilita. Abbiamo recentemente dimostrato che i telomeri svolgono un ruolo importante nella regolazione dell...
Divulgazioni
Riconoscimenti
Vorremmo ringraziare il Dott. Carolyn Prezzo per discussioni scientifiche e suggerimenti penetranti. Questo lavoro è supportato dal NIH concedere AI066095 (PI: Bibo Li).
Materiali
| Material Name | Type | Company | Catalogue Number | Comment |
|---|---|---|---|---|
| Name | Company | Catalog Number | Comments | |
| SeaKem LE Agarose | Lonza | 50004 | ||
| Agarose Type VII (low melting agarose) | Sigma | A4018 | ||
| Proteinase K, recombinant, PCR grade | Roche Diagnostic | 03 115801001 | ||
| Exo I | NEB | M0293 | ||
| Exo T | NEB | M0265 | ||
| T7 exonuclease | NEB | M0263 | ||
| T4 DNA polymerase | NEB | M0203 | ||
| QIAquick nucleotide removal kit | Qiagen | 28304 |
Riferimenti
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon