Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Die Untersuchung der Telomere G-Überhang Struktur in Trypanosoma brucei
In diesem Artikel
Zusammenfassung
Telomere sind unerlässlich für das Chromosom Stabilität und die Telomer-G-Überhang Struktur ist essentiell für Telomerase-vermittelte Telomererhaltung. Wir haben vor kurzem zwei Verfahren zum Nachweis der Telomer-G-Überhang Struktur in verabschiedet Trypanosoma brucei Sind die einheimischen In-Gel-Hybridisierung und Ligation-vermittelte Primer-Extension, die beschrieben werden.
Zusammenfassung
Die Telomer-G-Überhang Struktur hat sich in vielen Eukaryonten, einschließlich Hefe, Wirbeltieren und Trypanosoma brucei identifiziert worden. Es dient als Substrat für die Telomerase für die de novo Telomere DNA-Synthese und ist daher für Telomererhaltung wichtig. T. brucei ist ein einzelliger Parasit, dass die Schlafkrankheit bei Menschen und Nagana bei Rindern verursacht. Einmal infiziert Säugerwirt, T. brucei Zelle regelmäßig wechselt seinen Oberflächen-Antigen auf das Immunsystem des Wirts Angriff auszuweichen. Wir haben kürzlich gezeigt, dass die T. brucei Telomer-Struktur spielt eine wesentliche Rolle bei der Regulation der Oberflächen-Antigen Genexpression, die kritisch für T. ist brucei Pathogenese. Allerdings T. brucei Telomer-Struktur noch nicht ausführlich untersucht worden aufgrund der Begrenzung von Methoden zur Analyse dieser speziellen Struktur. Wir haben nun erfolgreich die einheimische In-Gel-Hybridisierung und Ligation-vermittelte Primer-Extension-Methoden zur Untersuchung der Telomer-G-Überhang-Struktur und eine Adaptorligation Methode zur Bestimmung der Telomere terminalen Nukleotids in T. angenommen brucei Zellen. Hier werden wir die Protokolle im Detail zu beschreiben und vergleichen ihre verschiedenen Vor-und Nachteile.
Protokoll
1. Detect T. brucei Telomere G-Überhang mit Native In-Gel-Hybridisierung 3
Prinzip (Abbildung 1): Unter nativen Zustand ein Ende-markierten (CCCTAA) 4 (TELC) Oligo-Sonde kann nur die einsträngige Telomer G-Überhang Region hybridisieren. Die Hybridisierung Intensität ist proportional zur Länge des Überhangs. Nach der Denaturierung und Neutralisation, wird die gleiche Sonde in der Lage sein, die ganze Telomer Region hybridisieren. Die Hybridisierung Intensität stellt die Summe der Telomere DNA-Menge und kann als Ladekontrolle verwendet werden. Zwei Standard-Steuerungen für dieses Experiments sind Hybridisierung mit Ende-markierten (TTAGGG) 4 (TELG) Oligo-Sonde, die nicht nachgeben sollte jedes Signal als T. brucei Zelle nicht Telomer C-Überhang Struktur und die genomische DNA mit 3'-spezifischen einzelsträngigen Exonukleasen wie Exo I oder Exo T vor der Hybridisierung zu behandeln, die dann den einheimischen TELC Hybridisierungssignal zu beseitigen.
Schritt 1. Bereiten DNA-Stecker für Pulsed Field Gel-Elektrophorese (PFGE)
Intakten Chromosomen werden durch PFGE getrennt werden. Daher ist genomischer DNA als DNA-Steckern vorbereitet.
- Beginnen Sie mit 100 ml Blut Zellen bilden (bei 1,5 -2 Millionen Zellen / ml) oder 20 ml prozyklischen Zellen (bei 10 Millionen Zellen / ml).
- Lösen Sie 1,6% mit niedrigem Schmelzpunkt (LMP) Agarose in L-Puffer (0,01 M Tris • CL pH 7,6, 0,02 M NaCl, 0,1 M EDTA pH 8,0) und warm halten bei 50 ° C.
- Ernten Sie die Zellen durch Zentrifugation bei 1,5 krpm, 4 ° C für 10 min. Überstand entfernen und Zellpellet resuspendieren mit L-Puffer zu einer Endkonzentration von 5 Millionen Zellen / ml.
- Inkubieren Zellsuspension bei 42-50 ° C für 10 min.
- Add 1 Volumen LMP Agarose auf 1 Volumen der Zellen. Gut mischen, aber schnell.
- Aliquot 85 ul Zellsuspension in jede Vertiefung einer Wegwerf-Stecker Form (BioRad).
- Nach 5-10 min oder bis der Stecker sind verfestigt, Transfer-Stecker in ein konisches 15 ml-Tube mit 5 ml L-Puffer / 1% Sarkosyl. Geben Sie 100 ul von 250 mg / ml Proteinase K oder eine Prise Proteinase K Pulver, gut mischen und inkubieren bei 50 ° C für 48 Stunden.
- Entsorgen Sie die Puffer waschen Stecker mit 5 ml L-Puffer zweimal wiederholen Proteinase K-Verdau für weitere 48 Stunden. Wash-Stecker mit frischem L-Puffer wieder. Shop-Stecker auf 4 ° C in L-Puffer. Diese Stecker sollte innerhalb von 2 Wochen verwendet werden.
Schritt 2. Separate intakt T. brucei Chromosomen mit Pulsed Field Gelelektrophorese
- Bereiten Agarosegel
- Bereiten Sie 2-3 l von 0,5 x TBE-Puffer und Transfer zum Gel läuft Kammer eines CHEF DRII (BioRad)-Einheit. Stellen Sie die Betriebstemperatur, die Pumpe, dann starten Sie das Kühlaggregat.
- Bereiten Sie 100 mL einer 1,2% igen Agarosegel in 0,5 x TBE-Puffer. Coole Agarose bei der Einrichtung der Gelträger.
- Entscheiden Sie, die Probe zu bestellen. Denken Sie daran, die DNA-Standard gehören. Dry den Kamm und legen Sie es auf eine ebene Fläche. Platzieren Sie jede DNA-Stecker auf den entsprechenden Zahn. Überschüssiges Puffer um den DNA-Stecker durch Aspiration so den Stecker in die Zahn-Stick wird.
- Gießen Sie das Agarosegel ohne Einlegen der Kamm. Lassen Sie es abkühlen auf 40-50 ° C (wenige Minuten), schieben Sie den Kamm mit den beiliegenden Stopfen in das Gel. Stellen Sie sicher, dass die Stecker nicht abrutschen den Kamm. Lassen Sie das Gel erstarren. Entfernen Kamm aus Gel langsam.
- Pulsed Field Gelelektrophorese
Run Gel in einer CHEF DRII Einheit: 700 s-1500 s Impulse, 2,5 V / cm, bei 12 ° C für 120 Stunden.
Schritt 3. Stain und entfärben Agarosegel
Stain das Gel mit 1 g / ml Ethidiumbromid in 0,5 x TBE bei RT für 1 h dann in 0,5 x TBE ohne Ethidiumbromid für 1 Stunde unter leichtem Schütteln. Machen Sie ein Foto des Gels.
Schritt 4. Trocknen Sie die Agarosegel
Legen Sie die Agarosegel auf zwei Schichten von 3 mm Whatman-Papier und decken das Gel mit Plastikfolie. Trocknen Sie das Gel bei RT (sollte das Gel nicht beheizt mehr als 50 ° C zu vermeiden Denaturierung der DNA-Proben werden). Ersetzen Sie die nassen Whatman Papiere mit trockenem solche, die nach 2 und 4 Stunden zum Trocknen bzw.. Keep Trocknen, bis das Gel vollständig trocken ist. Dies kann über Nacht dauern, je nach dem Trockner.
Schritt 5. Beschriften Sie die Oligo-Sonden
Mix 1 ul 50 ng / ul TELC oder TELG Oligo mit 1 ul 10 x Polynukleotid-Kinase (PNK)-Puffer, 5 ul γ [32P] ATP, 2 &mgr; l ddH 2 O und 1 ul T4 PNK. Bei 37 ° C für 1 Stunde. Add 90 &mgr; l TNES Puffer (10 mM Tris Cl, pH 8,0, 100 mM NaCl, 10 mM EDTA, pH 8,0, 1% SDS) zum Reaktionsgemisch.
Während die Kennzeichnung der oligoes, bereiten Sie einen G-25-Säule: put einige Glaswolle in eine 3 ml-Spritze und stopfen es sich eng mit einer Pipettenspitze. Füllen Sie die Spritze mit Sephadex G-25 fein (autoclAved in TE), um die 3 ml-Markierung. Lassen Sie es Gew. begleichen. Zur Reinigung der markierten Sonde: Laden Sie die Sonde auf der Säule. Waschen mit 700 ul TNES Puffer und eluiert mit 600 ul der TNES Puffer. Alternativ zu reinigen der markierten Sonde mit einem Mini Quick Spin ™-Säule (Roche Applied Science).
Schritt 6: Hybridisierung
- Kurz nass das getrocknete Gel in destilliertem Wasser und wäscht alle Whatman-Papier auf das Gel stecken
- Legen Sie das Gel in eine Hybridisierung Tasche und 20 ml hybridiztion Puffer (0,25 M Na 2 HPO 4 pH 7,2, 1 mM EDTA, 7% SDS, 1% BSA, durch 0,22 um-Filter filtriert). Inkubieren im Wasserbad bei 50 ° C unter leichtem Schütteln für mindestens 1 Stunde.
- Entfernen Sie die Pre-Hybridisierungspuffer. Kochen Sie die markierte Sonde für 5 min und fügen Sie ihn auf 25 ml frischem Hybridisierungspuffer, Filter-sterilisiert die Mischung mit einem 0,22 um Spritzenfilter und fügen Sie die Hybridisierung Mix direkt auf die Hybridisierung Tasche. Seal die Tasche und in einen flachen Behälter mit warmem Wasser. Platzieren Sie den gesamten Behälter in das Wasserbad. Inkubation bei 50 ° C über Nacht unter leichtem Schütteln.
- Schneiden Sie eine kleine Öffnung in der Hybridisierung Tasche. Gieße so viel wie möglich der Hybridisierung mischen und speichern Sie sie in einem 50 ml konischen Rohr. Halten Sie die Sonde bei -20 ° C eingefroren Entfernen Sie das Gel aus der Hybridisierung Tasche und steckte es in einen Container.
Bei diesem Schritt ALLES ist heiß und NEEDS umfangreiche Reinigung (BENCH, SCREEN, SCHERE, ETC. UNBEDINGT Doppelschichten Handschuhe zu tragen! - Wash Gel in 4 x SSC für 30 min bei 50 ° C. Ersetzen Sie mit frischem Waschpuffer und zweimal wiederholen.
- Wash Gel in 4 x SSC/0.1% SDS bei 50 ° C.
- Legen Sie das Gel auf einem Stück von 3 mm Whatman-Papier und leicht trockene Gel.
- Wickeln Sie das Gel mit Plastikfolie und aussetzen Phosporimager für ~ 3 Tage
- Scan der Phosporimager
- Inkubieren des Gels in denaturierenden Puffer (1,5 M NaCl, 0,5 M NaOH) bei RT für 30 min
- Wash Gel in Neutralisationspuffer (3 M NaCl, 0,5 M Tris • / Cl pH 7,0) bei RT für 30 min
- Spülen in destilliertem Wasser für 3 min
- Bei 55 ° C Pre-Hybridisierung für 1 Stunde
- Wiederholen Sie die Hybridisierung mit der gleichen Sonde enthaltenden Hybridisierung Mix bei 55 ° C über Nacht.
- Wie oben beschrieben waschen beschrieben, außer bei 55 ° C.
- Wickeln Sie das Gel und aussetzen für ca. 2 Std. Phosporimager.
- Scan der Phosporimager. Normalisieren der Hybridisierung Signal vor Denaturierung mit, dass nach Denaturierung gewonnen wird.
2. Bestimmen Sie die Telomere Terminal-Nucleotide durch Adaptor Ligation 6
Prinzip (Abb. 2A und 2B): A 5 Ende des G-Überhang-Ende-markierten einzigartige Oligonukleotid ist die 3 ligiert. " Dies wird durch Glühen auf bestimmte ergänzende Anleitung Oligo durchgeführt. Nur die einmalige / guide Oligo-Adapter tragenden Sequenzen kompatibel zu den Telomeren G-Überhang terminalen Sequenzen wird ligiert werden. Für T. brucei, kann die Telomere in einem der sechs verschiedenen Nukleotide der TTAGGG wiederholt zu kündigen. Daher sechs verschiedene Führer oligoes verwendet werden (TG1-TG6, Abbildung 2a). Nach der Ligation wird die DNA mit Restriktionsendonukleasen verdaut und aufgelöst auf einem Agarosegel.
- Kinase-Behandlung der einzigartigen Oligo.
Mix 6 ul von 10 pmol / ul einzigartige Oligo mit 3 uL 10x PNK-Puffer, 5 ul γ [32 P] ATP, 14 uL ddH 2 O und 2 ul (10 U) T4 PNK. Inkubieren Sie die Mischung bei 37 ° C für 1 Stunde. - Entfernen unincorporated heißen Nukleotid.
Verwenden Qiaquick Nukleotid Demontagesatz die unincorporated heißen ATP zu entfernen und eluieren Ende markierte Oligos mit 60 ul Elutionspuffer (Endkonzentration wäre ca. 1 pmol / ul). - Glühen einzigartige oligo zu führen Oligo (um den Adapter zu erstellen).
Fügen Sie 10 ul des gereinigten einzigartige Oligo bis 2 ul jeder Führung Oligo-und 1 ul Annealingpuffer (200 mM NaCl, 100 mM Tris-HCl pH 8,0). Setzen Sie Rohre in ein 85 ° C Hitze blockieren und schalten Sie die Hitze zu blockieren. Lassen Sie die Röhrchen und Heizblock cool, RT. Das dauert ein paar Stunden. Wenn es eilig, Rohrweiche nach jeweils 5 min auf 65 ° C, dann 37 ° C und dann abkühlen lassen auf RT. - Ligieren Adapter gesamten genomischen DNA.
Um 2,5 ul intakter genomischer DNA, fügen Sie 4 ul 5 x Ligase-Puffer, 10 uL geglüht Oligos, 2,5 ul ddH 2 O und 1 ul T4 DNA Ligase. Inkubation bei 16 ° C über Nacht. - Restriktionsendonukleaseverdau
Um 20 uL Ligationsprodukt add 3 ul von 10 x NEB-Puffer 4, 1,5 ul AluI und MboI jeder, und 4 ul ddH 2 O. Inkubieren Sie die Mischung bei 37 ° C über Nacht. - Elektrophorese.
Add 3 ul von 10 x Orange G Farbstoff (50% Glycerin, 0,5% Orange G) zu jeder Probe. Laden Sie die DNA-Proben auf einem 20x20 cm 0,7% Agarosegel in 0,5 x TBE mit 0,2 ug / ml Ethidiumbromid. Run bei 30V für 30 min, dann ciederholt mit höherer Spannung, aber weniger als 120 auf insgesamt 1000 v hr. Machen Sie ein Foto des Gels mit einem Lineal neben der DNA-Marker. - Dry-Gel und freizulegen.
Legen Sie die Agarosegel auf einer Schicht aus DE81 Filterpapier und zwei Schichten von 3 mm Whatman-Papier und decken das Gel mit Plastikfolie. Dry-Gel bei RT. Expose das getrocknete Gel über Nacht Phosporimager.
3. Ligation vermittelte Primer Extension 6
Prinzip (Abbildung 2A & 2C): Ein einzigartiges Oligonukleotid ist es, die 3 'G-Überhang mit bestimmten terminalen Sequenz ligiert, bestimmt durch ihre komplementären Sequenzen in den Adapter (Führer) Oligo. Nach der Reinigung wird der markierte Führung Oligo dass geglüht, um die G-overhang/unique Oligo bleibt Primer erweitert, um die ss-ds Kreuzung mit Hilfe der T4-DNA-Polymerase, die Strand-Displacement-und 5'-3 'Exonuklease-Aktivitäten fehlt. Die Primer-Extension-Produkte daher geben die Länge des G-Überhang.
- Kinase-Behandlung der Führung und einzigartige oligoes
- Mix 20 ul von 10 pmol / ul einzigartige Oligo mit 10 ul 10 x PNK-Puffer, 20 ul 5 x Ligase-Puffer (enthält 10 mM ATP), 3 ul (30 U) T4 PNK, und 47 ul der ddH 2 O . Inkubieren Sie die Mischung bei 37 ° C für 60 Minuten.
- Mix 1 ul von 10 pmol / ul Führung Oligo mit 1 ul 10 x PNK-Puffer, 2 ul der γ [32 P] ATP, 1 ul (10 U) T4 PNK, und 5 ul ddH 2 O. Inkubieren Sie die Mischung bei 37 ° C für 60 Minuten.
- Glühen Führung Oligo-und einzigartige Oligo
Add 10 l (20 pmol) der phosphorylierten einzigartige Oligo-und 10 l (10 pmol) der Ende-markierten Oligo Führung in einem 1,5 ml Eppendorf-Röhrchen (das 2:1-Verhältnis von einzigartigen Oligo Guide sorgt dafür, dass alle Führer Oligo geglüht wird ). Folgen Sie dem gleichen Verfahren in Protokoll II zum Glühen oligoes beschrieben. - Ligieren des geglüht guide / einzigartige Oligo-Adapter an Telomeren endet
Folgen Sie dem gleichen Verfahren in Protokoll II zur DNA-Ligation beschrieben. - Niederschlag DNA mit 1 / 10 Volumen Natriumacetat und 2,5 Volumen eiskaltem Ethanol bei -80 ° C für 30 min. Spin down DNA-Pellet bei 12 krpm, 4 ° C für 15 min. Wash-DNA mit 70% Ethanol und lösen Palette in 10 ul ddH 2 O.
- Primer Extension
Um jeweils 10 ul DNA-Probe add 2 ul von 10 x T4-DNA-Polymerase-Puffer, 1 ul 1 mg / mL BSA, 1 ul 25 mM dATP, dCTP und dTTP je 1 ul (3 U) T4 DNA Polymerase, und 3 ul ddH 2 O.
Machen Sie einen Master-Mix mit der Polymerase, Puffer, etc. und aliquoten entsprechende Menge des Master-Mix für jede DNA-Probe. Inkubation bei 30 ° C für 30 min. Dann fügen Sie 1,2 ul 0,5 M EDTA sofort auf die Reaktion. - Elektrophorese
Run Extension-Produkte auf einem 10% Acrylamid / 7 M urea/1xTBE Gel. Last 4 ul jeder Probe. Kochen Sie die Proben für 10 Minuten vor dem Laden. Run auf 800V in 1x TBE, bis der blaue Farbstoff erreicht den Boden des Gels. Trocknen Sie das Gel bei 65 ° C für 30 min bei RT für 30 min und setzen für 2 Std. Phosporimager.
Zur Herstellung von 100 ml 10% Polyacrylamid-Gel, lösen sich 41 g Harnstoff und 21 ml 40% Acrylamid-Lösung (Acrylamid / Bisacrylamid 29:1) in 30 ml destilliertem Wasser, 10 ml 10x TBE. Montieren Glasplatten. Rechts vor dem Gießen des Gels, 1 mL 10% APS und 100 ul TEMED. Gießen Sie das Gel und versuchen, alle Blasen zu vermeiden.
4. Repräsentative Ergebnisse:
Detect T. brucei Telomer G-Überhang Struktur mit nativen in-Gel-Hybridisierung
Intact T. brucei Chromosomen durch PFGE getrennt sind in Abbildung 3A und 3B (linkes Bild) dargestellt. T. brucei Zellen enthalten in der Regel ca. 100 Kopien Minichromosomen, alle mit ähnlicher Größe (50-150 kb) und wandern in der gleichen Position auf dem Gel (MC). Aufgrund dieser Tatsache, nach Hybridisierung mit TELC Oligo-Sonde, die Telomer-G-Überhang Signal prominentesten auf Minichromosomen (Abbildung 3A, Mitte). Hybridisierung mit Oligo TELG Sonde in der Regel liefert keine Hybridisierungssignal (Abbildung 3B, Mitte), weil T. brucei Zellen nicht haben Telomer C-Überhang Struktur. Nach der Denaturierung und Neutralisation, Hybridisierung mit entweder TELC oder TELG Oligo-Sonde sollte Telomer-Signale auf allen Chromosomen-Enden (Abbildung 3A & 3B, rechts Panels) zu offenbaren.
Bestimmen Sie die Telomer-terminalen Nukleotid von Adaptorligation
Loading gleiche Menge an DNA in jeder Spur ist von wesentlicher Bedeutung für diesen Test, und dies wird durch Ethidiumbromid-Färbung des Gels gezeigt. Ein Beispiel ist in Abbildung 4A, linke Tafel dargestellt.
In diesem Protokoll werden die Ende-markierten Oligo einzigartig und führen Oligo-Adapter nur auf Chromosom Ende ligiert, wenn die Telomere G-Überhänge mit der Endung-Sequenzen, die mit der Führung oligo sind. Si nce die einzigartige Oligo ist endmarkiert, wird das Ligationsprodukt radioaktiv sein und ein starkes Signal. Wie in Abbildung 4A, rechte Tafel zeigt, gibt Spur 1 ein starkes Signal, was anzeigt, dass eine große Menge von unique/TG1 Adapter an die Telomer-Ende wurde unterbunden. TG1 hat eine Endsequenz von 5'CCCTAA3 ". Daher die Telomer-G-Überhänge an diesen Adapter Ende in 5'TTAGGG3 'ligiert. Keine signifikante Menge an Ligationsprodukte beobachtet mit anderen Führer oligoes, was darauf hinweist, dass die Telomere der Endung TTAGGG vorherrschende in T. sind brucei Zellen.
Die Behandlung der genomischen DNA mit Exo I oder Exo T, die 3 'Überhang Nukleasen sind, führte zu einem Verlust der Ligationsprodukte (Abbildung 4A, rechte Tafel, Spuren 2 & 3), was darauf hinweist, dass die Ligation der Tat ergab sich aus der Telomer-G- Überhang-Struktur
Ligation vermittelte Primer Extension
Ohne Ligation, ist das Ende-markierten Führung Oligo 22 nt lang. Loading endmarkiert Führung Oligo würde als negative Kontrolle und eine Größe Marker dienen. (Abbildung 4B, Spur 11, zeigt Sternchen Ende markierte Führung oligo). Wir in der Regel auch beobachten, eine Bande von ~ 44 nt in dieser Negativ-Kontrolle (Abb. 4B, Spur 11, offene Dreiecke), die am ehesten Rest nichtdenaturiertem Adapter sind. Nach Ligation an das Chromosom Ende spiegelt die Größe des erweiterten Produkt der Länge der G-Überhang Struktur. Die meisten T. brucei Telomer G-Überhänge Ende in 5'TTAGGG3 "(Abbildung 4A). Allerdings scheinen diese G-Überhänge sehr kurz (nur ~ 10 nt lang), als Produkte aus der ligiert TG1 Führung Oligo verlängert werden nicht viel länger als die Führung Oligo selbst (4B, Spur 10), aber sie erlauben Ligation TG1-Adapter (Abbildung 4A, rechte Tafel, Spur 1). Obwohl nur eine kleine Menge von TG4 Adapter an die Telomer-Enden (Abbildung 4A, rechte Tafel, Spur 6) ligiert wurde, sind Produkte aus ligiert TG4 erweiterten viel länger (Abb. 4B, Spur 7, Pfeilspitzen), mit der längsten Produkts ~ 55 nt. Daher beobachten wir zwei Arten von Telomer-G-Überhang in T. brucei Zellen. Der überwiegende Telomer G-Überhänge Ende in 5'TTAGGG3 'sind aber nur ~ 10 nt lang. Wenige Telomer G-Überhänge Ende in 5'GGGTTA3 ', kann aber bis zu 40 nt lang sein. Beide Arten von G-Überhang sind empfindlich gegen Exo T (oder Exo I)-Behandlung (Abbildung 4A, rechte Tafel, Spur 2, 3 und 7; 4B, Spur 1, Pfeile).

Abbildung 1. Das Prinzip der einheimischen In-Gel-Hybridisierung. Linken, unter den nativen Zustand, der Ende-markierten Oligo-Sonde TELC kann nur mit den einzel-G-reiche Telomer Überhang hybridisieren. Die Intensität der Hybridisierung spiegelt die Länge der Telomere G-Überhang. Richtig, nach Denaturierung wird die TELC-Oligo-Sonde mit allen Telomer-DNA hybridisieren. Die Intensität dieser Hybridisierung ist der Gesamtbetrag der Telomer-DNA und wird als Ladekontrolle verwendet, und die endgültige G-Überhang wird durch Division des nativen Hybridisierungssignal durch, dass nach Denaturierung erhalten wird, berechnet.
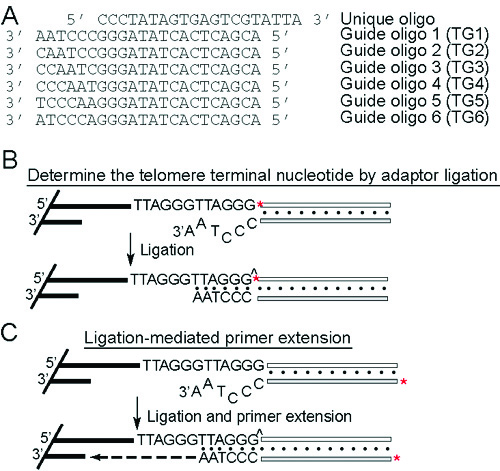
Abbildung 2. Das Prinzip der Adaptor-Ligation und Ligation vermittelte Primer-Extension. (A) Sequenzen der einzigartigen und sechs Führer oligoes. (B) Bestimmen Sie die Telomer-terminalen Nukleotid von Adaptorligation. Die einzigartige Oligo ist endmarkiert (markiert mit einem roten Sternchen), geglüht, um die Führung oligoes und ligiert, um das Telomer Ende. Nur wenn die einzigartige / guide-Adapter eine 3-Überhang, der kompatibel mit dem Terminal Telomersequenz wird der Adapter ligiert werden Bären. (C) In Ligation vermittelte Primer-Extension ist die Führung Oligo-endmarkierten (markiert mit einem roten Sternchen) und geglüht, um die einzigartige Oligo vor, um die Chromosomen-Enden ligiert. Nur die einmalige / guide-Adapter trägt einen 3-Überhang mit dem G-Überhang wird ligiert werden. ^ Zeigt die ligiert phophordiester Bindung. Nach dem Entfernen des unligierten Oligo-Paare werden Primer Extension mit T4-DNA-Polymerase, die Strand-Displacement-und 5'-3 'Exonuklease-Aktivitäten fehlt durchgeführt werden. Die endgültige Länge der erweiterten Führung Oligo spiegelt daher die Länge des G-Überhang.
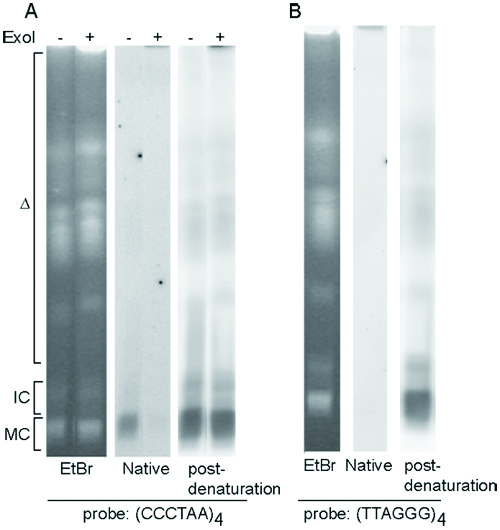
Abbildung 3. T. brucei Telomer G-Überhang Struktur analysiert, von Muttersprachlern in-Gel-Hybridisierung. (A) In-Gel-Hybridisierung mit TELC Oligo-Sonde. (B) In-Gel-Hybridisierung mit TELG Oligo-Sonde. In beiden (A) und (B) links, Ethedium Bromid gefärbt PFG. Middle, native Hybridisierung führen. Richtig, post-Denaturierung Hybridisierung führen. Wild-Typ T. brucei-Zellen verwendet wurden. Intakte oder Exo I behandelt genomische DNA wurden durch PFGE getrennt. Offene Dreieck steht für die Megabasen Chromosomen; IC, mittelgroße Chromosomen; MC, Minichromosomen.
"/>
Abbildung 4. T. brucei Telomer G-Überhang Struktur durch Ligation-vermittelte Primer-Extension analysiert. (A) Die meisten T. brucei Telomer G-Überhänge enden in 5'TTAGGG3 ". Left, Ethedium Bromid angefärbt DNA-Gel. Etwa die gleiche Menge DNA ist in jeder Spur geladen. Richtig, Exposition aufgrund der gleichen Gel nach dem Trocknen. Intakt, Exo I-behandelt oder Exo T-behandelter DNA wurde zu sechs verschiedene endmarkiert einzigartige / guide Oligo-Adapter ligiert, wie angegeben. (B) T. brucei Telomere haben kurze G-Überhänge. Intakte oder Exo T-behandelten genomischen DNA wurde zu sechs verschiedene einzigartige / guide Oligo-Adapter von Primer Extension mit Hilfe der T4-DNA-Polymerase, gefolgt ligiert. End-markierten TG4 Oligo wurde als negative Kontrolle (Spur 11) geladen. Die Oligo selbst läuft bei 22 nt (*), sondern gab auch ein schwächeres Signal bei ~ 44 nt (Δ). Nur unique/TG4 Adapter ergab Verlängerung Produkte deutlich länger als die Führung Oligo selbst (Pfeilspitzen, Spur 7).
Diskussion
Trypanosoma brucei verursacht Schlafkrankheit beim Menschen. Diese Krankheit, die unbehandelt unweigerlich tödlich. T. brucei Zellen in Säugetierwirten unterziehen antigene Variation regelmäßig, so dass das Immunsystem des Wirts Attacke 7 auszuweichen. Daher ist es sehr schwierig, T. beseitigen brucei Zellen einmal eine Infektion festgestellt wird. Wir haben kürzlich gezeigt, dass die Telomere eine wichtige Rolle spielen bei der Regulierung der Expression von T. br...
Offenlegungen
Danksagungen
Wir möchten Dr. Carolyn Preis für wissenschaftliche Diskussionen und aufschlussreiche Anregungen bedanken. Diese Arbeit wird durch NIH AI066095 (: Bibo Li PI) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| SeaKem LE Agarose | Lonza Inc. | 50004 | |
| Agarose Type VII (low melting agarose) | Sigma-Aldrich | A4018 | |
| Proteinase K, recombinant, PCR grade | Roche Group | 03 115801001 | |
| Exo I | New England Biolabs | M0293 | |
| Exo T | New England Biolabs | M0265 | |
| T7 exonuclease | New England Biolabs | M0263 | |
| T4 DNA polymerase | New England Biolabs | M0203 | |
| QIAquick nucleotide removal kit | Qiagen | 28304 |
Referenzen
- Munoz-Jordan, J. L., Cross, G. A., de Lange, T., Griffith, J. D. t-loops at trypanosome telomeres. EMBO J. 20, 579-588 (2001).
- Li, B., Espinal, A., Cross, G. A. M. Trypanosome telomeres are protected by a homologue of mammalian TRF2. Mol Cell Biol. 25, 5011-5021 (2005).
- Wellinger, R. J., Wolf, A. J., Zakian, V. A. Saccharomyces telomeres acquire single-strand TG1-3 tails late in S phase. Cell. 72, 51-60 (1993).
- McElligott, R., Wellinger, R. J. The terminal DNA structure of mammalian chromosomes. EMBO J. 16, 3705-3714 (1997).
- Harrington, L. A., Greider, C. W. Telomerase primer specificity and chromosome healing. Nature. 353, 451-454 (1991).
- Jacob, N. K., Skopp, R., Price, C. M. G-overhang dynamics at Tetrahymena telomeres. Embo J. 20, 4299-4308 (2001).
- Barry, J. D., McCulloch, R. Antigenic variation in trypanosomes: enhanced phenotypic variation in a eukaryotic parasite. Adv Parasitol. 49, 1-70 (2001).
- Yang, X., Figueiredo, L. M., Espinal, A., Okubo, E., Li, B. RAP1 is essential for silencing telomeric variant surface glycoprotein genes in Trypanosoma brucei. Cell. 137, 99-109 (2009).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten