Method Article
RNAi detección de los factores del huésped implicados en Vaccinia Infección del virus utilizando Drosophila Celdas
En este artículo
Resumen
Nuevos factores de acogida implicados en la infección viral puede ser identificado a través de celulares basados en el genoma pérdida de la función de detección de RNAi. A Drosophila modelo de cultivo es particularmente susceptible a este enfoque debido a la facilidad y la eficiencia de RNAi. Aquí se demuestra que esta técnica con Vaccinia Virus como un ejemplo.
Resumen
Patógenos virales representan una amenaza para la salud pública, no sólo puede causar epidemias de virus natural de la enfermedad humana, pero su uso potencial en bioterrorismo es también una preocupación. Una mejor comprensión de los factores celulares que la infección por el impacto que faciliten el desarrollo de la muy necesaria la terapéutica. Los avances recientes en el ARN de interferencia (RNAi), junto con la tecnología de secuenciación completa del genoma de varios organismos ha llevado a la optimización de las pantallas de todo el genoma, a base de células de pérdida de función. Células de Drosophila son particularmente susceptibles a las pantallas a escala del genoma debido a la facilidad y eficacia de RNAi en este sistema 1. Es importante destacar que una amplia variedad de virus que pueden infectar a las células Drosophila, incluyendo un número de virus de mamíferos de importancia médica y agrícola 2,3,4. Anterior pantallas de RNAi en Drosophila se han identificado los factores del huésped que se requieren para diversas etapas de la infección por virus, incluida la entrada, la traducción y la replicación del ARN 5. Por otra parte, muchos de los factores celulares necesarios para la replicación viral en cultivo de células de Drosophila también son limitantes en las células humanas infectadas con estos virus 4,6,7,8, 9. Por lo tanto, la identificación de los factores del huésped cooptado durante la infección viral se presentan nuevos objetivos para la terapia antiviral. Aquí se presenta un protocolo generalizado de una pantalla de alto rendimiento RNAi para identificar los factores celulares implicados en la infección viral, utilizando virus vaccinia como un ejemplo.
Protocolo
Parte 1: RNAi en 384 placas
- La estandarización de los reactivos utilizados para la investigación es esencial. Probamos lotes individuales de los medios de comunicación de Schneider, suero fetal bovino, y reactivos de coloración. Luego alícuota y el uso del mismo lote de toda la pantalla.
- Para cualquier ensayo, lo mejor es probar varias líneas celulares diferentes para determinar cuál es más susceptible a la biológica de lectura. Por lo general el uso de la línea S2-DRSC.
- Se cultivan las células Drosophila a 25 ° C en los medios de comunicación completa de Schneider.
- Schneider medios de comunicación
- 10% de SFB inactivado por calor
- 1X L-glutamina
- 1X penicilina / estreptomicina los antibióticos
- Las células de Drosophila son semi-adherente y se pasan sin tripsinización cada cuatro días con la pipeta las células de los frascos y la dilución de las células de 1:10 en un matraz fresco. Las células deben estar en fase de registro para los experimentos.
- Placa a placa y la variabilidad del día a día se reducen al mínimo mediante el uso de manejo de líquidos automatizados para todas las adiciones a varios líquidos y placas. Utilizamos un WellMate (Matrix).
- Preparar tubos WellMate.
- Spray campana y WellMate con etanol al 70%.
- Remueva el tubo de empaque y rociado con etanol al 70%.
- Inserte un cassette tubería en la dispensación de la posición en la WellMate.
- El primer tubo con 25 ml de etanol al 70%.
- Esterilice los tubos en etanol durante 15 minutos, el entonces primer con otro de 25 ml de etanol.
- Enjuague la tubería de la preparación con 50 ml de PBS estéril.
- Separar las células del frasco y el recuento. Precipitar las células para que tenga un 25% más de lo necesario (300xg, 5 min). El número de células sembradas por pocillo debe ser optimizada de acuerdo a la longitud de ensayo. Al llevar a cabo análisis automatizado de imágenes, una sola monocapa sin grumos de células es ideal para la segmentación de la imagen.
- Volver a suspender las células sedimentadas en el suero libre de Schneider en los medios de comunicación 1.7x10 6 células / ml.
- Realizamos la pantalla en 384 placas pre-alícuotas con una biblioteca disponible en el mercado de dsRNA a una concentración de 250ng/well. Varios pozos de cada placa se deja en blanco para los controles, que se añaden manualmente. Usamos dsRNA a luciferasa como control negativo. Usamos dsRNA contra Thread (DIAP) como control de sólidos RNAi. Derribar los resultados de este factor anti-apoptóticos de la muerte celular dramático y es una manera rápida visual para evaluar la calidad de la derribarlo. Por último, se utiliza un dsRNA viral contra el periodista, en este caso de beta-galacosidase, como un control positivo para validar que pueden inhibir la infección viral con nuestro ensayo de lectura. Si el goteo dsRNA en el plato en el momento de la siembra de células, una alícuota de dsRNA l en la esquina del pozo para permitir una mejor visualización, centrifugar dsRNA contra el fondo del pozo (300xg, 1 min) antes de añadir las células.
- Use WellMate añadir 10 celdas l preparada anteriormente a cada pocillo.
- Girar las células en las placas mediante la centrifugación de 300xg, 1 min.
- Incubar en 25 ° C incubadora durante 45 minutos.
- Añadir 20 l completo de Schneider medios de comunicación y centrifugar 300xg, 1 min.
- Coloque las placas en humidificado recipientes Tupperware cubierto con toallas de papel empapado en agua. El volumen de un 384 y es pequeña, por lo tanto, la evaporación puede tener grandes efectos en la biología, que a su vez puede conducir a cambios de artefactos en las salidas de lectura de los pozos de borde. Para superar esto, las placas deben mantenerse en condiciones de humedad alta.
- Incubar durante tres días para permitir la caída fuerte. No hay necesidad de cambiar los medios de comunicación o de abrir los contenedores humidificado durante este tiempo.
Parte 2: Infección con el virus vaccinia
- Esterilizar multi-canal de aspiración en Quatricide durante 15 minutos, luego enjuague con etanol al 70%.
- Prepare WellMate como se describió anteriormente.
- Prepare los medios de comunicación de Schneider con FBS al 2% para la infección mediante la dilución de 1:05 completar medios de comunicación en medio libre de suero.
- Acciones filtro de virus crudo a través de una jeringa 0,8 um filtro para eliminar los desechos celulares o el uso de virus purificado.
- Diluir el virus vaccinia en el suero de Schneider 2% los medios de comunicación para obtener la multiplicidad de infección (MOI), de 2.
- Quitar de la tubería WellMate PBS, e imprima con virus diluido hasta que el tubo esté libre de burbujas de aire.
- Use esterilizados multi-canal de aspiración y colector de vacío para extraer con cuidado los medios de comunicación de las placas de dsRNA tratados con células de Drosophila.
- Prescindir de 30 virus por pocillo en placas con el WellMate.
- Centrifugar las placas 1 minuto a 300xg.
- Esterilizar tubos WellMate. Primer con 25 ml de 10% bleach, espere 10 minutos, y la primera con otro de 25 ml de lejía al 10%.
- El primer tubo con 50 ml de agua, seguido de 50 ml de etanol al 70%. Volver a la tubería de su embalaje.
- Volver a las placas humidificado recipiente Tupperware. Se incuba a 25 ° C durante 48 horas.
Parte 3: Placas de tinción
- Fijar placas en 15 l de formaldehído al 4% / PBS durante 10 minutos. Líquido se elimina con un aspirador de multi-canal y colector de vacío a cada paso. Una vez que las placas se han fijado, equipos y reactivos estériles ya no son necesarios.
- Quitar reparar y utilizar WellMate añadir 30 l PBST (PBS + 0,1% Triton-X). Incubar 10 minutos y repita. Operar WellMate como se describió anteriormente, excepto el tubo no necesita ser esterilizado.
- Para detectar la infección por vaccinia se utiliza un reportero β-galactosidasa impulsado por un promotor temprano / tardío vaccinia.
- Eliminar las células de líquido y se incuba en el bloque (PBST con un 2% BSA) durante 10 minutos.
- Diluir el anticuerpo primario en el bloque, eliminar el líquido y añadir 15 l de anticuerpos a cada pipeta repetir así el uso de múltiples canales (Matrix).
- Girar las placas (300xg, 1 min), cubierta con una etiqueta clara, e incubar a 4 ° C durante la noche.
- Eliminar el anticuerpo primario, y el uso de la WellMate para lavar los pocillos 3 veces con PBST, a 10 minutos cada uno.
- Diluir el anticuerpo secundario marcado con fluorescencia (FITC) y la tinción nuclear en el bloque, y añadir 15 l de pozos utilizando una pipeta multicanal repetir.
- Incubar las placas 1 hora a temperatura ambiente, protegido de la luz.
- Lave los pozos en PBST 3 veces, 10 minutos cada uno con el WellMate.
- Selle la placa con la etiqueta engomada transparente, tapar y guardar a 4 ° C hasta tres semanas.
Parte 4: Imágenes y Análisis de Imágenes
- Utilizar un microscopio automatizado a 20X a las placas de la imagen.
- Capturar imágenes de los núcleos total (DAPI) y las células infectadas (FITC). Optimizar el tiempo de exposición para cada canal por separado con el fin de maximizar el rango dinámico.
- Ajustar la función de enfoque automático mediante la adopción de una pila Z de imágenes, 1um separados, que abarcan los aviones por encima y por debajo del plano de espera de atención. Una vez que el plano focal óptima se encuentra, ajustar la configuración de enfoque automático en consecuencia.
- La imagen de toda la placa, la captura de al menos tres imágenes por bien tanto en el canal de Hoechst (DAPI) y el canal de virus (FITC). Elegir los sitios de distintas regiones dentro de un pozo con el fin de capturar las imágenes que representan el bien en su conjunto.
- Use MetaXpress software para escribir un diario para segmentar la imagen y el uso de módulos de Conde núcleos para calcular el número total de células positivas para la tinción nuclear (el total de células) y la infección (FITC) para cada plato.
- Calcular la infección por ciento (100 * Las células FITC positivos / total) y el registro de transformar.
- Para identificar a los candidatos que utilizan un sólido puntaje Z basada en la mediana y rango intercuartil de cada plato. Este método no es sensible a los valores atípicos y los datos no simétrica, y por lo tanto proporciona una mayor potencia en la identificación de éxitos, incluso débil o moderada 10. Para cada plato, la mediana se resta de los datos de transformación logarítmica. A continuación, los valores resultantes se dividen por 0,74 veces el rango intercuartil (la diferencia entre los valores de la percentil 75 y el percentil 25). El factor 0,74 se utiliza para aproximar una distribución normal estándar.
- El robusto puntaje Z es una medida de la distancia en desviaciones estándar de la mediana de la placa. Por ejemplo, un robusto Z-score de 2 indica que la infección% del pozo es de ~ 2 desviaciones estándar de la mediana o la placa de p <0,05.
- Analizar el número de células para identificar a los candidatos citotóxicos. Calcular el puntaje Z de robusta cuenta nucleares. Los candidatos con una sólida Z <-2 en pantallas duplicadas son consideradas citotóxicos.
- Los pozos que tienen un sólido puntaje Z para la infección por ciento de> 2 o <-2 en pantallas duplicadas (p <0,0025), sin citotoxicidad se consideran candidatos potenciales de la pantalla principal.
- Candidatos positivos son validados utilizando reactivos independientes. dsRNAs se generan en contra de los genes de interés de otra región del ARNm y probado que el anterior.
- Los genes de los cuales dos dsRNAs independientes tienen el mismo fenotipo puede ser considerado para el estudio adicional.
Parte 5: Imágenes Representante e interpretación de las tasas de infección
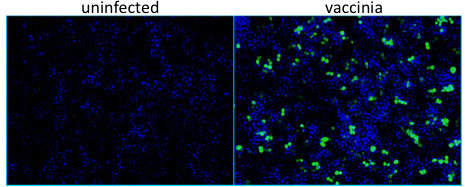
Los pozos de la figura 1. Infectadas no muestran manchas de proteínas de virus de la vacuna, mientras que un representante de infectados y contiene células con tinción positiva para la vacuna-expresó beta-galactosidasa (βgal) de proteínas, medida por microscopía de inmunofluorescencia.Virus = verde, azul = núcleos.
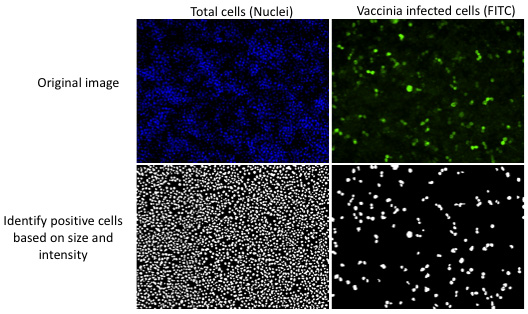
Figura 2. Software automatizado de análisis de imágenes cuantifica la infección en cada imagen con los parámetros establecidos por el usuario. En una imagen representativa, el número total de núcleos celulares y el número de células que expresan el antígeno viral se contabilizan en función del tamaño y la intensidad de la tinción por encima de la tinción de fondo local.

Figura 3. Derribo de hilo (DIAP) sirve como un control positivo para el agotamiento de RNAi robusta de los genes diana. Caída de este factor anti-apoptóticos conduce a la muerte celular dramático. Núcleos = azul.
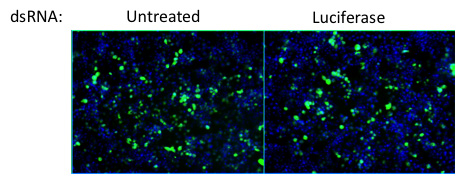
Derribo Figura 4. De la luciferasa sirve como control negativo para el efecto de la doble cadena de ARN en el tratamiento de la infección. El agotamiento de la luciferasa no tiene ningún efecto sobre la infección en relación con las células no se tratan. Virus = verde, azul = núcleos.

Figura 5. Derribo de la proteína βgal sirve como un control positivo de una reducción de la infección, ya que el ensayo utiliza los niveles de proteína βgal como una lectura de la infección. El agotamiento de los resultados βgal en una disminución en el porcentaje de células infectadas. Virus = verde, azul = núcleos.
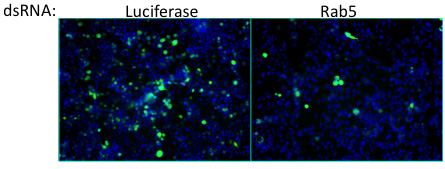
Figura 6. Derribo de celulares factor de Rab5 resulta en una disminución en el porcentaje de células infectadas. Desde Rab5 se sabe que participan en la endocitosis, este factor probablemente contribuye a la entrada del virus vaccinia. Esto representa un valor atípico ejemplo de la pantalla. Virus = verde, azul = núcleos.
Discusión
Todo el genoma de Drosophila RNAi de detección proporciona un método robusto y eficiente para el examen de los componentes celulares de la infección viral. Un número de virus que infectan a las células de mamíferos Drosophila, que puede ser utilizado para identificar los componentes de la respuesta inmune del huésped intrínseco, y los factores del huésped proteínas necesarias para la replicación viral que puede representar nuevas dianas terapéuticas. Antes de realizar una pantalla, el ensayo debe ser cuidadosamente optimizado para el tipo de células, el número de células, y el nivel de infección, y debe estar bien controlado, tanto positivos como negativos virales y dianas celulares 11. Es importante garantizar una separación suficiente entre los controles positivos y negativos previos a la selección para maximizar el rango dinámico de la prueba. Una vez que la pantalla se ha realizado, los candidatos se pueden dividir en categorías funcionales para determinar qué mecanismos de acogida contribuyen a la infección. De virus de mamíferos, tales como la vacuna, la contribución de los homólogos mamíferos de RNAi se debe determinar como parte del análisis secundario. En conjunto, estos métodos de detección robusta nos permitirá comprender mejor los complejos mecanismos por los cuales los virus interactúan con las células de su huésped.
Divulgaciones
Agradecimientos
Este trabajo fue apoyado por subvenciones del NIAID (R01AI074951, U54AI057168) y el Penn Genoma Fronteras Instituto de Carolina del Sur, de los NIH T32HG000046 de TSM, de los NIH y T32GM07229 T32AI007324 a LRS.
Materiales
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
Referencias
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions?. Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados