Method Article
Lo screening RNAi per fattori di accoglienza che partecipano Vaccinia Infezione da virus utilizzando Drosophila Celle
In questo articolo
Riepilogo
Fattori dell'ospite romanzo coinvolti nella infezione virale può essere identificato tramite cell-based genome-wide perdita della funzione di screening RNAi. A Drosophila modello di coltura è particolarmente suscettibile a questo approccio a causa della facilità ed efficienza di RNAi. Qui mostriamo questa tecnica utilizzando Vaccinia come esempio.
Abstract
Patogeni virali rappresentano un pericolo significativo per la salute pubblica, non solo può causare epidemie virus naturale della malattia umana, ma il loro uso potenziale nella bioterrorismo è anche una preoccupazione. Una migliore comprensione dei fattori cellulari che l'infezione impatto avrebbe facilitato lo sviluppo di terapie molto bisogno. I recenti progressi nella interferenza dell'RNA (RNAi), la tecnologia accoppiata con il sequenziamento completo del genoma di organismi diversi ha portato alla ottimizzazione del genoma, a base di cellule perdita di funzione di schermi. Cellule di Drosophila sono particolarmente suscettibili di genoma scala schermi a causa della facilità ed efficienza di RNAi in questo sistema 1. Soprattutto, una grande varietà di virus possono infettare le cellule di Drosophila, compreso un certo numero di virus dei mammiferi di interesse medico e agricolo 2,3,4. Precedenti schermi RNAi in Drosophila hanno identificato fattori dell'ospite che sono richiesti per varie fasi dell'infezione da virus, compresa l'iscrizione, la traduzione e la replicazione dell'RNA 5. Inoltre, molti dei fattori cellulari necessari per la replicazione virale nelle cellule in coltura Drosophila sono anche limitante nelle cellule umane infettate con questi virus 4,6,7,8, 9. Pertanto, l'identificazione di fattori dell'ospite cooptati durante l'infezione virale presenta nuovi obiettivi per la terapia antivirale. Qui vi presentiamo un protocollo generalizzata per un high-throughput schermo RNAi per identificare fattori cellulari coinvolti nella infezione virale, utilizzando vaccinia virus come esempio.
Protocollo
Parte 1: RNAi in 384 piastre
- Standardizzazione dei reagenti utilizzati per lo screening è essenziale. Testiamo singoli lotti di media Schneider, siero fetale bovino, e reagenti colorazione. Poi abbiamo aliquota e utilizzare lo stesso lotto per l'intero schermo.
- Per ogni test, si consiglia di testare diverse linee cellulari differenti per determinare quale è più riconducibile al biologico lettura. Usiamo tipicamente la S2-DRSC linea.
- Crescere le cellule di Drosophila a 25 ° C in mezzi di comunicazione completa di Schneider.
- Schneider mezzi di comunicazione
- 10% di calore FBS inattivato
- 1X L-glutammina
- 1X Pen / strep antibiotici
- Cellule di Drosophila sono semi-aderenti e sono diversi passaggi senza tripsinizzazione ogni quattro giorni pipettando le cellule fuori i palloni e diluendo le cellule 1:10 in un pallone fresca. Le cellule devono essere in fase di log per esperimenti.
- Piastra-to-plate e giorno per giorno variabilità sono ridotti al minimo attraverso l'utilizzo di gestione automatica dei liquidi per tutte le aggiunte liquido multi-pozzetti. Usiamo un WellMate (Matrix).
- Preparare tubi WellMate.
- Spray hood e WellMate con il 70% di etanolo.
- Rimuovere il tubo dalla confezione e spruzzare con il 70% di etanolo.
- Inserire cassetta tubi di erogazione posizione sul WellMate.
- Tubi primo con 25 ml di etanolo al 70%.
- Sterilizzare tubi in etanolo per 15 minuti, allora primo con un altro 25 ml di etanolo.
- Sciacquare tubi di priming con 50 ml di PBS sterile.
- Staccare le cellule dal pallone e contare. Celle a pellet in modo da avere 25% in più del necessario (300xg, 5 min). Il numero di cellule seminate in ogni pozzetto deve essere ottimizzata in base alla lunghezza del saggio. Quando si esegue l'analisi automatizzata delle immagini, un monostrato unico senza aggregazione cellulare è l'ideale per la segmentazione delle immagini.
- Risospendere le cellule in pellet nel siero senza mezzi di comunicazione di Schneider a 1.7x10 6 cellule / ml.
- Eseguiamo lo schermo in 384 piastre pre-aliquotati con una libreria disponibile in commercio del dsRNA ad una concentrazione di 250ng/well. Diversi pozzi di ogni piatto vengono lasciati vuoti per i controlli, che vengono aggiunti manualmente. Noi usiamo dsRNA a luciferasi come controllo negativo. Noi usiamo dsRNA contro Thread (DIAP) come controllo per la robusta RNAi. Abbattere di questo anti-apoptotica risultati fattore di morte cellulare drammatico ed è un modo rapido visivo per valutare la qualità del abbattere. Infine, usiamo un dsRNA virale contro il giornalista, in questo caso i beta-galacosidase, come controllo positivo per confermare che siamo in grado di inibire l'infezione virale con il nostro test di lettura. Se spotting dsRNA nel piatto al momento della semina delle cellule, aliquota 1 dsRNA microlitri in un angolo del pozzo per consentire la facile visualizzazione, quindi si centrifuga dsRNA verso il fondo del pozzo (300xg, 1 min) prima di aggiungere le cellule.
- Usa WellMate per aggiungere 10 microlitri cellule preparate in precedenza per ciascun pozzetto.
- Spin cellule su piastre da centrifugazione 300xg, 1 min.
- Incubare a 25 ° C incubatore per 45 minuti.
- Aggiungere 20 l completa di Schneider media e centrifuga 300xg, 1 min.
- Piastre posto in umidificata contenitori Tupperware foderato con impregnato d'acqua tovaglioli di carta. Il volume di un 384 è ben piccolo: così, l'evaporazione può avere grandi effetti sulla biologia, che a sua volta può portare a cambiamenti artefatta nel read-out dei pozzi di bordo. Per ovviare a questo, piastre devono essere mantenuti sotto umidità elevata.
- Incubare per tre giorni per consentire knockdown robusto. Non c'è bisogno di cambiare i mezzi di comunicazione o di aprire i contenitori umidificata in questo periodo.
Parte 2: Infezione con virus vaccinico
- Sterilizzare multicanale aspiratore in Quatricide per 15 minuti, poi sciacquare in etanolo al 70%.
- Preparare WellMate come descritto sopra.
- Preparare Schneider mezzi di comunicazione con il 2% FBS relative alle infezioni da diluire multimediale completo 1:5 in siero media liberi.
- Filtro di riserva virus greggio attraverso siringa 0,8 um filtro per rimuovere detriti cellulari o utilizzare virus purificato.
- Diluire virus vaccinico nel 2% dei media siero Schneider ad essere molteplicità di infezione (MOI) di 2.
- Rimuovere PBS da tubi WellMate, e il primo con virus diluito fino a quando il tubo è libero di bolle d'aria.
- Usa sterilizzati multi-canale collettore di aspirazione e aspiratore per rimuovere delicatamente i media dalle tavole di dsRNA cellule trattate con Drosophila.
- Dispensare 30 microlitri per virus e sulle piastre con il WellMate.
- Centrifugare piastre 1 minuto a 300xg.
- Sterilizzare tubi WellMate. Il primo con 25 ml di 10% Bleach, attendere 10 minuti, e il primo con un altro candeggina 25 ml al 10%.
- Tubi primo con 50 mL di acqua seguita da 50 mL di etanolo al 70%. Ritorna tubi al suo confezionamento.
- Ritorna piatti per umidificare Tupperware. Incubare a 25 ° C per 48 ore.
Parte 3: Lastre di colorazione
- Fissare le piastre in 15 microlitri di formaldeide al 4% / PBS per 10 minuti. Liquido viene rimosso utilizzando un multi-canale collettore di aspirazione e vuoto ad ogni passo. Una volta che le piastre sono stati risolti, attrezzature sterili e reagenti non sono più necessari.
- Rimuovere fix e utilizzare WellMate aggiungere 30 microlitri PBST (PBS + 0,1% Triton-X). Incubare 10 minuti e ripetere. WellMate operare come descritto sopra, ad eccezione di tubi non ha bisogno di essere sterilizzato.
- Per rilevare l'infezione vaccinia usiamo un β-galattosidasi giornalista guidato da un inizio / fine promotore vaccinia.
- Rimuovere le cellule di liquido e incubare in blocco (PBST con il 2% BSA) per 10 minuti.
- Diluire l'anticorpo primario in blocco, rimuovere il liquido e aggiungere 15 microlitri di anticorpi ad ogni pozzetto usando un pipettatore multicanale ripetizione (Matrix).
- Spin down piastre (300xg, 1 min), coprire con un adesivo trasparente, e incubare a 4 ° C durante la notte.
- Rimuovere l'anticorpo primario, e usare il WellMate per lavare i pozzi in PBST 3 volte, a 10 minuti ciascuno.
- Diluire anticorpo secondario fluorescente (FITC) e nucleari macchia in blocco, e aggiungere 15 microlitri di pozzi con un pipettatore multicanale ripetere.
- Incubare le piastre 1 ora a temperatura ambiente, al riparo dalla luce.
- Lavare i pozzetti in PBST 3 volte, a 10 minuti ciascuno utilizzando la WellMate.
- Guarnizione piastra con adesivo trasparente, coprire e conservare a 4 ° C fino a tre settimane.
Parte 4: Imaging and Image Analysis
- Utilizzare un microscopio automatico a 20X di ingrandimento alle piastre immagine.
- Catturare le immagini dei nuclei totale (DAPI) e le cellule infettate (CIC). Ottimizzare il tempo di esposizione per ogni canale singolarmente al fine di massimizzare la gamma dinamica.
- Ottimizzare la funzione di messa a fuoco automatica prendendo una pila Z di immagini, 1um distanziate, che attraversano i piani sopra e sotto il piano previsto di messa a fuoco. Una volta che il piano focale ottimale si trova, regolare le impostazioni di messa a fuoco automatica conseguenza.
- Immagine l'intera piastra, catturando almeno tre immagini al bene sia nel canale Hoechst (DAPI) e il canale di virus (CIC). Scegliere i siti da diverse regioni diverse all'interno di un bene al fine di catturare immagini che rappresentano il bene nel suo complesso.
- Usa MetaXpress software per scrivere un diario di segmentare l'immagine e l'utilizzo dei moduli Nuclei Conte per calcolare il numero totale di cellule positive per la colorazione nucleare (cellule totali) e infezioni (CIC) per ogni piatto.
- Calcolare l'infezione per cento (100 celle * FITC positivi / totale) e log trasformare.
- Per identificare i candidati che usiamo un punteggio Z robusta basata sulla gamma media e interquartile di ogni piatto. Questo metodo è insensibile ai valori anomali e dati non simmetrico, e fornisce quindi una maggiore potenza a identificare anche colpi deboli o moderati 10. Per ogni piatto, la mediana viene sottratto dai dati di log-trasformati. Poi i valori risultanti sono divisi di 0,74 volte il range interquartile (la differenza tra i valori del percentile 75 ° e il 25 ° percentile). Il fattore 0,74 viene utilizzato per approssimare una distribuzione normale standard.
- Il punteggio robusto Z è una misura della distanza in deviazioni standard dalla media del piatto. Ad esempio, un robusto Z-score di 2 indica l'infezione% del pozzo è ~ 2 deviazioni standard dalla mediana piatto o p <0,05.
- Analizzare il numero di cellule per identificare i candidati citotossici. Calcolare il punteggio Z robusto dei conteggi nucleare. I candidati con un robusto Z <-2 in schermi doppio sono da considerarsi citotossici.
- Pozzi che hanno un punteggio robusto Z per cento delle infezioni> 2 o <-2 nelle schermate duplicato (p <0,0025) senza citotossicità sono considerati potenziali candidati dalla schermata principale.
- Candidati positivi sono convalidati utilizzando reagenti indipendente. dsRNAs sono generati contro i geni di interesse da un'altra regione del mRNA e testato come sopra.
- Questi geni per il quale due dsRNAs indipendenti hanno lo stesso fenotipo può considerare per ulteriori studi.
Parte 5: immagini rappresentative e interpretazione dei tassi di infezione
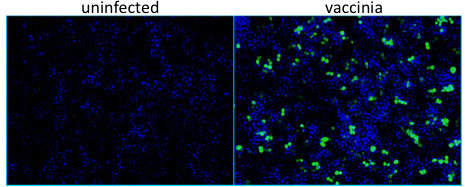
Pozzi Figura 1. Infette non mostrano alcuna colorazione per le proteine del virus vaccinico, mentre un rappresentante e contiene le cellule infettate colorazione positiva per vaccinia-espresso beta-galattosidasi (βgal) di proteine, come misurato da microscopia di immunofluorescenza.Virus = verde, nuclei = blu.
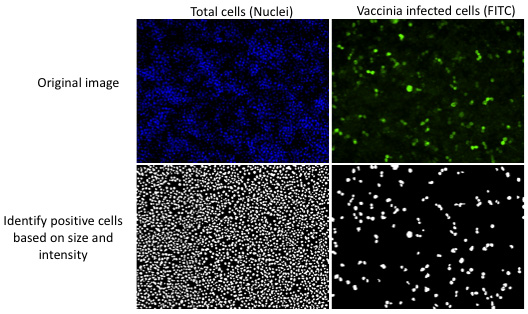
Figura 2. Automatizzato software di analisi dell'immagine quantifica l'infezione in ogni immagine utilizzando i parametri impostati dall'utente. In un'immagine rappresentativa, il numero totale dei nuclei delle cellule e il numero di cellule che esprimono l'antigene virale sono conteggiati in base alle dimensioni e l'intensità della colorazione al di sopra della colorazione di fondo locale.

Figura 3. Knockdown di filo (DIAP) serve come controllo positivo per la robusta riduzione RNAi di geni bersaglio. Knockdown di questo anti-apoptotica fattore porta alla morte cellulare drammatico. Nuclei = blu.
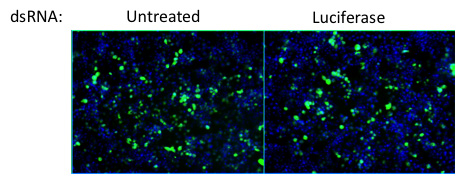
Knockdown Figura 4. Luciferasi della funge da controllo negativo per l'effetto del doppio filamento di RNA su trattamento delle infezioni. L'esaurimento delle luciferasi non ha alcun effetto su infezioni rispetto alle cellule non trattate. Virus = verde, nuclei = blu.

Figura 5. Knockdown di proteine βgal serve come controllo positivo per una riduzione di infezione, dato che il test utilizza livelli di proteina βgal come una lettura di infezione. Esaurimento dei risultati βgal una diminuzione della percentuale di cellule infette. Virus = verde, nuclei = blu.
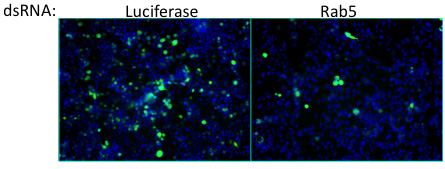
Figura 6. Knockdown di cellulari fattore Rab5 si traduce in una diminuzione della percentuale di cellule infette. Dal momento che Rab5 è noto per partecipare a endocitosi, questo fattore contribuisce probabile ingresso del virus vaccinico. Ciò rappresenta un outlier esempio dallo schermo. Virus = verde, nuclei = blu.
Discussione
Genoma di screening RNAi in Drosophila fornisce un metodo robusto ed efficiente per l'esame della componente cellulare di infezione virale. Un certo numero di virus di infettare le cellule di mammifero Drosophila, che può essere utilizzato per identificare i componenti della risposta immunitaria ospite intrinseca, e fattori proteici ospitante è richiesta per la replicazione virale, che possono rappresentare nuovi bersagli terapeutici. Prima di eseguire uno schermo, il dosaggio deve essere attentamente ottimizzati per il tipo cellulare, numero di cellulare, e il livello di infezione, e deve essere ben controllato, sia positivi che negativi virali e obiettivi cellulari 11. E 'importante garantire una sufficiente separazione tra controlli positivo e negativo prima dello screening per massimizzare la gamma dinamica del test. Una volta che lo schermo è stato effettuato, i candidati possono essere suddivisi in categorie funzionali per determinare quali meccanismi ospitare contribuiscono alle infezioni. Per i virus sui mammiferi, come vaccino, il contributo di omologhi mammiferi da RNAi dovrebbero essere determinati come parte di analisi secondaria. Presi insieme, questi metodi di screening robusti ci permetterà di ottenere informazioni sui meccanismi complessi da cui i virus interagiscono con i loro cellule ospiti.
Divulgazioni
Riconoscimenti
Questo lavoro è stato supportato anche da finanziamenti NIAID (R01AI074951, U54AI057168) e la Penn frontiere Genome Institute di SC; dal NIH T32HG000046 di TSM, dal NIH T32GM07229 e T32AI007324 di LRS.
Materiali
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
Riferimenti
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions? Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon