Method Article
に関与する宿主因子のためのRNAiスクリーニングワクウイルス感染ショウジョウバエセル
要約
ウイルス感染に関与する新規の宿主因子は、機能のRNAiスクリーニングの細胞ベースのゲノムワイドな損失を介して識別できます。 Aショウジョウバエ細胞培養モデルでは、RNAi実験の容易さと効率のためにこのアプローチに特に適している。ここでは、使用してこのテクニックを示していますワクウイルス。
要約
だけでなく、ウイルスは、ヒトの病気の自然な流行を引き起こす可能性がありますが、バイオテロの潜在的な利用も懸念される。ウイルス性病原体は、重大な公衆衛生上の脅威を表す。衝撃の感染が待望の治療薬の開発を促進することを細胞因子の理解を深める。いくつかの生物の塩基配列の完全なゲノムとの結合RNA干渉(RNAi)技術の最近の進歩により、ゲノムワイドな、セルベースの機能喪失の画面の最適化につながっている。 ショウジョウバエの細胞ためのゲノムスケールの画面には特に敏感に反応このシステム1におけるRNAiの使いやすさと効率性。重要なのは、ウイルスの多種多様な医療や農業の大切さ2,3,4の哺乳類のウイルスの数を含む、 ショウジョウバエの細胞を、感染することができます。 ショウジョウバエの前のRNAiスクリーニングは、エントリ、翻訳とRNA複製5を含むウイルス感染におけるさまざまなステップに必要な宿主因子を同定した。また、 ショウジョウバエの細胞培養でウイルス複製に必要な細胞因子の多くは、これらのウイルス4,6,7,8、9に感染したヒトの細胞に制限されています。したがって、ウイルス感染時の共存を選んだ宿主因子の同定は、抗ウイルス治療のための新しいターゲットを示します。ここでは例として、 ワクシニアウイルスを使用して、ウイルス感染に関与する細胞因子を特定するハイスループットRNAiスクリーニングのための一般的なプロトコルを提示する。
プロトコル
パート1:384ウェルプレートでのRNAi
- スクリーニングに用いる試薬の標準化が不可欠です。我々はシュナイダーのメディアの個々のたくさんの、ウシ胎児血清、および染色試薬をテストします。その後、我々は、分注して、画面全体のための同一ロットを使用してください。
- 任意のアッセイの場合、それは、読み出して生物学的に最も適しているかを判断するためにいくつかの異なる細胞株をテストするのが最適です。我々は通常、S2 - DRSCラインを使用してください。
- 25℃で完全なシュナイダーのメディアでショウジョウバエの細胞を成長する。
- シュナイダーのメディア
- 10%の熱不活化FBS
- 1X L -グルタミン
- 1Xのペン/連鎖球菌抗生物質
- ショウジョウバエの細胞は半接着性であり、フラスコから細胞をピペッティングし、新鮮なフラスコに1時10分、細胞を希釈して4日ごとにトリプシン処理することなく継代されています。細胞は実験の対数増殖期にある必要があります。
- プレート間のプレートと日々の変動は、マルチウェルプレートにすべての液体の追加のための自動液体ハンドリングを使用することによって最小限に抑えられています。我々はWellMate(マトリックス)を使用してください。
- WellMateチューブを準備します。
- 70%エタノールでスプレーフードとWellMate。
- 70%エタノールで包装し、スプレーのチューブを取り外します。
- WellMate上の位置を調剤にチューブカセットを挿入します。
- 70%エタノール25mLの素チューブ。
- エタノールの別の25 mLのその後素数15分、のためのエタノールのチューブを滅菌する。
- 滅菌PBS 50 mLでプライミングによってチューブを洗浄します。
- フラスコとカウントから細胞を取り除きます。細胞をペレット化するために必要なよりも25%以上を持つように(300xg、5分)。ウェルあたり播種する細胞の数は、アッセイの長さに応じて最適化する必要があります。自動画像解析を行う際に、無細胞の凝集を持つ単一の単層は、画像のセグメンテーションに最適です。
- 1.7 × 10 6細胞/ mLで無血清シュナイダーのメディアでペレット化した細胞を再懸濁します。
- 我々は、384ウェルプレート250ng/wellの濃度でdsRNAの市販のライブラリーが事前に小分けに画面を実行します。各プレートのいくつかのウェルを手動で追加されたコントロール、のために空のままにされています。我々は、ネガティブコントロールとしてルシフェラーゼにdsRNAを使用。我々は、堅牢なRNAi実験のコントロールとして(dIAP)スレッドに対するdsRNAを使用してください。劇的な細胞死でこの抗アポトーシス因子の結果のノックダウンとダウンノックの品質を評価するために迅速に視覚的な方法です。最後に、我々は我々のアッセイを読んでアウトを使用してウイルス感染を阻害することができることを検証するためにポジティブコントロールとして、この場合はβ- galacosidaseで、ウイルスのレポーターに対してdsRNAを使用してください。一定分量1μlのdsRNAが簡単に可視化を可能にするためにものコーナーに、細胞播種の時にプレートにdsRNAをスポッティングした場合は、セルを追加する前に、井戸の底(300xg、1分)にdsRNAを遠心してください。
- 各ウェルに上記で調製した10μLの細胞を追加するWellMateを使用してください。
- 300xg、1分を遠心分離することによってプレート上に細胞を回転させる。
- 45分間25℃インキュベーターでインキュベートする。
- 20μLの完全シュナイダーのメディアを追加し、300xg、1分遠心してください。
- 加湿タッパーウェアの容器に入れプレートは水浸状のペーパータオルを敷いた。 384の量は十分小さく、したがって、蒸発は順番に、エッジの井戸のリードアウトの人為的な変化につながることができる生物学、大きな影響を持つことができます。これを克服するために、プレートは、高湿度下で維持されるべきである。
- 堅牢なノックダウンを可能にするために三日間インキュベートする。この期間中にメディアまたはオープン加湿容器を変更する必要はありません。
パート2: ワクシニアウイルスで感染する
- 15分間Quatricideでマルチチャンネルの吸引器を殺菌し、70%エタノールでリンス。
- 上記のようにWellMateを準備します。
- 無血清培地での完全なメディア1:5に希釈して感染を2%FBSとシュナイダーのメディアを準備します。
- 細胞残渣を除去または精製されたウイルスを使用するフィルタ0.8 UMシリンジを介してフィルタ原油ウイルスストック。
- 2の感染の多重度(MOI)を取得するには、2%血清シュナイダーのメディアにワクシニアウイルスを希釈する。
- WellMateチューブからPBSを外し、チューブが気泡の自由になるまで希釈したウイルスと素数。
- 静かにdsRNAを処理したショウジョウバエの細胞のプレートから培地を除去する滅菌マルチチャンネルアスピレーターと吸引マニホールドを使用してください。
- WellMateを使用してプレートにウェルあたり30μLのウイルスを分注する。
- 300xgでプレート1分間遠心します。
- WellMateチューブを滅菌する。 10%のブリー25mLの素チャンネル、10分待ってから別の25 mLの10%の漂白剤と素数。
- 50mLの70%エタノール、続い50mLの水素チューブ。その包装にチューブを返します。
- 加湿タッパーウェアの容器にプレートを返します。 48時間25℃でインキュベートする。
パート3:染色プレート
- 10分15μLを4%ホルムアルデヒド/ PBSでプレートを固定してください。液体は、各ステップでマルチチャンネルの吸引器と吸引マニホールドを使用して削除されます。一度プレートが固定されている、滅菌された器具や試薬が不要になります。
- 削除修正して30μLPBST(PBS + 0.1%トリトン- X)を追加するWellMateを使用してください。 10分とリピートをインキュベートする。チューブは滅菌する必要がない場合を除き、上記のようWellMate動作する。
- ワクシニア感染を検出するために我々は、初期/後期ワクシニアプロモーターによって駆動されるβ-ガラクトシダーゼのレポーターを使用してください。
- 10分間ブロック(2%BSAを含むPBST)で液体とインキュベートセルを削除します。
- ブロック内で一次抗体を希釈して、液体を除去し、各ウェルのマルチチャネルを用いた繰り返しピペッター(マトリックス)に15μLの抗体を加える。
- プレート(300xg、1分)スピンダウン、明確なステッカーでカバーし、4℃で一晩インキュベートする。
- 一次抗体を削除し、PBSTで3回、10分ごとに井戸を洗浄するWellMateを使用してください。
- 蛍光標識二次抗体(FITC)と核ブロックで染色、およびマルチチャンネルのリピートピペッターを使用してウェルに15μLを追加して希釈する。
- プレートを、室温で1時間は、光から保護。
- WellMateを使用してPBSTで3回、10分ごとに井戸を洗浄する。
- 4で明確なステッカー、カバー、およびストアでプレートをシール° Cまで3週間。
パート4:イメージングと画像解析
- イメージングプレートに20倍の倍率で自動化された顕微鏡を使用してください。
- 合計核(DAPI)と感染細胞(FITC)の画像をキャプチャ。ダイナミックレンジを最大化するために、個別に各チャネルごとに露光時間を最適化する。
- 焦点の予想される平面の上下面にまたがる画像を、間隔をあけ1um、のZスタックを取ることによってオートフォーカス機能を微調整します。最適な焦点面が配置されると、それに応じてオートフォーカスの設定を調整します。
- 画像全体のプレート、ヘキストチャンネル(DAPI)とウイルスのチャンネル(FITC)の両方に1ウェルあたり、少なくとも3つの画像をキャプチャする。全体として十分に表すイメージをキャプチャするためにも一つの中にいくつかの異なる地域からのサイトを選択してください。
- セグメントにイメージをジャーナルを書き、それぞれのプレートのための核染色(総細胞)や感染(FITC)のための陽性細胞の合計数を計算するには、Countの核のモジュールを使用するソフトウェアをMetaXpress使用。
- パーセントの感染症(100 * FITC陽性/総細胞)を計算し、変換ログオンします。
- 候補者を識別するために我々は、各プレートの中央値と四分位範囲に基づいて、堅牢なZスコアを使用してください。このメソッドは、外れ値や非対称データに鈍感であり、そのためにも弱または中程度のヒット10を特定するのに大きな力を提供します。各プレートについて、中央値は、対数変換したデータから減算されます。次に、得られた値は、0.74倍の四分位範囲(75パーセンタイルの値と25 パーセンタイルの間の差)によって分けられています。 0.74係数が近似標準正規分布に使用されます。
- 堅牢なZスコアは、プレートの中央値から標準偏差の距離の尺度です。たとえば、2の堅牢なZ -スコアは、井戸の%の感染がプレートの中央値またはp <0.05から〜2標準偏差であることを示します。
- 細胞毒性の候補を識別するために、細胞数を分析する。核カウントの堅牢なZスコアを計算する。重複画面で堅牢なZ <-2を持つ候補者は細胞毒性とみなされます。
- 細胞毒性なし、重複画面で> 2または<-2(P <0.0025)のパーセント感染の堅牢なZスコアを持っている井戸は、一次画面からの潜在的候補と見なされます。
- 肯定的な候補者は、独立した試薬を用いて検証されます。 dsRNAはmRNAの別の領域から興味のある遺伝子に対して生成され、上記のように試験されています。
- 二つの独立したdsRNAは、同じ表現型を所持している遺伝子は、さらなる研究のために考えることができる。
パート5:代表画像と感染率の解釈
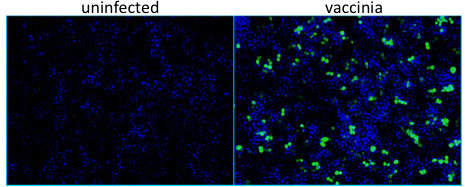
よく感染の代表がのような免疫蛍光顕微鏡で測定したワクシニア発現するβ-ガラクトシダーゼ(βgal)蛋白質、のために陽性染色細胞を含んでいるときに、 図1。非感染の井戸は、 ワクシニアウイルスのタンパク質のいずれかの染色を示していない。ウイルス=緑、核=青。
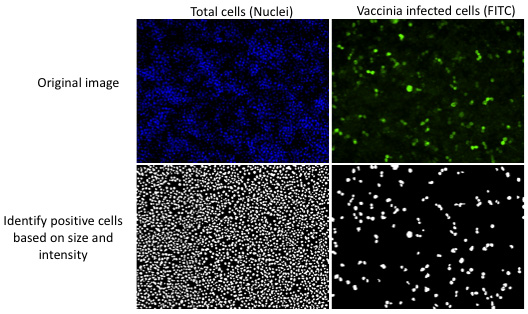
図2自動画像解析ソフトウェアは、ユーザが設定したパラメータを用いて各画像に感染を定量化する。代表的なイメージでは、細胞核やウイルス抗原を発現する細胞の数の合計数は、サイズおよびローカルバックグラウンド染色の上に染色の強度に基づいてカウントされます。

図3。スレッド(dIAP)のノックダウンは、標的遺伝子の堅牢なRNAiの枯渇のためのポジティブコントロールとして機能します。この抗アポトーシス因子のノックダウンは、劇的な細胞死につながる。核=青。
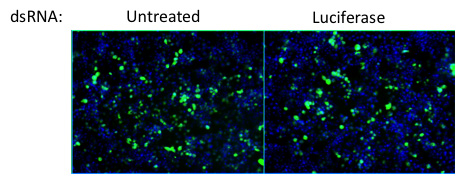
図4。ルシフェラーゼのノックダウンは、感染に対する二本鎖RNAの治療の効果のためのネガティブコントロールとして機能します。ルシフェラーゼの枯渇は、放置細胞への相対的な感染には影響しません。ウイルス=緑、核=青。

感染から読み出さとしてアッセイはβgalタンパク質レベルを使用するので、 図5。βgalタンパク質のノックダウンは、感染症の減少のためのポジティブコントロールとして機能します。感染細胞の割合の減少にβgal結果の枯渇。ウイルス=緑、核=青。
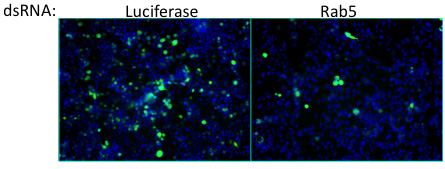
図6。細胞因子のノックダウン感染細胞の割合の減少のRab5の結果。 Rab5のエンドサイトーシスに関与することが知られているので、この要因は、おそらくワクシニアウイルスのエントリーに貢献しています。これは、画面から例の外れ値を表します。ウイルス=緑、核=青。
ディスカッション
ショウジョウバエのゲノムワイドなRNAiスクリーニングでは、ウイルス感染の細胞成分を調べるための、堅牢で効率的な方法を提供する。哺乳類のウイルスの数は、ホスト固有の免疫応答の構成要素を識別するために使用できるショウジョウバエの細胞を、感染、および新たな治療標的を表す可能性があるウイルス複製に必要なタンパク質因子を開催。画面を実行する前に、アッセイは、慎重に細胞型、細胞数、および感染のレベルに合わせて最適化しなければならない、とも正と負のウイルスおよび細胞標的11の両方を含む、制御しなければならない。アッセイのダイナミックレンジを最大にする前にスクリーニングにポジティブコントロールとネガティブコントロールとの間の十分な分離を確保することが重要です。画面が実行されたら、候補者は、ホストのメカニズムは、感染症に寄与するかを決定するために機能的なカテゴリに分けることができます。 ワクシニアなどの哺乳動物ウイルスの場合は、RNAiによる哺乳動物のホモログの寄与は、二次分析の一環として決定されるべきである。一緒に、これらの堅牢なスクリーニング法は、私たちは、ウイルスが宿主細胞と相互作用することによって、複雑なメカニズムを把握することができます。
開示事項
謝辞
この作品は、NIAID(R01AI074951、U54AI057168)とSCにペンゲノムフロンティア研究所からの補助金によって支えられて、NIH T32HG000046からTSMに、NIH T32GM07229とT32AI007324からLRSに。
資料
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
参考文献
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions? Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved