Method Article
Dépistage ARNi pour les facteurs de l'hôte impliqués dans Vaccine aide Drosophile Cellules
Dans cet article
Résumé
Facteurs de l'hôte roman impliqués dans l'infection virale peuvent être identifiés par la cellule basée échelle du génome perte de fonction ARNi dépistage. Une Drosophile Modèle de culture cellulaire est prête particulièrement bien à cette approche en raison de la facilité et l'efficacité de l'ARNi. Ici nous montrer cette technique en utilisant Vaccine Virus comme un exemple.
Résumé
Pathogènes viraux représentent une menace de santé publique important, non seulement peut provoquer des épidémies des virus naturels de la maladie humaine, mais leur utilisation potentielle dans le bioterrorisme est aussi une préoccupation. Une meilleure compréhension des facteurs cellulaires que l'infection impact serait de faciliter le développement de thérapies bien nécessaire. Les progrès récents dans l'interférence ARN (ARNi) couplée à la technologie du génome séquençage complet de plusieurs organismes a conduit à l'optimisation de l'ensemble du génome, basé sur des cellules de perte de fonction des écrans. Cellules de drosophile sont particulièrement favorables à l'échelle du génome écrans en raison de la la facilité et l'efficacité de l'ARNi dans ce système 1. Surtout, une grande variété de virus peuvent infecter les cellules de drosophile, y compris un certain nombre de virus de mammifères d'importance médicale et agricole 2,3,4. Précédente écrans ARNi chez la drosophile ont identifié des facteurs d'hôte qui sont nécessaires pour les différentes étapes de l'infection du virus y compris l'entrée, la traduction et de réplication de l'ARN 5. Par ailleurs, bon nombre des facteurs cellulaires nécessaires à la réplication virale en culture cellulaire chez la drosophile sont également de limiter dans les cellules humaines infectées par ces virus 4,6,7,8, 9. Par conséquent, l'identification des facteurs de l'hôte co-opté au cours infection virale présente de nouvelles cibles pour des traitements antiviraux. Nous présentons ici un protocole généralisé pour un écran haut débit ARNi pour identifier les facteurs cellulaires impliqués dans l'infection virale, à l'aide virus de la vaccine comme un exemple.
Protocole
Partie 1: ARNi dans Plaques 384 puits
- Standardisation des réactifs utilisés pour le dépistage est essentiel. Nous testons des lots individuels de média de Schneider, sérum de veau fœtal et réactifs de coloration. Ensuite, nous aliquote et utiliser le même lot pour la totalité de l'écran.
- Pour tout dosage, il est préférable de tester plusieurs lignées cellulaires différentes afin de déterminer qui est le plus prête à votre biologiques lecture. Nous utilisons généralement la ligne S2-DRSC.
- Cultiver des cellules de drosophile à 25 ° C dans des milieux complets de Schneider.
- Schneider médias
- FBS inactivé chaleur de 10%
- 1X L-glutamine
- 1X Pen / strep antibiotiques
- Les cellules de drosophile sont semi-adhérent et sont passées sans trypsination tous les quatre jours par pipetage des cellules hors des flacons et de diluer les cellules dans un flacon de 01h10 fraîche. Les cellules doivent être en phase logarithmique pour des expériences.
- Plate-to-plate et au jour le jour la variabilité sont minimisés par l'utilisation de la manipulation de liquides automatisée pour tous les ajouts liquides pour plaques multi-puits. Nous utilisons un WellMate (Matrix).
- Préparer tube WellMate.
- Capote et WellMate avec de l'éthanol à 70%.
- Retirez l'emballage et du tube de pulvérisation avec de l'éthanol à 70%.
- Insérez la cassette dans le tuyau de distribution position sur le WellMate.
- Premier tube avec 25 ml d'éthanol à 70%.
- Stériliser tube dans de l'éthanol pendant 15 minutes, alors premier avec un autre de 25 ml d'éthanol.
- Rincez tube par amorçage avec 50 ml de PBS stérile.
- Déloger les cellules du flacon et à compter. Pellet cellules de sorte que vous avez 25% de plus que nécessaire (300xg, 5 min). Le nombre de cellules ensemencées par puits doit être optimisée en fonction de la longueur de dosage. Lors de la réalisation d'analyse d'image automatique, une seule monocouche sans agglutination cellulaire est idéal pour la segmentation d'images.
- Resuspendre culot cellulaire dans un milieu sans sérum de Schneider au 1.7x10 6 cellules / ml.
- Nous effectuons l'écran en 384 plaques ainsi pré-aliquotés avec une bibliothèque disponible dans le commerce d'ARNdb à une concentration de 250ng/well. Plusieurs puits de chaque plaque sont laissés vides pour les contrôles, qui sont ajoutés manuellement. Nous utilisons des ARNdb à la luciférase comme contrôle négatif. Nous utilisons des ARNdb contre-fil (DPAI) comme contrôle pour robustes ARNi. Faites tomber les résultats de ce facteur anti-apoptotique de la mort cellulaire dramatique et est un moyen visuel rapide pour évaluer la qualité de l'assommer. Enfin, nous utilisons un ARNdb contre le journaliste virale, dans ce cas, le bêta-galacosidase, comme un contrôle positif à valider que l'on peut inhiber l'infection virale à l'aide de notre test de lecture. Si spotting ARNdb dans la plaque au moment de l'ensemencement des cellules, une partie aliquote ARNdb ul dans le coin du puits pour permettre la visualisation plus facile, puis centrifuger ARNdb dans le fond du puits (300xg, 1 mn) avant d'ajouter les cellules.
- Utilisez WellMate d'ajouter 10 uL cellules préparé ci-dessus à chaque puits.
- Spin cellules sur des plaques par centrifugation 300xg, 1 min.
- Incuber à 25 ° C incubateur pendant 45 minutes.
- Ajouter 20 uL médias complet de Schneider et centrifuger 300xg, 1 min.
- Plaques Placer dans des contenants Tupperware humidifié bordée de serviettes en papier imbibées d'eau. Le volume d'une 384 est bien petite, ainsi, l'évaporation peut avoir des effets importants sur la biologie, qui à son tour peut conduire à des changements dans les aboutissants artéfactuelle lecture des puits de pointe. Pour y remédier, les plaques doivent être maintenues sous une forte humidité.
- Incuber pendant trois jours pour permettre knockdown robuste. Il n'est pas nécessaire de changer les médias ou d'ouvrir les conteneurs humidifié pendant ce temps.
Partie 2: infecter avec le virus vaccine
- Stériliser multi-canal d'aspiration dans les Quatricide pendant 15 minutes, puis rincer dans de l'éthanol à 70%.
- Préparer WellMate comme décrit plus haut.
- Préparez le support de Schneider avec 2% de FBS à l'infection par dilution totale des médias 01h05 dans le sérum des médias libres.
- Filtre stock de virus brut par 0,8 um seringue filtre pour éliminer les débris cellulaires ou de l'utilisation de virus purifié.
- Diluer virus de la vaccine dans 2% des médias du sérum de Schneider d'obtenir multiplicité d'infection (MOI) de 2.
- Retirer du PBS à partir de tubes WellMate, et le premier avec des virus dilué jusqu'à ce que le tube est libre de bulles d'air.
- Utilisez stérilisés multi-canal d'aspiration et collecteur à vide pour enlever délicatement les médias à partir des plaques de cellules de drosophile ARNdb-traitée.
- Distribuer 30 virus par puits sur des plaques en utilisant les WellMate.
- Centrifuger 1 minute à plaques 300xg.
- Stériliser tube WellMate. Premier avec 25 ml d'Blea 10%ch, attendre 10 minutes, et le premier avec un autre agent de blanchiment de 25 ml à 10%.
- Premier tube 50 ml d'eau suivi de 50 ml d'éthanol à 70%. Retour tube à son emballage.
- Retour aux plaques contenant Tupperware humidifié. Incuber à 25 ° C pendant 48 heures.
Partie 3: Plaques de coloration
- Fixer les plaques dans 15 uL formaldéhyde à 4% / PBS pendant 10 minutes. Le liquide est retiré à l'aide d'un aspirateur multi-canal et le collecteur d'aspiration à chaque étape. Une fois les plaques ont été fixés, matériel stérile et des réactifs ne sont plus nécessaires.
- Retirer fixer et utiliser WellMate d'ajouter 30 uL PBST (PBS + 0,1% de Triton-X). Incuber 10 minutes et répéter. Utilisez WellMate comme décrit plus haut, sauf le tuyau n'a pas besoin d'être stérilisé.
- Pour détecter l'infection vaccine nous utilisons un journaliste β-galactosidase entraînée par un promoteur précoce / tardif vaccine.
- Retirer les cellules de liquide et laisser incuber dans le bloc (PBST avec 2% de BSA) pendant 10 minutes.
- Diluer l'anticorps primaire dans le bloc, enlever le liquide et ajouter 15 uL d'anticorps à chaque pipeteur répétez bien en utilisant un canal multi-(Matrix).
- Isoler plaques (300xg, 1 mn), couvrir avec un autocollant clair, et incuber à 4 ° C pendant la nuit.
- Retirer anticorps primaire, et d'utiliser les WellMate pour laver les puits dans PBST 3 fois, 10 minutes chacune.
- Diluer anticorps secondaire marqué par fluorescence (FITC) et colorant nucléaire dans le bloc, et ajouter 15 ul de puits à l'aide d'une pipette multi-canaux répéter.
- Incuber les plaques pendant 1 heure à température ambiante, protégé de la lumière.
- Laver les puits de PBST 3 fois, 10 minutes chacune utilisant la WellMate.
- Joint de la plaque avec l'autocollant clair, couvrir et conserver à 4 ° C jusqu'à trois semaines.
Partie 4: imagerie et d'analyse d'images
- Utiliser un microscope automatisé à un grossissement de 20x sur des plaques de l'image.
- Capturez des images de noyaux totale (DAPI) et les cellules infectées (ITCF). Optimiser le temps d'exposition pour chaque canal, afin de maximiser la plage dynamique.
- Peaufinez la fonction mise au point automatique en prenant une pile d'images Z, 1um espacées, qui couvrent les avions au-dessus et en dessous du plan prévu de se concentrer. Une fois le plan de focale optimale se situe, d'ajuster les paramètres de mise au point automatique en conséquence.
- L'image de la plaque entière, capturant au moins trois images par puits dans les deux canaux de Hoechst (DAPI) et le canal du virus (ITCF). Choisissez les sites de plusieurs régions différentes au sein d'un bien afin de capturer des images qui représentent le bien comme un tout.
- Utilisez MetaXpress logiciel pour écrire un journal pour segmenter l'image et utiliser des modules noyaux Comte de calculer le nombre total de cellules positives pour la coloration nucléaire (nombre total de cellules) et l'infection (ITCF) pour chaque plaque.
- Calculer l'infection pour cent (100 cellules * FITC positifs / total) et une transformation logarithmique.
- Pour identifier les candidats, nous utilisons un score Z robuste basé sur la médiane et interquartile de chaque assiette. Cette méthode est insensible aux valeurs aberrantes et des données non symétrique, et fournit ainsi une plus grande puissance dans l'identification, même faible ou modérée frappe 10. Pour chaque plaque, la médiane est soustrait des données log-transformées. Puis les valeurs obtenues sont divisées par 0,74 fois l'écart interquartile (la différence entre les valeurs de la 75 e percentile et le 25 e percentile). Le facteur 0,74 est utilisé pour approcher une distribution normale standard.
- Le score Z robuste est une mesure de distance en écarts-types de la médiane de la plaque. Par exemple, un robuste Z-score de 2 indique l'infection% du puits est ~ 2 déviations standards de la médiane plaque ou p <0,05.
- Analyser le nombre de cellules pour identifier les candidats cytotoxiques. Calculer le score Z robuste de comptages nucléaires. Les candidats ayant une solide Z <-2 dans les écrans dédoublés sont considérés comme cytotoxiques.
- Les puits qui ont un score Z robuste pour une infection pour cent de> 2 ou <-2 dans les écrans de reproduire (p <0,0025), sans cytotoxicité sont considérés comme des candidats potentiels de l'écran principal.
- Candidats positifs sont validés à l'aide des réactifs indépendants. dsRNA sont générés contre les gènes d'intérêt d'une autre région de l'ARNm et testé comme ci-dessus.
- Ces gènes, pour lequel deux dsRNA indépendants ont le même phénotype peut être envisagée pour une étude plus approfondie.
Partie 5: images représentatives et interprétation des taux d'infection
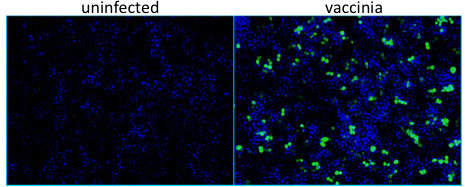
Puits Figure 1. Non infectées ne montrent aucune coloration pour les protéines du virus de la vaccine, tandis qu'un représentant ainsi infectés contient des cellules coloration positive pour la vaccine a exprimé la bêta-galactosidase (ßgal) des protéines, tel que mesuré par immunofluorescence.Virus = vert, les noyaux = bleu.
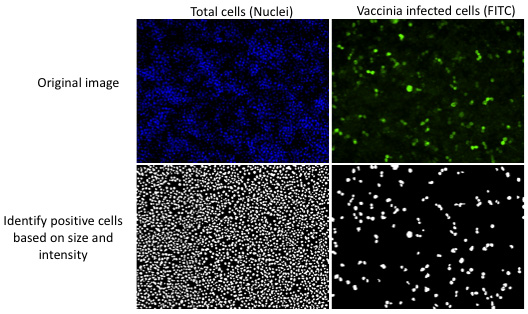
Figure 2. Logiciel automatisé d'analyse d'images permet de quantifier l'infection dans chaque image en utilisant les paramètres définis par l'utilisateur. Dans une image représentative, le nombre total de noyaux de cellules et le nombre de cellules exprimant des antigènes viraux sont comptés selon la taille et l'intensité de la coloration au-dessus du bruit de fond local.

Figure 3. Knockdown de fil (DPAI) sert de contrôle positif pour l'ARNi déplétion robuste de gènes cibles. Knockdown de ce facteur anti-apoptotique conduit à la mort cellulaire dramatique. Noyaux = bleu.
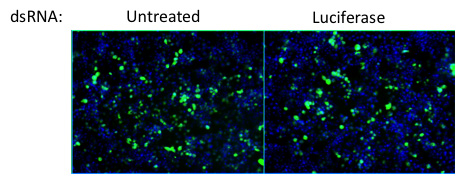
Knockdown Figure 4. De la luciférase sert de contrôle négatif de l'effet de double brin d'ARN du traitement de l'infection. L'appauvrissement de la luciférase n'a aucun effet sur l'infection par rapport aux cellules non traitée. Virus = vert, les noyaux = bleu.

Figure 5. Knockdown de protéines ßgal sert de contrôle positif pour une réduction de l'infection, car le dosage utilise des niveaux de protéines ßgal comme une lecture de l'infection. Épuisement des résultats ßgal une diminution du pourcentage de cellules infectées. Virus = vert, les noyaux = bleu.
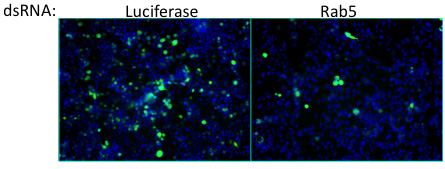
Figure 6. Knockdown des cellulaires facteur Rab5 résulte en une diminution du pourcentage de cellules infectées. Depuis Rab5 est connu pour participer à l'endocytose, ce facteur contribue probablement à l'entrée du virus vaccine. Cela représente une valeur aberrante par exemple de l'écran. Virus = vert, les noyaux = bleu.
Discussion
L'échelle du génome de dépistage ARNi chez la drosophile fournit une méthode robuste et efficace pour examiner la composante cellulaire de l'infection virale. Un certain nombre de virus de mammifères infectent les cellules de drosophile, qui peut être utilisé pour identifier les composants de l'hôte réponse immunitaire intrinsèque, et les facteurs de protéine de l'hôte nécessaires à la réplication virale qui peut représenter de nouvelles cibles thérapeutiques. Avant de procéder à un écran, le dosage doit être soigneusement optimisée pour le type de cellule, le nombre de cellules, et le niveau d'infection, et doivent être bien contrôlés, y compris à la fois positifs et négatifs virale et les cibles cellulaires 11. Il est important d'assurer une séparation suffisante entre les contrôles positifs et négatifs avant le dépistage afin d'optimiser la gamme dynamique du dosage. Une fois que l'écran a été effectué, les candidats peuvent être divisés en catégories fonctionnelles afin de déterminer quels sont les mécanismes d'accueil contribuent à l'infection. Pour les virus de mammifères tels que la vaccine, la contribution des homologues chez les mammifères par RNAi devraient être déterminés dans le cadre d'une analyse secondaire. Pris ensemble, ces méthodes de dépistage robustes va nous permettre de mieux comprendre les mécanismes complexes par lesquels les virus interagissent avec leurs cellules hôtes.
Déclarations de divulgation
Remerciements
Ce travail a été soutenu par des subventions du NIAID (R01AI074951, U54AI057168) et l'Institut Penn Génome Frontières à SC; du NIH T32HG000046 à TSM; du NIH et de T32GM07229 T32AI007324 au LRS.
matériels
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
Références
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions?. Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon