Method Article
RNAi Screening für Host beteiligten Faktoren in Vaccinia Virus-Infektion mit Drosophila Cells
In diesem Artikel
Zusammenfassung
Novel Wirtsfaktoren in Virusinfektion beteiligt durch zell-basierte genomweite Verlust der Funktion RNAi-Screening identifiziert werden. A Drosophila Zellkultur-Modell ist besonders zugänglich, diesen Ansatz durch die Leichtigkeit und Effizienz von RNAi. Hier zeigen wir diese Technik mit Vaccinia Virus als ein Beispiel.
Zusammenfassung
Virale Erreger stellen eine erhebliche Bedrohung der öffentlichen Gesundheit, nicht nur Viren verursachen Epidemien von Krankheiten, aber ihre mögliche Verwendung in Bioterrorismus ist auch ein Anliegen. Ein besseres Verständnis der zellulären Faktoren, die Auswirkungen Infektion wäre die Entwicklung von dringend benötigten Therapien zu erleichtern. Jüngste Fortschritte in der RNA-Interferenz (RNAi)-Technologie mit vollständiger Sequenzierung des Genoms von mehreren Organismen gekoppelt ist, um die Optimierung der genomweiten, zellbasierte loss-of-function-Bildschirme geführt. Drosophila-Zellen sind besonders zugänglich genomweite Bildschirme wegen der Benutzerfreundlichkeit und Effizienz von RNAi in diesem System 1. Wichtig ist, dass eine Vielzahl von Viren infizieren Drosophila-Zellen, darunter eine Reihe von Säugetier-Viren der medizinischen und landwirtschaftlichen Bedeutung 2,3,4. Vorherige RNAi-Screens in Drosophila haben Host-Faktoren, die für die verschiedenen Schritte in Virus-Infektion, einschließlich Ein-, Übersetzungs-und RNA-Replikation 5 erforderlich sind identifiziert. Darüber hinaus sind viele der zellulären Faktoren für die virale Replikation in Drosophila Zellkultur benötigt auch die Begrenzung in menschlichen Zellen mit diesen Viren 4,6,7,8, 9 infiziert. Daher stellt die Identifizierung von Host-Faktoren während der viralen Infektion kooptiert neue Targets für eine antivirale Therapie. Hier präsentieren wir eine verallgemeinerte Protokoll für ein High-throughput RNAi Bildschirm, um zelluläre Faktoren in Virusinfektion beteiligt sind, mit Vaccinia-Virus als ein Beispiel.
Protokoll
Teil 1: RNAi in 384 Well Platten
- Standardisierung der Reagenzien für das Screening verwendet unerlässlich. Wir testen einzelne Lose von Schneider Medien, Rinderfötenserum und Färbereagenzien. Dann haben wir Aliquot und verwenden die gleiche Menge für den gesamten Bildschirm.
- Für jeden Test wird es am besten, verschiedene Zelllinien zu bestimmen, welche ist am wirkungsvollsten zu Ihrem biologischen read-out-Test. Wir verwenden in der Regel die S2-DRSC Linie.
- Wachsen Drosophila-Zellen bei 25 ° C in völliger Schneider Medien.
- Schneider Medien
- 10% hitzeinaktiviertem FBS
- 1X L-Glutamin
- 1X Pen / Strep Antibiotika
- Drosophila-Zellen sind semi-Anhänger und sind ohne Trypsinierung passagiert alle vier Tage mit einer Pipette die Zellen aus dem Fläschchen und Verdünnen der Zellen 1.10 in eine frische Flasche. Die Zellen sind in Log-Phase für Experimente sein.
- Plate-to-Plate-und Tag-zu-Tag Variabilität durch den Einsatz von automatisierten Liquid-Handling für alle flüssigen Zusätze zu Multi-Well-Platten minimiert. Wir verwenden eine WellMate (Matrix).
- Bereiten WellMate Schlauch.
- Sprayhood und WellMate mit 70% Ethanol.
- Entfernen Schläuche aus der Verpackung und Spray mit 70% Ethanol.
- Das Rohr Kassette in Abgabeposition auf dem WellMate.
- Prime Tubing mit 25 ml 70% Ethanol.
- Sterilisieren Schlauch in Ethanol für 15 Minuten, dann grundieren mit weiteren 25 ml Ethanol.
- Spülen Schlauch durch Grundierung mit 50 ml sterilem PBS.
- Verdrängen Zellen aus Kolben und zählen. Pellet-Zellen so, dass Sie 25% mehr als nötig (300xg, 5 min). Die Anzahl der Zellen pro Vertiefung ausgesät muss nach Assay Länge optimiert werden. Bei der Durchführung von automatisierten Bildanalyse, ist eine einzige Monolage ohne Zelle Verklumpung ideal für Bildsegmentierung.
- Resuspendieren pelletierten Zellen in serum-freien Schneider Medien 1.7x10 6 Zellen / ml.
- Wir führen den Bildschirm in 384-Well-Platten mit einem kommerziell erhältlichen Bibliothek von dsRNA in einer Konzentration von 250ng/well pre-aliquotiert. Mehrere Brunnen jeder Platte sind leer für die Kontrollen, die manuell hinzugefügt wurden. Wir verwenden dsRNA Luciferase als negative Kontrolle. Wir verwenden dsRNA gegen Thread (DIAP) als Kontrolle für robuste RNAi. Knock down dieses anti-apoptotischen Faktor ergibt sich in dramatischen Zelltod und ist eine schnelle visuelle Weg, um die Qualität der knock down beurteilen. Schließlich verwenden wir eine dsRNA gegen das virale Reporter, in diesem Fall beta-Galactosidase, als positive Kontrolle, um zu überprüfen, dass wir Virusinfektion mit unserem Test Auslesen zu verhindern. Wenn Spotting dsRNA in Platte zum Zeitpunkt der Zellaussaat, Aliquot 1 ul dsRNA in die Ecke des Brunnens, um eine einfachere Visualisierung zu ermöglichen, dann Zentrifuge dsRNA auf Boden gut (300xg, 1 min) vor der Zugabe Zellen.
- Verwenden Sie WellMate bis 10 ul Zellen über jeweils gut vorbereitet hinzuzufügen.
- Spin-Zellen auf Platten durch Zentrifugieren 300xg, 1 min.
- Inkubieren in 25 ° C Inkubator für 45 Minuten.
- Add 20 uL komplette Schneider Medien-und Zentrifuge 300xg, 1 min.
- Legen Platten in feuchter Tupperware Behälter ausgekleidet mit Wasser getränkten Papiertüchern. Das Volumen einer 384-Well ist klein, und so, Verdunstung kann große Auswirkungen auf die Biologie, die wiederum zu artifactual Veränderungen in der read-outs der Kante Brunnen führen können. Um dies zu überwinden, sollten Platten unter hoher Luftfeuchtigkeit gehalten werden.
- Inkubieren für drei Tage bis zur robusten knockdown ermöglichen. Es besteht keine Notwendigkeit, die Medien ändern, oder öffnen Sie befeuchteten Behälter während dieser Zeit.
Teil 2: Infektion mit Vaccinia Virus
- Sterilisieren Multi-Channel-Aspirator in Quatricide für 15 Minuten, dann in 70% Ethanol spülen.
- Bereiten WellMate wie oben beschrieben.
- Bereiten Schneider Medien mit 2% FBS für eine Infektion durch Verdünnen komplette Medien 1:5 in serumfreien Medien.
- Filter Rohöl Virus Lager bis 0,8 um Spritzenfilter um Zelltrümmer zu entfernen oder gereinigte Virus.
- Verdünnen Vaccinia-Virus in 2% Serum Schneider Medien Multiplizität der Infektion (MOI) von 2 erhalten.
- Entfernen PBS aus WellMate Schlauch, Grundanstrich mit verdünnten Virus bis der Schlauch frei von Luftblasen.
- Gebrauch sterilisiert Multi-Channel-Aspirator und Vakuumkammer zu entfernen sanft Medien von den Platten der dsRNA behandelten Drosophila-Zellen.
- Dispense 30 ul Virus pro Well auf Platten mit dem WellMate.
- Centrifuge Platten 1 Minute bei 300xg.
- Sterilisieren WellMate Schlauch. Prime mit 25 ml 10% Bleach 10 Minuten warten, Grundanstrich mit weiteren 25 mL 10% Bleichmittel.
- Prime Schläuche mit 50 ml Wasser, gefolgt von 50 ml 70% Ethanol. Zurück Schlauch an der Verpackung.
- Zurück Platten befeuchtet Tupperware Behälter. Inkubation bei 25 ° C für 48 Stunden.
Teil 3: Färbung Plates
- Fix Platten in 15 ul 4% Formaldehyd / PBS für 10 Minuten. Die Flüssigkeit wird entfernt mit einem Multi-Channel-Aspirator und Vakuumkammer bei jedem Schritt. Sobald Platten wurden behoben, sterile Geräte und Reagenzien sind nicht mehr notwendig.
- Entfernen Sie fix und verwenden WellMate zu 30 ul PBST (PBS + 0,1% Triton-X) hinzu. Inkubieren 10 Minuten und wiederholen. Operate WellMate wie oben beschrieben, außer Schlauch muss nicht sterilisiert werden.
- Zum Nachweis von Vaccinia-Infektion verwenden wir ein β-Galactosidase-Reporter von einem frühen / späten Vaccinia-Promotor angetrieben.
- Nehmen Sie Flüssigkeit und inkubieren Zellen in Block (PBST mit 2% BSA) für 10 Minuten.
- Verdünnen primärer Antikörper in Block, flüssiges und fügen 15 ul Antikörper in jede Vertiefung mit einem Multi-Channel wiederholen Pipette (Matrix).
- Spin down-Platten (300xg, 1 min), mit einem klaren Aufkleber decken und Inkubation bei 4 ° C über Nacht.
- Entfernen primärer Antikörper, und verwenden Sie die WellMate, um Brunnen in PBST 3 mal, jeweils 10 Minuten abwaschen.
- Verdünnen fluoreszenzmarkierten sekundären Antikörper (FITC) und Kernfärbung in Block und fügen Sie 15 ul in die Vertiefungen mit einer Multi-Channel wiederholen Pipette.
- Die Platten 1 Stunde bei Raumtemperatur, vor Licht geschützt.
- Wash Brunnen in PBST 3 mal, jeweils 10 Minuten mit dem WellMate.
- Seal Platte mit klaren Aufkleber, Abdeckung, und lagern bei 4 ° C bis zu drei Wochen.
Teil 4: Imaging and Image Analysis
- Verwenden Sie ein automatisiertes Mikroskop bei 20-facher Vergrößerung auf Bild-Platten.
- Machen Sie Bilder von insgesamt Kerne (DAPI) und infizierte Zellen (FITC). Optimieren Belichtungszeit für jeden Kanal individuell um Dynamikbereich zu maximieren.
- Feinabstimmung der automatischen Fokus-Funktion, indem sie Z-Stapel von Bildern, Abstand 1um auseinander, dass die Flugzeuge span oberhalb und unterhalb der erwarteten Fokusebene. Sobald die optimale Fokusebene befindet, passen Autofokus-Einstellungen entsprechend an.
- Bild die gesamte Platte, die Erfassung von mindestens drei Bildern pro Vertiefung sowohl in der Hoechst-Kanal (DAPI) und das Virus Kanal (FITC). Wählen Sie Websites aus verschiedenen Regionen innerhalb eines gut um Bilder, die gut vertreten als Ganzes zu erfassen.
- Verwenden Sie MetaXpress Software, um eine Zeitschrift zu segmentieren schreiben Sie das Image und die Verwendung Count Kerne Module, um die Gesamtzahl der Zellen positiv für die Kernfärbung (insgesamt Zellen) und Infektionen (FITC) für jede Platte zu berechnen.
- Berechnen Prozent Infektion (100 * FITC positive / insgesamt Zellen) und log zu verwandeln.
- Zur Identifizierung Kandidaten, die wir verwenden eine robuste Z berechnet sich aus dem Median und Quartilsabstand jeder Platte basiert. Diese Methode ist unempfindlich gegenüber Ausreißern und unsymmetrischen Daten, und sorgt so für mehr Leistung bei der Identifizierung auch schwacher oder mäßiger hits 10. Für jede Platte ist der Median aus den Log-transformierten Daten subtrahiert. Dann werden die so ermittelten Werte werden um 0,74 fachen Quartilsabstand (die Differenz zwischen den Werten der 75-Perzentil und das 25-Perzentil) unterteilt. Die 0,74-Faktor ist die Angleichung einer Standard-Normalverteilung verwendet.
- Die robuste Z-Score ist ein Maß für die Entfernung in Standardabweichungen vom Mittelwert der Platte. Zum Beispiel zeigt eine robuste Z-Score von 2% der Infektion der gut ist ~ 2 Standardabweichungen von der Platte Median-oder p <0,05.
- Analysieren Zellzahl auf zytotoxische Kandidaten zu identifizieren. Berechnen Sie die robuste Z-Scores der Kernenergie zählt. Kandidaten mit einem robusten Z <-2 in doppelte Bildschirme gelten als zytotoxisch.
- Wells, die eine robuste Z-Scores für die Prozent-Infektion von> 2 oder <-2 in doppelte Bildschirme (p <0,0025) haben, ohne Zytotoxizität gelten als potenzielle Kandidaten aus dem primären Bildschirm.
- Positive Kandidaten werden validiert durch unabhängige Reagenzien. dsRNAs gegen die Gene von Interesse aus einer anderen Region der mRNA generiert und getestet, wie oben.
- Die Gene für die zwei unabhängigen dsRNAs haben die gleiche Phänotyp kann für weitere Untersuchungen in Betracht gezogen werden.
Teil 5: Repräsentative Bilder und Interpretation der Infektionsraten
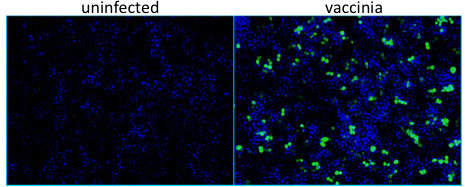
Abbildung 1. Infizierte Brunnen zeigen keine Färbung für Vaccinia-Virus-Proteine, während ein Vertreter infiziert und enthält Zellen Färbung für Vaccinia-Ausdruck beta-Galaktosidase (&bgr; Gal)-Protein, wie Immunfluoreszenzmikroskopie gemessen positiv.Virus = grün, Zellkerne = blau.
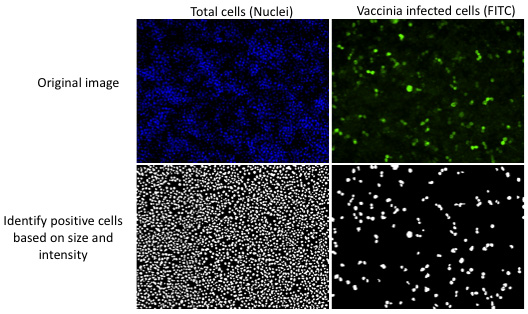
Abbildung 2. Automatische Bildanalyse-Software quantifiziert Infektion in jedem Bild mit Hilfe von Parametern durch den Benutzer festgelegt. In einer repräsentativen Bildes sind die Gesamtzahl der Zellkerne und die Anzahl der Zellen, die Virus-Antigen auf Größe und die Intensität der Färbung über den lokalen Hintergrundfärbung gezählt.

Abbildung 3. Knockdown des Fadens (DIAP) dient als positive Kontrolle für robuste RNAi Depletion von Zielgenen. Knockdown dieser anti-apoptotischen Faktors führt zu dramatischen Zelltod. Zellkerne = blau.
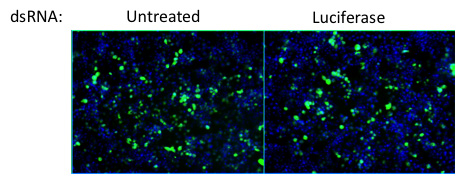
Abbildung 4. Knockdown Luciferase dient als negative Kontrolle für die Wirkung von doppelsträngigen RNA-Behandlung bei Infektion. Depletion von Luciferase hat keinen Einfluss auf Infektionen bezogen auf Zellen unbehandelt. Virus = grün, Zellkerne = blau.

Abbildung 5. Knockdown von &bgr; Gal-Protein dient als positive Kontrolle für eine Verringerung der Infektion, da die Assay verwendet &bgr; Gal-Protein-Spiegel als Auslesen der Infektion. Depletion von &bgr; Gal führt zu einer Abnahme des Anteils der infizierten Zellen. Virus = grün, Zellkerne = blau.
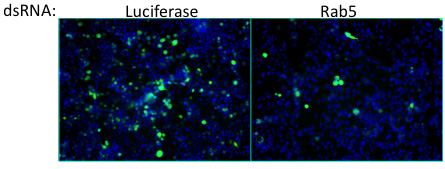
Abbildung 6. Knockdown von zellulären Faktor Rab5 führt zu einer Abnahme des Anteils der infizierten Zellen. Da Rab5 ist bekannt, dass in der Endozytose beteiligt, dieser Faktor trägt wahrscheinlich zur Vaccinia-Virus-Eintrag. Dies stellt ein Beispiel Ausreißer aus dem Bildschirm. Virus = grün, Zellkerne = blau.
Diskussion
Genomweiten RNAi-Screening in Drosophila bietet eine robuste und effiziente Methode für die Untersuchung der zellulären Komponente der Virusinfektion. Eine Reihe von Säugetieren infizieren Drosophila-Zellen, die verwendet werden, um Komponenten des Host intrinsische Immunantwort und Wirtsprotein Faktoren für die virale Replikation, dass neue therapeutische Targets darstellen kann erforderlich identifiziert werden können. Vor der Durchführung eines Bildschirms, muss der Test sorgfältig Zelltyp, Zellzahl und Befall optimiert werden, und muss gut kontrolliert werden, die sowohl positive als auch negative virale und zelluläre Angriffspunkte 11. Es ist wichtig, um eine ausreichende Trennung zwischen positiven und negativen Kontrollen vor dem Screening zu gewährleisten, um den Dynamikbereich des Tests zu maximieren. Sobald der Bildschirm durchgeführt wurde, können die Kandidaten in funktionale Kategorien eingeteilt werden, um festzustellen, welche Host-Mechanismen der Infektion beitragen. Für Säugetiere Viren wie Vaccinia, sollte der Beitrag von Säugetier-Homologe von RNAi als Teil der sekundären Analyse bestimmt werden. Zusammen genommen werden diese robusten Screening-Methoden ermöglichen es uns, Einblicke in komplexe Mechanismen, durch die Viren interagieren mit ihren Wirtszellen zu gewinnen.
Offenlegungen
Danksagungen
Diese Arbeit wurde durch Zuschüsse aus NIAID (R01AI074951, U54AI057168) und der Penn Genome Frontiers Institut SC unterstützt; von NIH T32HG000046 zu TSM; von NIH T32GM07229 und T32AI007324 zu LRS.
Materialien
| Name | Company | Catalog Number | Comments |
| Cell culture | |||
| Schneider’s media | GIBCO, by Life Technologies | 11720 | |
| GlutaMAX 100X | GIBCO, by Life Technologies | 35050 | |
| Penicillin/streptomycin (pen/strep) | GIBCO, by Life Technologies | 15140 | |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F6178 | |
| Screening | |||
| Genome-wide dsRNA library | Ambion | ||
| 384 well plates, black with clear bottom | Corning | 3712 | |
| Aluminum seals | Corning | 6569 | |
| Clear seals | Denville Scientific | B1212-4 | |
| Well Mate | Matlab | 201-10001 | |
| 24 pin manifold | Drummond Scientific | 3-000-101 | |
| H–chst 33342 | Sigma-Aldrich | B2261 | |
| Fluorescently-labeled secondary antibodies | Invitrogen | ||
| ImageXpressMicro automated microscope | Molecular Devices | ||
| MetaXpress image analysis software | Molecular Devices |
Referenzen
- Ramadan, N., Flockhart, I., Booker, M., Perrimon, N., Mathey-Prevot, B. Design and implementation of high-throughput RNAi screens in cultured Drosophila cells. Nat Protoc. 2, 2245-2264 (2007).
- Chotkowski, H. L. West Nile virus infection of Drosophila melanogaster induces a protective RNAi response. Virology. 377, 197-206 (2008).
- Blondel, D., Petitjean, A. M., Dezelee, S., Wyers, F. Vesicular stomatitis virus in Drosophila melanogaster cells: regulation of viral transcription and replication. J Virol. 62, 277-284 (1988).
- Sessions, O. M. Discovery of insect and human dengue virus host factors. Nature. 458, 1047-1050 (2009).
- Cherry, S. Genomic RNAi screening in Drosophila S2 cells: what have we learned about host-pathogen interactions?. Curr Opin Microbiol. 11, 262-270 (2008).
- Hao, L. Drosophila RNAi screen identifies host genes important for influenza virus replication. Nature. 454, 890-893 (2008).
- Cherry, S. COPI activity coupled with fatty acid biosynthesis is required for viral replication. PLoS Pathog. 2, e102-e102 (2006).
- Cherry, S. Genome-wide RNAi screen reveals a specific sensitivity of IRES-containing RNA viruses to host translation inhibition. Genes Dev. 19, 445-452 (2005).
- Moser, T. S., Jones, R. G., Thompson, C. B., Coyne, C. B., Cherry, S. A Kinome RNAi Screen Identified AMPK as Promoting Poxvirus Entry through the Control of Actin Dynamics. PLoS Pathog. 6, e1000954-e1000954 (2010).
- Zhang, J. H., Chung, T. D., Oldenburg, K. R. A Simple Statistical Parameter for Use in Evaluation and Validation of High Throughput Screening Assays. J Biomol Screen. 4, 67-73 (1999).
- Echeverri, C. J., Perrimon, N. High-throughput RNAi screening in cultured cells: a user's guide. Nat Rev Genet. 7, 373-384 (2006).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten