Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
La purificación y la visualización de lipopolisacárido de bacterias Gram-negativas por Hot acuosa de fenol-Extracción
En este artículo
Resumen
Se describe una modificación acuosa caliente-fenol método de extracción para la purificación de lipopolisacárido (LPS) de bacterias Gram-negativas. Una vez extraído, el LPS puede ser seguidamente analizadas por SDS-PAGE y se visualizaron mediante tinción directa o inmunotransferencia Western.
Resumen
Lipopolisacárido (LPS) es un componente importante de bacterias Gram-negativas membranas externas de bacterias. Es una molécula tripartito compuesto de lípido A, que está incrustada en la membrana exterior, un núcleo de oligosacáridos y repetición de O-antígeno unidades que se extienden hacia fuera desde la superficie de la célula 1, 2. LPS es una molécula inmunodominante que es importante para la virulencia y patogénesis de muchas especies de bacterias, incluyendo Pseudomonas aeruginosa, especies de Salmonella, Escherichia coli y 3-5, y las diferencias en forma LPS composición antígeno O-la base para serotipado de las cepas. LPS está implicado en la unión a las células huésped en el inicio de la infección y proporciona protección contra la muerte mediada por complemento; cepas que carecen de LPS puede ser atenuada para la virulencia 6-8. Por estas razones, es importante visualizar LPS, particularmente a partir de aislados clínicos. LPS Visualización de los patrones de bandas y el reconocimiento por una específicantibodies pueden ser herramientas útiles para identificar linajes de tensión y para caracterizar distintos mutantes.
En este informe, se describe un acuosa caliente-fenol método para el aislamiento y purificación de LPS de bacterias Gram-negativas células bacterianas. Este protocolo permite la extracción de LPS de distancia a partir de ácidos nucleicos y proteínas que pueden interferir con la visualización de LPS que se produce con más cortos, los métodos de extracción menos intensivas 9. LPS preparado de esta manera se pueden separar por dodecil sulfato sódico (SDS)-poliacrilamida electroforesis en gel (PAGE) y se tiñeron utilizando directamente carbohidrato / glicoproteína manchas o estándar métodos de tinción de plata. Muchos anti-sueros a LPS contienen anticuerpos que reaccionan de forma cruzada con las proteínas de membrana externa u otros objetivos antigénicos que pueden obstaculizar la reactividad observados después de inmunotransferencia Western de SDS-PAGE-separados lisados celulares crudos. Tratamiento de la proteasa de lisados celulares crudos por sí solos no siempre es una manera eficaz de eliminar estede fondo de usar este u otros métodos de visualización. Además, el tratamiento de la proteasa extensa en un intento de eliminar este fondo puede llevar a LPS de baja calidad que no está bien resueltas por cualquiera de los métodos antes mencionados. Por estas razones, creemos que el protocolo siguiente, adaptado de Westpahl y Jann 10, es ideal para la extracción de LPS.
Protocolo
1. Preparación de las bacterias para la extracción de LPS
- Empezar un cultivo de una noche en 5 ml de caldo Luria (LB), suplementado con antibióticos si es necesario. Cultive hasta el día (12-18 horas) en una incubadora de agitación a 37 ° C y 200 rpm.
- Diluir la 1:10 de cultivo con LB y tener un 600 OD lectura en un espectrofotómetro. Sobre la base de los 600 OD lectura, hacer una suspensión de 1,5 ml a las bacterias a un OD 600 de 0,5.
- Sedimentar las bacterias en una microcentrífuga a 10.600 x g durante 10 minutos. Retire y deseche el sobrenadante. El sedimento puede ser almacenado a -20 ° C, si LPS no va a ser extraído inmediatamente.
2. Extracción de LPS
- En primer lugar, preparar 2x tampón SDS. Hacer una solución de 50 ml (BME) 4% β-mercaptoetanol, 4% de SDS y 20% de glicerol en 0,1 M Tris-HCl, pH 6,8. Añadir una pizca de azul de bromofenol para la coloración de la solución. Haga una 1x SDS-tampón de stock de diluir 2X stac 1:1 en agua destilada estéril H 2 O. Esto se puede almacenar a temperatura ambiente.
- Haga tres separadas 10 mg / ml de soluciones de DNasa I, RNasa y proteinasa K en agua destilada estéril H 2 O.
- Resuspender las bacterias sedimentadas desde el paso 1.3 en 200 l de 1x SDS-tampón. Asegurarse de que el sedimento se resuspendió completamente pipeteando la solución arriba y hacia abajo lentamente. No vórtice.
- Hervir las bacterias en suspensión en un baño de agua durante 15 minutos. Dejar que la solución se enfríe a temperatura ambiente durante 15 minutos.
- Añadir 5 l de ambos DNasa I y RNasa soluciones preparadas en 2,2. Se incuban las muestras a 37 ° C durante 30 minutos. * Este paso es opcional, no hay diferencia mínima en la calidad de LPS entre las muestras de nucleasa tratados y no tratados.
- Añadir 10 ml de la solución de proteinasa K preparado en 2,2. Se incuban las muestras a 59 ° C durante 3 horas. Este paso puede realizarse durante la noche, si hay limitaciones de tiempo.
- Para cada muestra, anunciod 200 l de helado de Tris-fenol saturado (agua saturada con fenol puede utilizarse como un sustituto si Tris-fenol saturado no está disponible). Asegúrese de que las tapas de los tubos estén bien cerradas, y cada muestra de vórtice por aproximadamente 5 a 10 segundos.
- Se incuban las muestras a 65 ° C durante 15 minutos, agitación ocasional. Después de incubar enfriar a temperatura ambiente, a continuación, añadir 1 ml de éter dietílico a temperatura ambiente a cada muestra y agitar durante 5 a 10 segundos. Asegúrese de realizar el éter mango en una campana extractora, ya que es volátil.
- Centrifugar las muestras a 20.600 x g durante 10 minutos. Retire con cuidado las muestras de la centrífuga y extraer la capa azul abajo. Asegúrese de evitar la capa superior, claro. Dejando atrás una pequeña cantidad de la capa azul es preferible a la contaminación con la capa superior.
- Vuelva a extraer las muestras repitiendo los pasos 2,7 a 2,9. Dos extracciones suelen ser suficientes. Si las muestras de aspecto turbio, más extracciones pueden ser Realizamosd. Añadir 200 l de SDS-2x tampón a cada una de las muestras extraídas antes de separar por SDS-PAGE. Las muestras se pueden ejecutar en 8% -15% de SDS-geles de poliacrilamida. De cinco a quince l de LPS preparados con este método suele ser suficiente para la visualización.
3. Los resultados representativos
LPS muestras preparadas como anteriormente pueden ser visualizadas por tinción directa después de la separación en SDS-PAGE usando un estándar de tinción con plata protocolo o un kit de LPS tinción disponibles comercialmente. Alternativamente, el LPS separados en un gel de poliacrilamida puede ser transferido a una membrana de nitrocelulosa y se sometieron a inmunotransferencia usando LPS-específicos anti-sueros. Para este protocolo, se utilizó el Pro-Q Esmeralda 300 Kit de Gel Stain lipopolisacárido (Molecular Probes), y siguió las instrucciones del fabricante.
Se muestra en la Figura 1 es un Pro-Q Esmeralda 300 manchado 12% de SDS-gel de poliacrilamida. Cada carril contiene 15 l de LPS preparados a partir de didiferentes cepas de Burkholderia dolosa aislado de muestras de esputo de los pacientes con fibrosis quística. LPS diferentes patrones de bandas reflectantes escalera, de diferentes números de O-antígeno unidades repetitivas unidos al núcleo oligosacárido, son evidentes con este método, por ejemplo, las muestras en el carril 1 y 6 tienen similares patrones de bandas entre sí (en caja en rojo).
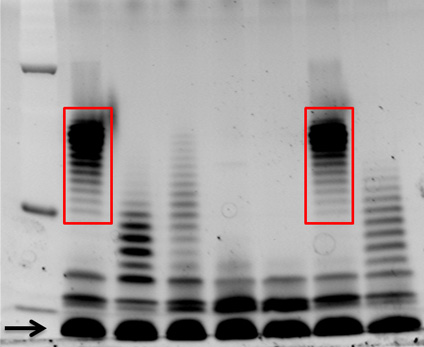
Figura 1. Pro-Q Esmeralda 300 LPS teñidos de Burkholderia dolosa aislamientos clínicos. LPS de siete B. cepas dolosa de un brote en pacientes con fibrosis quística fueron aislados como se describe en este protocolo. Quince l fueron separados en un 12% de SDS-gel de poliacrilamida, y se tiñeron con Pro-Q Esmeralda 300, según las instrucciones del fabricante. LPS central se indica una flecha, y LPS muestran similares patrones de bandas de repeticiones O-antígeno se empacan en cajas de color rojo. M = marcador de peso molecular.
Discusión
Hemos descrito un método de purificación de LPS de distancia de otros componentes celulares, incluyendo los ácidos nucleicos y proteínas. Este método proporciona alta calidad de LPS que pueden ser utilizados en un número de métodos de visualización diferentes, incluyendo tinción de carbohidratos de geles de SDS-PAGE, como se muestra en la Figura 1. Este método puede ser utilizado al serotipo LPS de una variedad de cepas, utilizando anticuerpos específicos anti-sueros, o para mostrar la relación entre los ais...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue apoyado por becas de los Institutos Nacionales de Salud y la Fundación de Fibrosis Quística.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| DNasa I recombinante, libre de RNasa | Roche | 04716728001 | |
| RNasa A | Roche | 10109169001 | |
| Proteinasa K | Pescador | BP1700 | |
| Tris-fenol saturado | Pescador | BP1750-100 | |
| Éter dietílico | Thomas Scientific | C313K31 | |
| Pro-Q Esmeralda 300 lipopolisacárido Kit de Gel Stain | Molecular Probes | P20495 |
Referencias
- Raetz, C. R., Whitfield, C. Lipopolysaccharide endotoxins. Annu. Rev. Biochem. 71, 635-700 (2002).
- Valvano, M. A. Export of O-specific lipopolysaccharide. Front. Biosci. 8, 452-471 (2003).
- Pier, G. B. Pseudomonas aeruginosa lipopolysaccharide: a major virulence factor, initiator of inflammation and target for effective immunity. Int. J. Med. Microbiol. 297, 277-295 (2007).
- Freudenberg, M. A. Role of lipopolysaccharide susceptibility in the innate immune response to Salmonella typhimurium infection: LPS, a primary target for recognition of Gram-negative bacteria. Microbes Infect. 3, 1213-1222 (2001).
- Jann, K., Jann, B. Polysaccharide antigens of Escherichia coli. Rev. Infect. Dis. 9, S517-S526 (1987).
- McCallum, K. L., Schoenhals, G., Laakso, D., Clarke, B., Whitfield, C. A high-molecular-weight fraction of smooth lipopolysaccharide in Klebsiella serotype O1:K20 contains a unique O-antigen epitope and determines resistance to nonspecific serum killing. Infect. Immun. 57, 3816-3822 (1989).
- Hancock, R. E. Pseudomonas aeruginosa isolates from patients with cystic fibrosis: a class of serum-sensitive, nontypable strains deficient in lipopolysaccharide O side chains. Infect. Immun. 42, 170-177 (1983).
- Gupta, S. K., Masinick, S., Garrett, M., Hazlett, L. D. Pseudomonas aeruginosa lipopolysaccharide binds galectin-3 and other human corneal epithelial proteins. Infect. Immun. 65, 2747-2753 (1997).
- Hitchcock, P. J., Brown, T. M. Morphological heterogeneity among Salmonella lipopolysaccharide chemotypes in silver-stained polyacrylamide gels. J. Bacteriol. 154, 269-277 (1983).
- Westphal, O., Jann, K. Bacterial lipopolysaccharides. Methods Carbohyr. Chem. 5, 83 (1965).
- Lieberman, T. D. Parallel bacterial evolution within multiple patients ties novel genes to pathogenesis. Nat Genet. , (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados