Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Procesamiento de tejido primario Tumor cerebral para ensayos con células madre y flujo de Ordenación
En este artículo
Resumen
La identificación de células tumorales cerebrales iniciador (BTICs), las células raras dentro de una heterogéneos tumorales que poseen propiedades de células madre, proporciona nuevos conocimientos sobre la patogénesis humana tumor cerebral. Hemos mejorado las condiciones de cultivo específicos para enriquecer BTICs, y nosotros usamos la citometría de flujo para enriquecer aún más a estas poblaciones. De auto-renovación ensayos y análisis de transcripción por una sola célula de RT-PCR posteriormente se puede realizar en estas células aisladas.
Resumen
Los tumores cerebrales son típicamente compuesto de células morfológicamente distintas que expresan una variedad de marcadores de linaje neural. Sólo una fracción relativamente pequeña de las células en el tumor con propiedades de células madre, denominadas células tumorales cerebrales iniciador (BTICs), poseen una capacidad de diferenciarse a lo largo de múltiples linajes, auto-renovación, e iniciar los tumores in vivo. Se aplicaron condiciones de cultivo usadas originalmente para normales células madre neurales (NSC) a una variedad de tumores cerebrales humanos y se ha encontrado que este método de cultivo selecciona específicamente para como vástago-poblaciones. Medio libre de suero (NSC) permite el mantenimiento de un estado indiferenciado de las células madre, y la adición de bFGF y EGF permite la proliferación de multi-potente, auto-renovación, y tumorspheres expandibles.
Para caracterizar aún más la población BTIC cada tumor, se evalúa marcadores de superficie celular por citometría de flujo. También podemos ordenar las poblaciones de interés para characteriz más específicosación. De auto-renovación ensayos se realizan en BTICs individuales ordenados en placas de 96 pocillos; la formación de tumorspheres tras la incubación a 37 ° C indica la presencia de un tallo o células progenitoras. Varios números de celulares de una población en particular también se pueden clasificar en diferentes pozos para limitar el análisis de dilución, para analizar la capacidad de auto-renovación. También se puede estudiar la expresión diferencial de genes dentro de una población celular particular mediante el uso de células individuales de RT-PCR.
Los protocolos siguientes describen los procedimientos para la disociación y el cultivo de muestras humanas primarias para enriquecer poblaciones BTIC, así como la disociación de tumorspheres. También se incluyen los protocolos de tinción para el análisis de citometría de flujo o de la clasificación, los ensayos de auto-renovación, y única célula de RT-PCR.
Introducción
Los tumores cerebrales son algunos de los cánceres más agresivos y heterogéneos conocidos en los seres humanos. A pesar de su detección temprana y el diagnóstico han sido facilitados por la moderna tecnología de neuroimagen, todavía carecemos de terapias curativas para muchos tumores cerebrales, en particular para los difusas, invasivos o localizados profundamente en el cerebro.
Los tumores cerebrales representan la principal causa de mortalidad por cáncer en los niños debido a su naturaleza altamente agresivo e incurable a menudo. El glioblastoma (GBM), el tumor cerebral primario más común en los adultos, es uno de los cánceres humanos más agresivos, temían por su pronóstico uniformemente fatal 1. Este tumor altamente maligno astrocytic (OMS grado 4) por lo general ocurre en los hemisferios cerebrales de los adultos, y también puede ocurrir en niños pequeños y bebés. Su crecimiento es rápido y infiltrante y diagnósticos características patológicas incluyen pleomorfismo nuclear, la proliferación microvascular, necrosis y 2,3. Para los adults con diagnóstico reciente de GBM, la supervivencia media rara vez se extiende más allá de 12 meses 1, con respuestas generalmente pobres a todas las modalidades terapéuticas. Hemos observado que hay muchas similitudes funcionales y genéticos compartidos por las células madre somáticas y células cancerosas, y que las vías moleculares que regulan el desarrollo normal del cerebro están a menudo alteradas en cáncer. En la aplicación de paradigmas madre de biología celular para el estudio de los tumores cerebrales, que fueron los primeros investigadores para identificar prospectivamente y purificar una subpoblación de células de GBMs humanos que presentaban las propiedades de células madre de la proliferación, la auto-renovación y diferenciación in vitro y en 4 5 in vivo. Se aplicaron condiciones de cultivo y ensayos originalmente utilizados para caracterizar normales las células madre neurales (NSC) in vitro 6,7 a múltiples tumores cerebrales pediátricos y adultos, y enriquecido para estas células madre-como por clasificación de células para el marcador de superficie celular CD133 progenitora neural 8 ,9. El CD133 + fracción tumor cerebral contenía células que tenían una frecuencia mucho más alta de la iniciación del tumor que la fracción CD133-en-SCID NOD cerebros de ratón 5,10. Este formalmente establecido que sólo un raro subconjunto de las células tumorales del cerebro con propiedades de células madre son iniciadoras del tumor, ganándose el nombre de "tumor cerebral células iniciadoras" o "BTICs". La identificación novela de BTICs proporciona nuevos conocimientos sobre la tumorigénesis cerebro humano, dando un fuerte apoyo a la hipótesis de las células madre del cáncer 10-13 como base para muchos tumores sólidos, y establece una nueva diana celular de terapias más eficaces contra el cáncer 14-20. Las terapias que se centran en matar a la mayor parte del tumor puede pasar por alto la rara madre como fracción, permitiendo que el tumor siga creciendo. Las terapias que se centran en matar a la célula madre del cáncer pueden proporcionar un mejor tratamiento y pronóstico de los pacientes con tumores cerebrales.
Con el fin de estudiar las poblaciones BTIC, hemos mejorado nuestra culture protocolos para seleccionar específicamente para las poblaciones de células dentro de los tumores cerebrales humanos, que poseen propiedades de células madre. Libre de suero, células madre neurales (NSC) medio permite el mantenimiento de un estado indiferenciado de las células madre, y la adición de factor de crecimiento de fibroblastos básico (bFGF), factor de crecimiento epidérmico (EGF), y factor inhibidor de leucemia (LIF) permite la proliferación de multi-potente, la auto-renovación y ampliable tumorspheres humanos. A continuación, se describen los métodos empleados en el procesamiento de los tumores cerebrales primarios y cultivarlas en un medio de NSC para enriquecer a las poblaciones BTIC. Hemos llamado a nuestro sistema modelo experimental "aislados de pacientes BTIC" para enfatizar el hecho de que estas células se cultivan en sólo mínimamente tallo las condiciones para la celda para seleccionar poblaciones de células madre. immunolabelling posterior de poblaciones BTIC clave para marcadores de células madre tales como CD133 y CD15 y el análisis de citometría de flujo también se describe. A continuación se discute el análisis de dilución limitante,que ayuda en el estudio del potencial de auto-renovación de BTICs. Por último, se explora el análisis de la expresión génica de estas células raras clasificar las células individuales en portaobjetos AmpliGrid y la realización de una sola célula de RT-PCR. Estas técnicas son también aplicables a otros tumores cerebrales, tales como meduloblastoma, ependimoma y gliomas pediátricos.
Protocolo
1. Cultivo de tejido del tumor cerebral
- Añadir 200 l Liberase descongelado (Roche Applied Science) a 15 ml de líquido cefalorraquídeo artificial (LCRa-ver Tabla 1) y ponerlo en un baño a 37 ° C. Liberase TM es una mezcla de enzimas proteolíticas utilizadas para disociar muestras de tejidos primarios, así como tumorspheres cultivadas. A diferencia de tripsina-EDTA, el método Liberase conserva el antígeno CD133 superficie. Para obtener una muestra de tejido de aproximadamente 0,5 cm 3, usamos 200 l de Liberase. Si el tejido es más pequeño, se utiliza 100 ul.
- Llevar la solución de cloruro de amonio (tecnologías de células madre) a temperatura ambiente. Solución de cloruro amónico suavemente lisa las células rojas de la sangre con un mínimo efecto sobre otras células. No contiene un fijador.
- En el gabinete de seguridad biológica estéril, añadir 5 ml de muestra a LCRa recipiente en forma de remolino para lavar tejidos, luego de pipetear. Este paso sirve para eliminar los glóbulos rojos (RBC).
- Transferencia del tejido de tumor cerebral a un estéril 100 mm Petri dish.
- Con unas tijeras finas o bisturís y pinzas, desagregar tejido de consistencia de lechada.
- Recoja la muestra con una pipeta 10 ml regular o fórceps y fragmentos de transferencia en el tubo que contiene precalentado LCRa con Liberase.
- Colocar en una incubadora agitador (30 rpm) y se puso a 37 ° C, durante 15 min.
- Filtrar el lisado de tejido a través de 70 micras filtro de células en un tubo Falcon de 50 ml.
- Haga girar el filtrado abajo a 280 xg durante 5 min.
- Eliminar con cuidado el sobrenadante y evaluar el tamaño y el color de la sedimento celular resultante: gránulos que son de color rosa o rojo indican números crecientes de células rojas de la sangre.
- Resuspender el sedimento en 1 ml de PBS.
- Añadir una cantidad apropiada de solución de cloruro de amonio (4-12 ml) en base a tamaño de los gránulos y la contaminación de eritrocitos (la solución de cloruro de amonio es muy suave y cantidades aumentadas no son perjudiciales para las células distintas de las células rojas).
- Incubar a temperatura ambiente durante 5 min.
- Células Girohacia abajo a 280 xg durante 5 min.
- Lavar una vez con 10 ml de PBS estéril.
- Resuspender en 5 ml de medio NSC completa (Tabla 2) y la transferencia a un ultra-baja unión de 60 mm placa de cultivo de tejidos (Corning). Utilizamos ultra-bajo placas de cultivo de unión con superficies de hidrogel unido covalentemente que son hidrófilos y con carga neutra, para minimizar la adhesión celular mediada por diferenciación.
Durante los primeros días en la cultura, no cambian los medios de comunicación: top-up sólo con 1-2 ml de medio, según sea necesario, y luego continuar a observar los medios de cultivo y el cambio en el color de los medios de comunicación se vuelve ligeramente amarillo.
2. Tumorsphere disociación de Citometría de Flujo
- Evaluar tumorspheres bajo microscopio: si el tamaño de la esfera es> 100 mm, la disociación se recomienda como esferas más grandes pueden llegar a ser necrótico en el centro.
- Traslado cultura a 15 ml tubo cónico.
- Añadir 2-3 ml estéril PBS para enjuagar la placa de fondo y añadir al tubo cónico.
- Centrifugar a 280 xg durante 5 min.
- Remover el sobrenadante y resuspender en 1 a 2 ml de PBS estéril.
- Añadir 10 l Liberase.
- Incubar en baño a 37 ° C durante 3 min. Retire y evaluar visualmente la suspensión, si matas múltiples visto, triturar suavemente con una punta de pipeta de 1.000 l.
- Si persiste la aglutinación, continuar la incubación durante otras 1-2 min.
- Lavar las células mediante la adición de 5 ml de PBS estéril y centrifugación a 280 xg durante 5 min.
- Remover el sobrenadante y se resuspende en PBS estéril 500-1,000 l EDTA 2 mM.
- Evaluar el número de células y la viabilidad utilizando azul de tripano.
- Ajustar el recuento de células a 1 millón / ml en PBS EDTA 2 mM.
3. Tinción de la superficie
- Transferir 100 l de suspensión celular 1x10 6 / ml por prueba, tubos de 12x75 mm de caudal.
- Añadir la cantidad apropiada de anticuerpos. Conjunto combinado de controles de isotipo se debe utilizar para cadaanticuerpo (véase la Tabla 3).
- Incubar durante 30 min en hielo.
- Añadir 2 ml de PBS 2 mM EDTA a cada tubo de flujo.
- Centrifugar a 200 xg durante 4 min, se decanta y blot.
- Resuspender el pellet en 300 l de PBS 2 mM EDTA.
- Añadir 10 l de tinte viabilidad 7AAD a cada tubo y se incuba durante al menos 15 min en hielo.
- Analizar mediante citometría de flujo.
4. Citometría de flujo de Adquisición y Análisis
Los detalles específicos de adquisición y análisis de datos de flujo son instrumento-dependiente. Representativas muestras negativas se ejecutan y ajustes del instrumento establecido para Adelante (tamaño) y lateral (granularidad) de dispersión. Este patrón de dispersión permite que el usuario final para ver todas las células en la muestra, incluyendo los desechos. Una región es por lo general dibujado alrededor de las células de interés. (Figura 4a) La configuración se establece entonces para todos los detectores de fluorescencia requerida para colocar las células negativas dentro de la primera década del afparcela intensidad luorescence. Cuando más de un colorante o fluorocromo se utiliza, controles individuales teñidas se debe ejecutar para establecer los valores de compensación de color (sustracción de interferir las emisiones de fluorescencia). Cuando se ejecuta células vivas, un colorante de viabilidad tal como 7-AAD (7-amino-actinomicina D - 488/emission excitación 655) se utiliza y una segunda región dibujado para excluir las células muertas (Figura 4b). Ambas de estas regiones se aplican a cualquier otro análisis de las muestras.
5. Auto-renovación Ensayo
El ensayo de clave que se facilita en gran medida por el crecimiento clonal esfera es la prueba in vitro de la capacidad de auto-renovación, el ensayo de dilución limitante. Tumorspheres se disocian y se distribuye en diluciones a una única célula por pocillo, y la tasa de formación de esferas posterior durante 7 días en cultivo se calcula. En el día 7, el porcentaje de pozos que no contiene esferas para cada densidad de placas de células (F 0) se calcula y se trazacontra el número de células por pocillo (x). El número de células necesarias para formar al menos un tumorsphere en cada pocillo se determina desde el punto en el cual la línea cruza el nivel de 0,37 (F = 0 e-x). En una distribución de Poisson de las células, F 0 = 0,37 corresponde a la dilución de una célula por pocillo, de modo que este cálculo (el 37% se cruzan) refleja la frecuencia de clonogénicas células madre en la población de células enteras (Figura 5).
6. Single Cell RT-PCR
Las células aisladas de diferentes poblaciones están ordenadas por Beckman Coulter MoFlo XDP en los sitios de reacción en una diapositiva AmpliGrid (Beckman Coulter cat # AG480F). Individual de células RT-PCR se realiza usando AmpliGrid individual One Step RT-PCR (Beckman Coulter cat # OAX04515) de acuerdo con las especificaciones del fabricante. En resumen, una sola célula se deposita en cada sitio de reacción de una diapositiva AmpliGrid; la presencia de una única célula en cadasitio de reacción confirmó por microscopía. Transcripción reversa (RT) se lleva a cabo inmediatamente después de la clasificación para evitar que la muestra se vea comprometida. La mezcla RT maestro está preparado fresco, de acuerdo con las instrucciones del kit (Tabla 4). Se utilizó 0,03 l de 25 cebadores aleatorios mu M (Promega) por reacción; 1 l de mezcla maestra se añade a la centro de cada sitio de reacción. Esta es inmediatamente recubierto con 5 l de solución de sellado (Beckman Coulter cat # OAX04503). Uso de una diapositiva AmpliSpeed ciclador (Beckman Coulter; cat # OAX04101), los portaobjetos se incubaron a 25 ° C durante 10 min, 42 º C durante 10 min, y 55 ° C durante 45 min. Después de la transcripción inversa, 3 l de DNasa / RNasa libre de agua se añade a cada sitio de reacción, y la mezcla entera se transfirió a un tubo de PCR (1 tubo / sitio). En una placa de PCR, un volumen de reacción de 10 l se utiliza para la PCR cuantitativa: 8 l qPCR mezcla maestra (5 l SYBR Green (Quanta), 1 l de agua, 1 l de 10 mM de avance y mezcla de cebador inverso) más 2 μ l de la mezcla de RT. Para la cuantificación en tiempo real, las muestras se ejecutan en un termociclador BioRad usando el siguiente programa: 95 º C durante 10 min, 45 ciclos de 95 ° C durante 30 seg, 60 ° C durante 60 seg, 72 ° C durante 60 segundos, seguido por 10 min a 72 ° C y el análisis de curva de fusión. Los valores de Ct se determinaron usando Opticon Monitor 3 (BioRad). Tres genes pueden ser evaluados por célula, incluyendo GAPDH como gen de mantenimiento. La expresión génica se normalizó a la expresión de GAPDH, de acuerdo a la 2-? Ct. Por lo menos una docena de células individuales de la misma población de la misma especie se puede utilizar como repeticiones biológica para compensar la falta de repeticiones técnica.
7. Los resultados representativos
La figura 1 muestra una imagen de resonancia magnética (IRM) de un paciente con un representante GBM. Muestras de tumores cerebrales son obtenidos a partir de pacientes que consienten inmediatamente después de la cirugía, según lo aprobado por las Ciencias de la Salud de Hamilton / McMaster Health Sciences Junta Ética de Investigación. Una parte de cada muestra se le da a la neuropatólogo para el diagnóstico clínico de rutina. La muestra restante se procesa como se muestra en la Figura 2. Las células tumorales, una vez sembradas en medios completos de células madre neuronales con factores de crecimiento, tumorspheres forma como se muestra en la Figura 3.
Las células tumorales están marcados con marcadores neuronales de superficie de células madre CD133 y CD15 y se analizaron por citometría de flujo como se muestra en la Figura 4.
Las células individuales de diferentes poblaciones para, por ejemplo, CD15 + / CD133 +, CD15 + / CD133-, CD15-/CD133 +, CD15-/CD133- continuación, se ordenan en placas de 96 pocillos (figura 5a) o en diapositivas AmpliGrid (Figura 6a).
En la placa de 96 pocillos, las células individuales que poseen la capacidad de auto-renovación (por ejemplo células CD133 +), (Figura 5b), pueden formar tumorspheres (Figura 5c) tras incubación. Un gráfico de la auto-renovación puede ser trazado (Figura 5d) contando el número de esferas formadas por pocillo. Las células individuales ordenados en el sitio de reacción de la corredera AmpliGrid (Figura 6a). El ARN extraído a partir de células individuales se pueden transcribe de forma inversa usando el AmpliSpeed Advalytix (Figura 6b). El cDNA obtenido se utiliza para realizar en tiempo real RT-PCR (Figura 6c).
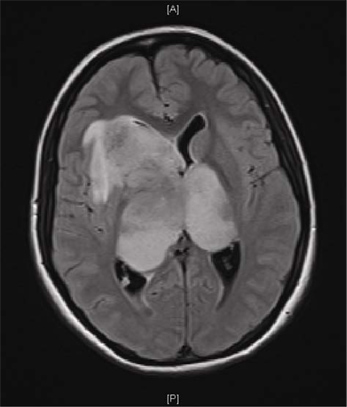
Figura 1. Imagen por resonancia magnética (MRI) de un paciente GBM. Resonancia magnética del cerebro de un joven de 16 años de edad con una historia de un mes de dolor de cabeza y una historia de una semana de vómitos, letargo y visión borrosa. Secuencia FLAIR axial muestra una gran bi-hemisférico ("mariposa") GBM.
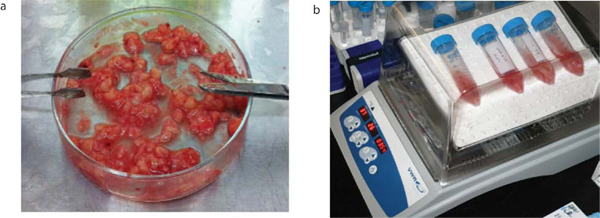
Figura 2. Tratamiento de tumores cerebrales. Muestras de tumores cerebrales son obtaINED consentimiento de los pacientes inmediatamente después de la cirugía. Son disociado mecánicamente como se muestra (a) y son entonces enzimáticamente disociarse en aCSF con Liberase por incubación en una incubadora de 30 rpm balanceo a 37 º C durante 15 min (b).

Figura 3. Poblaciones BTIC formar tumorspheres en cultivo. Disociadas las células tumorales del cerebro sembradas en medios de comunicación completas de células madre neuronales con factores de crecimiento forman tumorspheres.
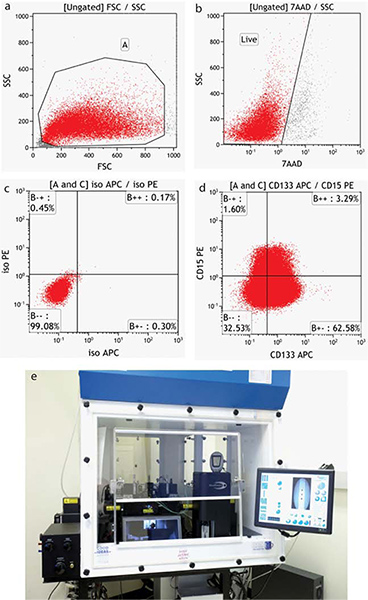
Figura 4. Análisis de citometría de flujo de poblaciones BTIC. (A) hacia delante frente a las propiedades de dispersión lateral proporcionan una imagen de todas las células, incluyendo los desechos (b) Las células que se tiñen con el colorante de viabilidad 7-AAD se excluyen del análisis. (C) La posición de los cuadrantes estadísticos se determina usando controles apropiados isotipo (d) Las células tumorales teñidas con superficie markers CD15-PE y APC-CD133. (D) MoFlo clasificador de células XDP. Haga clic aquí para ampliar la cifra .
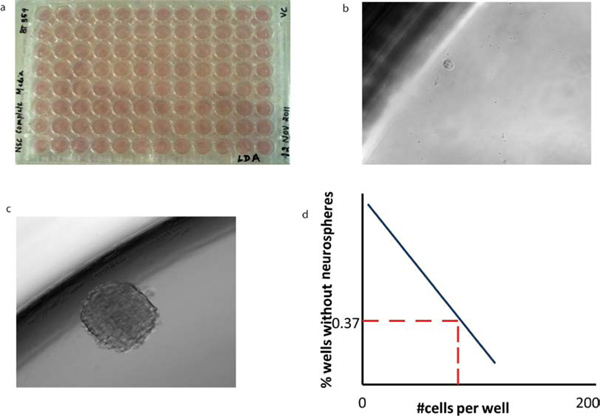
Figura 5. Limitando el análisis de dilución de BTICs revela su capacidad de auto-renovación. (A) Células de diluciones variables se ordenan en 96 pocillos que contienen 200 l de NSC a (b) una única célula por pocillo. (C) Después de un período de incubación de 7 días, se forma un tumorsphere. La morfología de tumorspheres secundarios es idéntica a la de tumorspheres primarios. (D) Los medios de intercepción x-valores se pueden calcular a partir de análisis de dilución limitante para cada subtipo de tumor para revelar el número de células necesarias para formar al menos un tumorsphere por pocillo.

Figura 6. Análisis de expresión génica de una célula única BTICes posible usar sola célula RT-PCR tecnología. (a) células individuales de una población BTIC se pueden ordenar en portaobjetos AmpliGrid. (B) la síntesis de ADNc se realizó en las células individuales en las diapositivas AmpliGrid utilizando el AmpliSpeed Advalytix. (C) Quantitative PCR en tiempo real se realiza en el cDNA sintetizado a partir de células individuales BTIC.
| 2 M NaCl | 62 ml |
| 1 M KCl | 5 ml |
| 1 M MgCl 2 | 3,2 ml |
| 155 mM NaHCO 3 | 169 ml |
| La glucosa 1M | 10 ml |
| 108 mM CaCl 2 | 0,9256 ml |
| Purelab Ultra H 2 0 | 749,84 ml |
Tabla 1 fluido cerebroespinal artificial (ACSF) -. 1.000 ml.
| Basales de los medios de comunicación | 500 ml |
| 01:01 DMEM: F12 | 480 ml |
| N2 suplemento | 5 ml |
| HEPES 1 M | 5 ml |
| Glucosa | 3,0 g |
| N-acetilcisteína (60 mg / ml) | 1 ml |
| Factor-1 de la supervivencia neuronal (NSF-1) | 10 ml |
Tabla 2. Neuronales medios de células madre.
NSC completos medios de comunicación (recién hechas antes de su uso):
NSC basales medios + 20 ng / ml de EGF (factor de crecimiento epidérmico) + 20 ng / ml de bFGF (factor de crecimiento de fibroblastos básico) + 10 ng / ml de LIF (factor inhibidor de la leucemia) * + 10 l / ml de antibiótico-antimicótico
* Factor inhibidor de la leucemia (LIF): Este reactivo de Millipore contiene una interleuquina 6 citoquina de clase, un prOtein que suprime la diferenciación espontánea de células madre que promueven el mantenimiento a largo plazo de los cultivos de células madre.
| Anticuerpos | Proveedor / CAT # | l / prueba (en 100 l) |
| CD133 / 2 APC (293C3) | Miltenyi Biotec/130-090-854 | 5 |
| IgG2b APC (control de isotipo) | Miltenyi Biotec/130-092-217 | 5 |
| CD15PE | Beckman Coulter/IM1954U | 5 |
| IgG2a PE (Isotipo control) | Beckman Coulter/A09141 | 5 |
| 7AAD | Beckman Coulter/A07704 | 10 |
Tabla 3. Anticuerpos de flujo.
| Componente | Volumen (1 reacción) | Volumen (48 reacción) |
| 2x Single Cell tampón de reacción RT | 0,50 l | 30,0 l |
| Inhibidor de RNasa (10 U / l) | 0,02 l | 1,2 l |
| 5X Single Cell RT potenciador | 0,15 l | 9,00 l |
| Single Cell RT Enzyme Mix | 0,04 l | 2,4 l |
| Advablue (OAX04227) | L 0,1 | 6,00 l |
| Nucleasa libre de agua | completar hasta 1 l | llevar hasta 60 l |
Tabla 4. Composición de RT mezcla maestra para RT-PCR Single Cell (cat # OAX04515).
Discusión
La hipótesis de las células madre de cáncer 10, basado en el trabajo en la leucemia 21, 11 de cáncer de mama y cáncer cerebral 4,5, sugiere que sólo una fracción relativamente pequeña de las células en el tumor, denominadas células madre de cáncer, poseen la habilidad de proliferar extensivamente y auto -renovación. La mayoría de las células tumorales pierden la capacidad de proliferar y de auto-renovación, ya que se diferencian en células que se convierten en la firma fe...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Este trabajo fue financiado por el Instituto de Ontario de Investigación del Cáncer (OICR), la Fundación Terry Fox y la Asociación Americana de Cirujanos Neurológicos.
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Número de catálogo | |
| 01:01 DMEM: F12 | Invitrogen | 11320-082 | |
| N2 suplemento | Invitrogen | 17502-048 | |
| HEPES 1 M | Wisent | 330-050-EL | |
| Glucosa | Invitrogen | 15023-021 | |
| N-acetilcisteína | Sigma Aldrich | A9165-25G | |
| Factor de supervivencia neuronal -1 (NSF-1) | Lonza Clonetics | CC-4323 | |
| Factor de crecimiento epidérmico (EGF) | Sigma Aldrich | E9644 | |
| Factor de crecimiento básico de fibroblastos (bFGF) | Invitrogen | PHG0261 | |
| Factor inhibidor de leucemia (LIF) | Millipore | LIF1010 | |
| Antibiótico / micótica | Wisent | 450-115-EL | |
| Liberase TM | Roche | 05 401 119 001 | |
| Solución de cloruro de amonio | Stem Cell Technologies | 07850 |
Referencias
- Ohgaki, H., Kleihues, P. Epidemiology and etiology of gliomas. Acta Neuropathol. 109, 93 (2005).
- Huse, J. T., Holland, E. C. Targeting brain cancer: advances in the molecular pathology of malignant glioma. 10, 319 (2010).
- Wechsler-Reya, R., Scott, M. P. The developmental biology of brain tumors. Annu. Rev. Neurosci. 24, 385 (2001).
- Singh, S. K. Identification of a cancer stem cell in human brain tumors. Cancer Res. 63, 5821 (2003).
- Singh, S. K. Identification of human brain tumour initiating cells. Nature. 432, 396 (2004).
- Reynolds, B. A., Weiss, S. Clonal and population analyses demonstrate that an EGF-responsive mammalian embryonic CNS precursor is a stem cell. Dev. Biol. 175, 1 (1996).
- Tropepe, V. Distinct neural stem cells proliferate in response to EGF and FGF in the developing mouse telencephalon. Dev. Biol. 208, 166 (1999).
- Yin, A. H. AC133, a novel marker for human hematopoietic stem and progenitor cells. Blood. 90, 5002 (1997).
- Yu, Y., Flint, A., Dvorin, E. L., Bischoff, J. AC133-2, a novel isoform of human AC133 stem cell antigen. J. Biol. Chem. 277, 20711 (2002).
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414, 105 (2001).
- Al-Hajj, M. Prospective identification of tumorigenic breast cancer cells. Proc. Natl. Acad. Sci. U. S. A. 100, 3983 (2003).
- Bonnet, D., Dick, J. E. Human acute myeloid leukemia is organized as a hierarchy that originates from a primitive hematopoietic cell. Nat. Med. 3, 730 (1997).
- Matsui, W. Characterization of clonogenic multiple myeloma cells. Blood. 103, 2332 (2004).
- Bao, S. Targeting cancer stem cells through L1CAM suppresses glioma growth. Cancer Res. 68, 6043 (2008).
- Bao, S. Glioma stem cells promote radioresistance by preferential activation of the DNA damage response. Nature. 444, 756 (2006).
- Bao, S. Stem cell-like glioma cells promote tumor angiogenesis through vascular endothelial growth factor. Cancer Res. 66, 7843 (2006).
- Beier, D. Temozolomide preferentially depletes cancer stem cells in glioblastoma. Cancer Res. 68, 5706 (2008).
- Piccirillo, S. G. Distinct pools of cancer stem-like cells coexist within human glioblastomas and display different tumorigenicity and independent genomic evolution. Oncogene. 28, 1807 (2009).
- Piccirillo, S. G. morphogenetic proteins inhibit the tumorigenic potential of human brain tumour-initiating cells. Nature. 444, 761 (2006).
- Rich, J. N., Bao, S. Chemotherapy and cancer stem cells. Cell Stem Cell. 1, 353 (2007).
- Lapidot, T. A cell initiating human acute myeloid leukaemia after transplantation into SCID mice. Nature. 367, 645 (1994).
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414, 105 (2001).
- Fuchs, E., Segre, J. A. Stem cells: a new lease on life. Cell. 100, 143 (2000).
- Weissman, I. L. Stem cells: units of development, units of regeneration, and units in evolution. Cell. 100, 157 (2000).
- Reynolds, B. A., Weiss, S. Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system. Science. 255, 1707 (1992).
- Reynolds, B. A., Tetzlaff, W., Weiss, S. A multipotent EGF-responsive striatal embryonic progenitor cell produces neurons and astrocytes. J. Neurosci. 12, 4565 (1992).
- Hulett, H. R., Bonner, W. A., Barrett, J., Herzenberg, L. A. Cell sorting: automated separation of mammalian cells as a function of intracellular fluorescence. Science. 166, 747 (1969).
- Kohler, G., Milstein, C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature. 256, 495 (1975).
- Barrett, L. E. Self-renewal does not predict tumor growth potential in mouse models of high-grade glioma. Cancer Cell. 21, 11 (2012).
- Venugopal, C. Bmi1 marks intermediate precursors during differentiation of human brain tumor initiating cells. Stem Cell Res. 8, 141 (2012).
- Gerlinger, M. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N. Engl. J. Med. 366, 883 (2012).
- Gilbertson, R. J. Medulloblastoma: signalling a change in treatment. Lancet. Oncol. 5, 209 (2004).
- Zhu, Y., Parada, L. F. The molecular and genetic basis of neurological tumours. Nat. Rev. Cancer. 2, 616 (2002).
- Maher, E. A. Malignant glioma: genetics and biology of a grave matter. Genes Dev. 15, 1311 (2001).
- Blake, W. J., KAErn, M., Cantor, C. R., Collins, J. J. Noise in eukaryotic gene expression. Nature. 422, 633 (2003).
- Elowitz, M. B., Levine, A. J., Siggia, E. D., Swain, P. S. Stochastic gene expression in a single cell. Science. 297, 1183 (2002).
- Maheshri, N., O'Shea, E. K. Living with noisy genes: how cells function reliably with inherent variability in gene expression. Annu. Rev. Biophys. Biomol. Struct. 36, 413 (2007).
- Raj, A. Stochastic mRNA synthesis in mammalian cells. PLoS Biol. 4, e309 (2006).
- Ross, I. L., Browne, C. M., Hume, D. A. Transcription of individual genes in eukaryotic cells occurs randomly and infrequently. Immunol. Cell Biol. 72, 177 (1994).
- Kubista, M. The real-time polymerase chain reaction. Mol. Aspects Med. 27, 95 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1, 1559 (2006).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados