Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Traitement des tissus de tumeur cérébrale primaire pour les essais sur les cellules souches et les flux de tri
Dans cet article
Résumé
L'identification des cellules tumorales du cerveau d'amorçage (BTICs), les cellules rares, dans un tumorales hétérogènes possédant des propriétés de cellules souches, offre de nouvelles perspectives sur la pathogénie des tumeurs cérébrales humaines. Nous avons amélioré les conditions de culture spécifiques pour enrichir BTICs, et nous avons l'habitude d'utiliser la cytométrie en flux pour enrichir davantage ces populations. D'auto-renouvellement des tests et de l'analyse par transcription seule cellule RT-PCR peut ensuite être effectuée sur ces cellules isolées.
Résumé
Les tumeurs cérébrales sont typiquement constitués de cellules morphologiquement différents qui expriment une variété de marqueurs de lignage neural. Seule une fraction relativement faible de cellules dans la tumeur avec des propriétés de cellules souches, appelées cellules tumorales du cerveau d'amorçage (BTICs), possèdent une capacité à différencier selon plusieurs lignées, auto-renouvellement, et d'initier des tumeurs in vivo. Nous avons appliqué les conditions de culture utilisés à l'origine pour les normales des cellules souches neurales (NSC) à une variété de tumeurs cérébrales humaines et a constaté que cette méthode de culture sélectionne spécifiquement pour les souches de type populations. Un milieu sans sérum (NSC) permet le maintien d'un état de cellules souches non différenciées, et l'addition de bFGF et EGF permet la prolifération des multi-puissant, auto-renouvellement, et tumorspheres expansibles.
Pour caractériser davantage la population BTIC chaque tumeur, nous évaluons marqueurs de surface cellulaire par cytométrie en flux. Nous pouvons également trier les populations d'intérêt pour characteriz plus spécifiquestion. L'auto-renouvellement des essais sont réalisés sur BTICs simples triés dans des plaques à 96 puits, la formation d'tumorspheres après incubation à 37 ° C indique la présence d'une tige ou d'une cellule progénitrice. Le nombre de cellules multiples d'une population donnée peut également être triées dans différents puits pour limiter l'analyse de dilution, d'analyser la capacité d'auto-renouvellement. Nous pouvons également étudier expression différentielle des gènes dans une population cellulaire particulière en utilisant seule cellule RT-PCR.
Les protocoles suivants décrivent les procédures pour la dissociation et de culture primaire d'échantillons humains pour enrichir des populations BTIC, ainsi que la dissociation de tumorspheres. Sont également inclus les protocoles de coloration pour l'analyse de cytométrie de flux ou tri, l'auto-renouvellement des essais, et seule cellule RT-PCR.
Introduction
Les tumeurs cérébrales sont parmi les cancers les plus agressifs et hétérogènes connus chez l'homme. Bien que leur détection précoce et le diagnostic ont été facilitées par la neuro-imagerie moderne de la technologie, nous manquons encore de traitement curatif pour les tumeurs cérébrales nombreuses, en particulier pour diffuses, celles envahissantes ou celles situées profondément dans le cerveau.
Les tumeurs cérébrales représentent la première cause de mortalité par cancer chez les enfants en raison de leur caractère très agressif et souvent incurable. Le glioblastome (GBM), la tumeur cérébrale primaire la plus fréquente chez l'adulte, est l'un des cancers les plus agressifs de l'homme, craint pour son pronostic toujours fatale 1. Cette tumeur très maligne astrocytaire (OMS grade 4) se produit généralement dans les hémisphères cérébraux des adultes, et peut également se produire chez les jeunes enfants et les nourrissons. Sa croissance est rapide et infiltrantes et de diagnostic des caractéristiques pathologiques comprennent pléomorphisme nucléaire, la prolifération microvasculaire et une nécrose 2,3. Pour adults avec glioblastome nouvellement diagnostiqué, la médiane de survie dépasse rarement 12 mois 1, avec des réponses généralement pauvres à toutes les modalités thérapeutiques. Nous avons remarqué qu'il ya beaucoup de similitudes fonctionnelles et génétiques partagées par les cellules souches somatiques et les cellules cancéreuses, et que les voies moléculaires qui régulent le développement normal du cerveau sont souvent dérégulée dans le cancer. En appliquant les paradigmes en biologie des cellules souches à l'étude des tumeurs du cerveau, nous étions les premiers chercheurs à identifier de manière prospective et purifier une sous-population de cellules de GBM de l'homme qui présentaient les propriétés des cellules souches de la prolifération, l'autorenouvellement et la différenciation in vitro 4 et dans vivo 5. Nous avons appliqué les conditions de culture et les dosages utilisés à l'origine pour caractériser les cellules souches neurales normales (NSC) in vitro à de multiples 6,7 tumeurs cérébrales pédiatriques et adultes, et enrichi par ces cellules souches comme par tri cellulaire pour le marqueur de surface des cellules progénitrices neurales CD133 8 ,9. Le CD133 + fraction tumeur au cerveau contient des cellules qui ont une fréquence beaucoup plus élevée de l'initiation tumorale que le CD133-fraction dans les cerveaux de souris NOD-SCID 5,10. Cette formellement établi que seul un sous-ensemble rare de cellules tumorales du cerveau avec des propriétés de cellules souches sont des tumeurs initier, de les gagner le nom de «tumeur au cerveau cellules initiatrices» ou «BTICs". L'identification roman de BTICs offre de nouvelles perspectives sur la tumorigenèse cerveau humain, accordant son soutien résolu à l'hypothèse des cellules souches cancéreuses 10-13 comme base pour de nombreuses tumeurs solides, et établit une nouvelle cible cellulaire pour des thérapies plus efficaces contre le cancer 14-20. Thérapies qui mettent l'accent sur les tuant la majeure partie de la tumeur risquent de rater le rare tige-comme fraction, permettant à la tumeur de continuer à croître. Thérapies qui mettent l'accent sur la destruction des cellules souches du cancer peut offrir un meilleur traitement et le pronostic des patients atteints de tumeurs cérébrales.
Afin d'étudier les populations BTIC, nous avons affiné notre culture protocoles spécifiquement sélectionnées pour les populations de cellules au sein des tumeurs cérébrales humaines qui possèdent des propriétés de cellules souches. Sans sérum, les cellules souches neurales (NSC) permet moyen pour le maintien d'un état de cellules souches non différenciées, et l'addition de facteur de croissance des fibroblastes basique (bFGF), le facteur de croissance épidermique (EGF), le facteur inhibiteur de leucémie et (LIF) permet de la prolifération des multi-puissant, d'auto-renouvellement et extensibles tumorspheres de l'homme. Ici, nous décrivons les méthodes impliquées dans le traitement des tumeurs cérébrales primaires et leur culture dans un milieu de NSC pour enrichir les populations BTIC. Nous avons appelé notre modèle expérimental "isolats de patients BTIC» pour souligner le fait que ces cellules sont très peu cultivées dans des souches conditions de la cellule pour sélectionner des populations de cellules souches. immunomarquage ultérieur des populations BTIC pour les principaux marqueurs de cellules souches telles que CD133 et CD15 et l'analyse par cytométrie en flux est également décrit. Nous discutons ensuite l'analyse de dilution limitante,qui aide à étudier le potentiel d'auto-renouvellement de BTICs. Enfin, nous explorons l'analyse de l'expression des gènes de ces cellules rares en triant les cellules simples sur des lames AmpliGrid et l'exécution seule cellule RT-PCR. Ces techniques sont également applicables aux autres tumeurs cérébrales telles que le médulloblastome, épendymome et les gliomes pédiatriques.
Protocole
1. Culture de tissus tumeur au cerveau
- Ajouter 200 ul Libérase décongelés (Roche Applied Science) à 15 ml de LCR artificiel (ACSF-voir le tableau 1), puis placer en bain à 37 ° C de l'eau. Liberase TM est un mélange d'enzymes protéolytiques utilisés pour dissocier les échantillons de tissus primaires, ainsi que tumorspheres cultivées. Contrairement à la trypsine-EDTA, la méthode Libérase préserve l'antigène de surface CD133. Pour un échantillon de tissu d'environ 0,5 cm 3, nous utilisons 200 pi de Libérase. Si le tissu est plus petit, nous utilisons 100 ul.
- Apportez une solution de chlorure d'ammonium (technologies de cellules souches) à température ambiante. Solution de chlorure d'ammonium lyse des globules rouges délicatement avec un effet minimal sur les autres cellules. Il ne contient pas de fixateur.
- Dans le cabinet de sécurité biologique stérile, ajouter 5 ml de aCSF de récipient d'échantillon, agiter pour rincer le tissu, puis pipeter hors tension. Cette étape permet d'éliminer les globules rouges (RBC).
- Transfert des tissus tumeur au cerveau à un stérile 100 mm Petri dish.
- Avec des ciseaux ou des scalpels et des fines pinces, désagréger le tissu de consistance d'un coulis.
- Prélever un échantillon à l'aide d'une pipette 10 ml régulière ou une pince et des fragments de transfert dans le tube contenant préchauffé aCSF avec Libérase.
- Lieu de l'incubateur secoueur (30 min) et réglé à 37 ° C, pendant 15 min.
- Filtrer le lysat tissulaire à 70 um tamis cellulaire dans un tube Falcon de 50 ml.
- Faites tourner le filtrat vers le bas à 280 xg pendant 5 min.
- Retirer le surnageant attentivement et évaluer la taille et la couleur du culot cellulaire résultant: granulés qui sont roses ou rouges indiquent un nombre croissant de globules rouges.
- Remettre en suspension le culot dans 1 ml de PBS.
- Ajouter une quantité appropriée de la solution de chlorure d'ammonium (4-12 ml) en fonction de la taille et de la contamination culot de globules rouges (solution de chlorure d'ammonium est très doux et des quantités accrues sont pas dangereux pour les cellules autres que les cellules rouges).
- Incuber à température ambiante pendant 5 min.
- Cellules dérivéesvers le bas à 280 xg pendant 5 min.
- Laver une fois avec 10 ml de PBS stérile.
- Remettre en suspension dans 5 ml de milieu complet NSC (tableau 2) et le transfert d'un ultra-faible pouvoir de fixation plaque 60 mm culture tissulaire (Corning). Nous utilisons ultra-basse des plaques de culture de liaison avec des surfaces d'hydrogel liés de façon covalente qui sont hydrophiles et de charge neutre, afin de minimiser l'attachement cellulaire médiée par la différenciation.
Durant les premiers jours de culture, ne modifiez pas les médias: top-up uniquement avec 1-2 ml de milieu au besoin, puis continuer à observer les milieux de culture et le changement lorsque la couleur des médias devient légèrement jaune.
2. Tumorsphere dissociation de cytométrie en flux
- Évaluer tumorspheres sous le microscope: si la taille de la sphère est> 100 um, la dissociation est recommandé que les grandes sphères peuvent se nécroser dans le centre.
- Transfert culture à 15 ml tube conique.
- Ajouter 2-3 ml stérile PBS de rincer soigneusement la plaque et ajouter à tube conique.
- Centrifuger à 280 xg pendant 5 min.
- Retirer le surnageant et remettre en suspension dans 1 à 2 ml de PBS stérile.
- Ajouter 10 ul Libérase.
- Incuber à 37 ° C bain d'eau pendant 3 min. Retirez et évaluer visuellement la suspension, si des amas de multiples vu, triturer doucement à l'aide d'une pointe de pipette 1000 pi.
- Si l'agglutination persiste, passez d'incubation pour une période supplémentaire 1-2 min.
- Laver les cellules en ajoutant 5 ml de PBS stérile et centrifugation à 280 xg pendant 5 min.
- Retirer le surnageant et remettre en suspension dans du PBS stérile 500-1000 ul d'EDTA 2 mM.
- Évaluer le nombre et la viabilité cellulaire en utilisant du bleu trypan.
- Réglez le nombre de cellules à 1 million / ml dans du PBS EDTA 2 mM.
3. La coloration de surface
- Transférer 100 ul de suspension cellulaire 1x10 6 / ml par test 12x75 mm tubes d'écoulement.
- Ajouter la quantité appropriée d'anticorps. Contrôles isotypiques appariés doivent être utilisés pour chaqueanticorps (voir tableau 3).
- Incuber pendant 30 min sur la glace.
- Ajouter 2 ml de PBS + 2 mM EDTA à chaque tube d'écoulement.
- Centrifuger à 200 xg pendant 4 minutes, décanter et éponger.
- Resuspendre le culot dans 300 ul de PBS EDTA 2 mM.
- Ajouter 10 ul colorant viabilité 7AAD dans chaque tube et incuber pendant au moins 15 min sur la glace.
- Analyse par cytométrie en flux.
4. Acquisition et analyse de cytométrie en flux
Les détails de l'acquisition et de l'analyse des données de flux sont des instruments-dépendante. Représentatifs des échantillons négatifs sont gérés et réglages de l'instrument mis en place pour l'avenir (taille) et latérale (granularité) de dispersion. Ce modèle de diffusion permet à l'utilisateur final de visualiser toutes les cellules dans l'échantillon, y compris les débris. Une région est habituellement établi autour des cellules d'intérêt. (Figure 4a) Les réglages sont alors mis en place pour tous les détecteurs de fluorescence nécessaires pour positionner les cellules négatives au sein de la première décennie du afparcelle intensité luorescence. Lorsque plus d'un colorant ou fluorochrome est utilisé, simples contrôles colorés doit être utilisé pour établir des valeurs de compensation de couleur (soustraction d'interférer émissions de fluorescence). Lors de l'exécution cellules vivantes, un colorant tel que la viabilité 7-AAD (7-amino-actinomycine D - 655 488/emission excitation) est utilisé et une seconde zone établie afin d'exclure les cellules mortes (figure 4b). Ces deux régions sont appliqués à une analyse plus poussée des échantillons.
5. Test d'auto-renouvellement
Le test clé qui est grandement facilité par la croissance clonale sphère est le test in vitro de la capacité d'auto-renouvellement, le test de dilution limite. Tumorspheres sont dissociés et distribués à des dilutions vers le bas à une seule cellule par puits, et le taux de formation sphère subséquente de plus de 7 jours de culture est calculée. Au jour 7, le pourcentage de puits ne contenant pas de sphères de chaque cellule densité de placage (F 0) est calculée et tracéeen fonction du nombre de cellules par puits (x). Le nombre de cellules nécessaires pour former au moins une tumorsphere dans chaque puits est déterminée à partir du point où la ligne croise le niveau 0,37 (F 0 = e-x). Dans une distribution de Poisson de cellules, F 0 = 0,37 correspond à la dilution d'une cellule par puits, de sorte que ce calcul (les 37% se croisent) reflète la fréquence des cellules souches clonogéniques dans la population de cellules entières (Figure 5).
6. Single Cell RT-PCR
Les cellules individuelles de différentes populations sont triés par Beckman Coulter XDP MoFlo sur les sites de réaction sur une diapositive AmpliGrid (Beckman Coulter cat # AG480F). Seule cellule RT-PCR est réalisée en utilisant AmpliGrid Simple One Step RT-PCR System (Beckman Coulter cat # OAX04515) selon les spécifications du fabricant. En bref, une seule cellule est déposée dans chaque site de réaction d'un coulisseau AmpliGrid; la présence d'une cellule unique dans chaquesite de réaction confirmée par microscopie. La transcription inverse (RT) est réalisée immédiatement après le tri pour empêcher l'échantillon ne soit compromise. Le mélange master RT est préparé frais, selon les instructions du kit (tableau 4). Nous avons utilisé 0,03 ul de 25 uM des amorces aléatoires (Promega) par réaction; 1 pi de mélange maître est ajouté au centre de chaque site de réaction. Ceci est immédiatement recouvert avec 5 pi de solution d'étanchéité (Beckman Coulter cat # OAX04503). En utilisant une lame AmpliSpeed cycleur (Beckman Coulter; cat # OAX04101), les lames sont incubées à 25 ° C pendant 10 min, 42 ° C pendant 10 min et 55 ° C pendant 45 min. Après une transcription inverse, 3 pl de DNase / RNase eau est ajouté à chaque site de réaction, et la totalité du mélange transféré dans un tube PCR (1 tube / le site). Dans une plaque de PCR, un volume de réaction de 10 ul est utilisé pour la PCR quantitative: 8 pl qPCR master mix (5 pi SYBR Green (Quanta), 1 pl d'eau, 1 pl de 10 uM avant et mélanger amorce inverse) plus 2 μ l du mélange RT. Pour quantification en temps réel, les échantillons sont analysés sur un cycleur BioRad utilisant le programme suivant: 95 ° C pendant 10 min, 45 cycles de 95 ° C pendant 30 secondes, 60 ° C pendant 60 secondes, 72 ° C pendant 60 secondes, puis par 10 min à 72 ° C et l'analyse des courbes de fusion. Les valeurs de Ct ont été déterminées à l'aide Opticon Monitor 3 (BioRad). Trois gènes peut être évaluée par cellule, y compris GAPDH comme gène de ménage. L'expression des gènes est normalisée à l'expression de GAPDH, selon 2-ΔCt. Au moins une douzaine de cellules individuelles de la même population de la même espèce peuvent être utilisés comme biologique reproduit pour compenser le manque de technique répétitions.
7. Les résultats représentatifs
La figure 1 montre une imagerie par résonance magnétique (IRM) d'un patient avec un représentant de GBM. Échantillons de tumeurs du cerveau sont obtenues à partir de patients consentants immédiatement après la chirurgie, tel qu'approuvé par le Hamilton Health Sciences / S McMaster Healthciences éthique de la recherche. Une partie de chaque échantillon est donnée à la neuropathologiste pour le diagnostic clinique de routine. L'échantillon restant est traité comme le montre la figure 2. Les cellules tumorales, une fois immatriculés dans les médias complets de cellules souches neurales de facteurs de croissance, tumorspheres comme montré dans la figure 3.
Les cellules tumorales sont marquées avec des marqueurs de surface de cellules souches neurales CD133 et CD15 et analysées par cytométrie en flux comme le montre la figure 4.
Les cellules individuelles provenant de populations différentes, par exemple, CD15 + / CD133 +, CD15 + / CD133-, CD15-/CD133 +, CD15-/CD133- sont ensuite triées dans des plaques de 96 puits (figure 5a) ou dans des diapositives AmpliGrid (Figure 6a).
Dans la plaque de 96 puits, des cellules individuelles qui possèdent la capacité d'auto-renouvellement (par exemple, les cellules CD133 +), (figure 5b), peuvent former tumorspheres (figure 5c) après incubation. Un graphe d'auto-renouvellement peuvent être tracées (Figure 5d) en comptant le nombre de sphères formées par puits. Cellules individuelles triées dans le site de réaction de la lame AmpliGrid (Figure 6a). Les ARN extrait de cellules simples peuvent être une transcription inverse en utilisant la AmpliSpeed Advalytix (Figure 6b). L'ADNc obtenu est utilisé pour effectuer en temps réel RT-PCR (figure 6c).
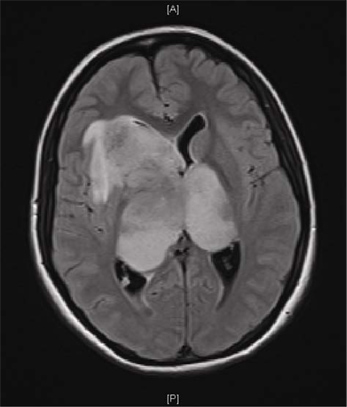
Figure 1. Imagerie par résonance magnétique (IRM) d'un GBM patient. IRM du cerveau d'une jeune fille de 16 ans avec une histoire d'un mois de maux de tête et une histoire d'une semaine de vomissements, léthargie et confusion visuelle. Séquence FLAIR axial montre une grande bi-hémisphérique ("papillon") GBM.
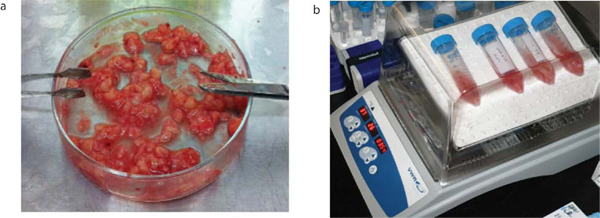
Figure 2. Traitement des tumeurs cérébrales. Échantillons de tumeurs cérébrales sont bénéficier du droit àminé des patients consentants immédiatement après la chirurgie. Ils sont dissociés mécaniquement comme indiqué (a) et sont ensuite dissociées enzymatiquement en aCSF avec Liberase par incubation dans un incubateur 30 min à bascule à 37 ° C pendant 15 min (b).

Figure 3. Populations BTIC former tumorspheres en culture. Dissociées cellules tumorales du cerveau immatriculés dans les médias complets de cellules souches neurales de facteurs de croissance former tumorspheres.
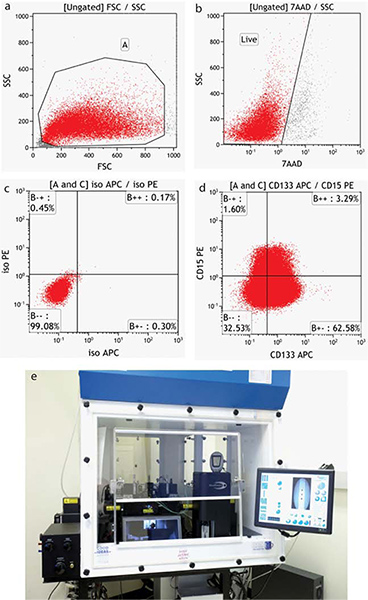
Figure 4. L'analyse par cytométrie en flux des populations BTIC. (A) par rapport à l'avenir propriétés de diffusion latérales offrent une image de toutes les cellules, y compris les débris (b) Les cellules qui se colorent avec le colorant viabilité 7-AAD sont exclus de l'analyse. (C) La position des quadrants statistiques est déterminée à l'aide des contrôles isotypiques appropriés (d) Les cellules tumorales colorées avec ma surfacerkers CD15 et CD133-PE-APC. (D) trieur de cellules MoFlo XDP. Cliquez ici pour agrandir la figure .
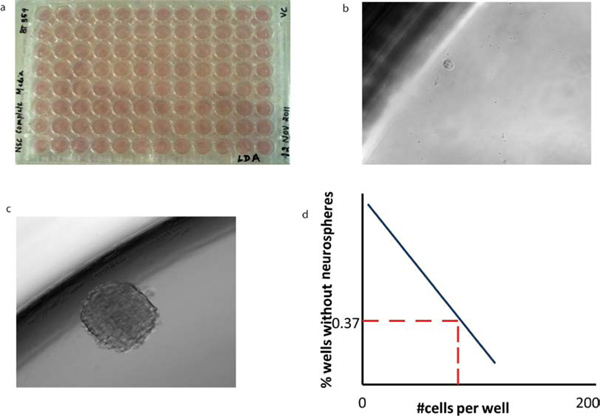
Figure 5. Limiter l'analyse dilution de BTICs révèle leur capacité d'auto-renouvellement. (A) Cellules de dilutions variables sont classées dans 96 puits contenant 200 pl de NSC (b) une seule cellule par puits. (C) Après une période d'incubation de 7 jours, un tumorsphere est formé. La morphologie de tumorspheres secondaire est identique à celle de tumorspheres primaires. (D) Les valeurs moyennes de x-interception valeurs peuvent être calculées à partir de l'analyse de limiter la dilution pour chaque sous-type de tumeur à révéler le nombre de cellules nécessaires pour former au moins une tumorsphere par puits.

Figure 6. Analyse de l'expression des gènes d'une cellule unique BTICest possible en utilisant une seule cellule technologie RT-PCR. (a) Les cellules individuelles d'une population BTIC peuvent être triées sur des lames AmpliGrid. (B) la synthèse d'ADNc est effectuée sur des cellules individuelles sur des lames AmpliGrid l'aide de l'AmpliSpeed Advalytix. (C) La PCR quantitative en temps réel est effectuée sur le ADNc synthétisé à partir des cellules individuelles BTIC.
| 2M NaCl | 62 ml |
| 1M KCl | 5 ml |
| 1M MgCl 2 | 3,2 ml |
| 155 mM NaHCO 3 | 169 ml |
| Le glucose 1M | 10 ml |
| 108 mM de CaCl 2 | 0,9256 ml |
| Purelab Ultra H 2 0 | 749,84 ml |
Tableau 1 liquide céphalo-rachidien artificiel (ACSF) -. 1.000 ml.
| Média basales | 500 ml |
| 01:01 DMEM: F12 | 480 ml |
| Supplément N2 | 5 ml |
| HEPES 1M | 5 ml |
| Glucose | 3,0 g |
| La N-acétylcystéine (60 pg / ml) | 1 ml |
| Factor-1 la survie neuronale (NSF-1) | 10 ml |
Tableau 2. Neuronaux médias de cellules souches.
Médias NSC complets (fait frais avant utilisation):
Médias NSC basales + 20 ng / ml d'EGF (epidermal growth factor) + 20 ng / ml de bFGF (basic fibroblast growth factor) + 10 ng / ml LIF (facteur inhibiteur de leucémie) * + 10 ul / ml antibiotique-antimycotique
* Facteur inhibiteur de leucémie (LIF): Ce réactif contient de Millipore une cytokine interleukine 6 classe, un protein qui supprime la différenciation spontanée des cellules souches promotion d'entretien à long terme des cultures de cellules souches.
| Anticorps | Fournisseur / CAT # | ul / test (dans 100 pl) |
| CD133 / 2 APC (293C3) | Miltenyi Biotec/130-090-854 | 5 |
| IgG2b APC (contrôle isotypique) | Miltenyi Biotec/130-092-217 | 5 |
| CD15PE | Beckman Coulter/IM1954U | 5 |
| IgG2a PE (isotype contrôle) | Beckman Coulter/A09141 | 5 |
| 7AAD | Beckman Coulter/A07704 | 10 |
Tableau 3. Anticorps débit.
| Composant | Volume (1 réaction) | Volume (48 réactions) |
| 2x tampon de réaction cellulaire RT | 0,50 ul | 30,0 ul |
| RNase Inhibitor (10 U / pl) | 0,02 ul | 1,2 pl |
| 5X Single Cell RT enhancer | 0,15 ul | 9,00 ul |
| Single Cell RT Enzyme Mix | 0,04 ul | 2,4 pl |
| Advablue (OAX04227) | 0,1 pl | 6,00 ul |
| Nucléase sans eau | compléter à 1 ul | faire jusqu'à 60 pi |
Tableau 4. Composition du master mix RT RT-PCR pour cellule unique (cat # OAX04515).
Discussion
L'hypothèse de cellules souches du cancer 10, basé sur le travail dans la leucémie 21, le cancer du sein 11 et 4,5 le cancer du cerveau, suggère que seule une fraction relativement faible de cellules dans la tumeur, appelées cellules souches cancéreuses, possèdent une capacité à proliférer et largement auto renouveler. La plupart des cellules tumorales perdent leur capacité à proliférer et à se renouveler car ils se différencient en cellules qui deviennent l...
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été financé par l'Institut ontarien de recherche sur le cancer (IORC), la Fondation Terry Fox et l'American Association of Neurological Surgeons.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| 01:01 DMEM: F12 | Invitrogen | 11320-082 | |
| Supplément N2 | Invitrogen | 17502-048 | |
| HEPES 1M | Wisent | 330-050 EL- | |
| Glucose | Invitrogen | 15023-021 | |
| N-acétylcystéine | Sigma Aldrich | A9165-25g | |
| Facteur de survie des neurones -1 (NSF-1) | Lonza Clonetics | CC-4323 | |
| Facteur de croissance épidermique (EGF) | Sigma Aldrich | E9644 | |
| Facteur de croissance basique des fibroblastes (bFGF) | Invitrogen | PHG0261 | |
| Facteur inhibiteur de leucémie (LIF) | Millipore | LIF1010 | |
| Antibiotique / antimycotique | Wisent | 450-115 EL- | |
| Libérase TM | Roche | 05 401 119 001 | |
| Une solution de chlorure d'ammonium | Technologies des cellules souches | 07850 |
Références
- Ohgaki, H., Kleihues, P. Epidemiology and etiology of gliomas. Acta Neuropathol. 109, 93 (2005).
- Huse, J. T., Holland, E. C. Targeting brain cancer: advances in the molecular pathology of malignant glioma. 10, 319 (2010).
- Wechsler-Reya, R., Scott, M. P. The developmental biology of brain tumors. Annu. Rev. Neurosci. 24, 385 (2001).
- Singh, S. K. Identification of a cancer stem cell in human brain tumors. Cancer Res. 63, 5821 (2003).
- Singh, S. K. Identification of human brain tumour initiating cells. Nature. 432, 396 (2004).
- Reynolds, B. A., Weiss, S. Clonal and population analyses demonstrate that an EGF-responsive mammalian embryonic CNS precursor is a stem cell. Dev. Biol. 175, 1 (1996).
- Tropepe, V. Distinct neural stem cells proliferate in response to EGF and FGF in the developing mouse telencephalon. Dev. Biol. 208, 166 (1999).
- Yin, A. H. AC133, a novel marker for human hematopoietic stem and progenitor cells. Blood. 90, 5002 (1997).
- Yu, Y., Flint, A., Dvorin, E. L., Bischoff, J. AC133-2, a novel isoform of human AC133 stem cell antigen. J. Biol. Chem. 277, 20711 (2002).
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414, 105 (2001).
- Al-Hajj, M. Prospective identification of tumorigenic breast cancer cells. Proc. Natl. Acad. Sci. U. S. A. 100, 3983 (2003).
- Bonnet, D., Dick, J. E. Human acute myeloid leukemia is organized as a hierarchy that originates from a primitive hematopoietic cell. Nat. Med. 3, 730 (1997).
- Matsui, W. Characterization of clonogenic multiple myeloma cells. Blood. 103, 2332 (2004).
- Bao, S. Targeting cancer stem cells through L1CAM suppresses glioma growth. Cancer Res. 68, 6043 (2008).
- Bao, S. Glioma stem cells promote radioresistance by preferential activation of the DNA damage response. Nature. 444, 756 (2006).
- Bao, S. Stem cell-like glioma cells promote tumor angiogenesis through vascular endothelial growth factor. Cancer Res. 66, 7843 (2006).
- Beier, D. Temozolomide preferentially depletes cancer stem cells in glioblastoma. Cancer Res. 68, 5706 (2008).
- Piccirillo, S. G. Distinct pools of cancer stem-like cells coexist within human glioblastomas and display different tumorigenicity and independent genomic evolution. Oncogene. 28, 1807 (2009).
- Piccirillo, S. G. morphogenetic proteins inhibit the tumorigenic potential of human brain tumour-initiating cells. Nature. 444, 761 (2006).
- Rich, J. N., Bao, S. Chemotherapy and cancer stem cells. Cell Stem Cell. 1, 353 (2007).
- Lapidot, T. A cell initiating human acute myeloid leukaemia after transplantation into SCID mice. Nature. 367, 645 (1994).
- Reya, T., Morrison, S. J., Clarke, M. F., Weissman, I. L. Stem cells, cancer, and cancer stem cells. Nature. 414, 105 (2001).
- Fuchs, E., Segre, J. A. Stem cells: a new lease on life. Cell. 100, 143 (2000).
- Weissman, I. L. Stem cells: units of development, units of regeneration, and units in evolution. Cell. 100, 157 (2000).
- Reynolds, B. A., Weiss, S. Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system. Science. 255, 1707 (1992).
- Reynolds, B. A., Tetzlaff, W., Weiss, S. A multipotent EGF-responsive striatal embryonic progenitor cell produces neurons and astrocytes. J. Neurosci. 12, 4565 (1992).
- Hulett, H. R., Bonner, W. A., Barrett, J., Herzenberg, L. A. Cell sorting: automated separation of mammalian cells as a function of intracellular fluorescence. Science. 166, 747 (1969).
- Kohler, G., Milstein, C. Continuous cultures of fused cells secreting antibody of predefined specificity. Nature. 256, 495 (1975).
- Barrett, L. E. Self-renewal does not predict tumor growth potential in mouse models of high-grade glioma. Cancer Cell. 21, 11 (2012).
- Venugopal, C. Bmi1 marks intermediate precursors during differentiation of human brain tumor initiating cells. Stem Cell Res. 8, 141 (2012).
- Gerlinger, M. Intratumor heterogeneity and branched evolution revealed by multiregion sequencing. N. Engl. J. Med. 366, 883 (2012).
- Gilbertson, R. J. Medulloblastoma: signalling a change in treatment. Lancet. Oncol. 5, 209 (2004).
- Zhu, Y., Parada, L. F. The molecular and genetic basis of neurological tumours. Nat. Rev. Cancer. 2, 616 (2002).
- Maher, E. A. Malignant glioma: genetics and biology of a grave matter. Genes Dev. 15, 1311 (2001).
- Blake, W. J., KAErn, M., Cantor, C. R., Collins, J. J. Noise in eukaryotic gene expression. Nature. 422, 633 (2003).
- Elowitz, M. B., Levine, A. J., Siggia, E. D., Swain, P. S. Stochastic gene expression in a single cell. Science. 297, 1183 (2002).
- Maheshri, N., O'Shea, E. K. Living with noisy genes: how cells function reliably with inherent variability in gene expression. Annu. Rev. Biophys. Biomol. Struct. 36, 413 (2007).
- Raj, A. Stochastic mRNA synthesis in mammalian cells. PLoS Biol. 4, e309 (2006).
- Ross, I. L., Browne, C. M., Hume, D. A. Transcription of individual genes in eukaryotic cells occurs randomly and infrequently. Immunol. Cell Biol. 72, 177 (1994).
- Kubista, M. The real-time polymerase chain reaction. Mol. Aspects Med. 27, 95 (2006).
- Nolan, T., Hands, R. E., Bustin, S. A. Quantification of mRNA using real-time RT-PCR. Nat. Protoc. 1, 1559 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon