Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Iterativo de optimización de dúplex de ADN para la cristalización de SEQA-ADN complejos
En este artículo
Resumen
Estructura cristalina de los complejos de proteínas de ADN puede proporcionar información sobre la función de proteínas, el mecanismo, así como, la naturaleza de la interacción específica. Aquí reportamos cómo optimizar la longitud, la secuencia y los extremos del ADN dúplex en co-cristalización con Escherichia coli SEQA, un regulador negativo de la iniciación de la replicación.
Resumen
Escherichia coli SEQA es un regulador negativo de la replicación del ADN que impide prematuros eventos reiniciación por secuestrantes GATC grupos hemimethylated dentro del origen de replicación 1. Más allá del origen, SEQA se encuentra en las horquillas de replicación, donde se organiza ADN recién replicado en mayores estructuras ordenadas 2. Asociados SEQA sólo débilmente con las secuencias GATC individuales, sino que forma complejos de alta afinidad con dúplex de ADN que contienen múltiples sitios GATC. La unidad mínima funcional y estructural de SEQA es un dímero, lo que explica el requisito de al menos dos secuencias GATC para formar un complejo de alta afinidad con el ADN hemimethylated 3. Además, la arquitectura SEQA, con la oligomerización y dominios de unión al ADN separados por un enlazador flexible, permite la unión a repeticiones GATC separados por hasta tres vueltas helicoidales. Por lo tanto, la comprensión de la función de SEQA a nivel molecular estructural requiere la analisis de SEQA unido a múltiples secuencias GATC. En la cristalización de proteínas-ADN, el ADN puede tener ninguno a un efecto excepcional en las interacciones de embalaje en función de los tamaños relativos y la arquitectura de la proteína y el ADN. Si la proteína es más grande que el ADN o huellas de la mayoría del ADN, el cristal de embalaje es principalmente mediada por interacciones proteína-proteína. A la inversa, cuando la proteína es del mismo tamaño o más pequeño que el ADN o sólo cubre una fracción de las interacciones ADN, ADN-ADN y ADN-proteína dominar cristal de embalaje. Por lo tanto, la cristalización de proteínas de ADN complejos requiere el cribado sistemático de ADN de longitud y 4 extremos de ADN (romo o saliente) 5-7. En este reporte se describe cómo diseñar, optimizar, purificar y cristalizar hemimethylated dúplex de ADN que contienen repeticiones en tándem GATC en el complejo con una variante dimérica de SEQA (SeqAΔ (41-59)-A25R) para obtener cristales adecuados para la determinación de la estructura.
Protocolo
1. Purificación de proteínas
El enlazador flexible que une el. N-(oligomerización) y C-terminal (de unión a ADN) dominios de SEQA SIDA el reconocimiento de hemimethylated repite GATC separados por uno a tres vueltas en el ADN Para este estudio, se utilizó una variante dimérica de SEQA (SeqAΔ (41-59)-A25R) con una mutación puntual en el dominio N-terminal que impide aún más la oligomerización y un enlazador acortado que restringe la unión al ADN en tándem para GATC repite separados exclusivamente por una vuelta en el ADN (Figura 1) 2,8.
- Transformar BL21 (DE3) células con el plásmido que codifica SEQA bajo el control del promotor T7,
- Placa de la reacción de transformación en placas de agar LB-incluyendo 100 mg / ml de ampicilina,
- Recoger colonias mixtas para inocular un cultivo durante la noche a pequeña escala (medio LB con 100 mg / ml de ampicilina),
- A la mañana siguiente inocular un 1 L de medios de comunicación usando una dilución 1:100 de la overnight cultura,
- Cultivar las células hasta una DO 600 de 0,7 ~ e inducir la producción de proteínas por adición de isopropil β-D-1-tiogalactopiranósido (IPTG) a una concentración final de 1 mM,
- Continuar la incubación durante 3 horas a 37 ° C con agitación orbital y entonces recoger las células por centrifugación (10 min a 3.300 g),
- Resuspender el sedimento celular en tampón de purificación A y lisar por sonicación,
- Borrar el lisado por centrifugación (40 min a 39.000 g) y cargar el sobrenadante en una columna de heparina equilibrada con tampón de purificación,
- SEQA Elute utilizando un gradiente lineal de NaCl 1 M (SEQA eluye a ~ NaCl 0,7 M),
- Piscina las fracciones que contienen SEQA juntos, se diluye para disminuir la fuerza iónica de la muestra y de la carga en una columna de cromatografía de intercambio catiónico equilibrada con tampón de purificación,
- Utilizando un gradiente lineal de sal, puro SEQA eluye a ~ NaCl 0,4 M,
- Piscina las fracciones que contienen SEQA juntos, concentrane (3 mg / ml) y almacenar en tampón de almacenamiento.
2. Purificación de ADN
- Solicitar oligonucleótidos complementarios unmethylated metilado y de su compañía favorita,
- Se disuelve 1 mol de cada cadena de ADN único liofilizado en 800 l de autoclave ddH 2 O, vórtice y dejar que repose durante 10-20 min,
- Añadir 800 l de tampón de carga 2X precalentado a cada oligonucleótido,
- Para oligonucleótidos 20-30 nucleótidos de largo, preparar una gran gel desnaturalizante 10% (160 x 250 x 1 mm):
* Mezclar bien 80 ml de la mezcla PÁGINA 10%, 80 l TEMED y 800 l por persulfato de amonio gel y verter,
* Una vez polimerizado, retire el peine y lave los pocillos con fondo ddH 2 O,
* Reúne los geles de reparto gel incluyendo placa de enfriamiento y luego llenar los depósitos superior e inferior con tampón de corrida (1X TBE),
* Pre-correr el gel a 700-750 V para calentar el gel hasta 55 ° C,
* Detener la carrera y enjuague el thoro pozosughly con tampón de corrida. - Calentar los oligonucleótidos a 90 º C durante 2 min,
- Vortex y centrifugar las muestras e inmediatamente antes de cargar el gel,
- Correr el gel a 700 V ~ y detenerlo una vez que su oligonucleótido se ha desplazado hasta la mitad. (Tenga en cuenta que en un gel de poliacrilamida al 10% de azul de bromofenol co-migra con oligonucleótidos ~ 20 bases de largo y xileno cianol FF con ~ 60 bases de largo),
- Detener el gel, se desmonte de la caja de gel y retirar los separadores,
- En una superficie plana, retire una placa de vidrio y cubrir el gel con una envoltura de plástico,
- Gire el gel alrededor, retire la placa de vidrio otra y se cubre con una envoltura de plástico,
- Marque las bandas con luz UV y una placa fluorescente detrás del gel para ver la sombra de ADN,
- Cortar la banda con una cuchilla de afeitar en trozos pequeños y los transfieren a un tubo estéril de 15 ml,
- Añadir 9 ml de tampón de elución y eluir durante la noche a 37 ° C con agitación,
- Transferir cuidadosamente la solucióna un tubo de centrífuga esterilizado en autoclave usando una pipeta de punta de carga de gel para evitar la transferencia de piezas de acrilamida y añadir 1 ml de acetato de sodio 3 M a pH 7 (dilución 1:10) y 25 ml de etanol enfriado 100% (2,5 volúmenes),
- Incubar a -20 º C durante al menos 3 h, se
- Decantar el sobrenadante y transferir a un tubo separado,
- Secar el pellet de la velocidad-vac a fuego medio,
- Resuspender el pellet en 400 l de autoclave ddH 2 O y transferir a un tubo nuevo,
- Añadir 40 l de acetato de sodio 3 M a pH 7 y 1 ml de etanol al 100%, mezclar bien (vórtice) e incubar 30 min a temperatura ambiente, seguido por 30 min a -20 ° C,
- Girar durante 15 minutos a 18.000 g y descartar el sobrenadante,
- Enjuague el precipitado con 100 l de etanol al 70% frío para eliminar la sal residual del sedimento y centrifugado durante 6 min a 18.000 g. Deseche el etanol y secar el pellet de la velocidad-vac,
- Resuspender el precipitado en un total de 100 l de autoclave ddH 2 O. Medir la concentración del oligonucleótido,
- Para reasociar los dúplex de ADN hemimethylated, mezclar concentraciones equimolares de las cadenas simples complementarias y calentar las mezclas a 95 ° C en un baño de agua durante 5 minutos y después se dejó enfriar lentamente a temperatura ambiente en el interior del baño de agua.
3. Proteína-ADN formación de complejos y Análisis
- Mezclar volúmenes iguales de SeqAΔ purificado (41-59)-A25 (81 mM) y ADN hemimetilado (81 mM),
- Incubar a temperatura ambiente durante 15 min y se almacena a 4 º C hasta que esté listo para usarlo,
- Pantalla de condiciones de cristalización utilizando escaso comerciales de matriz pantallas,
- Una vez pistas iniciales de cristalización se han identificado, optimizar las condiciones para hacer crecer cristales de difracción de calidad,
- Cryoprotect los resultantes de ADN SEQA-cristales, ya sea aumentando la cantidad de PEG 400 presente en la solución de cristalización a una concentración final de 25% (v / v) o la adición de glicerol al 20% (v / v) a la solución de cristalización,
- Recoger los cristales individuales con un lazo de nylon, y los flash-congelar en nitrógeno líquido,
- Pruebe el límite de difracción de cada cristal a 100 K.
4. Los resultados representativos
Para obtener la estructura cristalina de SeqAΔ (41-59)-A25R unido al ADN hemimetilado, que consecutivamente tres parámetros optimizados en el ADN: (i) la separación entre hemimethylated secuencias GATC, (ii) la longitud del dúplex, y la (iii) ausencia / presencia de salientes 5 '.
Electro-ensayos de cambio de movilidad indican que SeqAΔ (41-59)-A25R se une preferentemente a repeticiones GATC separadas por 9-10 pares de bases (Figura 1). Por lo tanto, inicialmente proyectado dúplex 23-24 pares de bases (pb) de largo que contiene dos secuencias GATC hemimethylated separados por 9 o 10 puntos básicos. Tres dúplex produjo cristales bien formados (Figura 2). Alaunque ninguno de los cristales de difracción de alta resolución, el 23 bps largo dúplex con los dos sitios GATC separadas por 9 bps rayos X difractados mejor que el resto, lo que indica que una separación GATC de 9 bps se prefirió para la cristalización. Por lo tanto, se fijó la distancia entre GATC a 9 puntos básicos para todas las pantallas posteriores.
En general, la cristalización se ve favorecida por ADN dúplex correspondientes a longitudes exactas vueltas helicoidales porque múltiples moléculas de ADN pueden apilar de cabeza a cola para formar un continuo B-DNA dentro del cristal 9. Por lo tanto, se acorta la longitud total de los dúplex a 21 bps (es decir, dos vueltas helicoidales). Mientras que un dúplex 21 bps con extremos romos no dió cristales de difracción de calidad (datos no mostrados), un dúplex 21 bps con un solo saliente 5 'de nucleótidos en cada extremo que dió cristales difractan a 5 A en nuestros fuente de origen. La mejora en el límite de difracción sugerido que de extremo a extremo dúplex asociación fue en efecto favorecerING cristal de embalaje.
Desde SEQA interactúa con secuencias GATC que están en la misma cara del ADN, la cara opuesta del ADN dúplex debe estar expuesto al disolvente. Para mejorar aún más los cristales del complejo, que modifica entonces la secuencia de los dúplex para incluir un dinucleótido CG entre los dos sitios GATC para promover la cadena principal de ranura interacciones con moléculas de ADN adyacentes en el cristal a través de la cara opuesta de la doble hélice, un método que se ha utilizado para mejorar la cristalización del ADN en el pasado 10. Sin embargo, el límite de difracción de los cristales crecidos con el dúplex que contiene CG era idéntica a las cultivadas con un dúplex de ADN similares que no contienen el dinucleótido CG (Figura 3). Este resultado indica que la cadena principal de ranura interacciones no fueron importantes en este caso. Estamos posteriormente optimizado la longitud de los salientes mediante la comparación de los cristales crecidos con un dúplex de ADN que tenía dos nucleótidos adicionales en cadaExtremo 5 '. Este cambio tuvo un efecto drástico en la morfología del cristal, así como, límite de difracción adicional que indica que el nucleótido cambiado drásticamente los contactos moleculares y organización cristalina mejorada (Figura 3).
La estructura cristalina de SeqAΔ (41-59)-A25R unido a esta última dúplex de ADN confirmó que la cara libre de la doble hélice de ADN no participa en contactos de cristal, como el esperado del efecto limitado de la introducción de un dinucleótido CG-. A pesar del tamaño relativo de las proteínas y el ADN, la mayoría de las interacciones entre los compañeros de simetría están mediadas por interacciones proteína-proteína y proteína-DNA-(Figura 4). Curiosamente, en este caso particular, el efecto beneficioso de un voladizo dinucleótido 5 'no es debido a la formación de un ADN de pseudo-continuo. En cambio, el extremo 5 'de los proyectos de cadena metilada de distancia de los ejes del ADN e interacciona con la molécula SEQA proximal del complejo, que explica por qué dos nucleótidos w ere estrictamente necesario para cambiar el cristal embalaje 8,11.
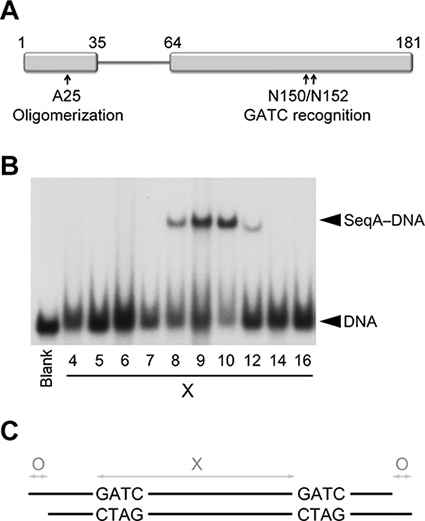
Figura 1. La unión de SeqAΔ (41-59)-A25R a hemimethylated ADN. (A) Representación esquemática de los dominios de oligomerización SEQA que median la proteína y unión de ADN. (B) la movilidad electroforética cambio de ensayo de SeqAΔ (41-59)-A25R con los ADN que contienen dos secuencias GATC hemimethylated separados por un número cada vez mayor de pares de bases ( X). El carril de más a la izquierda (con la etiqueta en blanco) contiene una mezcla equimolar de los ADN con 5, 7, 12, 21, 25 y 34 pares de bases entre las dos secuencias GATC en ausencia de SeqAΔ (41-59)-A25R. (C) Diagrama que representa las tres variables optimizados en los dúplex de ADN para obtener cristales de difracción de calidad.
upload/4266/4266fig2.jpg "fo: content-width =" 6in "fo: src =" / files/ftp_upload/4266/4266fig2highres.jpg "/>
Figura 2. Efecto de la variación de la distancia inter-GATC. Resumen de los oligonucleótidos utilizados y los cristales obtenidos con 23-24 pb de largo dúplex que contienen dos sitios GATC hemimethylated separadas por 9 o 10 pares de bases. Todas las imágenes de los cristales fueron tomadas con el mismo aumento y la barra de escala indica 100 micras. El límite de resolución de cada SeqAΔ (41-59)-A25R-DNA cristal se basa en imágenes de difracción recogidos en un Rigaku RU-300 sistema de generador de rayos-X. Haga clic aquí para ampliar la cifra .

Figura 3. Efecto de la variación de los extremos de ADN. Resumen de tél oligonucleótidos utilizados y obtenidos cristales con 21 dúplex bps largos que contiene dos sitios GATC hemimethylated separadas por 9 pares de bases e incluyendo 0, 1 o 2 nucleótidos salientes 5 'finales. Todas las imágenes de los cristales fueron tomadas con el mismo aumento y la barra de escala indica 100 micras. El límite de resolución de cada SeqAΔ (41-59)-A25R-DNA cristal se basa en imágenes de difracción recogidos en líneas de luz y X12C X29 (NSLS, BNL). Haga clic aquí para ampliar la cifra .
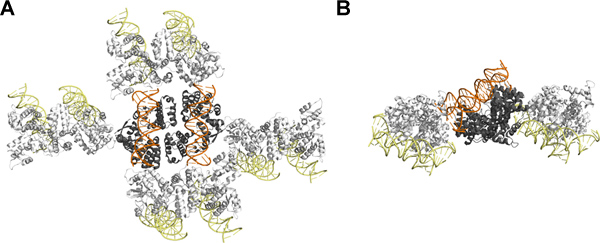
Figura 4. Embalaje de cristal de SeqAΔ (41-59)-A25R unido al ADN con saliente dinucleótido: Visto desde la parte superior (A) y lateral (B). La unidad asimétrica contiene dos SeqAΔ (41-59) de ADN A25R complejos donde se muestra la proteína en gris, mientras que el ADN se muestra en color naranja. Symmetria relacionada SeqAΔ (41-59)-A25R moléculas de ADN se muestran en blanco para la proteína y amarillo para el ADN. Esta cifra se preparó utilizando PyMOL 12. Esta cifra está relacionada con película 1. Haga clic aquí para ampliar la cifra .
Película 1. Haga clic aquí para ver la película .
Discusión
Uno de los mayores retos en macromolecular cristalografía de rayos X es la obtención de cristales de difracción de calidad. En el caso de complejos de proteínas o ADN-proteína, este reto se agrava debido a las variables adicionales que deben ser optimizados. Se cree ampliamente que la longitud del ADN y la presencia de voladizos pegajosas para mejorar la asociación de moléculas de ADN vecinos en un tiempo pseudo-duplex son los principales parámetros a optimizar. Sin embargo, hemos demostrado que la naturaleza y ...
Divulgaciones
No hay conflictos de interés declarado.
Agradecimientos
Los autores desean agradecer al personal PXRR al NSLS (Brookhaven National Laboratory) para la asistencia durante la recolección de datos y Pillon Mónica para ayuda en la purificación del ADN. Este trabajo fue financiado por los Institutos Canadienses de Investigación en Salud (RP 67189).
Materiales
| Name | Company | Catalog Number | Comments |
| Nombre del reactivo | Empresa | Catálogo # | Comentarios (opcional) |
| TRIS | Bioshop | TRS003.5 | |
| Ácido etilendiaminotetraacético (EDTA) | Fisher Scientific | E478-500 | |
| Dithiotheitrol (TDT) | Bio Basic Inc. | DB0058 | |
| NaCl | Bioshop | SOD002.10 | |
| Glicerol | Caledon | 5350-1 | |
| Sacarosa | Sigma-Aldrich | S5016-500G | |
| Dodecil sulfato de sodio (SDS) | Bioshop | SDS001.500 | |
| Urea | Bioshop | URE001.5 | |
| 40% Bis 29:1 / acrilamida | Bio Basic Inc. | A0007-500ml | Almacenar a 4 ° C |
| El ácido bórico | EMD | BX0865-1 | |
| Xileno FF cianol | Bio-Rad | 161-0423 | |
| Azul de bromofenol | Bioshop | BR0222 | |
| Sistema Dual Adjustable Gel Vertical | CBC Scientific Company Inc. | DASG-250 | |
| Índice de pantalla cristalización | Hampton Research | HR2-144 | Almacenar a 4 ° C |
| Asistente Yo pantalla cristalización | BioSystems Esmeralda | EBS-WIZ-1 | Almacenar a 4 ° C |
| Wizard II pantalla de cristalización | Emerald BioSystems | EBS-WIZ-2 | Almacenar a 4 ° C |
| Clásicos pantalla cristalización | Qiagen | 130701 | Almacenar a 4 ° C |
| Intelliplate bandejas | Art Robbins Instrumentos | 102-0001-00 | |
Soluciones Proteína tampón de purificación: 100 mM Tris pH 8, 2 mM EDTA, 2 mM DTT y 5% de glicerol. Proteína tampón de almacenamiento: 20 mM Tris pH 8, 150 mM NaCl, 5 mM DTT, 0,5 mM EDTA y 5% de glicerol. Gel mezcla de carga: Añadir 20 g de sacarosa, 25 mg de azul de bromofenol, 25 mg de xileno cianol FF, 1 ml de 10% w / v de SDS y 10 ml de TBE 10X a 70 ml de ddH autoclave 2 O. Se agita con calentamiento suave hasta que la sacarosa se disuelve y ajustar la finalvolumen a 100 ml con ddH autoclave 2 O. Almacenar a 4 ° C. Tampón de carga 2X: Añadir 11 g de urea a 10 ml de mezcla de carga de gel. Se agita sobre una placa caliente hasta que se disuelve la urea. Dividir en partes alícuotas en tubos de 2 ml y se almacena a 4 º C. Mezcla PÁGINA 10X: Mix 420,4 g de urea, 100 ml de TBE 10X (autoclave), 250 ml de 40% 29:1 Bis / acrilamida en ddH 2 O. Agitar hasta que esté totalmente disuelto y ajustar el volumen a 1 litro. Almacene en botellas oscuras a 4 ° C. 10X TBE: Disolver 108 g de TRIS, 55 g de ácido bórico y 9,3 g de EDTA en 1 litro de ddH 2 O. Autoclave y almacenar a temperatura ambiente. Tampón de elución: Diluir 8 ml de 5 M NaCl, 2 ml de 1 M Tris pH 7,5, 0,4 ml de 0,5 M EDTA pH 8 en 200 ml de ddH 2 O. Autoclave y almacenar a temperatura ambiente. |
Referencias
- Campbell, J. L., Kleckner, N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell. 62, 967-979 (1990).
- Guarné, A. Crystal structure of a SeqA-N filament: implications for DNA replication and chromosome organization. Embo. J. 24, 1502-1511 (2005).
- Brendler, T., Austin, S. Binding of SeqA protein to DNA requires interaction between two or more complexes bound to separate hemimethylated GATC sequences. Embo. J. 18, 2304-2310 (1999).
- Jordan, S. R., Whitcombe, T. V., Berg, J. M., Pabo, C. O. Systematic variation in DNA length yields highly ordered repressor-operator cocrystals. Science. 230, 1383-1385 (1985).
- Tan, S., Hunziker, Y., Pellegrini, L., Richmond, T. J. Crystallization of the yeast MATalpha2/MCM1/DNA ternary complex: general methods and principles for protein/DNA cocrystallization. J. Mol. Biol. 297, 947-959 (2000).
- Rice, P. A., Yang, S., Mizuuchi, K., Nash, H. A. Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn. Cell. 87, 1295-1306 (1996).
- Yang, W., Steitz, T. A. Crystal structure of the site-specific recombinase gamma delta resolvase complexed with a 34 bp cleavage site. Cell. 82, 193-207 (1995).
- Chung, Y. S., Brendler, T., Austin, S., Guarne, A. Structural insights into the cooperative binding of SeqA to a tandem GATC repeat. Nucleic Acids Res. , (2009).
- Anderson, J., Ptashne, M., Harrison, S. C. Cocrystals of the DNA-binding domain of phage 434 repressor and a synthetic phage 434 operator. Proc. Natl. Acad. Sci. U.S.A. 81, 1307-1311 (1984).
- Timsit, Y., Moras, D. DNA self-fitting: the double helix directs the geometry of its supramolecular assembly. Embo J. 13, 2737-2746 (1994).
- Chung, Y. S., Guarne, A. Crystallization and preliminary X-ray diffraction analysis of SeqA bound to a pair of hemimethylated GATC sites. Acta Crystallogr Sect F Struct Biol Cryst Commun. 64, 567-571 (2008).
- DeLano, W. L. . The PyMOL Molecular Graphic Systems. , (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados