È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Ottimizzazione iterativa di duplex di DNA per cristallizzazione di Seqa-DNA Complessi
In questo articolo
Riepilogo
Struttura cristallina di proteine-DNA può fornire indicazioni in funzione della proteina, meccanismo, così come la natura dell'interazione specifica. Qui, segnaliamo come ottimizzare la lunghezza, la sequenza e le estremità del DNA duplex per la co-cristallizzazione con Escherichia coli Seqa, un regolatore negativo dell'inizio della replicazione.
Abstract
Escherichia coli Seqa è un regolatore negativo della replicazione del DNA che impedisce prematuri eventi reinitiation sequestrando cluster hemimethylated GATC entro l'origine di replicazione 1. Al di là della provenienza, Seqa si trova a le forche di replicazione, dove organizza DNA appena replicato in un aumento delle strutture ordinate 2. Associa Seqa solo debolmente con singole sequenze GATC, ma forma complessi ad alta affinità con duplex di DNA contenenti più siti GATC. L'unità minima funzionale e strutturale di Seqa è un dimero, spiegando così il requisito di almeno due sequenze GATC per formare un complesso ad alta affinità con DNA hemimethylated 3. Inoltre, l'architettura Seqa, con l'oligomerizzazione e DNA-legame domini separati da un linker flessibile, consente il collegamento di ripetizioni GATC separati da un massimo di tre spire elicoidali. Pertanto, comprendere la funzione di Seqa a livello molecolare richiede strutturale analisi di Seqa legata a più sequenze GATC. In proteina-DNA cristallizzazione, DNA può avere nessuno ad un eccezionale effetto sulle interazioni di imballaggio a seconda delle dimensioni relative e l'architettura della proteina e del DNA. Se la proteina è più grande della DNA o impronte maggior parte del DNA, l'imballaggio cristallo è mediata principalmente da interazioni proteina-proteina. Viceversa, quando la proteina è la stessa dimensione o minore del DNA o copre solo una frazione delle interazioni DNA, DNA-DNA e DNA-proteina dominano imballaggio cristallo. Pertanto, la cristallizzazione della proteina-DNA complessi richiede lo screening sistematico di 4 DNA lunghezza e termina DNA (smussato o sporgenza) 5-7. In questo rapporto, descriviamo come progettare, ottimizzare, purificare e cristallizzare duplex di DNA contenenti hemimethylated tandem ripete GATC in complesso con una variante dimerica di Seqa (SeqAΔ (41-59)-A25R) per ottenere cristalli adatti per la determinazione della struttura.
Protocollo
1. Protein Purification
Il linker flessibile che collega la N-(oligomerizzazione) e C-terminale (DNA binding) dominii Seqa aiuti riconoscimento dei hemimethylated ripetizioni GATC separate da 1-3 giri sul DNA. Per questo studio, abbiamo utilizzato una variante dimerica della Seqa (SeqAΔ (41-59)-A25R) con una mutazione puntiforme nel dominio N-terminale che impedisce l'ulteriore oligomerizzazione e un linker accorciata che limita legante il DNA a tandem GATC ripete separati esclusivamente un giro sul DNA (Figura 1) 2,8.
- Trasformare BL21 (DE3) con il plasmide codificante Seqa sotto il controllo del promotore T7,
- Piastra la reazione di trasformazione in piastre di agar LB-tra 100 pg / ml ampicillina,
- Selezionare colonie miste per inoculare una piccola coltura overnight (media LB con 100 pg / ml ampicillina),
- La mattina dopo inoculare un 1 l di media utilizzando una diluizione 1:100 del overniGHT cultura,
- Crescere le cellule ad una OD 600 di ~ 0,7 e indurre la produzione di proteine per aggiunta di isopropil β-D-1-tiogalattopiranoside (IPTG) ad una concentrazione finale di 1 mM,
- Continuare l'incubazione per 3 ore a 37 ° C con agitazione orbitale e quindi raccogliere le cellule per centrifugazione (10 'a 3300 g),
- Risospendere il pellet di cellule in tampone A e purificazione lisi mediante sonicazione,
- Azzerare il lisato tramite centrifugazione (40 min a 39.000 g) e caricare il surnatante su una colonna di eparina equilibrata con tampone di purificazione,
- Eluiscono Seqa usando un gradiente lineare di NaCl 1 M (Seqa eluisce a ~ 0,7 M NaCl),
- Pool Seqa le frazioni contenenti insieme, diluire per abbassare la forza ionica del campione e di carico in una colonna a scambio cationico cromatografia equilibrata con tampone di purificazione,
- Usando un gradiente lineare di sale, puro Seqa eluisce a ~ 0,4 M NaCl,
- Pool i Seqa contenenti frazioni insieme, concentrate (3 mg / ml) e memorizzare in tampone di conservazione.
2. Purificazione del DNA
- Ordine oligonucleotidi complementari non metilato e metilati dalla vostra azienda preferita,
- Sciogliere 1 pmol di ogni singolo filamento di DNA liofilizzato in 800 ml di autoclavato DDH 2 O, vortice e lasciate riposare per 10-20 min,
- Aggiungere 800 pl di tampone di caricamento 2X preriscaldato a ciascun oligonucleotide,
- Oligonucleotidi per 20-30 nucleotidi, preparare una grande gel 10% denaturante (160 x 250 x 1 mm):
* Mescolare bene 80 ml di miscela PAGINA 10%, 80 TEMED ul e 800 ul persolfato di ammonio per gel e versare,
* Una volta polimerizzato, rimuovere il pettine e lavare i pozzetti con DDH 2 O a fondo,
* Montare i gel su gel colato tra piastra di raffreddamento e riempire i serbatoi superiore e inferiore con tampone di corsa (1X TBE),
* Pre-eseguire il gel a 700-750 V per riscaldare il gel fino a 55 ° C,
* Fermare la corsa e sciacquare il Thoro pozziughly con tampone di corsa. - Riscaldare gli oligonucleotidi a 90 ° C per 2 min,
- Vortex e centrifugare i campioni e subito prima di caricare il gel,
- Attivare il gel a ~ 700 V e fermarlo una volta che il oligonucleotide è migrata a metà strada. (Si noti che su un gel di poliacrilammide 10% blu di bromofenolo co-migra con oligonucleotidi ~ 20 basi di lunghezza e xilene cyanol FF con ~ 60 basi di lunghezza),
- Arrestare il gel, smontarlo dal box gel e rimuovere i distanziatori,
- Su una superficie piana, rimuovere una piastra di vetro e coprire il gel con pellicola trasparente,
- Girare il gel intorno, rimuovere l'altra piastra di vetro e coprire con pellicola trasparente,
- Contrassegnare le bande utilizzano la luce UV e una piastra fluorescente dietro il gel per vedere l'ombra del DNA,
- Tagliare la band con una lama di rasoio in piccoli pezzi e trasferirli in una provetta sterile 15 ml,
- Aggiungere 9 ml di tampone di eluizione ed eluire la notte a 37 ° C con agitazione,
- Attenzione trasferire la soluzionead un tubo da centrifuga sterilizzato utilizzando un gel-loading punta della pipetta per evitare pezzi acrilammide trasferimento e aggiungere 1 ml di acetato di sodio 3 M a pH 7 (diluizione 1:10) più 25 ml di etanolo al 100% refrigerati (2,5 volumi),
- Incubare a -20 ° C per almeno 3 ore,
- Centrifugare e trasferire il surnatante in un tubo separato,
- Asciugare il pellet sulla velocità-vac a fuoco medio,
- Risospendere il pellet in 400 ml di autoclavato DDH 2 O e il trasferimento in una nuova provetta,
- Aggiungere 40 ml di acetato di sodio 3 M a pH 7 e 1 ml di etanolo al 100%, mescolare bene (vortex) e incubare per 30 min a temperatura ambiente, seguito da 30 minuti a -20 ° C,
- Centrifugare per 15 minuti a 18.000 g ed eliminare il surnatante,
- Risciacquare il precipitato con 100 ml di etanolo al 70% freddo per rimuovere residui di sale dal pellet e centrifuga per 6 minuti a 18.000 g. Eliminare l'etanolo e asciugare il pellet sulla velocità-vac,
- Risospendere il pellet in un totale di 100 pl di autoclavato DDH 2 O. Misurare la concentrazione del oligonucleotide,
- Per temprare i duplex di DNA hemimethylated, mescolare concentrazioni equimolari dei singoli filamenti complementari e le miscele riscaldare a 95 ° C in un bagno d'acqua per 5 minuti e poi lasciar raffreddare lentamente a temperatura ambiente all'interno del bagno d'acqua.
3. Proteina-DNA Complex Formazione e analisi
- Miscelare volumi uguali di acqua di SeqAΔ (41-59)-A25 (81 mM) e DNA hemimethylated (81 mM),
- Incubare a temperatura ambiente per 15 minuti e conservare a 4 ° C fino al momento di usarlo,
- Schermo per condizioni di cristallizzazione che utilizzano commerciali sparse schermi a matrice,
- Una volta conduce cristallizzazione iniziali sono stati identificati, di ottimizzare le condizioni di crescere cristalli qualità diffrazione,
- Cryoprotect le risultanti Seqa-DNA cristalli, o aumentando la quantità di PEG 400 presenti nella soluzione di cristallizzazione ad una concentrazione finale del 25% (v / v) o aggiunta di glicerolo al 20% (v / v) alla soluzione di cristallizzazione,
- Scoop singoli cristalli con un anello di nylon, e li flash-congelamento in azoto liquido,
- Verificare il limite di diffrazione di ogni cristallo a 100 K.
4. Risultati rappresentativi
Per ottenere la struttura cristallina di SeqAΔ (41-59)-A25R legato al DNA hemimethylated, abbiamo ottimizzato consecutivamente tre parametri della DNA: (i) separazione tra hemimethylated sequenze GATC; (ii) lunghezza del duplex, e (iii) l' assenza / presenza di 5 'sporgenze.
Electro-mobilità saggi spostamento indicano che SeqAΔ (41-59)-A25R si lega preferenzialmente ripete GATC separati da 9-10 paia di basi (Figura 1). Pertanto, inizialmente proiettato su due piani 23-24 paia di basi (pb) lunga che contiene due sequenze hemimethylated GATC separati da 9 o 10 bps. Tre appartamenti su due piani prodotto cristalli ben sagomate (Figura 2). Alse nessuno dei cristalli diffratta di alta risoluzione, il 23 bps duplex lungo con i due siti GATC separati da 9 bps diffratta raggi X migliore del resto, che indica che una separazione GATC di 9 bps è preferito per la cristallizzazione. Pertanto, abbiamo fissato l'inter-GATC spaziatura a 9 bps per tutte le schermate successive.
Generalmente, cristallizzazione DNA è favorito per lunghezze duplex corrispondenti esatte spire elicoidali perché molecole di DNA più possibile impilare testa-coda per formare un continuo B-DNA all'interno del cristallo 9. Pertanto, abbiamo ridotto la lunghezza complessiva dei duplex a 21 bps (cioè due spire elicoidali). Mentre un duplex 21 bps con estremità smussata non ottenere cristalli qualità diffrazione (dati non mostrati), un duplex 21 bps con una sola sporgenza 5 'nucleotide su ciascuna estremità produssero cristalli che diffratta a 5 Å nella nostra sorgente casa. Il miglioramento del limite di diffrazione suggerito che end-to-end duplex associazione è stata effettivamente favorireing cristallo imballaggio.
Poiché Seqa interagisce con sequenze GATC che sono sulla stessa faccia del DNA, la faccia opposta del duplex DNA deve essere esposta al solvente. Per migliorare ulteriormente i cristalli del complesso, abbiamo poi modificato la sequenza del duplex per includere un dinucleotide CG tra i due siti GATC promuovere scanalatura-backbone interazioni con molecole di DNA adiacenti nel cristallo attraverso la faccia opposta del duplex, un metodo che è stato usato per migliorare cristallizzazione DNA in passato 10. Tuttavia, il limite di diffrazione dei cristalli cresciuti con la CG-duplex contenente era identico a quelli coltivati con un duplex DNA simile che non conteneva il dinucleotide CG (Figura 3). Questo risultato indica che scanalatura-dorsale interazioni non sono importanti in questo caso. Successivamente abbiamo ottimizzato la lunghezza delle sporgenze confrontando i cristalli cresciuti con un duplex DNA che aveva due nucleotidi addizionali ad ogni5 '. Questo cambiamento ha avuto un effetto drastico sulla morfologia cristallina, così come, limite di diffrazione che indica che il nucleotide complementare drammaticamente cambiato i contatti molecolari e organizzazione cristallo maggiore (Figura 3).
La struttura cristallina di SeqAΔ (41-59)-A25R legato a questo duplex DNA scorsa ha confermato che la faccia libera del DNA duplex non è attiva nei contatti di cristallo, come previsto dal limitato effetto di introdurre una CG-dinucleotide. Nonostante la dimensione relativa di proteine e DNA, la maggior parte delle interazioni tra compagni di simmetria sono mediati dalla proteina-proteina e proteina-DNA interazioni (Figura 4). È interessante notare, in questo caso particolare, l'effetto benefico di sbalzo un dinucleotide 5 'non è dovuto alla formazione di una pseudo-continuo DNA. Invece, l'estremità 5 'del filamento progetti metilate distanza dagli assi DNA e interagisce con la molecola Seqa prossimale del complesso, spiegando perché due nucleotidi w ere strettamente necessario per modificare il cristallo di imballaggio 8,11.
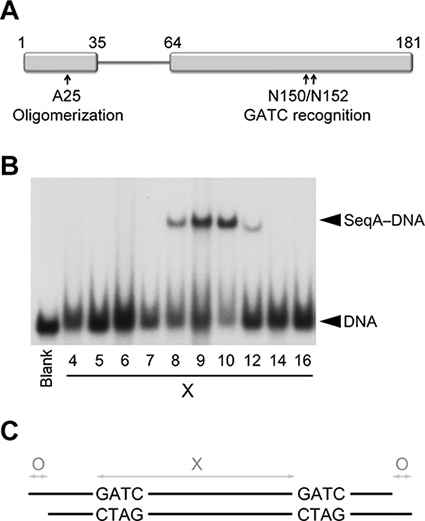
Figura 1. Il legame di SeqAΔ (41-59)-A25R al DNA hemimethylated. (A) Rappresentazione schematica dei domini di Seqa che oligomerizzazione di proteine mediato e legame al DNA. (B) elettroforetica spostamento saggio mobilità dei SeqAΔ (41-59)-A25R con DNA che contengono due sequenze hemimethylated GATC separati da un numero crescente di coppie di basi ( X). La corsia più a sinistra (etichettato Bianche) contiene una miscela equimolare dei DNA con 5, 7, 12, 21, 25 e 34 coppie di basi tra le due sequenze GATC in assenza di SeqAΔ (41-59)-A25R. (C) diagramma illustrante le tre variabili ottimizzati sulle duplex di DNA per ottenere cristalli di qualità diffrazione.
upload/4266/4266fig2.jpg "fo: content-width =" 6in "fo: src =" / files/ftp_upload/4266/4266fig2highres.jpg "/>
Figura 2. Effetto di variare la distanza tra GATC. Sintesi degli oligonucleotidi utilizzati e cristalli ottenuti con 23-24 appartamenti su due piani bp lunghi contenenti due siti hemimethylated GATC separati da 9 o 10 paia di basi. Tutte le immagini dei cristalli sono state scattate allo stesso ingrandimento e la barra della scala indica 100 micron. Il limite di risoluzione di ogni SeqAΔ (41-59)-A25R-DNA cristallo si basa su immagini di diffrazione raccolti su di un RU-300 Rigaku raggi X sistema generatore. Clicca qui per ingrandire la figura .

Figura 3. Effetto della variazione della DNA termina. Sintesi di tegli oligonucleotidi utilizzati e cristalli ottenuti con 21 bps lunghi duplex contenente due siti hemimethylated GATC separati da 9 coppie di basi e di cui 0, 1 o 2 nucleotidi 5 'sporgenze finali. Tutte le immagini dei cristalli sono state scattate allo stesso ingrandimento e la barra della scala indica 100 micron. Il limite di risoluzione di ogni SeqAΔ (41-59)-A25R-DNA cristallo si basa su immagini di diffrazione raccolti a linee di luce e X12C X29 (NSLS, BNL). Clicca qui per ingrandire la figura .
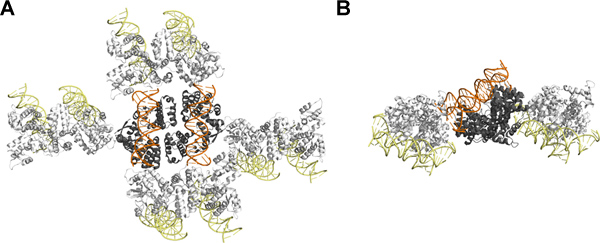
Figura 4. Imballaggio di cristallo di SeqAΔ (41-59)-A25R legato al DNA con sbalzo dinucleotide: Visto di (A) in alto e il lato (B). L'unità asimmetrica contiene due SeqAΔ (41-59)-A25R-DNA in cui viene mostrato la proteina in grigio mentre il DNA è indicato in arancione. Simmetricory correlato SeqAΔ (41-59)-A25R-DNA molecole sono presenti in bianco per la proteina e giallo per il DNA. Questo dato è stato preparato utilizzando PyMOL 12. Questo dato è legato al filmato 1. Clicca qui per ingrandire la figura .
Movie 1. Clicca qui per visualizzare filmati .
Discussione
Una delle maggiori sfide nel macromolecolare cristallografia a raggi X è l'ottenimento di cristalli di qualità di diffrazione. Nel caso di complessi proteici o proteine-DNA, questa sfida è aggravato a causa delle variabili aggiuntive che devono essere ottimizzati. Si crede che la lunghezza del DNA e la presenza di sbalzi appiccicose per migliorare associazione di molecole di DNA più vicini in un pseudo-duplex sono i principali parametri per ottimizzare. Tuttavia, abbiamo dimostrato che la natura e la lunghezza d...
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Gli autori desiderano ringraziare il personale PXRR al NSLS (Brookhaven National Laboratory) per l'assistenza durante la raccolta di dati e Monica Pillon aiuto per la purificazione del DNA. Questo lavoro è stato sostenuto dal Canadian Institutes of Health Research (MOP 67189).
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Catalogo # | Commenti (opzionale) |
| TRIS | Bioshop | TRS003.5 | |
| Acido etilendiamminotetraacetico (EDTA) | Fisher Scientific | E478-500 | |
| Dithiotheitrol (DTT) | Bio Base Inc. | DB0058 | |
| NaCl | Bioshop | SOD002.10 | |
| Glicerina | Caledon | 5350-1 | |
| Saccarosio | Sigma-Aldrich | S5016-500G | |
| Di sodio dodecil solfato (SDS) | Bioshop | SDS001.500 | |
| Urea | Bioshop | URE001.5 | |
| 40% Bis 29:1 / acrilammide | Bio Base Inc. | A0007-500ml | Conservare a 4 ° C |
| Acido borico | EMD | BX0865-1 | |
| Xilene cyanol FF | Bio-Rad | 161-0423 | |
| Blu bromofenolo | Bioshop | BR0222 | |
| Doppio sistema di regolazione verticale Gel | CBC Scientific Company Inc. | DASG-250 | |
| Indice cristallizzazione schermo | Hampton ricerca | HR2-144 | Conservare a 4 ° C |
| I cristallizzazione guidata schermo | Emerald BioSystems | EBS-WIZ-1 | Conservare a 4 ° C |
| Wizard II cristallizzazione schermo | Emerald BioSystems | EBS-WIZ-2 | Conservare a 4 ° C |
| Classics cristallizzazione schermo | Qiagen | 130701 | Conservare a 4 ° C |
| Intelliplate vassoi | Art Robbins Instruments | 102-0001-00 | |
Soluzioni Proteine tampone purificazione: 100 mM TRIS pH 8, 2 mM EDTA, 2 mM DTT e 5% glicerolo. Proteine tampone di conservazione: 20 mM TRIS pH 8, 150 mM NaCl, 5 mM DTT, 0,5 mM EDTA e il 5% glicerolo. Gel mix caricamento: Aggiungere 20 g di saccarosio, 25 mg di blu di bromofenolo, 25 mg di xilene cyanol FF, 1 ml di 10% w / v SDS e 10 ml di TBE 10X a 70 ml di autoclavato DDH 2 O. Mescolare con blando riscaldamento fino al saccarosio è sciolto e regolare la finaleil volume a 100 ml con autoclavato DDH 2 O. Conservare a 4 ° C. Loading buffer 2X: Aggiungere 11 g di urea a 10 ml di miscela di gel loading. Mescolare su una piastra calda fino urea si scioglie. Aliquotare in 2 provette ml e conservare a 4 ° C. PAGINA 10 volte mix: Mix 420,4 g di urea, 100 ml di TBE 10X (autoclave), 250 ml di 40% 29:1 Bis / acrilammide in DDH 2 O. Mescolare fino a quando completamente sciolto e regolare il volume a 1 litro. Conservare in bottiglie scure a 4 ° C. TBE 10X: sciogliere 108 g di TRIS, 55 g di acido borico e 9,3 g di EDTA in 1 litro di DDH 2 O. Autoclave e conservare a temperatura ambiente. Tampone di eluizione: Diluire 8 ml di NaCl 5 M, 2 ml di 1 M Tris pH 7,5, 0,4 ml di 0,5 M EDTA pH 8 su 200 ml di DDH 2 O. Autoclave e conservare a temperatura ambiente. |
Riferimenti
- Campbell, J. L., Kleckner, N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell. 62, 967-979 (1990).
- Guarné, A. Crystal structure of a SeqA-N filament: implications for DNA replication and chromosome organization. Embo. J. 24, 1502-1511 (2005).
- Brendler, T., Austin, S. Binding of SeqA protein to DNA requires interaction between two or more complexes bound to separate hemimethylated GATC sequences. Embo. J. 18, 2304-2310 (1999).
- Jordan, S. R., Whitcombe, T. V., Berg, J. M., Pabo, C. O. Systematic variation in DNA length yields highly ordered repressor-operator cocrystals. Science. 230, 1383-1385 (1985).
- Tan, S., Hunziker, Y., Pellegrini, L., Richmond, T. J. Crystallization of the yeast MATalpha2/MCM1/DNA ternary complex: general methods and principles for protein/DNA cocrystallization. J. Mol. Biol. 297, 947-959 (2000).
- Rice, P. A., Yang, S., Mizuuchi, K., Nash, H. A. Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn. Cell. 87, 1295-1306 (1996).
- Yang, W., Steitz, T. A. Crystal structure of the site-specific recombinase gamma delta resolvase complexed with a 34 bp cleavage site. Cell. 82, 193-207 (1995).
- Chung, Y. S., Brendler, T., Austin, S., Guarne, A. Structural insights into the cooperative binding of SeqA to a tandem GATC repeat. Nucleic Acids Res. , (2009).
- Anderson, J., Ptashne, M., Harrison, S. C. Cocrystals of the DNA-binding domain of phage 434 repressor and a synthetic phage 434 operator. Proc. Natl. Acad. Sci. U.S.A. 81, 1307-1311 (1984).
- Timsit, Y., Moras, D. DNA self-fitting: the double helix directs the geometry of its supramolecular assembly. Embo J. 13, 2737-2746 (1994).
- Chung, Y. S., Guarne, A. Crystallization and preliminary X-ray diffraction analysis of SeqA bound to a pair of hemimethylated GATC sites. Acta Crystallogr Sect F Struct Biol Cryst Commun. 64, 567-571 (2008).
- DeLano, W. L. . The PyMOL Molecular Graphic Systems. , (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon