Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Iterative Optimierung der DNA-Duplexes für Kristallisation von SeqA-DNA-Komplexen
In diesem Artikel
Zusammenfassung
Kristallstruktur von Protein-DNA-Komplexe können Einblick in Proteinfunktion, Mechanismus, sowie, die Natur der spezifischen Wechselwirkung bereitzustellen. Hier berichten wir, wie die Länge, Sequenz und Enden der DNA-Doppelstrang für Co-Kristallisation zu optimieren mit Escherichia coli SeqA, ein negativer Regulator der Replikation Initiation.
Zusammenfassung
Escherichia coli SeqA ist ein negativer Regulator der DNA-Replikation, die vorzeitige Reinitiation Ereignisse durch Maskierungsmittel hemimethylierte GATC Cluster verhindert innerhalb der Replikationsursprung ein. Jenseits der Herkunft, wird SeqA an den Replikationsgabeln, wo es neu replizierte DNA organisiert in höhere geordneten Strukturen 2 gefunden. SeqA Mitarbeiter nur schwach mit einzelnen GATC-Sequenzen, aber es bildet eine hohe Affinität Komplexe mit DNA-Duplexe mit mehreren GATC Seiten. Die minimale funktionelle und strukturelle Einheit SeqA ein Dimer, wodurch Erläuterung der Anforderung von wenigstens zwei GATC-Sequenzen, um eine hohe Affinität Komplex mit DNA hemimethylierte 3 zu bilden. Zusätzlich erlaubt die SeqA Architektur, mit der Oligomerisierung und DNA-bindenden Domänen durch einen flexiblen Linker getrennt sind, die Bindung an GATC Repeats durch bis zu drei Wendelgänge getrennt. Daher Verständnis der Funktion SeqA auf molekularer Ebene erfordert die strukturelle anaLyse SeqA mehrere GATC Sequenzen gebunden. In Protein-DNA Kristallisation kann DNA keine haben eine außergewöhnliche Wirkung auf die Verpackung Wechselwirkungen in Abhängigkeit von den relativen Größen und Architektur des Proteins und der DNA. Wenn das Protein größer ist als die DNA oder Fußabdrücke meisten der DNA wird die Kristallpackung primär durch Protein-Protein-Wechselwirkungen vermittelt. Umgekehrt, wenn das Protein die gleiche Größe oder kleiner als der DNA ist, oder es deckt nur einen Teil der DNA-, DNA-DNA-und DNA-Protein-Wechselwirkungen Kristallpackung dominieren. Daher erfordert Kristallisation von Protein-DNA-Komplexe die systematische Screening von DNA-Länge 4 und DNA-Enden (blunt oder Überhang) 5-7. In diesem Bericht beschreiben wir, wie zu entwerfen, zu optimieren, zu reinigen und zu kristallisieren hemimethylierte DNA-Duplexe mit Tandem-GATC Wiederholungen im Komplex mit einem dimeren Variante SeqA (SeqAΔ (41-59)-A25R), um geeignete Kristalle für die Strukturaufklärung erhalten.
Protokoll
Ein. Protein Purification
Der flexible Linker, das N-(Oligomerisierung) und C-terminalen (DNA Bindung) Domänen von SeqA Hilfsmittel die Erkennung hemimethylierte GATC Repeats durch ein bis drei Windungen auf der DNA getrennt. Für diese Studie verwendeten wir eine dimere Variante SeqA (SeqAΔ (41-59)-A25R) mit einer Punktmutation in der N-terminalen Domäne, die weitere Oligomerisierung und eine verkürzte DNA Linkers, der die Bindung an GATC Tandem wiederholt ausschließlich durch abgetrennt einschränkt verhindert eine Umdrehung auf der DNA (Abbildung 1) 2,8.
- Transformieren BL21 (DE3)-Zellen mit dem Plasmid kodiert SeqA unter der Kontrolle des T7-Promotors,
- Platte die Transformationsreaktion in LB-Agarplatten mit 100 ug / ml Ampicillin,
- Wählen gemischten Kolonien eine kleine Nacht angeimpft (LB-Medium mit 100 ug / ml Ampicillin)
- Am nächsten Morgen inokulieren einen 1 l Medium mit einer 1:100 Verdünnung der overnight Kultur,
- Wachsen die Zellen bis zu einer OD 600 von ~ 0,7 und induzieren Proteinproduktion durch Zugabe von Isopropyl-β-D-1-thiogalactopyranosid (IPTG) zu einer Endkonzentration von 1 mM,
- Fahren Sie mit der Inkubation für 3 Stunden bei 37 ° C unter kreisförmigem Schütteln und dann ernten die Zellen durch Zentrifugation (10 min bei 3.300 g),
- Zellpellet in Reinigungspuffer A und durch Beschallung lysiert,
- Deaktivieren des Lysats durch Zentrifugation (40 min bei 39.000 g) und Laden des Überstands auf eine Säule mit Heparin Reinigungspuffer,
- Eluieren SeqA Verwendung eines linearen Gradienten bis 1 M NaCl (SeqA eluiert bei ~ 0,7 M NaCl),
- Pool die SeqA enthaltenden Fraktionen zusammen, verdünnen, um die Ionenstärke der Probe und Last in eine Kationenaustauschchromatographie-Säule, äquilibriert mit Reinigungspuffer abzusenken,
- Verwendung eines linearen Salzgradienten eluiert reinen SeqA bei ~ 0,4 M NaCl,
- Pool die SeqA-haltigen Fraktionen zusammen, konzentrierte (3 mg / ml) und abspeichern im Pufferspeicher.
2. DNA Purification
- Bestellen komplementäre methylierten und methylierten Oligonukleotide aus Ihrem Lieblings-Unternehmen,
- Lösen Sie 1 umol jedes lyophilisierten Einzelstrang-DNA in 800 ul autoklaviert ddH 2 O, Wirbel und lassen Sie es für 10-20 min zu sitzen,
- Fügen Sie 800 ul vorgeheizten 2X Ladepuffer jedem Oligonukleotid,
- Für 20-30 Nukleotide lange Oligonukleotide, bereiten eine große 10% denaturierenden Gel (160 x 250 x 1 mm):
* Gut mischen 80 ml 10% PAGE-Mix, 80 ul TEMED und 800 ul Ammoniumpersulfat pro Gel und gießen,
* Sobald polymerisiert, nehmen Sie den Kamm und spülen Sie die Vertiefungen mit ddH 2 O gründlich,
* Setzen Sie die Gele auf Gel-Besetzung einschließlich Kühlplatte und füllen Sie dann die oberen und unteren Reservoirs mit Laufpuffer (1X TBE),
* Vorlauf des Gels bei 700-750 V, um das Gel erwärmen bis zu 55 ° C,
* Halten Sie den Lauf und spülen Sie den Brunnen thoroughly mit Laufpuffer. - Erhitzen der Oligonukleotide auf 90 ° C für 2 min,
- Vortex und drehen Sie die Proben und unmittelbar vor dem Laden des Gels,
- Führen Sie das Gel bei ~ 700 V und stoppen Sie ihn, sobald Ihr Oligonukleotid halbwegs migriert wurde. (Man beachte, dass auf einem 10% Polyacrylamidgel Bromphenolblau wandert Zusammenarbeit mit Oligonukleotiden ~ 20 Basen lang und Xylencyanol FF mit ~ 60 Basen lang),
- Stoppen Sie das Gel, zerlegen sie aus dem Gel und entfernen Sie die Abstandshalter,
- Auf einer ebenen Fläche, entfernen Sie eine Glasplatte und decken das Gel mit Plastikverpackung,
- Schalten Sie das Gel um, entfernen Sie die andere Glasplatte und bedecken Sie es mit Plastikverpackung,
- Die Bänder mit UV-Licht und eine fluoreszierende Platte hinter dem Gel, um die DNA Schatten sehen
- Schneiden Sie das Band mit einer Rasierklinge in kleine Stücke und übertragen sie in einem sterilen 15 ml Tube,
- 9 ml Elutionspuffer und eluieren Nacht bei 37 ° C unter Rühren,
- Transferieren es vorsichtig in die Lösungzu einem autoklavierten Zentrifugenröhrchen mit einem Gel-loading Pipettenspitze Übertragung Acrylamid Stücke zu vermeiden und 1 ml 3 M Natriumacetat bei pH 7 (1:10 Verdünnung) plus 25 ml gekühlt 100% Ethanol (2,5 Bände),
- Inkubation bei -20 ° C für mindestens 3 Stunden,
- Spin-down und übertragen den Überstand in ein separates Rohr,
- Trocknen Sie die Pellets auf dem Speed-Vac bei mittlerer Hitze,
- Das Pellet in 400 ul autoklaviert ddH 2 O und übertragen in ein frisches Röhrchen,
- Fügen Sie 40 ul 3 M Natriumacetat bei pH 7 und 1 ml 100% Ethanol; gut mischen (Vortex) und inkubieren 30 min bei Raumtemperatur, gefolgt von 30 min bei -20 ° C,
- Spin für 15 min bei 18.000 g und den Überstand verwerfen,
- Spülen Sie das Pellet mit 100 ul 70% kaltem Ethanol, um das restliche Salz aus dem Pellet und Spin zu entfernen für 6 min bei 18.000 g. Entsorgen Sie das Ethanol und Trocknen des Pellets auf Speed-Vac,
- Das Pellet in einer Gesamtmenge von 100 ul autoklaviertem ddH 2 O. Messung der Konzentration des Oligonukleotids,
- Die DNA-Duplexe hemimethylierte annealen, mischen äquimolaren Konzentrationen der komplementären Einzelstränge und erhitzen die Mischungen auf 95 ° C im Wasserbad 5 min und dann abkühlen lassen langsam auf Raumtemperatur innerhalb der Wasserbad.
3. Protein-DNA-Komplex Bildung und Analyse
- Mix gleichen Volumina an gereinigtem SeqAΔ (41-59)-A25 (81 uM) und hemimethylierte DNA (81 &mgr; M)
- Inkubation bei Raumtemperatur für 15 min und bei 4 ° C bis Sie bereit sind, es zu benutzen,
- Screen für Kristallisationsbedingungen mit kommerziellen Sparse-Matrix-Bildschirme,
- Nach dem ersten Kristallisation führt identifiziert wurden, optimieren die Bedingungen der Beugung Qualität Kristalle wachsen,
- Cryoprotect die resultierenden Kristalle SeqA-DNA entweder durch Erhöhung der Menge an PEG 400 in der Kristallisationslösung auf eine Endkonzentration von 25% (v / v) oder Zugabe von 20% Glycerin (Vol / Vol) zu der Kristallisationslösung,
- Scoop einzelnen Kristalle mit einem Nylon-Schleife, und Flash-frieren sie in flüssigem Stickstoff
- Testen der Beugungsgrenze jedes Kristalls bei 100 K.
4. Repräsentative Ergebnisse
Die Kristallstruktur der SeqAΔ (41-59)-A25R gebunden hemimethylierte DNA zu erhalten, man nacheinander drei Parameter optimiert auf der DNA: (i) Trennung zwischen hemimethylierte GATC-Sequenzen, (ii) Länge des Duplexes, und (iii) das Fehlen / Vorhandensein 5'-Überhänge.
Electro Mobility-Shift-Assays zeigen, dass SeqAΔ (41-59)-A25R bindet bevorzugt GATC Wiederholungen von 9-10 Basenpaare (Abbildung 1) getrennt. Deshalb haben wir zunächst abgeschirmt Duplexe 23-24 Basenpaaren (bps) lang mit zwei hemimethylierte GATC-Sequenzen entweder 9 oder 10 bps getrennt. Drei Duplexe ergab schön geformten Kristalle (Abbildung 2). Alobwohl keiner von den Kristallen gebeugt, um hochauflösende, Basenpaare der 23 langen Duplex mit den beiden GATC Seiten um 9 Basispunkte gebeugten Röntgenstrahlen besser als der Rest abgetrennt, was darauf hinweist, dass ein GATC Trennung von 9 bps für die Kristallisation bevorzugt wurde. Daher haben wir die inter-GATC Abstand bis 9 bps für alle nachfolgenden Bildschirmen.
Im Allgemeinen wird DNA Kristallisation für Duplex Längen entsprechend genaue Wendelgänge begünstigt, da mehrere DNA-Moleküle Kopf-Schwanz-stapeln kann, um einen kontinuierlichen B-DNA innerhalb des Kristalls 9 zu bilden. Daher wir die Gesamtlänge der Duplexe zu 21 bps (dh zwei Wendelgänge) verkürzt. Während eine 21 bps Duplex mit stumpfen Enden nicht nachgeben Beugungsqualität Kristalle (Daten nicht gezeigt), einen 21 bps Duplex mit einem einzelnen 5'-Überhang an jedem Ende Nucleotid hat Kristalle zu ergeben, dass gebeugtes bis 5 Å im Hause Quelle. Die Verbesserung gegenüber der Beugungsgrenze vorgeschlagen, dass Ende-zu-Ende-Duplex Assoziation wurde tatsächlich begünstigenten Kristallpackung.
Da SeqA interagiert mit GATC-Sequenzen, die auf der gleichen Fläche der DNA sind, sollte die gegenüberliegende Fläche des DNA-Duplex zu dem Lösungsmittel ausgesetzt werden. Zur weiteren Verbesserung der Kristalle des Komplexes, dann wir die Sequenz des Duplex modifiziert, um eine CG-Dinukleotid zwischen den beiden Seiten zu GATC rillenförmigen Backbone Wechselwirkungen mit benachbarten DNA-Moleküle in der Kristallstruktur zu fördern, durch die gegenüberliegende Seite des Duplex, ein Verfahren beinhalten Das wurde verwendet, um DNA Kristallisation in den letzten 10 verbessern. Jedoch war die Beugungsgrenze der Kristalle mit dem CG-enthaltenden Duplex gezüchtet identisch zu denen mit einer ähnlichen DNA-Duplex, der nicht enthielt das CG Dinucleotid (Abbildung 3) aufgewachsen. Dieses Ergebnis zeigt, dass die Nut-Rückgrat-Wechselwirkungen waren nicht in diesem Fall wichtig. Wir anschließend optimiert die Länge der Überhänge durch Vergleichen der Kristalle mit einem DNA-Doppelstrang, der zwei zusätzliche Nukleotide an jeder hatte gezüchtet5'-Ende. Diese Veränderung hatte eine drastische Wirkung auf Kristallmorphologie, sowie, was anzeigt, dass die Beugungsgrenze zusätzliches Nukleotid dramatisch die molekularen Kontakte und verstärkter Kristallorientierung Organisation (Abbildung 3) verändert.
Die Kristallstruktur von SeqAΔ (41-59)-A25R zu dieser letzten DNA-Duplex bestätigt, dass die freie Fläche der DNA-Doppelstrang nicht in Kristallkontakte eingreifen gebunden, wie aus der begrenzten Wirkung des Einführens eines CG-Dinukleotid erwartet. Trotz der relativen Größe der Protein-und DNA, sind die meisten Wechselwirkungen zwischen Symmetrieverknüpfungen durch Protein-Protein-und Protein-DNA-Wechselwirkungen (Fig. 4) vermittelt. Interessanterweise sind in diesem speziellen Fall ist die vorteilhafte Wirkung von einer 5'-Überhang Dinucleotid nicht durch die Bildung eines pseudo-kontinuierliche DNA. Stattdessen wirkt das 5'-Ende der methylierten Strang wegragt aus den DNA-Achsen und mit dem proximalen SeqA Molekül des Komplexes, zu erklären, warum zwei Nucleotide w ere unbedingt erforderlich, um die Kristallpackung 8,11 ändern.
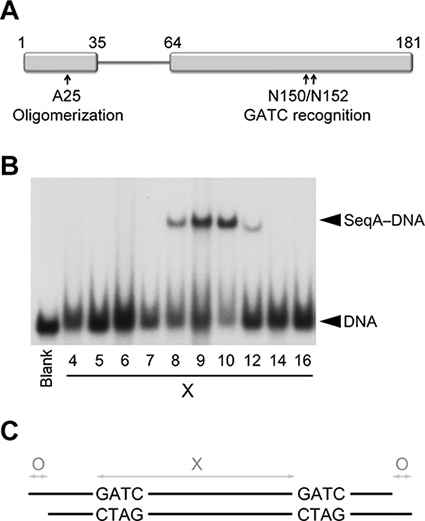
Abbildung 1. Bindung SeqAΔ (41-59)-A25R um hemimethylierte DNA. (A) Schematische Darstellung der Domänen SeqA, die den viralen Protein Oligomerisierung und DNA-Bindung. (B) Elektrophoretische Mobilitäts-Shift-Assay von SeqAΔ (41-59)-A25R mit DNAs mit zwei hemimethylierte GATC-Sequenzen von einer zunehmenden Zahl von Basenpaaren getrennt ( X). Die äußerst linken Spur (beschriftet Blank) eine äquimolare Mischung von DNAs mit 5, 7, 12, 21, 25 und 34 Basenpaare zwischen den beiden GATC Sequenzen in Abwesenheit von SeqAΔ (41-59)-A25R. (C) Schema der drei Variablen auf die DNA-Duplexe optimiert Beugungsqualität Kristalle zu erhalten.
upload/4266/4266fig2.jpg "fo: content-width =" 6in "fo: src =" / files/ftp_upload/4266/4266fig2highres.jpg "/>
Abbildung 2. Wirkung der Variation der inter-GATC erreichbar. Zusammenfassung der verwendeten Oligonukleotide und Kristalle mit 23-24 bp langen Duplexe mit zwei hemimethylierte GATC Websites von 9 oder 10 Basenpaare getrennt erhalten. Alle Bilder von Kristallen wurden bei der gleichen Vergrößerung aufgenommen und die Waage zeigt an 100 um. Die Auflösungsgrenze der einzelnen SeqAΔ (41-59)-A25R-DNA-Kristall auf der Beugung von Bildern auf einem Rigaku RU-300 X-ray-Generator-System gesammelt wurden. Klicken Sie hier für eine größere Abbildung zu sehen .

Abbildung 3. Effekt der Variation der DNA-Enden. Zusammenfassung von ter verwendeten Oligonukleotide und Kristalle mit 21 bps langen Duplexe mit zwei hemimethylierte GATC Stellen durch 9 Basenpaare getrennte und einschließlich 0, 1 oder 2 Nukleotid 5'-Ende Überhänge. Alle Bilder von Kristallen wurden bei der gleichen Vergrößerung aufgenommen und die Waage zeigt an 100 um. Die Auflösungsgrenze der einzelnen SeqAΔ (41-59)-A25R-DNA-Kristall auf Beugungsbilder an Strahlführungen X12C und X29 (NSLS, BNL) gesammelt wurden. Klicken Sie hier für eine größere Abbildung zu sehen .
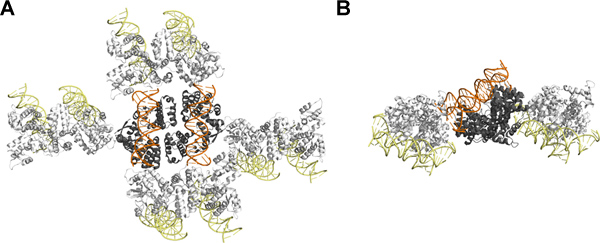
Abbildung 4. Kristallpackung SeqAΔ (41-59)-A25R DNA mit Dinukleotid Überhang gebunden: Gesehen von der (A) nach oben und von der Seite (B). Die asymmetrische Einheit enthält zwei SeqAΔ (41-59)-A25R-DNA-Komplexe, wo das Protein in grau dargestellt wird, während die DNA in orange dargestellt. Symry verwandten SeqAΔ (41-59)-A25R-DNA-Moleküle sind in weiß für das Protein und Gelb für die DNA dargestellt. Diese Zahl wurde unter Verwendung PyMOL 12. Diese Zahl wird auf Movie 1 verwandt. Klicken Sie hier für eine größere Abbildung zu sehen .
Movie 1. Klicken Sie hier, um Film anzusehen .
Access restricted. Please log in or start a trial to view this content.
Diskussion
Eine der größten Herausforderungen in der makromolekularen Röntgenkristallographie ist der Erhalt Beugungsqualität Kristalle. Im Falle von Protein-oder Protein-DNA-Komplexe, wird diese Herausforderung verschärft durch die zusätzlichen Variablen optimiert werden müssen. Es wird allgemein angenommen, dass die Länge der DNA und die Anwesenheit von klebrigen Überhänge zur Vereinigung von benachbarten DNA-Moleküle in einer längeren Pseudo-Duplex werden die wesentlichen Parameter zur Optimierung verbessern. Wir ha...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Die Autoren möchten die PXRR Mitarbeiter an der NSLS (Brookhaven National Laboratory) danken für die Unterstützung während der Datensammlung und Monica Pillon für die Hilfe bei DNA-Aufreinigung. Diese Arbeit wurde von der kanadischen Institutes of Health Research (MOP 67.189) unterstützt.
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| Name des Reagenzes | Firma | Katalog # | Kommentare (optional) |
| TRIS | Bioshop | TRS003.5 | |
| Ethylendiamintetraessigsäure (EDTA) | Fisher Scientific | E478-500 | |
| Dithiotheitrol (DTT) | Bio Grundlegende Inc. | DB0058 | |
| NaCl | Bioshop | SOD002.10 | |
| Glycerol | Caledon | 5350-1 | |
| Saccharose | Sigma-Aldrich | S5016-500G | |
| Natriumdodecylsulfat (SDS) | Bioshop | SDS001.500 | |
| Harnstoff | Bioshop | URE001.5 | |
| 40% 29:1 Bis / Acrylamid | Bio Grundlegende Inc. | A0007-500ml | Lagerung bei 4 ° C |
| Borsäure | EMD | BX0865-1 | |
| Xylencyanol FF | Bio-Rad | 161-0423 | |
| Bromphenolblau | Bioshop | BR0222 | |
| Dual Adjustable Vertical Gel System | CBC Scientific Company Inc. | DASG-250 | |
| Index Kristallisation Bildschirm | Hampton Research | HR2-144 | Lagerung bei 4 ° C |
| Wizard I Kristallisation Bildschirm | Smaragd BioSystems | EBS-WIZ-1 | Lagerung bei 4 ° C |
| Wizard II Kristallisation Bildschirm | Emerald BioSystems | EBS-WIZ-2 | Lagerung bei 4 ° C |
| Classics Kristallisation Bildschirm | Qiagen | 130701 | Lagerung bei 4 ° C |
| Intelliplate Trays | Art Robbins Instruments | 102-0001-00 | |
Lösungen Proteinreinigung Puffer: 100 mM TRIS pH 8, 2 mM EDTA, 2 mM DTT und 5% Glycerin. Protein Lagerungspuffer: 20 mM Tris pH 8, 150 mM NaCl, 5 mM DTT, 0,5 mM EDTA und 5% Glycerin. Gelbeladung Mix: 20 g Saccharose, 25 mg Bromphenolblau, 25 mg Xylencyanol FF, 1 ml von 10% w / v SDS und 10 ml 10X TBE bis 70 ml autoklaviertem ddH 2 O. Rühren Sie mit leichtem Erwärmen bis Saccharose aufgelöst und stellen die endgültigeVolumen auf 100 ml mit autoklaviertem ddH 2 O. Lagerung bei 4 ° C. 2X Ladepuffer: In 11 g Harnstoff in 10 ml Gelbeladung Mix. Stir auf einer Heizplatte bis Harnstoff auflöst. Aliquot in 2 ml Röhrchen und bei 4 ° C. 10X PAGE-Mix: Mix 420,4 g Harnstoff, 100 ml 10X TBE (autoklaviert), 250 ml von 40% 29:1 Bis / Acrylamid in ddH 2 O. Rühren, bis vollständig gelöst und die Lautstärke auf 1 Liter. Bewahren Sie in dunklen Flaschen bei 4 ° C. 10X TBE: auflösen 108 g TRIS, 55 g Borsäure und 9,3 g EDTA in 1 Liter ddH 2 O. Autoklaven und bei Raumtemperatur lagern. Elutionspuffer: Verdünnen 8 ml 5 M NaCl, 2 ml 1 M Tris pH 7,5, 0,4 ml 0,5 M EDTA pH 8 auf 200 ml ddH 2 O. Autoklaven und bei Raumtemperatur lagern. |
Referenzen
- Campbell, J. L., Kleckner, N. E. coli oriC and the dnaA gene promoter are sequestered from dam methyltransferase following the passage of the chromosomal replication fork. Cell. 62, 967-979 (1990).
- Guarné, A. Crystal structure of a SeqA-N filament: implications for DNA replication and chromosome organization. Embo. J. 24, 1502-1511 (2005).
- Brendler, T., Austin, S. Binding of SeqA protein to DNA requires interaction between two or more complexes bound to separate hemimethylated GATC sequences. Embo. J. 18, 2304-2310 (1999).
- Jordan, S. R., Whitcombe, T. V., Berg, J. M., Pabo, C. O. Systematic variation in DNA length yields highly ordered repressor-operator cocrystals. Science. 230, 1383-1385 (1985).
- Tan, S., Hunziker, Y., Pellegrini, L., Richmond, T. J. Crystallization of the yeast MATalpha2/MCM1/DNA ternary complex: general methods and principles for protein/DNA cocrystallization. J. Mol. Biol. 297, 947-959 (2000).
- Rice, P. A., Yang, S., Mizuuchi, K., Nash, H. A. Crystal structure of an IHF-DNA complex: a protein-induced DNA U-turn. Cell. 87, 1295-1306 (1996).
- Yang, W., Steitz, T. A. Crystal structure of the site-specific recombinase gamma delta resolvase complexed with a 34 bp cleavage site. Cell. 82, 193-207 (1995).
- Chung, Y. S., Brendler, T., Austin, S., Guarne, A. Structural insights into the cooperative binding of SeqA to a tandem GATC repeat. Nucleic Acids Res. , (2009).
- Anderson, J., Ptashne, M., Harrison, S. C. Cocrystals of the DNA-binding domain of phage 434 repressor and a synthetic phage 434 operator. Proc. Natl. Acad. Sci. U.S.A. 81, 1307-1311 (1984).
- Timsit, Y., Moras, D. DNA self-fitting: the double helix directs the geometry of its supramolecular assembly. Embo J. 13, 2737-2746 (1994).
- Chung, Y. S., Guarne, A. Crystallization and preliminary X-ray diffraction analysis of SeqA bound to a pair of hemimethylated GATC sites. Acta Crystallogr Sect F Struct Biol Cryst Commun. 64, 567-571 (2008).
- DeLano, W. L. The PyMOL Molecular Graphic Systems. , DeLano Scientific. (2002).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten