Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Purificación y replegamiento de amiloide fibrillas de (Su)
En este artículo
Resumen
A two-step chromatographic method is described for the purification of recombinant Shadoo protein expressed as inclusion bodies in Escherichia coli, as well as a protocol to fibrillate purified Shadoo into amyloid structures.
Resumen
El sistema de expresión de Escherichia coli es una poderosa herramienta para la producción de proteínas recombinantes eucariotas. Lo utilizamos para producir Shadoo, una proteína perteneciente a la familia de priones. Un método cromatográfico para la purificación de (His) 6-etiquetados Shadoo recombinantes expresadas como cuerpos de inclusión se describe. Los cuerpos de inclusión se solubilizan en 8 M urea y unidos a una columna de Ni 2+ -charged para llevar a cabo la cromatografía de afinidad de iones. Las proteínas unidas se eluyen mediante un gradiente de imidazol. Las fracciones que contienen la proteína Shadoo son sometidas a cromatografía de exclusión por tamaño para obtener una proteína altamente purificada. En la etapa final de Shadoo purificada se desala para eliminar las sales, urea e imidazol. Shadoo proteína recombinante es un reactivo importante para los estudios biofísicos y bioquímicos de los trastornos de la conformación de proteínas que ocurren en las enfermedades priónicas. Muchos informes han demostrado que las enfermedades neurodegenerativas causadas por priones originan a partir de la deposición de puñaladale ordenó fibrillas amiloides. / Se presentan valores de pH básicos protocolos de ejemplo que describen cómo fibrilar Shadoo en fibrillas amiloides en ácido y neutro. Los métodos sobre cómo producir y fibrilar Shadoo puede facilitar la investigación en laboratorios que trabajan sobre las enfermedades priónicas, ya que permite la producción de grandes cantidades de proteína en una manera rápida y de bajo coste.
Introducción
Enfermedades neurodegenerativas causadas por priones, que incluyen la encefalopatía espongiforme bovina en el ganado, la tembladera en las ovejas y la enfermedad de Creutzfeldt-Jakob en humanos, son fatales e incurable. Las enfermedades priónicas se caracterizan por cambios conformacionales de la proteína priónica celular compuestos principalmente de alfa-hélices, en el β-hoja de cruz enriquecido amiloide confórmero 1,2. Cruz-ß-estructuras contienen densas y ß-hojas-cristalinas como altamente ordenadas estabilizados con enlaces de hidrógeno 3,4. Amiloides priónicas puede auto-replicarse debido a los beta-capítulos en el borde de crecimiento que proporcionan una plantilla para el reclutamiento y la conversión de una unidad de proteína monomérica.
De acuerdo con la hipótesis de "única proteína", el confórmero de amiloide de la proteína prión es el único agente infeccioso. Sin embargo, se han propuesto algunas otras biomoléculas ser esencial para los trastornos por priones. Por ejemplo, las membranas celulares se cree que ser un lugar donde conversien su lugar y algunos toma lípidos cargados negativamente se ha demostrado para mejorar el proceso de conversión de 5,6. Además, algunas proteínas también pueden estar implicados en patologías priónicas. En concreto, hay otros dos miembros de la familia de proteínas de priones: Doppel y Shadoo 7,8. Doppel tiene una estructura relativamente rígida estabilizado con puentes di-sulfuro y no autopolimerizarse y el agregado de fibrillas amiloides 9. En contraste, similar a la proteína prión, Shadoo puede adoptar una estructura enriquecida en β-lámina y autoasociarse en estructuras de fibrillas amiloides. Se demostró que Shadoo puede fibrilar en condiciones nativas o tras la unión membranas cargadas negativamente 10,11. La conversión de Shadoo en fibras de tipo amiloide puede estar asociada con patologías. De hecho, los mecanismos por los cuales las estructuras de tipo amiloide causan la enfermedad son poco conocidos.
Shadoo tiene un dominio hidrófobo (HD) y una serie de tándem Arg / Gly repite t similareso la parte N-terminal de la proteína prión (Figura 1A). A medida que la parte N-terminal de la proteína prión, Shadoo está altamente cargado positivamente y parece ser una proteína nativa no estructurada 11,12. Shadoo parece ser funcionalmente relacionadas con los trastornos por priones, ya que puede unirse directamente la proteína del prión. Además, su expresión es regulada hacia abajo durante la patología prión 13,14. Sin embargo, el papel de Shadoo en la enfermedad priónica todavía no se ha establecido.
Hemos desarrollado un plásmido que porta la secuencia de codificación del gen Shadoo ratón. El plásmido se utilizó para transformar E. coli para la producción de N-terminal Su proteína Shadoo 6 de fusión. Este sistema de expresión está bien establecida en nuestro laboratorio y se utiliza comúnmente en nuestros proyectos en curso 6,15,16. Una cuestión importante para la expresión de Shadoo es la elección de la E. coli cepa. Mientras cepa bacteriana BL21 competente se utiliza comúnmente para la expresión de pro recombinanteproteínas Shadoo se expresó y se purifica sólo de la cepa bacteriana transformada SoluBL21 éxito. SoluBL21 E. competente coli es un mutante mejorada de cepa huésped BL21 desarrollado para la producción de proteínas cuya expresión en la BL21 padre no dio producto soluble detectable. Una expresión de alto nivel de Shadoo en SoluBL21 E. coli conduce a la acumulación de la proteína en cuerpos de inclusión. Como característica general, cuando una proteína no nativa es altamente expresado en E. coli, esta proteína tiende a acumularse en los cuerpos de inclusión insolubles. Shadoo probablemente agregados a través de interacciones hidrófobas no covalentes o iónicos (o una combinación de ambos) para estructuras dinámicas altamente enriquecido formadas por la proteína plegada en diversos grados. En consecuencia, la purificación comprende al menos dos etapas: (i) una separación selectiva de la proteína a partir de otras biomoléculas en un medio de desnaturalización, y (ii) una renaturalización de la proteína purificada utilizando en el plegamiento in vitrotécnicas.
La separación selectiva de Shadoo se logró en una solución tampón que contiene urea 8 M (o, alternativamente, guanidina-HCl 6 M). La eliminación de urea y de la renaturalización de la proteína se puede hacer mediante la aplicación de diversos protocolos de: (i) la renaturalización en una solución de pH ácido para obtener una proteína Shadoo monomérica no estructurada, o (ii) la renaturalización en una solución de pH ≥ 7 para obtener Shadoo polimerizado en fibras amiloides estables con motivos característicos cruz β-hoja.
Protocolo
1. Generación de plásmido y Shadoo Expresión
Nota: La proteína murina Shadoo gen que codifica (Shadoo 25 a 122), se subclonó en el vector de expresión pET-28 11. Este clon se transformó en E. SoluBL21 coli cepa bacteriana, para expresar la proteína Shadoo con una etiqueta de hexahistidina N-terminal, (HHHHHHHHHHSSGHIDDDDKHMKGGRGGARGSARGVRGGARGASRVRVRP APRYGSSLRVAAAGAAAGAAAGVAAGLATGSGWRRTSGPGELGLEDDENGAMGGNGTDRGVYSYWAWTSG) como se explicó previamente 11. El plásmido se puede solicitar a los autores. El peso molecular de Shadoo calculado a partir de su secuencia de aminoácidos es 12,243.2 Da.
Nota: Realice todas las manipulaciones con bacterias bajo una campana de flujo laminal o cerca de Bunsen llama.
- Transformar bacterias SoluBL21 competentes con el 28-Su plásmido pET -Shadoo 6, utilizando un protocolo de choque térmico estándar. Para esta mezcla 1 a 5 l de la plásmido (típicamente 10 a 100 pgng) en 50 l de las bacterias en un tubo. Realice el choque térmico a 42 ° C durante 45 segundos.
- Cultura bacterias transformadas a 37 ° C hasta 600 A de 0,5 a 0,7 en un medio Luria-Bertani suplementado con 40 mg / ml de kanamicina (Figura 1B).
- Inducir Shadoo expresión O / N mediante la adición de 240 mg / ml de isopropil-β-D-tiogalactopiranósido, ITPG, a la cultura. Cultura de las bacterias bajo agitación a 150 rpm a 37 ° C. Nota: Por lo general, la cultura bacteriana alcanza un 600 de 3 O / N.
Nota: Transformado bacterias pueden ser almacenadas a -80 ° C para la futura producción de proteínas. Para ello, agregue el 10% -50% (v / v) de glicerol estéril en alícuotas de cultivo bacteriano y congelar a -80 ° C.

Figura 1. Esquema de Shadoo construcción de expresión, las bacterias transformación y la inducción de la expresión Shadoo (como se describe en el paso 1). (A) El constructo de expresión para Shadoo codifica una etiqueta de hexahistidina fusionado al extremo N-terminal de la proteína, Nt. El esquema muestra que la proteína Shadoo contiene repeticiones básicos arginina / glicina (cilindro verde), un dominio hidrófobo (HD), y un único sitio de N-glicosilación (CHO), seguido de un terminal C (Ct). (B) pET-28-Su 6 -Shadoo plásmido se transforma en bacterias SoluBL21 por choque térmico de 45 segundos a 42 ° C. Bacterias transformadas se sembraron en agar selectivo que contenía 40 mg / ml de kanamicina. Una sola colonia de la placa se utiliza para inocular un cultivo en un matraz de agitador de 1.000-3.000 ml. La expresión de la proteína recombinante se indujo con IPTG 1 mM. Por favor haga clic aquí para ver una versión más grande de esta figura.
2. Purificación de Shadoo proteínas por baja presión cromatografía líquida (LPLC)
- La lisis celular e Inconclusión preparación cuerpos
- Recoger la suspensión bacteriana y la transfiere en tubos de centrifugación (Figura 2).
- Giran a 2500 xg, durante 20 min a 4 ° C. Eliminar el sobrenadante por decantación y pellets de resuspender en 15 ml de tampón (Tris 50 mM, EDTA 10 mM, pH 8 con cóctel anti-proteasa) para cada pellet derivado de 500 ml de cultivo celular.
Nota: Las bolitas resuspendidas pueden ser congelados y almacenados a -20 ° C durante varios días. La congelación facilita la lisis de células bacterianas. - Compruebe la sobre-expresión de la proteína recombinante utilizando bacterias crudo gránulos mediante análisis de SDS-PAGE y tinción de Coomassie.
- Para ello, de centrífuga de 1 ml de cultivo bacteriano para sedimentar las células. Añadir 100 l solución de Laemmli de desnaturalización (mM Tris-HCl 277,8, pH 6,8, 4,4% de SDS, 44,4% w / v de glicerol, 0,02% azul de bromofenol) para sedimentar y se calienta a 100 ° C durante 5 min.
- Cargar 10 l de la muestra a un gel de acrilamida al 12%, ejecute SDS-PAGE y visualize separa proteínas por tinción de Coomassie.
- Añadir Triton X-100 hasta una concentración final 0,5% al sedimento resuspendido completamente a partir de 2.1.2).
- Se incuba la suspensión en baño de agua a 37 ° C durante 30 min.
- Sonicar la suspensión durante 5 min a 6-12 micras amplitud pico a pico en agua enfriada con hielo para lisar células bacterianas.
- Transferencia sonicó la suspensión en 50 ml tubos de centrifugación limpio.
- Tubos centrífuga a 15.000 xg durante 15 min a 4 ° C.
- Eliminar el sobrenadante por decantación y añadir 5 ml de tampón de unión (20 mM de tampón Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea, 5 mM imidazol) por tubo.
- Colocar los tubos en la rotación de la rueda O / N para volver a suspender por completo las proteínas a partir de cuerpos de inclusión.
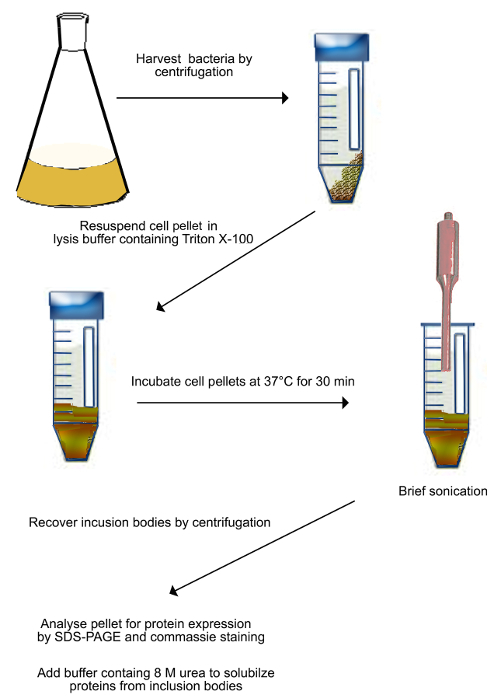
Figura 2. Esquema de purificación de inclusióncuerpos (como se describe en el paso 2.1). transformadas bacterias que expresan Shadoo se recogen por centrifugación y se resuspendieron en el tampón de lisis. La suspensión se incubó a 37 ° C durante 30 min, y luego se sometió a ultrasonidos para romper mecánicamente las células bacterianas. La sonicación se lleva a cabo en hielo para evitar el calentamiento de la muestra. Células rotas se recogieron por centrifugación y se resuspendieron en un tampón que contiene urea 8 M para solubilizar proteínas a partir de cuerpos de inclusión. Por favor haga clic aquí para ver una versión más grande de esta figura. - Cromatografía de Ni 2+ -affinity
Nota: La cromatografía se realiza en un sistema de FPLC usando Ni2 + -charged, columna quelante de 5 ml de Sepharose (Figura 3A). Todas las soluciones se cargan en la columna con un caudal de 1 ml / min.- Inyectar 10 ml de solución 4 agua 0,2 M NiSO _ en la columna de Sepharose con el fin de cargarlacon iones de Ni 2+.
- Equilibrar la columna cargada con Ni 2+ iones mediante la inyección de un 30-50 ml de tampón de unión que contiene la una baja concentración de imidazol (20 mM de tampón Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea, 5 mM imidazol).
- Cargar las proteínas de urea-solubilizado con un bucle de inyección de 50 ml.
- Recuperar la fracción de flujo a través lavando a fondo la columna con tampón de 10 ml bajo imidazol de unión (20 mM de tampón Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea, 5 mM imidazol) con el fin de eliminar un máximo de proteínas no unidas de la columna.
- Lavar la columna con 50-100 ml de tampón de lavado (20 mM Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea, 80 mM imidazol) para eluir las proteínas unidas de forma no específica a la columna.
- Aplicar un gradiente lineal 15 min de 80 a 800 mM imidazol en el tampón que contiene 20 mM Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea. Recoger fracciones de 1 ml durante la aplicación del gradiente. Nota: Las proteínas etiquetadas-Su se eluyen ingeniogradiente ha de tampón que contiene imidazol (usado como competidor).
- Tomar una alícuota de 8 l de cada fracción y añadir 4 l solución de desnaturalización 4x Laemmli (ver 2.1.3.1). Soluciones de calor a 100 ° C durante 5 min.
- Cargar 10 l de tamaño proteína marcadora y 10 l de cada muestra en el gel de acrilamida al 12%, correr SDS-PAGE y visualizar proteínas separadas por tinción de Coomassie. Nota: El peso molecular calculado de Shadoo formar su secuencia de aminoácidos es 12,243.2 Da.
- Piscina todas las fracciones que contienen Shadoo.
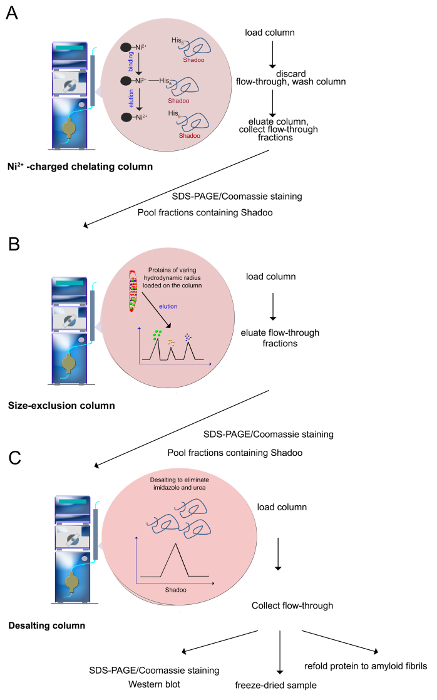
Figura 3. procedimiento de purificación (tal como se describe en los pasos 2.2-2.4). (A) Ni 2+ iones pueden ser coordinados por los residuos de histidina, lo que permite la purificación selectiva de proteínas polihistidina-etiquetado. El imidazol se utiliza para eluir la proteína, a través desu capacidad de coordinar con el 2+ -ion Ni y desplazar la proteína unida. (B) Las proteínas de diferente radio hidrodinámico que fueron retenidos en el Ni2 + -columna están separados en una columna de exclusión de tamaño. Las proteínas se eluyen de la más grande a los más pequeños. (C) Antes de su uso, las fracciones que contenían Shadoo se reúnen y se desalaron para eliminar la urea, imidazol y sales. Ensayos de control de calidad incluyen SDS-PAGE / tinción Coomassie, Western Blot. Purificada Shadoo puede ser liofilizado. Liofilizado Shadoo es estable durante meses cuando se almacena a -20 ° C. Por favor haga clic aquí para ver una versión más grande de esta figura. - Cromatografía de exclusión por tamaño
Nota: Una segunda etapa de purificación consiste en la cromatografía de exclusión por tamaño que separa la proteína Shadoo de las otras proteínas co-eluidos durante la primera etapa de purificación(Figura 3B).- Equilibrar la columna Superdex de 120 ml de volumen total del lecho mediante la inyección de 250 ml de 20 mM de tampón Tris-HCl, pH 7,4, NaCl 0,5 M, 8 M urea en la velocidad de flujo de 1,5 ml / min.
- Cargar fracciones reunidas que contienen Shadoo de 2.2.9 en la columna equilibrada. Recoger fracciones de 1 ml después de la volumen vacío de la columna de 40 ml sobre el flujo constante del tampón anterior.
- Tomar una alícuota de 10 l de cada fracción y le realizará un análisis de SDS-PAGE y tinción con Coomassie como se describió anteriormente para saber qué contienen fracciones Shadoo.
- Fracciones que contienen Shadoo piscina.
- Desalación
Nota: procedimiento de desalación elimina sales, imidazol y urea para obtener la proteína recombinante Shadoo en un tampón adecuado para posteriores estudios biofísicos y bioquímicos (Figura 3C). El mismo resultado se puede obtener muestras de diálisis contra el tampón.- Equilibrar una columna de desalación de 53 ml con 150 mlde 10 mM de tampón de acetato de amonio, pH 5, a un caudal de 5 ml / min.
- Cargar agruparon fracciones Shadoo en la columna. Tras la inyección del tampón de acetato de amonio recoger manualmente fracciones que muestran un aumento de la señal a 280 nm en la salida del detector de UV.
- Liofilizar la Shadoo desalada y almacenarla a -20 ° C.
- Disolver Shadoo liofilizado en un tampón adecuado, mM tampón tal como acetato de sodio 10, pH 5, antes de su uso.
Nota: La etiqueta de polihistidina se puede eliminar con enteroquinasa.
3. En Vitro Montaje de Shadoo en amiloide fibrillas en Physiological pH
Nota: Los agregados de proteínas Shadoo y espontáneamente forman fibrillas a pH ≥ 7 11.

Figura 4. Esquema de Shadoo replegamiento a las fibrillas de amiloide (como se descricama en el paso 3). purificada Shadoo convierte espontáneamente a las fibrillas de amiloide cuando se disuelve en tampón de pH ≥ 7. En una solución ácida, Shadoo convierte a las fibrillas de amiloide tras la incubación con M Gdn-HCl 1 mientras se agitaba. Ensayos de control de calidad incluyen la microscopía electrónica y tinción ThT. Por favor haga clic aquí para ver una versión más grande de esta figura.
- Diluir Shadoo a una concentración final de 2 mM en un tampón mM Tris-HCl 20, pH 7,4. Medir la concentración midiendo la densidad óptica de la solución a 280 nm y usando el coeficiente de extinción deducida a partir de la composición de aminoácidos Shadoo de 20.970.
- Incubar 500 l de la solución en un tubo de plástico cónico a 4 ° C durante un par de semanas, para permitir la conversión espontánea de proteínas a las estructuras amiloides (Figura 4).
- Agregar recién preparada tioflavina T (ThT) a un conce definitivantration de 10 micras a una alícuota de 100 l de la solución Shadoo. Incubar la solución durante 5 min a RT. Medida de emisión de fluorescencia 460-520 nm usando una longitud de onda de excitación de 435 nm para verificar la presencia de estructuras amiloides 17.
4. En Vitro Montaje de Shadoo de amiloide fibrillas en ácida pH
- Diluir Shadoo en 1 M GdnHCl, 20 mM de tampón Na-acetato, pH 5 a una concentración final de 2 mM.
- Incubar 500 l de esta solución en un tubo cónico con agitación continua a 37 ° C, durante 3-7 días.
- Compruebe la presencia de fibrillas amiloides añadiendo ThT a una alícuota de la solución como en 3.3.
- Dializar suspensión obtenida contra 10 mM de tampón Na-acetato, pH 5,0, para purificar las fibrillas de amiloide Shadoo.
- Guarde fibrillas purificados a 4 ° C.

Figura 5. Los resultados representativos. Las fracciones proteicas eluidas de Ni 2+ -charged columna quelante de (A) y la columna de exclusión por tamaño (B) se analizaron mediante SDS-PAGE y tinción de Coomassie. Identificación de Shadoo purificado se evaluó a través de anticuerpo anti-Shadoo SPRN (C). Por favor, haga clic aquí para ver una versión más grande de esta figura.
Resultados
Acerca de 5-10 mg de proteína purificada Shadoo se obtiene por litro de cultivo bacteriano. Se necesita una purificación de dos pasos para obtener Shadoo recombinante de alta pureza. El primer paso se lleva a cabo mediante cromatografía de afinidad con la columna -charged una de Ni 2+ que conserva sus-proteínas etiquetadas. Las fracciones eluidas se sometieron a SDS-PAGE y se tiñeron con azul brillante de Coomassie (Figura 5A). Las fracciones que contenían la proteína Shadoo se reúnen...
Discusión
Se presenta un protocolo fácil y eficiente tanto para la expresión y purificación de una gran cantidad de proteína Shadoo recombinante de ratón. El método descrito permite la solubilización y purificación de (Su 6) proteína recombinante Shadoo-etiquetados éxito. Es importante, como se ha señalado en el título, que cuando se expresa en una bacteria procariota E. coli Shadoo se acumula en cuerpos de inclusión. Antes de la purificación Shadoo, las bacterias tienen que ser lisadas por sonic...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
The authors would like to acknowledge Natalie Doude and David Westerway (University of Alberta) for providing us Sho cDNA, Christophe Chevalier (INRA) for plasmid construction, Michel Brémont (INRA) for providing us BL21Sol bacteria; Christine Longin (INRA) for TEM assistance and Edith Pajot-Augy (INRA) for comments and proofreading.
Materiales
| Name | Company | Catalog Number | Comments |
| AKTA FPLC | GE, Amersham | # 1363 | |
| HiLoad 16/60 Superdex | GE Healthcare | 17-1069-01 | |
| HiTrap IMAC | GE Healthcare | GE17-0920-05 | |
| HiPrep26/10 Desalting column | GE Healthcare | GE17-5087-01 | |
| SoluBL21 Competent E. coli | Genlantis | C700200 | |
| pET-28b+ | Novogen | 69865-3 | |
| IPTG | Sigma-Aldrich | I6758 | |
| LB-Broth medium | Sigma-Aldrich | L3022 | |
| Eppendorf Biophotometar | Eppendorf | 550507804 | |
| Trito X-100 | Life Technologies | 85111 | |
| NiSO4 | Sigma-Aldrich | 656895 | |
| EDTA | Sigma-Aldrich | E6758 | |
| Tris-HCl | Sigma-Aldrich | RES3098T | |
| Protease Inhibitor Cocktail Tablets | Roche | 4693116001 | |
| NaCl | Sigma-Aldrich | 746398 | |
| Imidazol | Sigma-Aldrich | I5513 | |
| Gdn-HCl | Sigma-Aldrich | G4505 | |
| protein size marker | Thermo Fisher Scientific | 26620 |
Referencias
- Biasini, E., Turnbaugh, J. A., Unterberger, U., Harris, D. A. Prion protein at the crossroads of physiology and disease. Trends in Neurosciences. 35, 92-103 (2012).
- Colby, D. W., Prusiner, S. B. De novo generation of prion strains. Nature Reviews. Microbiology. 9, 771-777 (2011).
- Chiti, F., Dobson, C. M. Protein misfolding, functional amyloid, and human disease. Annual Review of Biochemistry. 75, 333-366 (2006).
- Nelson, R., et al. Structure of the cross-beta spine of amyloid-like fibrils. Nature. 435, 773-778 (2005).
- Murphy, R. M. Kinetics of amyloid formation and membrane interaction with amyloidogenic proteins. Biochimica et Biophysica Acta. 1768, 1923-1934 (2007).
- Steunou, S., Chich, J. F., Rezaei, H., Vidic, J. Biosensing of lipid-prion interactions: insights on charge effect, Cu(II)-ions binding and prion oligomerization. Biosensors & Bioelectronics. 26, 1399-1406 (2010).
- Watts, J. C., Westaway, D. The prion protein family: diversity, rivalry, and dysfunction. Biochimica et Biophysica Acta. 1772, 654-672 (2007).
- Westaway, D., Daude, N., Wohlgemuth, S., Harrison, P. The PrP-like proteins Shadoo and Doppel. Topics in Current Chemistry. 305, 225-256 (2011).
- Baillod, P., Garrec, J., Tavernelli, I., Rothlisberger, U. Prion versus doppel protein misfolding: new insights from replica-exchange molecular dynamics simulations. Biochemistry. 52, 8518-8526 (2013).
- Daude, N., et al. Wild-type Shadoo proteins convert to amyloid-like forms under native conditions. Journal of Neurochemistry. 113, 92-104 (2010).
- Li, Q., et al. Shadoo binds lipid membranes and undergoes aggregation and fibrillization. Biochemical and Biophysical Research Communications. 438, 519-525 (2013).
- Watts, J. C., et al. The CNS glycoprotein Shadoo has PrP(C)-like protective properties and displays reduced levels in prion infections. The EMBO Journal. 26, 4038-4050 (2007).
- Watts, J. C., et al. Protease-resistant prions selectively decrease Shadoo protein. PLoS Pathogens. 7, e1002382 (2011).
- Westaway, D., et al. Down-regulation of Shadoo in prion infections traces a pre-clinical event inversely related to PrP(Sc) accumulation. PLoS Pathogens. 7, e1002391 (2011).
- Chevalier, C., et al. PB1-F2 influenza A virus protein adopts a beta-sheet conformation and forms amyloid fibers in membrane environments. The Journal of Biological Chemistry. 285, 13233-13243 (2010).
- Tarus, B., et al. Oligomerization paths of the nucleoprotein of influenza A virus. Biochimie. 94, 776-785 (2012).
- Biancalana, M., Koide, S. Molecular mechanism of Thioflavin-T binding to amyloid fibrils. Biochimica et Biophysica Acta. 1804, 1405-1412 (2010).
- Minic, J., Sautel, M., Salesse, R., Pajot-Augy, E. Yeast system as a screening tool for pharmacological assessment of g protein coupled receptors. Current Medicinal Chemistry. 12, 961-969 (2005).
- Schein, C. H. A cool way to make proteins. Nature Biotechnology. 22, 826-827 (2004).
- Sorensen, H. P., Mortensen, K. K. Advanced genetic strategies for recombinant protein expression in Escherichia coli. Journal of Biotechnology. 115, 113-128 (2005).
- Zhang, J., et al. Disruption of glycosylation enhances ubiquitin-mediated proteasomal degradation of Shadoo in Scrapie-infected rodents and cultured cells. Molecular Neurobiology. 49, 1373-1384 (2014).
- Englund, P. T. The structure and biosynthesis of glycosyl phosphatidylinositol protein anchors. Annual Review of Biochemistry. 62, 121-138 (1993).
- Puig, B., Altmeppen, H., Glatzel, M. The GPI-anchoring of PrP: implications in sorting and pathogenesis. Prion. 8, 11-18 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados