Method Article
Proyectos, Embalaje y Entrega de alto título de CRISPR Retro y lentivirus vía estereotáxica Inyección
En este artículo
Resumen
El sistema / Cas9 CRISPR ofrece el potencial para que la edición del genoma específico accesible y asequible para la comunidad científica. Este protocolo está destinado a demostrar cómo crear virus que Knockout un gen de interés utilizando el sistema / Cas9 CRISPR y, a continuación, inyectarlos estereotáxica en el cerebro del ratón adulto.
Resumen
Replication defective lentiviruses or retroviruses are capable of stably integrating transgenes into the genome of an infected host cell. This technique has been widely used to encode fluorescent proteins, opto- or chemo-genetic controllers of cell activity, or heterologous expression of human genes in model organisms. These viruses have also successfully been used to deliver recombinases to relevant target sites in transgenic animals, or even deliver small hairpin or micro RNAs in order to manipulate gene expression. While these techniques have been fruitful, they rely on transgenic animals (recombinases) or frequently lack high efficacy and specificity (shRNA/miRNA). In contrast, the CRISPR/Cas system uses an exogenous Cas nuclease which targets specific sites in an organism's genome via an exogenous guide RNA in order to induce double stranded breaks in DNA. These breaks are then repaired by non-homologous end joining (NHEJ), producing insertion and deletion (indel) mutations that can result in deleterious missense or nonsense mutations. This manuscript provides detailed methods for the design, production, injection, and validation of single lenti/retro virus particles that can stably transduce neurons to express a fluorescent reporter, Cas9, and sgRNAs to knockout genes in a model organism.
Introducción
Para el estudio de la base de la fisiología normal y patología de la enfermedad, existe una necesidad de manipular con precisión la expresión génica en organismos modelo. Para organismos modelo de mamíferos, esta se centra en gran medida en la creación y desarrollo de ratones transgénicos en el que un elemento genético de interés está flanqueado por sitios reconocidos por una recombinasa. Esto puede resultar en una manipulación específica del sitio de estos genes flanqueados. Si bien esto ha sido una estrategia exitosa, es tiempo y requiere muchos recursos; por ejemplo, la creación de un animal transgénico de triple que expresara un gen floxed, recombinasa Cre, y un gen indicador Cre requiere múltiples apareamientos y validación. En contraste, la inyección estereotáxica de la replicación de partículas virales defectuosas que codifican una proteína fluorescente y la recombinasa en un animal gen floxed no requiere la genotipificación complejo o estrategias de reproducción 1. Además, si una proteína fluorescente y expresar Cre virus es co-inyectados con un segundo virus encoding una proteína fluorescente diferente, a continuación, esto proporciona un control dentro de los tejidos para la manipulación genética dirigida. Si bien esta estrategia todavía requiere el uso de animales knock-in, las estrategias de ARN basado mediadas por virus eludir la necesidad de animales transgénicos. Por ejemplo, la inyección estereotáxica de virus deficientes en replicación que codifican una proteína fluorescente y un ARN de horquilla corta (ARNhc) puede utilizar la maquinaria de RNAi endógeno de la célula para dar lugar a una potente reducción de la transcripción de un gen de interés. Sin embargo, las estrategias de shRNA producen genes sutil derribos menudo resultan en fenotipos celulares modestas 2. Mientras que un knock-down puede ser fisiológicamente más relevantes para la disfunción gen heterocigoto, su robustez disminuido en comparación con un knock-out no es lo ideal para el descubrimiento de nuevos genes fenotípica.
Una tercera técnica que ha surgido recientemente, el CRISPR (agrupado regularmente espaciadas Repita palindrómico corto) / Cas9 (CRISPR-asociadoproteína 9) del sistema, se basa en la expresión tanto de un pequeño ARN exógeno y una enzima de corte de ADN. El sistema / Cas9 CRISPR es una adaptación del sistema inmunológico procariotas que se desarrolló un método de identificación de extranjero, la invasión de ADN de los virus y la orientación para la degradación a través de la enzima Cas9 3,4. Esta poderosa técnica de edición genoma se puede usar para la creación de deleciones específicas, inserciones y mutaciones; y el siguiente protocolo describirá cómo hacer deleciones en un gen de interés con el fin de knockout su expresión in vivo. La enzima Cas9 debe ser expresada con una guía de ARN homólogo a la región de interés y contigua con un ARN andamio. Knockout de un gen utilizando esta técnica requiere la orientación Cas9 a una región específica en el genoma utilizando guía RNAs sintéticos (sgRNA), y la inducción de roturas de doble cadena (DSBs) en un sitio de interés. Estos DSBs son entonces reparados por la maquinaria de la célula de reparación de endógeno a través de extremos no homólogos de unión (NHEJ), que leanuncio a indeles que pueden producir pérdida de sentido o sin sentido mutaciones y por lo tanto pueden crear una pérdida de la expresión de la proteína funcional 5. Debido a que este sistema produce alteraciones genómicas, que sólo requiere la expresión transitoria de la Cas9 y sgRNA. Sin embargo, es deseable para un indicador fluorescente estable para identificar las células y su progenie manipulados de esta manera.
Lenti- y retrovirus tienen la ventaja de integrar de forma estable de ADN de interés en las células huésped que mantienen la expresión a largo plazo y se transmiten a las células hijas durante la mitosis. Este protocolo describe el diseño y la producción de dos tipos de replicación defectuosa, los retrovirus alto título: el virus de la inmunodeficiencia humana partículas lentivirales derivados (lentivirus) y los basados en virus murina Maloney Leucemia (retrovirus). Mientras tanto de estos virus son capaces de soportar grandes expresando estable de los transgenes, las partículas retrovirales sólo se integran en el genoma dula división celular anillo con la degradación de la envoltura nuclear, y por lo tanto se puede utilizar como una herramienta para etiquetar y células fecha de nacimiento 6. Mientras que los lentivirus tienen una reputación de ser relativamente baja titulación 7, esta metodología, incluyendo el uso de la cafeína durante la recogida de 8 viral, habitualmente produce títulos de 10 y 9 10 10 partículas / ml. Otra ventaja de lenti- y los retrovirus es la tolerancia en caso de grandes insertos. La siguiente colección de protocolos describe el procedimiento para diseñar un lenti o retrovirus que codifica un informador fluorescente, sgRNAs, y Cas9 para utilizar el sistema / Cas9 CRISPR para modificar ADN, así como expresar una proteína fluorescente.
Neurocirugía estereotáxica ratón es un método valioso para la inyección de virus in vivo para estudiar la morfología, función y conectividad de las neuronas infectadas. La infección viral en las neuronas se puede utilizar para manipular los niveles de expresión durante un período prolongado de time, como en todo el desarrollo y la expresión pueden ser controlados con precisión mediante el uso de diversos sistemas inducibles de drogas y expresión específica impulsada Cre. Este protocolo particular explica cómo inyectar un virus que expresa un sgRNA y Cas9 a octavos de final de un gen de interés en el cerebro de un ratón adulto. Los ratones se recuperan muy rápidamente de este procedimiento y la expresión del transgén viral se puede ver dentro de las 48 horas después de la inyección. Sin embargo, la expresión fluoróforo parece aumentar en el transcurso de semanas que resultan en niveles máximos cerca de 3 semanas después de la infección. Los ratones que se someten a inyección estereotáxica viral se pueden utilizar para el comportamiento, la electrofisiología, o estudios morfológicos. En general, el propósito de estos procedimientos es demostrar cómo un gen knockout en el cerebro de ratón adulto utilizando cirugía estereotáxica y un virus que expresa un sgRNA específico y Cas9.
Protocolo
Declaración de Ética: Todos los protocolos fueron aprobados por el Dartmouth Bioseguridad Institucional e Institucional de Cuidado de Animales y el empleo juntas de revisión.
1. Protocolo para el Diseño de una Guía de Strand (sgRNA) de CRISPR / Cas9 Retrovirus
NOTA: Hay muchos sitios web sin ánimo de lucro que se pueden utilizar para generar sgRNAs para dirigirse a un gen de interés (https://benchling.com/ y http://crispr.mit.edu/). El objetivo de este protocolo es crear y activar los oligonucleótidos de cadena simple de un proveedor comercial que se hibridan entre sí. Este oligo recocida se ligó en el vector de transferencia PXL. Ver vídeo suplementario 1 para un ejemplo de diseño de sgRNAs utilizando Benchling.
- Use un sitio web dedicado a diseñar sgRNAs.
NOTA: Por ejemplo, insertando una secuencia de codificación / exonic desde cerca del comienzo del gen de interés en el sitio web generará un sgRNA 20 nucleótidos. Utilice la secuencia de nucleótidos sgRNA 20 para diseñar el sentido y antisentido oligos que se ordenó para crear el sgRNA. - Después de generar el sgRNA, copiar esta secuencia en un procesador de textos. Añadir una G (guanina) hasta el comienzo de la secuencia de nucleótidos sgRNA 20 en el documento si no ya comienza con uno (es decir, G-20 secuencia de nucleótidos sgRNA). Esto es necesario para asegurar una buena transcripción fuera el promotor U6.
- Documentar el complemento inverso de esta secuencia de nucleótidos ahora 20-21. Para el oligo sentido, añadir "CACC" al extremo 5 'de la secuencia en el documento (es decir, CACC-G-20 secuencia de nucleótidos sgRNA). Este saliente se utiliza para ligar la secuencia en el vector PXL.
- Para el oligo antisentido, añadir AAAC al extremo 5 '. Utilice esta voladizo para ligar la secuencia en el vector PXL.
- Obtener los oligonucleótidos sentido y antisentido. Después de recibir los oligos, hacer 100 M existencias utilizando DNasa libre de agua. Mezclar 10 l cada uno de los 100 M oligos sentido y antisentido con 4 μl de 10x tampón de ORC 2, y 16 l de agua.
NOTA: No necesitan purificación página o 5'phosphorylation (como los voladizos son BbsI extremos cohesivos no compatibles).- Trae 200 ml de agua a ebullición en un vaso de precipitados de 500 ml, entonces flotar el tubo que contiene esta mezcla en un soporte de espuma "flotador". Permitir que el agua se enfríe lentamente desde 95 ° C durante 2 horas a temperatura ambiente.
- Se diluye la mezcla-recocidos oligo ahora 1: 1.000 en agua estéril y utilizar inmediatamente en la siguiente ligadura o almacenar la reacción restante a -20 ° C.
- Recopilación PXL, un derivado PX330 vector, con la enzima de restricción BbsI a 37 ° C durante 2 hr. Utilice una reacción de 40 l que contiene 2 g de PXL, 4 l de 10x tampón de ORC 2, 1 l de agua y BBS1 equilibrio. Se somete el-PXL digerido a purificación en gel de rutina utilizando un kit comercial. Asegúrese de que el rendimiento es de ~ 8,5 kb producto.
- El uso de un kit de unión comercial, ligar 1 l de la 1: 1000-oligos hibridados con 50 ng de digerido y purificado en gel-PXL. Transformar el producto de ligación en la recombinación competente deficiente E. coli (NEB 5-alfa, la eficiencia subclonación). Se tamizan los transformantes para la presencia de la guía correcta mediante secuenciación de ADN de plásmido.
- Digerir el U6, hebra guía, y los elementos de andamio RNA (sgRNA) de PXL usando enzimas de restricción BstB1 y PAC1 y se liga en el esqueleto viral proteína-T2A-Cas9 fluorescente digerido con las mismas enzimas de restricción. Transformar el producto de ligación (NEB máxima eficiencia 5-alfa). Las proteínas T2A-Cas9 plásmidos columna vertebral viral fluorescentes son plásmidos baja copia y protocolos por lo tanto baja copia deben ser utilizados.
NOTA: Una segunda sgRNA se puede introducir de manera similar por la digestión PacI del plásmido PXL otra guía de cadena y se ligó en el Pac1 digerido y fosfatasa intestinal de ternera tratada columna vertebral viral ya que contiene la primera hebra guía. El tratamiento con fosfatasa del plásmido de transferencia viral ayudaráreducir el número de transformantes de la auto-ligación de los plásmidos que no contienen el segundo guía. - Secuencia verificar el plásmido y maxi final de preparación utilizando el kit maxi prep Nucleobond Xtra y empaquetarla en un virus utilizando la siguiente "Protocolo de retro / Lentiviral Producción - CaPO4 el método".
2. Preparar 293FT / 293GP células para la transfección (Retro / Lentiviral Producción - Método CaPO4)
- (Día 1) El rápido deshielo 1 vial de células por 10 cm de placa de cultivo celular en un baño de agua a 37 ° C. Para el envasado lentiviral, utilice 293FT células. Para empaquetamiento retroviral, utilizar células 293GP (gag / pol).
- Pipetear todas las células descongeladas del tubo de cryo en un tubo cónico de 15 ml y añadir 2 ml de pre-calentado CO 2 equilibrada-completa de Iscove medio de Dulbecco modificado.
- Centrifugar las células durante 5 minutos a 500 xg para sedimentar. Aspirar el sobrenadante y resuspender el sedimento celular en 10 ml de se haya completadocala de medio de Dulbecco modificado. Placa de las células en una placa de cultivo de células de 10 cm. Se incuban las células durante la noche a 37 ° C en un incubador de dióxido de carbono 5%.
- (Día 2) 24 hr después de la siembra, cambio de medio en la placa mediante la aspiración de los medios de comunicación existentes y la adición de 10 ml de pre-calentado medio modificado de Dulbecco de Iscove a la placa.
- (Día 3-4) 24-48 h después del cambio de medio y una vez que las células se vuelven confluentes, divide las células hasta una confluencia de 2,5-3,0 x 10 6 células / placa de 10 cm (placa).
- Para dividir las células, aspirar los medios y lavar la placa con 5 ml de PBS. Añadir 1 ml de 0,25% de tripsina a la placa y se incuba a 37 ° C hasta que las células se despegan de la placa. Añadir 0,5 ml de medio modificado de Dulbecco de Iscove para neutralizar la reacción de tripsina 0,25% y pipetear las células en un tubo de 1,5 ml.
- Girar las células a 500 xg durante 5 min. Resuspender las células en 1 ml de Iscove medio de Dulbecco modificado. diluir 1081; l de las células en 90 l de PBS. Contar las células usando un hemocitómetro o un contador de células automatizado. Re-placa de 2,5-3,0 x 10 6 células / placa con completa de Iscove medio de Dulbecco modificado.
Nota: Las celdas serán ~ 50% de confluencia 24-34 horas después de la siembra. Transfectar las células cuando son ~ 50% de confluencia.
3. (día 5) CaPO4 y transfección de partículas Viral Collection
- Cambie la hr medios 2 antes de la transfección mediante la aspiración de los medios de comunicación existentes y la adición de 10 ml de pre-calentado medio de Dulbecco modificado de Iscove. Asegúrese de que hay exactamente 10 ml de medios de comunicación en la placa de 10 cm.
- Preparar los reactivos de transfección durante 2 platos con 2, 5 ml tubos de poliestireno de fondo redondo. Etiquetar el primer tubo "ADN" y el segundo tubo "2X HBS". Ajustar la concentración de DNA a 1μg / l en Tris-EDTA a pH 7,4.
- Para los lentivirus, añada lentamente 20 l de vector de transferencia (la viral construcción de interés), 13 l de CMVdelta8.9, 9 l de VSV-G, 860 l de grado biología molecular H2O y 100 l de CaCl2 2,5 M al primer tubo ( "ADN"), mientras que tapping continuamente en el tubo para mezclar.
- Para los retrovirus, omitir el plásmido CMVdelta8.9. Añadir 20 l vector de transferencia, 15 l VSV-g, 860 l Biología Molecular Grado H 2 O, y 100 l 2.5 M CaCl 2 al tubo etiquetado como "DNA".
- Añadir 1 ml de solución salina tamponada con HEPES 2x (pH 7,0; este pH es absolutamente crítico) al tubo etiquetado como "2X HBS".
- Añadir lentamente el contenido 1 ml del tubo "ADN" al tubo "2X HBS", una gota a la vez. Continuamente presiona el tubo "2X HBS" con el dedo índice o medio tiempo que añade el contenido del tubo de "ADN". Observe aparentes CaPO4 vesículas después de cada gota. Incubar el tubo en la oscuridad durante 30 min a temperatura ambiente.
- Añadir 1 ml de la transfección en las gotas lentas a cada placa de células 10 cm, a continuación, se incuba durante la noche a 37 ° C.
- (Día 6) Sustituir los medios de comunicación con 8 ml de Modified Medium + 0,5% de FBS de Dulbecco de Iscove y 40 mg de cafeína / 100 ml de medio. Si el vector de transferencia contiene un marcador fluorescente, a continuación, la expresión fluoróforo indica una transfección con éxito.
- (Día 7) Recoger el medio que contiene las partículas virales mediante el uso de una 10 ml pipeta serológica y dispensar en un tubo cónico de 50 ml. Almacenar el tubo cónico de 50 ml a 4 ° C. Añadir 8 ml de 0,5% FBS a cada placa.
- (Día 8) Una vez más, recoger los medios de comunicación que contiene las partículas virales y se combinan con la cosecha del día anterior en el tubo cónico de 50 ml.
4. Concentración y purificación del virus
- Hacer una solución de 5x de polietileno glicol 6000 mediante la adición de 40% de polietilenglicol 6000 y 1,5 M NaCl a ddH 2 O. Autoclave la solución deel ciclo de líquido durante 45 minutos a 121 ° C. Poco a poco mezclar la solución durante el enfriamiento.
NOTA: La solución se iniciará nublado y luego se aclarará cuando se enfría. - Centrifugar el tubo cónico de 50 ml que contiene ambas colecciones de sobrenadante viral a 2.000 xg durante 10 min con el fin de sedimentar el material insoluble. Purificar los medios de comunicación viral mediante la filtración a través de un filtro de jeringa de 0,45 micras de unión baja en proteínas (PES) o PVDF.
- Añadir polietilenglicol 5x 6000 solución a medios de comunicación (La concentración final debe ser de 8% de polietilenglicol 6.000 y 0,3 M NaCl). Mezclar invirtiendo los tubos varias veces (no las vórtice). Incubar la solución de glicol de polietileno que contiene viral a 4 ° C durante 12 horas o más, la remezcla de vez en cuando.
- (Día 9) Centrifugar la solución de glicol de polietileno que contiene viral en 2500 xg durante 45 min. Retirar y desechar el sobrenadante y girar de nuevo durante 2 minutos. Una vez más, retirar y desechar el sobrenadante.
- Resuspender el precipitado mediante la adición de 320 l desolución salina estéril tamponada con fosfato (los 320 l es 1/100 del volumen original del medio que contiene virus recogidos) y se incuba durante la noche a 4 ° C. Opcionalmente, resuspender el precipitado a temperatura ambiente en un agitador durante 30 min.
- Después de volver a suspender el sedimento, el virus de la alícuota (5 a 10 l por 0,5 ml de tubo) y congelar alícuotas a -80 ° C (congelación descongelación o el almacenamiento a 4 ° C reducirá drásticamente el título).
- Título de los virus utilizando protocolos estándar, es decir, llevar a cabo una serie de diluciones en una placa de 6 pocillos de células HEK293T y contar manualmente las colonias fluorescentes 48 horas más tarde.
5. Prueba de Eficacia del Virus de CRISPR
NOTA: los clones de secuencia utilizando los siguientes pasos para la prueba de producción de doble filamento se rompe reparados por NHEJ utilizando las células Neuro2A ratón. Esto tiene la ventaja sobre los ensayos de regimen en que se puede utilizar para determinar el porcentaje de células que se han modificado y la naturalezade los indeles resultantes de NHEJ.
- Escudo tales como matrigel una placa de cultivo de células 3,5 cm con una mezcla de proteína gelatinosa diluyó 1:50 en Iscove medio de Dulbecco modificado e incubar durante 30 min a 37 ° C. Aspirar la mezcla de proteína gelatinosa y la placa de las células Neuro2A.
- Después de que las células Neuro2A alcanzan 50% de confluencia, añadir el Lentivirus CRISPR o Retrovirus a una multiplicidad de infección de 10 (es decir, 10 partículas virales por célula).
NOTA: células Neuro2A no son como amigable a la infección como son células HEK293, por lo tanto, una sola infección no da lugar a 100% de las células infectadas con el lentivirus. Además, el retrovirus sólo infecta a células que se dividen y por lo tanto también no resultará en infección 100%. Con el fin de conseguir una velocidad de cerca de 100% de la infección, la adición de virus se puede repetir en varios días, la división de las células, según sea necesario. Alternativamente, las células positivas fluorescentes se pueden aislar usando activada por fluorescencia de células de clasificación. The forma más eficaz de lograr una tasa de infección del 100% con un lentivirus es llevar a cabo una sola infección seguido de aislamiento FACS. Para un retrovirus, lleve a cabo 4 rondas de infección de 24 horas de diferencia y seguir por el aislamiento FACS para asegurar 100% de infección. - Después de infectar las células durante 1 semana, expandir las células a cerca de la confluencia en una placa de 10 cm, aspirar los medios de comunicación, y lavar la placa con 5 ml de solución salina tamponada con fosfato. Añadir 1 ml de 0,25% de tripsina y se incuba a 37 ° C hasta que las células se despegan de la placa. Añadir 0,5 ml de medio modificado de Dulbecco de Iscove para neutralizar la reacción de tripsina 0,25% y pipetear las células en un tubo de 1,5 ml y sedimentar las células a 500 xg durante 5 min.
- Para aislar el ADN, añadir 100 l de hidróxido de potasio 50 mM, volver a suspender las células, y se incuba a 95 ° C durante 5 min. Neutralizar la solución con 10 l de 1 M Tris, pH = 8,0.
- Amplificar por PCR la región que flanquea la región genómica dirigida por el CRISPR sgRNAutilizando ~ 300 ng del ADN aislado en la etapa 5.5 usando una PCR de alta fidelidad mezcla maestra 4.
- Ejecutar la reacción de PCR en un gel de agarosa al 2,5% y gel de purificar el fragmento de tamaño apropiado utilizando un kit de purificación de gel, tal como un kit de recuperación de ADN en gel. Ligar en un vector de clonación de PCR como pGEM-T fácil y se transforman en células competentes 9.
- 24 horas más tarde, se añaden 2 ml de caldo LB en un tubos de 15 ml de fondo redondo, así como el antibiótico apropiado (ampicilina, neomicina, etc.). Inocular el tubo con una colonia individual mediante el uso de una punta de pipeta estéril o bucle para seleccionar una única colonia de la placa de LB-. Expandir y hacer crecer la colonia agitando el tubo a 250 rpm y 37 ° C durante 24 hr. Repita para el mayor número de colonias como se desee.
- El uso de un kit de mini-prep, aislar el ADN de la E. coli y la secuencia con los cebadores diseñados para amplificar la zona del gen diana por el sgRNA el fin de analizar la región Cas9 dirigida del genoma del ratón.
6. Protocolo de inyección estereotáxica para el ratón adulto
- Prepararse para la cirugía
NOTA: Es importante mantener las condiciones estériles durante cirugías de supervivencia. Esto se logra en este protocolo por el calor de esterilización de las herramientas de cirugía, usando betadine para esterilizar el lugar de la inyección, y la adición de pomada antibiótica al sitio de la incisión después de que se cierre. También es importante la utilización de guantes estériles, así como un fondo esterilizado, área de la cirugía dedicado.- Equipo para cirugía de calor esterilizan antes de su uso, ya sea en autoclave o utilizando un esterilizador de cuentas en caliente. Preparar cámara de recuperación y el área de la cirugía mediante la activación de almohadillas de calor para mantener la temperatura corporal durante la cirugía y la recuperación. Montar la broca utilizada para crear agujeros en el cráneo para inyección.
- Carga de 4 l de virus en aguja de inyección por la retirada de la virus directamente desde el tubo de PCR estéril. Brevemente quitar la alícuota viral de hielo. Mantener el virusen la jeringa a temperatura ambiente durante no más de 1 hora antes de la inyección.
7. La inyección estereotáxica
- Confirmar que la ventilación de la sala de cirugía está abierto para asegurar el flujo de aire adecuado. Preparar el ratón para la cirugía anestesiando con 4% de isoflurano en una cámara de inducción. Después de la anestesia, afeitarse la cabeza para preparar el sitio de la inyección.
- Coloque el ratón en el instrumento estereotáxico utilizando pinzas para mover la lengua hacia abajo y hacia un lado. Inserte la barra de bocado en la boca hasta que los dientes caen en la ranura, a continuación, fije las barras de oído. Asegúrese de que el cuerpo del ratón está sobre el cojín eléctrico y la nariz se encuentra en el cono de la nariz. Directo 4% de isoflurano en el cono de la nariz. Confirmar anestesia pellizcando el pie con las pinzas y la garantía de que no hay reflejo de sobresalto.
- Aplicar las lágrimas artificiales para lubricar los ojos. Finalizar la preparación del sitio de la inyección con un hisopado de la cabeza afeitada con la alternancia de ciclos de POVIdone-yodo y lidocaína.
- El uso de un bisturí, corte pequeña incisión a lo largo de centro de cuero cabelludo. Se seca el cráneo con un hisopo de peróxido de hidrógeno y si es necesario para ayudar a visualizar bregma.
- El uso de un microscopio de disección, localizar bregma en el cráneo y coloque la punta de la broca en el bregma. Cero (o registro) las coordenadas estereotácticas digitales en los planos x, y, z.
- Con el fin de garantizar que la cabeza es el nivel en el rostral a caudal eje Y, coloque la broca en lambda y nivelar la cabeza de modo que la coordenada z es aproximadamente igual en ambos bregma y lambda.
- Con el fin de asegurar que la cabeza es de nivel en el eje x, colocar la broca para y = 1/2 lambda. Asegúrese de que la coordenada z es igual a 1 mm en cada lado (x = +/- 1 mm) de la sutura sagital. Ajuste el cráneo si hay una diferencia en la coordenada z en 1 mm a la izquierda y derecha de lambda.
- Coloque el taladro sobre las coordenadas deseadas. Para el giro dentado, utilice la coordenadasy = -1,9 desde el bregma y x = +/- 1,1. Antes de perforar, rebajar el isoflurano al 2%, luego poco a poco y con cuidado, perfore a través del cráneo.
- Después de perforar todos los agujeros, colocar la jeringa llena en el instrumento estereotáxico. Centrar visualmente la jeringa sobre el agujero y poner a cero la coordenada z en el cráneo.
- baje lentamente la jeringa a lo más profundo de profundidad z. Para el giro dentado, las profundidades z son -2.5, -2.4, -2.3 y. Comience inyección a una velocidad de 0,25 l / min usando un inyector estereotáxica.
- Después de la inyección se ha completado con el menor profundidad Z, espere 1 minuto, y después subir al siguiente coordinar y comenzar la inyección de nuevo. Continuar este patrón hasta que se inyectan todas las coordenadas z de la inyección. Espere 2 min antes de retirar la jeringa después de la última inyección.
NOTA: No se observaron efectos adversos se han observado en el tejido mediante la inyección de hasta 2 l de virus por el hemisferio en el cerebro del ratón.
- Después de la inyección se ha completado con el menor profundidad Z, espere 1 minuto, y después subir al siguiente coordinar y comenzar la inyección de nuevo. Continuar este patrón hasta que se inyectan todas las coordenadas z de la inyección. Espere 2 min antes de retirar la jeringa después de la última inyección.
- la inyección de repetición para otros agujeros.
- Después de la inyección, retire el ratón desde el instrumento estereotáxico y suturar el cuero cabelludo con 6-0 suturas de seda. Aplicar lidocaína y una crema anti-bacteriana a la herida.
- Inyectar 0,8-1,0 ml de solución salina + ketoprofeno (3-5 mg / kg) por vía IP para controlar el dolor. A continuación, coloque el ratón en la cámara de recuperación climatizada. No dejar al animal sin vigilancia hasta que se haya recuperado el conocimiento suficiente para mantener decúbito esternal. No devolver el animal a la compañía de otros animales hasta que se haya recuperado por completo.
- En los días siguientes a la cirugía, se pesa el ratón diario y proporcionar alimentos y golosinas blandas. Compruebe la herida y tenga en cuenta la condición / comportamiento en general.
- Después de la inyección estereotáxica, los ratones puede ser sacrificado para la preparación rebanada electrofisiología 1 o perfundido y se extrajeron sus cerebros, en rodajas, y se tiñeron mediante inmunohistoquímica para el análisis 10.
Resultados
Tras el "Protocolo para el diseño de una Guía de Strand (sgRNA) para CRISPR / Cas9 Retrovirus", oligos dirigidas a una secuencia particular se insertan en el vector de clonación PXL aguas abajo del promotor Hu6 y aguas abajo de un andamio guía de ARN usando los sitios de clonación BbsI (Figura 1, paso 1). Este sgRNA se escinde luego de PXL y se inserta en la columna vertebral pRubi usando los sitios BstBI y PacI (Figura 1, paso 2). Por último, otro sgRNA clonado en PXL se puede colocar aguas abajo de la primera guía de pRubi-Guía 1 (figura 1, paso 3), dirigido a otra área de la genética y aumentar las posibilidades de un golpe de gracia a través de NHEJ. Verificación de los constructos correctos debe ser determinado por análisis de secuencia. Una vez hecha esta construcción, puede ser empaquetado en un virus siguiendo el "Protocolo para Retro / Lentiviral producción- CaPO4 el método". embalaje exitosa se confirmó por la infección de virus en células HEK293 con el fin de titular la virus. Si no hay expresión fluoróforo entonces no era probable un error durante el empaquetado del virus.
La Figura 2 es un resultado representativo de 2 retrovirus, uno que expresan GFP y el otro mCherry expresar, co-inyecta en el giro dentado del ratón neonatal (7 días de edad) y la imagen de 21 días después de la inyección. Etiquetado de neuronas con mCherry o GFP permite la evaluación morfológica de diversas manipulaciones genéticas en el mismo tejido, donde un virus puede expresar una CRISPR mediada / Cas9 KO y el otro un virus control, que expresan únicamente un fluoróforo. inyección estereotáxica permite selectividad anatómica precisa como se demuestra por la infección discreta de las coordenadas destinados, el giro dentado. Al analizar las secciones del cerebro para la infección, es importante para mantener el tejido circundante hasta que se determina que la región anatómica correcta estaba infectado. Si no hay ningún signo de infección, entonces es posible que la inyección se produjo en una región de un vecinod puede ser identificado en las secciones de los alrededores. También puede ser útil para localizar el trayecto de la aguja para encontrar la zona de inyección exacta. Pseudotyped virus VSVG rara vez se diseminan fuera de los márgenes de la circunvolución dentada cuando se inyecta in vivo, y tienden a extenderse a lo largo de las células de eje infectar caudal / rostrales a lo largo de todo el giro dentado, tal como se analizó por la reconstrucción 3D (Figura 3).
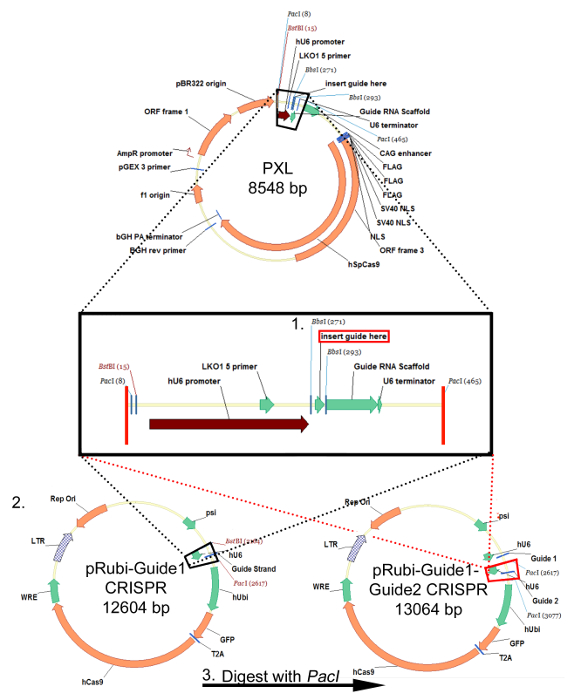
Figura 1:. Estrategia de clonación para el plásmido pRubi-Guía 1-Guía 2-retroviral CRISPR Esta estrategia es idéntica para los plásmidos de lentivirus basados-FU. oligos sgRNA se recuecen y se insertan en PXL usando los sitios de clonación BSBI. Después de la secuenciación para asegurar que el sgRNA se inserta con éxito en PXL, digerir el plásmido con BstBI y PacI. El inserto que se deja caer hacia fuera (cuadro negro) se clona en la espalda b viraluno (negro líneas de puntos) pRubi-Guía 1 CRISPR. Un segundo sgRNA también se puede insertar en PXL y se digirió a cabo utilizando la enzima PacI. Esto se clona en el vector de CRISPR pRubi-Guía 1 (líneas de puntos rojos), utilizando el sitio PacI. El plásmido resultante contiene entonces ambas cadenas de guía, así como los elementos necesarios virales, promotores y fluoróforos. Por favor, haga clic aquí para ver una versión más grande de esta figura.
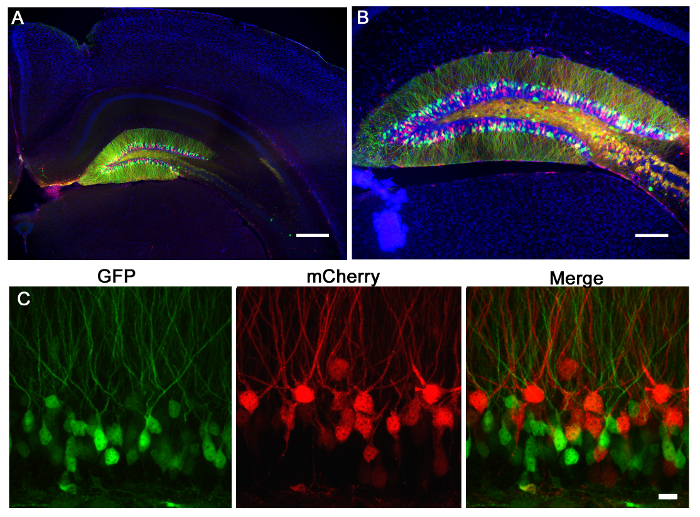
Figura 2:. De inyección retroviral del giro dentado del ratón Los retrovirus que expresan mCherry (rojo) o GFP (verde) se inyectaron en el giro dentado de un ratón p7. 21 días más tarde, los ratones fueron perfundidos y los cerebros se seccionaron y se tiñeron para GFP y mCherry. (A) Una imagen fluorescente de campo amplio 5X muestra el prECISIÓN de la inyección giro dentado y la especificidad de etiquetado dentado gyrus neuronas granulares. La morfología del hipocampo puede ser visto a través de la tinción (azul) Dapi. La barra de escala mide 200 micras. (B) Una imagen fluorescente 10X de gran campo demuestra que estos lentivirus alto título infectan un gran número de células cuya morfología se puede acceder a través de la expresión fluoróforo. La barra de escala mide 100 micras. (C) Los virus que expresan GFP o mCherry fueron co-inyectados en el giro dentado. El uso de un sistema de retrovirus, se puede utilizar un virus para hacer una manipulación genética marcada por GFP y otra manipulación marcado por mCherry, y luego evaluar los cambios individuales o aditivo debido a cada virus. La barra de escala mide 10 micras.
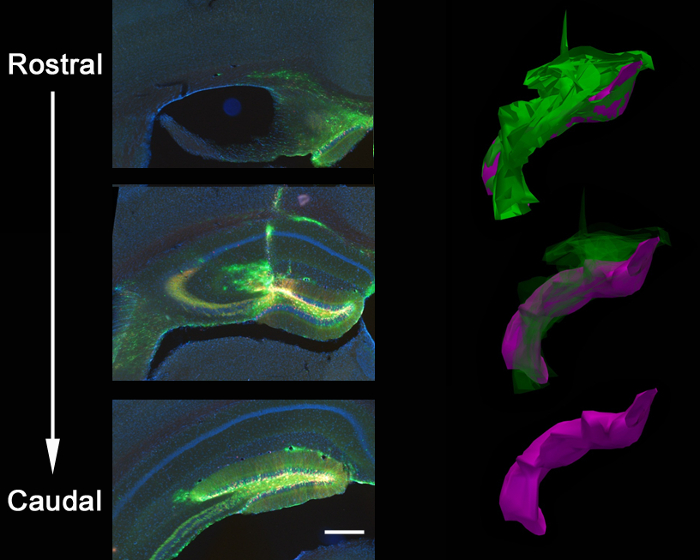
Figura 3:. Anatómico propagación de la inyección lentiviral co-inyección estereotáxica de un virus GFP-shPten y un virus de control mCherry en el cerebro de un adulto Pten loxP / + ratón dio como resultado una propagación viral a lo largo de todo el eje rostral / caudal del giro dentado del hipocampo. Esto se muestra en una reconstrucción en 3D de la extensión de la inyección en el que contornos cerrados de la propagación viral se trazaron más de 21 secciones en serie (Z = 50 micras / sección) utilizando software de reconstrucción. Los trazados de contorno fueron alineados para generar las imágenes en 3D para la cuantificación del volumen. La propagación viral total se muestra en verde (volumen = 54.730.800 m 3) y la propagación dentado-localizada se muestra en morado (volumen = 27.275.200 m 3). El virus se propaga a lo largo del trayecto de la aguja y el cuerpo calloso en la intersección de la pista de la aguja además de llenar el eje rostral / caudal del giro dentado. La barra de escala mide 200 micras.
Les / ftp_upload / 53783 / 53783video1frame.jpg "/>
Suplementario vídeo 1. Diseño de sgRNAs para clonar en estructura principal retroviral y lentiviral.
En este ejemplo de diseño RNA guía sintética (sgRNA), la secuencia genómica de ratón CHD8 se descarga desde NCBI. El codón de iniciación y la estructura exón se visualizaron después de Vector NTI. Esto nos permite copiar la región genómica alrededor del primer exón codificante y entramos en esta secuencia en Benchling. Benchling nos permite visualizar todas las sgRNAs potenciales en la región. Además, después de lo que indica la región genómica que tenemos de entrada, Benchling nos mostrará las puntuaciones en la diana y fuera de objetivo para cada guía de ARN. El usuario puede entonces seleccionar la guía de ARN con las puntuaciones más altas dentro y fuera de objetivo. Por favor, haga clic aquí para ver el vídeo. (Haga clic aquí para descargar.)
Discusión
Hay unos pocos pasos críticos que son importantes para el empaquetado viral exitosa. la salud de la célula es crítica antes y durante la transfección, tal como células enfermas gran medida a reducir la cantidad de virus producido. Si la transfección y envasado tienen éxito, entonces 100% de las células debe expresar el fluoróforo y las células deben formar un sincitio funcional. En el paso 3.2.4, golpeando ligeramente el tubo es necesario para la transfección de alto título de manera eficiente, y el pH de la solución salina tamponada con HEPES debe ser exacta. Los maxi-preps que producen los plásmidos necesarios para el empaquetado viral deben ser extremadamente puro. A este punto, es útil a etanol precipitar la elución de ADN final y volver a suspender en tampón Tris-EDTA. También es muy importante para reducir la cantidad de suero a 2% o menos en los medios de comunicación que la cafeína se añade a en el Día 6 (etapa 3.4) antes de la recogida viral. Si el suero no se reduce, entonces el virus purificado final contendrá una cantidad no deseable de prot sueroein. El uso de polietilenglicol 6000, cuando la precipitación de las partículas virales excluye la necesidad de ultracentrifugación. También es importante señalar que los CRISPR que contiene virus Cas9 típicamente tienen un título de alrededor de 10 veces menos que los virus que contienen únicamente un fluoróforo.
Para la cirugía estereotáxica, el uso de anestesia de inhalación permite el control rápido y preciso o la conciencia del animal en comparación con los anestésicos inyectables y permite la anestesia en un intervalo de edad más amplio. Es muy importante mantener los instrumentos quirúrgicos limpio y estéril, y la focalización reproducible requiere un posicionamiento preciso de la cabeza. Asegúrese de que no hay pitcheo o balanceo de la cabeza en el instrumento estereotáxico y que el cráneo se siente firmemente en su lugar. Puede ser útil para permitir que el cráneo se seque a fin de encontrar las suturas para determinar las coordenadas estereotáxica. Además, la velocidad y el volumen para cada estereotáxica coordenadas deben determinarse empíricamente.
Esta técnica es limitante en que la propagación de un retrovirus o lenti- está restringido, especialmente en comparación con los virus adeno-asociados (AAV) .Por lo tanto, estos virus son valiosas cuando infectar una región del cerebro discretos, pero no para la infección general asociado con AAV utilizado para el análisis del comportamiento en animales. El uso de la cafeína en este protocolo aumenta en gran medida el título de estos virus, pero todavía no son tan altos como los títulos obtenidos en el empaquetamiento de AAV. Además, la integración estable sólo es una ventaja de la expresión fluoróforo, como CRISPR / Cas9 forma ediciones genómica estable incluso cuando se transfectaron transitoriamente y es posible que en curso de la expresión de la Cas9 y sgRNA eventualmente puede producir efectos fuera de destino. La expresión transitoria del sistema / Cas9 CRISPR con AAV es suficiente para producir cambios genómicos que se propagan a lo largo de las divisiones celulares, sin embargo, no se mantuvo la expresión fluoróforo.
Creación de lenTi y retrovirus que utilizan el sistema de CRISPR / Cas9 impartirán la capacidad de dirigirse a cualquier nuevo gen en una amplia variedad de organismos. La eficiencia de la edición gen parece ser dependiente de la secuencia del RNA guía dirigidas a la escisión Cas9. Se ha determinado empíricamente que entre 10% y 80% de los clones contienen indeles después de la secuenciación infectado células Neuro2A. Actualmente se desconoce si las frecuencias indel calculados en células Neuro2A son un reflejo de aquellos en las neuronas. Guía de software de diseño de ARN tales como Benchling ahora incluyen una puntuación de "en-blanco" que pueden ser capaces de predecir la eficacia de una secuencia diana dada. Hasta qué punto estos índices "en la diana" son fiables las necesidades que se determinen empíricamente en las neuronas y otros tipos de células como el sistema CRISPR-Cas9 convierte implementadas más ampliamente.
la producción de animales transgénicos basados en lentivirus ha sido variable éxito con los informes de que los transgenes lentivirus entregado convierten SIlenced 11. CRISPR edición génica mediada de ADN se puede hacer pasar a través de la línea germinal para generar modelos de animales enteros. Por lo tanto, la edición genómica estable puede ser alcanzable, a pesar del silenciamiento de fluoróforos-virales y entregado transgenes Cas9. Esto puede proporcionar una plataforma eficiente para las alteraciones genómicas específicas. La entrega viral del sistema / Cas9 CRISPR, mientras que no requieren organismos transgénicos, es complementaria a esas técnicas. Por ejemplo, la inyección de tales partículas virales en un animal transgénico que expresa de manera inducible compuesto opto-o quimio-genética dependiente Cre y Cre transgenes debe facilitar los estudios complejos en la relación entre las manipulaciones genéticas y la actividad neuronal. Un segundo ejemplo es entregar estas partículas virales Cas9 / sgRNA en un knockout condicional en un intento de detectar interacciones gen-gen. Por último, otra vía interesante de esta investigación es la detección de fenotipos y compuestos terapéuticos en células derivadas del paciente, lo que puedeser utilizado para validar y descubrir las redes genéticas que se encuentran alteradas en diversas enfermedades.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el NINH conceder R01MH097949 y el autismo habla subvención piloto para 7359 BWL y el Centro de Cáncer Norris Cotton de imagen óptica compartida Instrumentación de Grant P30CA023108.
Materiales
| Name | Company | Catalog Number | Comments |
| List of Cell Culture Reagents | |||
| 293FT cell line | Life Technologies | R700-07 | For Lentivirus |
| 293GP cell line | Clontech | 631458 | For Retrovirus |
| Iscove's Modification of DMEM (IMDM) | Corning | 10-016-CV | Complete IMDM with 10% FBS, 1% NEAA, 1% L-Gln, and 1% P/S |
| Fetal Bovine Serum (FBS) | Corning | 35-011-CV | |
| MEM Nonessential Amino Acids (NEAA) | Corning | 25-025-CI | |
| L-Glutamine solution, 100x (L-Gln) | Corning | 25-005-CI | |
| Pennicillin/Streptomycin solution, 100x (P/S) | Corning | 30-002-CI | |
| Polystyrene 10 cm plate | USA Scientific | CC7682-3394 | |
| Trypsin EDTA 1x | Corning | 25-053-CI | |
| List of Transfection Reagents | |||
| 5 ml polystyrene tubes | Fisher Scientific | 352054 | |
| Calcium Chloride Dihydrate (CaCl2) | Fisher Scientific | C69-500 | Make a 2.5 M solution in ddH2O |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-3 | |
| HEPES | Fisher Scientific | BP2939-100 | |
| Sodium phosphate dibasic (Na2HPO4) | Fisher Scientific | S369-500 | |
| 2x HEPES Buffered Saline (HBS) | 500 ml: 8.2 g NaCl, 5.95 g HEPES, 0.106 g Na2HPO4, pH 7.01 (exact!) | ||
| Caffeine | Sigma-Aldrich | C0750-5G | |
| 0.22 µM syringe filter unit | EMD Millipore | SLGV033RS | |
| 0.45 µM syringe filter unit | EMD Millipore | SLHP033RS | |
| 60 cc L/L Syringe | Med-Vet International | MV60CCLL | |
| 50 ml Conical Tube | Corning | 352098 | |
| polyethylene glycol 6000 (PEG 6000) | Millipore | 528877 | |
| (10x) Phosphate Buffered Saline (PBS) | National Diagnostics | CL-253 | |
| 0.5 ml microcentrifuge tubes | USA Scientific | 1605-0000 | |
| Matrigel | Fisher Scientific | CB-40230A | |
| 6-well plate | Fisher Scientific | 353046 | |
| Paraformaldehyde | Fisher Scientific | AC41678-5000 | |
| Donor Horse Serum | Cellgro | 35-030-CV | |
| TritonX-100 | Sigma-Aldrich | X100-500ML | |
| 10 ml serological pipette | Fisher Scientific | 357551 | |
| anti-GFP, rabbit, 488 conjugate | Invitrogen | A21311 | |
| Stereotaxic Surgery Reagents | |||
| Vet-Syringe vet use T.B. Syringe only 1 cc Luer slip T.B. 100/bx | Med-Vet International | 1CCVLS | |
| Isoflurane | |||
| Stainless Steel Scalpel Blades, #10, 100-pk | Med-Vet International | JOR580S | |
| artificial tear ointment | Med-Vet International | RXPARALUBE-O | |
| PVP PrepSolution | Med-Vet International | HPIV108208H | |
| normal saline | Med-Vet International | DYND500MLSH | |
| cotton tipped applicators | Med-Vet International | CTA6 | |
| Triple antibiotic ointment | Med-Vet International | RXTRIP-OI15 | |
| MONOJECT® Needles Soft Pack 25 g x 5/8" | Med-Vet International | 25058 | |
| 6-0 silk sutures | Med-Vet International | MV-711 | |
Referencias
- Williams, M. R., DeSpenza, T., Li, M., Gulledge, A. T., Luikart, B. W. Hyperactivity of newborn Pten knock-out neurons results from increased excitatory synaptic drive. J Neurosci. 35 (3), 943-959 (2015).
- Luikart, B. W., et al. Pten knockdown in vivo increases excitatory drive onto dentate granule cells. J Neurosci. 31 (11), 4345-4354 (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol. 32 (4), 347-355 (2014).
- Luikart, B. W., et al. miR-132 mediates the integration of newborn neurons into the adult dentate gyrus. PLoS One. 6 (5), e19077 (2011).
- Nasri, M., Karimi, A., Allahbakhshian Farsani, M. Production, purification and titration of a lentivirus-based vector for gene delivery purposes. Cytotechnology. 66 (6), 1031-1038 (2014).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Fricano, C. J., et al. Fatty acids increase neuronal hypertrophy of Pten knockdown neurons. Front Mol Neurosci. 7, 30 (2014).
- Park, F. Lentiviral vectors: are they the future of animal transgenesis?. Physiol Genomics. 31 (2), 159-173 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados