Method Article
Progettazione, Packaging, e consegna di alto titolo CRISPR Retro e lentivirus via Stereotassica iniezione
In questo articolo
Riepilogo
Il sistema / Cas9 CRISPR offre il potenziale per fare l'editing genoma mirato accessibile e conveniente per la comunità scientifica. Questo protocollo ha lo scopo di dimostrare come creare virus che sarà ad eliminazione diretta di un gene di interesse utilizzando il sistema / Cas9 CRISPR, e poi iniettare stereotassica nel cervello di topo adulto.
Abstract
Replication defective lentiviruses or retroviruses are capable of stably integrating transgenes into the genome of an infected host cell. This technique has been widely used to encode fluorescent proteins, opto- or chemo-genetic controllers of cell activity, or heterologous expression of human genes in model organisms. These viruses have also successfully been used to deliver recombinases to relevant target sites in transgenic animals, or even deliver small hairpin or micro RNAs in order to manipulate gene expression. While these techniques have been fruitful, they rely on transgenic animals (recombinases) or frequently lack high efficacy and specificity (shRNA/miRNA). In contrast, the CRISPR/Cas system uses an exogenous Cas nuclease which targets specific sites in an organism's genome via an exogenous guide RNA in order to induce double stranded breaks in DNA. These breaks are then repaired by non-homologous end joining (NHEJ), producing insertion and deletion (indel) mutations that can result in deleterious missense or nonsense mutations. This manuscript provides detailed methods for the design, production, injection, and validation of single lenti/retro virus particles that can stably transduce neurons to express a fluorescent reporter, Cas9, and sgRNAs to knockout genes in a model organism.
Introduzione
Per studiare la base della normale fisiologia e patologia malattia, vi è la necessità di manipolare con precisione l'espressione genica in organismi modello. Per organismi modello mammiferi, questo è in gran parte incentrata sulla creazione e lo sviluppo di topi transgenici in cui un elemento genetico di interesse è affiancato da siti riconosciuti da un ricombinasi. Ciò può risultare in una determinata manipolazione sito di questi geni affiancate. Anche se questa è stata una strategia di successo, è tempo e risorse; per esempio, la creazione di un animale transgenico tripla che esprimesse un gene floxed, Cre ricombinasi, e un gene reporter Cre richiede più accoppiamenti e convalida. Al contrario, l'iniezione stereotassica di replicazione particelle virali difettose codificano una proteina fluorescente e la ricombinasi in un gene animale floxed non necessita genotipizzazione complesse o strategie di selezione 1. Inoltre, se una proteina fluorescente e Cre esprimere virus è co-iniettata con una seconda Enco virusding una proteina fluorescente diversa, allora questo fornisce un controllo all'interno dei tessuti per la manipolazione genetica mirata. Mentre questa strategia richiede ancora l'uso di animali knock-in, strategie di RNA virale basato mediate aggirare la necessità di animali transgenici. Ad esempio, l'iniezione stereotassica di virus replica-carenza che codificano una proteina fluorescente e un RNA breve hairpin (shRNA) può utilizzare macchinari endogena RNAi della cellula comportare una riduzione potenti della trascrizione di un gene di interesse. Tuttavia, le strategie shRNA producono sottile gene knock-down, spesso con conseguente modesti fenotipi cellulari 2. Mentre un knock-down può essere più fisiologicamente rilevanti per la disfunzione del gene eterozigote, la sua diminuzione robustezza rispetto ad un knock-out non è l'ideale per la scoperta di nuovi geni fenotipica.
Una terza tecnica che si è recentemente emerso, il CRISPR (cluster regolarmente intervallate Ripetere palindromo Short) / Cas9 (CRISPR-associataproteine 9) del sistema, si basa sulla espressione sia un piccolo RNA esogeno e un enzima taglio DNA. Il sistema / Cas9 CRISPR è stato adattato dal sistema immunitario procariote che si è evoluto un metodo per identificare estera, invadendo il DNA di virus e il targeting per la degradazione mediante l'enzima Cas9 3,4. Questa potente tecnica di montaggio del genoma può essere utilizzato per la creazione di eliminazioni mirate, inserimenti, e mutazioni; e il seguente protocollo illustrerà come fare delezioni in un gene di interesse per knockout sua espressione in vivo. L'enzima Cas9 deve essere espresso con un RNA guida omologa alla regione di interesse e contigua con un RNA ponteggio. Knockout di un gene utilizzando questa tecnica richiede mira Cas9 ad una regione specifica del genoma utilizzando RNA guida sintetici (sgRNA), e inducendo rotture a doppio filamento (DSB) in un sito di interesse. Questi DSB sono poi riparati dal macchinario cellulare di riparazione endogena via non omologa end-joining (NHEJ), che leannuncio a indels che possono produrre missenso o nonsense mutazioni e può quindi costituire una perdita di espressione della proteina funzionale 5. Poiché questo sistema produce alterazioni genomiche, richiede solo l'espressione transiente del Cas9 e sgRNA. Tuttavia, è desiderabile che un indicatore fluorescente stabile per identificare le cellule e la loro progenie manipolati in questo modo.
Lenti- e retrovirus hanno il vantaggio di stabile integrazione del DNA di interesse nelle cellule ospiti che mantengono l'espressione a lungo termine e si tramandano alle cellule figlie durante la mitosi. Questo protocollo descrive la progettazione e realizzazione di due tipi di replica difettoso, retrovirus alto titolo: il virus dell'immunodeficienza umana derivati particelle lentivirali (lentivirus) e quelli basati su murino virus Maloney leucemia (retrovirus). Mentre entrambi di questi virus sono in grado di supportare esprimere stabile di grandi transgeni, le particelle retrovirali possono integrare solo nel genoma dudivisione cellulare anello con la degradazione della membrana nucleare, e quindi può essere utilizzata come strumento per etichettare e cellule data di nascita 6. Mentre lentivirus hanno la reputazione di essere relativamente basso titolo 7, questa metodologia, compreso l'uso di caffeina durante la raccolta 8 virale, produce abitualmente titoli di 10 9 e 10 10 particelle / ml. Un altro vantaggio di lenti- e retrovirus è la tolleranza per grandi inserti. Il seguente insieme di protocolli delinea la procedura per la progettazione di un lenti o di retrovirus che codifica per un reporter fluorescente, sgRNAs, e Cas9 di utilizzare il sistema / Cas9 CRISPR di modificare il DNA, così come esprimere una proteina fluorescente.
Mouse neurochirurgia stereotassica è un metodo valido per l'iniezione di virus in vivo per studiare la morfologia, la funzione e la connettività dei neuroni infetti. L'infezione virale nei neuroni può essere usato per manipolare i livelli di espressione per un periodo prolungato di time, ad esempio durante lo sviluppo, e l'espressione possono essere controllati con precisione mediante l'uso di vari sistemi inducibili farmacologici e espressione specifica guidato Cre. Questo protocollo particolare spiega come iniettare un virus che esprime un sgRNA e Cas9 per knockout un gene di interesse nel cervello di un topo adulto. Topi recuperare molto rapidamente da questa procedura e espressione del transgene virale può essere visto entro 48 ore dopo l'iniezione. Tuttavia, l'espressione fluoroforo sembra aumentare nel corso di settimane conseguenti livelli massimi vicino da 3 settimane dopo l'infezione. I topi che subiscono l'iniezione stereotassica virale possono essere utilizzati per il comportamento, elettrofisiologia, o studi morfologici. Nel complesso, lo scopo di queste procedure è quello di dimostrare come knockout un gene nel cervello di topo adulto con radiochirurgia stereotassica e un virus che esprime un sgRNA specifico e Cas9.
Protocollo
Etica Dichiarazione: Tutti i protocolli sono stati approvati dai Dartmouth Istituzionale biosicurezza e istituzionale cura degli animali e del Comitato Usa revisione da stiro.
1. Protocollo per la progettazione di una guida Strand (sgRNA) per CRISPR / Cas9 Retrovirus
NOTA: Ci sono molti siti web senza scopo di lucro che possono essere utilizzati per generare sgRNAs per indirizzare un gene di interesse (https://benchling.com/ e http://crispr.mit.edu/). L'obiettivo di questo protocollo è quello di creare e ordinare singoli oligonucleotidi bloccati da un fornitore commerciale che sono ricotto gli uni agli altri. Questo oligo ricotto verrà ligato nel vettore di trasferimento PXL. Guarda il video supplementare 1 per un esempio di progettazione sgRNAs utilizzando Benchling.
- Utilizzare un sito web dedicato alla progettazione sgRNAs.
NOTA: Per esempio, inserendo una codifica / sequenza exonic da vicino all'inizio del gene di interesse nel sito web genererà un sgRNA 20 nucleotidi. Utilizzare la sequenza nucleotidica sgRNA 20 per progettare il senso e antisenso oligos che verrà ordinato per creare il sgRNA. - Dopo aver generato il sgRNA, copiare questa sequenza in un word processor. Aggiungere una G (guanina) all'inizio della sequenza nucleotidica sgRNA 20 nel documento, se non è già inizia con uno (cioè, G-20 sequenza nucleotidica sgRNA). Ciò è necessario per assicurare una buona trascrizione off promotore U6.
- Documentare il complemento inverso di questa ora 20-21 sequenza nucleotidica. Per l'oligo senso, aggiungere "CACC" per l'estremità 5 'della sequenza nel documento (cioè, CACC-G-20 sequenza nucleotidica sgRNA). Questa sporgenza viene utilizzata per legare la sequenza nel vettore PXL.
- Per l'oligo antisenso, aggiungere AAAC alla 'fine 5. Utilizzare questo sbalzo di legare la sequenza nel vettore PXL.
- Ottenere i oligonucleotidi senso e antisenso. Dopo aver ricevuto i oligonucleotidi, fare 100 micron scorte utilizzando DNAsi acqua libera. Mescolare 10 ml ciascuno dei 100 micron senso e antisenso oligonucleotidi con 4 μl di 10x NEB Buffer 2, e 16 ml di acqua.
NOTA: Non hanno bisogno di purificazione pagina o 5'phosphorylation (come gli sbalzi BBSI sono estremità coesive non compatibili).- Portare 200 ml di acqua a bollire in un bicchiere da 500 ml, poi galleggiare il tubo contenente il composto in una schiuma detentore "floater". Consentire l'acqua per raffreddare lentamente da 95 ° C per 2 ore a temperatura ambiente.
- Diluire il mix ora ricotto-oligo 1: 1.000 in acqua sterile ed utilizzare immediatamente seguente legatura o conservare la reazione rimanente a -20 ° C.
- Digest PXL, un derivato PX330 vettore, con l'enzima di restrizione BBSI a 37 ° C per 2 ore. Utilizzare una reazione di 40 ml contenente 2 mg di PXL, 4 pl di 10x NEB Buffer 2, 1 ml di acqua e BBS1 equilibrio. Sottoporre il digerito-PXL alla purificazione del gel di routine utilizzando un kit commerciale. Assicurarsi che la resa è ~ 8,5 kB prodotto.
- Utilizzando un kit di legatura commerciale, legare 1 ml di 1: 1.000ricotto-oligo con 50 ng di digerito e gel purificato-PXL. Trasformare il prodotto legatura in ricombinazione competente carente E. coli (NEB 5-alfa, efficienza subcloning). Schermo trasformanti per la presenza della guida corretta mediante sequenziamento di DNA plasmidico.
- Digest U6, guida filo, e gli elementi di RNA impalcatura (sgRNA) di PXL utilizzando BstB1 e PAC1 enzimi di restrizione e legare in fluorescente dorsale virale proteina-T2A-Cas9 digerito con gli stessi enzimi di restrizione. Trasformare il prodotto legatura (NEB 5-alfa massima efficienza). Le fluorescenti proteina-T2A-Cas9 plasmidi backbone virale sono bassi plasmidi di copia e protocolli in tal modo low copy devono essere utilizzati.
NOTA: Una seconda sgRNA può essere simile introdotto da Paci digestione di un altro PXL guida filone plasmide e ligato nel PAC1 digerito e fosfatasi vitello-intestinale trattata dorsale virale che contiene già il primo filone guida. trattamento fosfatasi del plasmide trasferimento virale aiuteràridurre il numero di trasformanti dalla auto-legatura di plasmidi che non contengono la seconda guida. - Sequenza verificare il plasmide e maxi finale di preparazione utilizzando il kit di preparazione maxi Nucleobond Xtra e confezionarlo in un virus utilizzando il "Protocollo per il Retro / lentivirali Produzione - Metodo CaPO4" in seguito.
2. Preparare 293FT / 293GP cellule per trasfezione (Retro / lentivirali Produzione - Metodo CaPO4)
- (1 ° giorno) Rapidamente scongelare 1 flacone di cellule per 10 centimetri piastra di coltura cellulare in un bagno d'acqua a 37 ° C. Per il confezionamento lentiviral, usare 293FT cellule. Per il confezionamento retrovirale, utilizzare 293GP (gag / pol) cellule.
- Pipettare tutte le cellule scongelate dal tubo crio in un tubo da 15 ml e aggiungere 2 ml di pre-riscaldato CO 2 equilibrata completamento Modified Media Dulbecco di Iscove.
- celle di centrifugazione per 5 min a 500 xg a pellet. Aspirare il surnatante e risospendere il pellet cellulare in 10 ml di completo èbaia di Modified Media Dulbecco. Piastra le cellule su un 10 centimetri piatto di coltura cellulare. Incubare le cellule notte a 37 ° C in un incubatore anidride carbonica 5%.
- (Day 2) 24 ore dopo la placcatura, cambiate multimediale su piastra aspirando media esistenti e aggiungendo 10 ml di pre-riscaldato Piano di Iscove Modified Dulbecco alla piastra.
- (Giorno 3-4) 24-48 ore dopo la modifica di supporto e una volta che le cellule diventano confluenti, dividere le cellule ad una confluenza di 2,5-3,0 x 10 6 cellule / piastra (10 cm Piatto).
- Per dividere le celle, aspirare media e lavare la piastra con 5 ml di PBS. Aggiungere 1 ml di 0,25% tripsina alla piastra e incubare a 37 ° C finché le cellule sollevare la piastra. Aggiungere 0,5 ml di mezzo di Iscove Modified Dulbecco per neutralizzare la reazione tripsina 0,25% e pipettare le cellule in una provetta da 1,5 ml.
- Spin le cellule a 500 xg per 5 min. Risospendere le cellule in 1 ml di Modified medio Dulbecco Iscove. Diluire 1081; l di cellule in 90 ml di PBS. Contare le cellule utilizzando sia un emocitometro o contatore di cellule automatizzato. Re-piatto 2,5-3,0 x 10 6 cellule / piastra con Modified Media Dulbecco completo di Iscove.
NOTA: Le cellule saranno ~ 50% confluenti 24-34 ore dopo la placcatura. Trasfezione le cellule quando sono ~ 50% confluenti.
3. (5 ° giorno) CaPO4 Transfection e Viral Collection particelle
- Modificare l'ora dei media 2 prima di trasfezione aspirando i mezzi di comunicazione esistenti e l'aggiunta di 10 ml di pre-riscaldato Modified Media Dulbecco di Iscove. Assicurarsi che vi sia esattamente 10 ml di media nella piastra 10 cm.
- Preparare i reagenti di trasfezione per 2 piatti a base di tubi in polistirolo fondo rotondo 2, 5 ml. Etichettare il primo tubo "DNA" e il secondo tubo "2X HBS". Regolare la concentrazione di DNA a 1 ug / ml in tampone Tris-EDTA a pH 7,4.
- Per lentivirus, aggiungere lentamente 20 ml di vettore di trasferimento (The Viral costrutto di interesse), 13 ml di CMVdelta8.9, 9 ml di VSV-g, 860 ml di biologia molecolare di grado H 2 O, e 100 ml di 2,5 M CaCl 2 al primo tubo ( "DNA"), mentre continua autofilettanti sul tubo per mescolare.
- Per i retrovirus, omettere il plasmide CMVdelta8.9. Aggiungere 20 ml di trasferimento vettore, 15 ml VSV-g, 860 ml Molecular Biology Grade H 2 O, e 100 ml 2,5 M CaCl 2 alla provetta con l'etichetta "DNA".
- Aggiungere 1 ml di soluzione fisiologica 2x HEPES-buffered (pH 7.0; questo pH è assolutamente fondamentale) alla provetta con l'etichetta "2X HBS".
- Aggiungere lentamente il contenuto 1 ml del tubo "DNA" al tubo "2X HBS", una goccia alla volta. Continuamente toccare il tubo "2X HBS" con il dito indice o medio, mentre l'aggiunta del contenuto del tubo "DNA". Osservare apparenti Capo 4 vescicole dopo ogni caduta. Incubare la provetta al buio per 30 minuti a temperatura ambiente.
- Aggiungere 1 ml di trasfezione in goccioline lenti a ciascuna piastra cella 10 cm incubare una notte a 37 ° C.
- (Giorno 6) Sostituire media con 8 ml di Modified Medium + 0,5% FBS Dulbecco di Iscove e 100 ml dei media 40 mg di caffeina /. Se il vettore di trasferimento contiene un marcatore fluorescente, quindi espressione fluoroforo indica una trasfezione di successo.
- (7 ° giorno) Raccogliere il supporto contenente le particelle virali utilizzando un 10 ml pipetta sierologica ed erogare in un tubo conico da 50 ml. Conservare il tubo conico da 50 ml a 4 ° C. Aggiungere 8 ml di 0.5% Mezzi di FBS per ogni piatto.
- (Giorno 8) Anche in questo caso, raccogliere i supporti contenenti le particelle virali e si combinano con la raccolta del giorno precedente nel tubo conico da 50 ml.
4. concentrazione e purificazione del virus
- Fare un 5x polietilene glicole 6000 con l'aggiunta del 40% di polietilene glicole 6000 e 1,5 M di NaCl per DDH 2 O. Autoclave la soluzione suil ciclo di liquido per 45 min a 121 ° C. Lentamente mescolare la soluzione durante il raffreddamento.
NOTA: La soluzione inizierà nuvoloso e poi diventato chiaro come si raffredda. - Centrifugare la provetta conica da 50 ml contenente entrambe le collezioni del surnatante virale a 2.000 xg per 10 min per pellet materiale insolubile. Purificare la media virali filtrando attraverso un filtro da 0,45 micron a basso contenuto proteico siringa legame (PES o PVDF).
- Aggiungere 5x polietilene glicole 6000 soluzione ai media (La concentrazione finale dovrebbe essere l'8% di polietilene glicole 6000 e 0,3 M NaCl). Miscelare invertendo i tubi diverse volte (non vortex). Incubare la soluzione di polietilenglicole virale contenente a 4 ° C per 12 o più ore, rimescolando di tanto in tanto.
- (Giorno 9) Centrifugare la soluzione contenente glicole polietilenico virale a 2.500 g per 45 min. Rimuovere e scartare il surnatante e far girare ancora per 2 minuti. Anche in questo caso, rimuovere ed eliminare il surnatante.
- Risospendere il pellet aggiungendo 320 ml disterile PBS (320 ml è 1/100 del volume originale di supporti contenenti virali raccolti) e incubare una notte a 4 ° C. Facoltativamente, risospendere il pellet a temperatura ambiente su un agitatore meccanico per 30 min.
- Dopo ri-sospensione il pellet, un'aliquota del virus (5-10 ml per 0,5 ml tubo) e congelare aliquote a -80 ° C (congelamento scongelamento o conservazione a 4 ° C ridurrà drasticamente il titolo).
- Titolo i virus utilizzando protocolli standard, vale a dire, eseguire una serie di diluizioni su un 6-pozzetti di cellule HEK293T e contare manualmente colonie fluorescenti 48 ore più tardi.
5. prove di efficacia del virus CRISPR
NOTA: i cloni sequenza usando le seguenti operazioni per testare per la produzione di rotture del doppio filamento riparati da NHEJ utilizzando le cellule del mouse Neuro2A. Questo ha il vantaggio rispetto saggi geometra in quanto può essere utilizzato per determinare la percentuale di cellule che sono stati modificati e la naturadei indels derivanti da NHEJ.
- Coat un piatto di coltura cellulare 3,5 centimetri con una miscela proteica gelatinosa come matrigel diluito 1:50 in Modified medio Dulbecco Iscove e incubare per 30 min a 37 ° C. Aspirare la miscela proteica gelatinosa e piastra le cellule Neuro2A.
- Dopo che le cellule Neuro2A raggiungono il 50% di confluenza, aggiungere il CRISPR Lentivirus o Retrovirus ad una molteplicità di infezione di 10 (cioè, 10 particelle virali per cella).
NOTA: le cellule Neuro2A non sono così amabile alle infezioni come lo sono HEK293 cellule, in tal modo, una singola infezione si traduce in 100% delle cellule che sono infettate con il lentivirus. Inoltre, il retrovirus infetta solo le cellule in divisione e quindi inoltre non provocare infezioni 100%. Al fine di raggiungere un tasso vicino al 100% di infezione, aggiunta di virus può essere ripetuta più giorni, dividendo le cellule come necessario. In alternativa, le cellule positive fluorescenti possono essere isolate utilizzando la fluorescenza-attivato cell-smistamento. the modo più efficace per raggiungere un tasso di infezione del 100% con un lentivirus è quello di eseguire una singola infezione seguita da isolamento FACS. Per un retrovirus, eseguire 4-cicli di infezione 24 ore a parte e seguire per l'isolamento FACS per garantire% di infezione 100. - Dopo infettare le cellule per 1 settimana, espandere le cellule a confluenza vicino su una piastra 10 cm aspirare i media, e lavare la piastra con 5 ml di soluzione salina tamponata con fosfato. Aggiungere 1 ml di 0,25% tripsina e incubare a 37 ° C finché le cellule sollevare la piastra. Aggiungere 0,5 ml di mezzo di Iscove Modified Dulbecco per neutralizzare la reazione tripsina 0,25% e pipettare le cellule in una provetta da 1,5 ml e agglomerare le cellule a 500 xg per 5 min.
- Per isolare DNA, aggiungere 100 ml di 50 mM di idrossido di potassio, risospendere le cellule, e incubare a 95 ° C per 5 min. Neutralizzare la soluzione con 10 ml di 1 M Tris, pH = 8,0.
- PCR amplifica la regione che fiancheggia la regione genomica di destinazione del CRISPR sgRNAutilizzando ~ 300 ng del DNA isolato in fase 5.5 utilizzando un alta fedeltà PCR Master Mix 4.
- Eseguire la reazione di PCR su un gel di agarosio 2,5% e gel purificare il frammento di dimensioni appropriate utilizzando un kit di purificazione gel come un kit gel recupero DNA. Legare in un vettore di clonaggio PCR come pGEM-t-facile e trasformarsi in cellule competenti 9.
- 24 ore più tardi, aggiungere 2 ml di brodo LB in un 15 ml a fondo rotondo tubi nonché l'antibiotico appropriato (ampicillina, neomicina, etc.). Inoculare il tubo con una colonia singola utilizzando una punta di pipetta sterile o loop per selezionare una singola colonia dalla LB-plate. Espandere e far crescere la colonia agitando la provetta a 250 RPM e 37 ° C per 24 ore. Ripetere l'operazione per il maggior numero di colonie, se lo desideri.
- Utilizzo di un mini-prep kit, isolare il DNA da E. coli e sequenza con primer disegnati per amplificare la regione del gene bersaglio dal sgRNA per analizzare la regione Cas9 mirata del genoma del topo.
6. Stereotassica iniezione protocollo per il topo adulto
- Preparare per la chirurgia
NOTA: E 'importante mantenere condizioni di sterilità durante interventi chirurgici di sopravvivenza. Ciò si ottiene in questo protocollo dal calore sterilizzare gli strumenti chirurgici, utilizzando betadine per sterilizzare il sito di iniezione, e l'aggiunta di pomata antibiotica al sito di incisione dopo averlo chiuso. E 'anche importante usare guanti sterili nonché un accuratamente sterilizzato, zona chirurgia dedicata.- Sterilizzare calore strumenti di chirurgia prima dell'uso da una autoclave o mediante sterilizzatore a caldo tallone. Preparare camera di recupero e zona chirurgia accendendo termofori per mantenere la temperatura corporea durante l'intervento chirurgico e il recupero. Montare il trapano usato per creare fori nel cranio iniettabile.
- Carico 4 ml di virus in ago per l'iniezione di ritirare il virus direttamente dalla provetta sterile PCR. Brevemente rimuovere l'aliquota virale da ghiaccio. Mantenere il virusnella siringa a temperatura ambiente per non più di 1 ora prima dell'iniezione.
7. Iniezione Stereotassica
- Verificare che la ventilazione per la suite di chirurgia è aperto per garantire il corretto flusso d'aria. Preparare il mouse per un intervento chirurgico per anestetizzare con il 4% isoflurano in una camera di induzione. Dopo l'anestesia, la barba la testa per preparare il sito di iniezione.
- Posizionare il mouse nello strumento stereotassico utilizzando pinzette per spostare la lingua verso il basso e di lato. Inserire la barra morso in bocca fino a quando i denti cadono nella fessura, quindi fissare le barre orecchio. Assicurarsi che il corpo del mouse è sulla rampa di riscaldamento e il naso si trova nel cono. Diretto 4% isoflurano nel cono naso. Confermare l'anestesia da pizzicare il piede con una pinzetta e garantire che non vi è alcun riflesso di trasalimento.
- Applicare lacrime artificiali per lubrificare gli occhi. Fine preparazione del sito di iniezione tamponando la testa rasata con alternanza di cicli di povIdone-iodio e lidocaina.
- Usando un bisturi, tagliare piccola incisione lungo il centro del cuoio capelluto. Asciugare il cranio con un perossido di idrogeno tampone e, se necessario, per aiutare a visualizzare bregma.
- Utilizzando un ambito dissezione, individuare bregma sul cranio e posizionare la punta trapano su bregma. Zero (o registrare) le coordinate stereotassica digitali sui piani x, ye z.
- Al fine di garantire che la testa sia in piano sul rostrale a caudale asse y, posizionare la punta su lambda e livellare la testa in modo che la coordinata z è approssimativamente uguale sia bregma e lambda.
- Al fine di garantire che la testa sia in piano sul l'asse x, posizionare la punta a y = 1/2 lambda. Assicurarsi che la coordinata z è uguale a 1 mm per lato (x = +/- 1 mm) della sutura sagittale. Regolare il cranio se vi è una differenza nel coordinata z a 1mm a sinistra ea destra di lambda.
- Posizionare il trapano sopra le coordinate desiderate. Per il giro dentato, utilizzare la coordinatasy = -1.9 dal bregma e x = +/- 1,1. Prima di forare, abbassare il isoflurano al 2%, poi lentamente e con attenzione, forare il cranio.
- Dopo aver praticato tutti i fori, apporre il pieno siringa sullo strumento stereotassico. Visivamente centrare la siringa sopra il foro e zero coordinata z il cranio.
- Abbassare lentamente la siringa a più profonda Z-profondità. Per il giro dentato, gli z fondali sono -2.5, -2.4, e -2.3. Iniziare iniezione ad una velocità di 0,25 ml / min usando un iniettore stereotassico.
- Dopo l'iniezione è completo a basso Z-profondità, attendere 1 minuto, quindi sollevare al prossimo coordinare e ricominciare iniezione. Continuare questo modello fino a quando tutte le coordinate Z-iniezione vengono iniettate. Attendere 2 minuti prima di rimuovere la siringa dopo l'ultima iniezione.
NOTA: Nessun effetto negativo sono stati notati sul tessuto iniettando fino a 2 ml di virus per dell'emisfero nel cervello di topo.
- Dopo l'iniezione è completo a basso Z-profondità, attendere 1 minuto, quindi sollevare al prossimo coordinare e ricominciare iniezione. Continuare questo modello fino a quando tutte le coordinate Z-iniezione vengono iniettate. Attendere 2 minuti prima di rimuovere la siringa dopo l'ultima iniezione.
- iniezione Ripetere l'operazione per gli altri fori.
- Dopo l'iniezione, rimuovere il mouse dallo strumento stereotassico e suturare il cuoio capelluto con 6-0 punti di sutura in seta. Applicare lidocaina e una crema antibatterica alla ferita.
- Iniettare 0,8-1,0 ml di soluzione fisiologica + Ketoprofene (3-5 mg / kg) per via IP per gestire il dolore. Poi posto del mouse nella camera di recupero riscaldato. Non lasciare l'animale incustodito fino a quando non ha ripreso conoscenza sufficiente a mantenere decubito sternale. Non rispedire l'animale verso la compagnia di altri animali fino a quando non ha pienamente recuperato.
- Nei giorni dopo l'intervento, pesare il mouse quotidiana e fornire cibo morbido e tratta. Controllare ferita e notare la generale condizione di / contegno.
- Dopo l'iniezione stereotassica, i topi possono essere eutanasia per fetta preparazione elettrofisiologia 1 o perfuso e il loro cervello rimosso, affettato, e macchiato tramite immunoistochimica per l'analisi 10.
Risultati
A seguito del "Protocollo per la progettazione di una guida Strand (sgRNA) per CRISPR / Cas9 Retrovirus", oligo di targeting una particolare sequenza vengono inseriti nel PXL clonazione vettore valle del promotore hU6 ea valle di un ponteggio guida RNA utilizzando i siti di clonazione BBSI (Figura 1, punto 1). Questo sgRNA viene poi asportato da PXL e inserito nella spina dorsale pRubi utilizzando i siti BstBI e Paci (Figura 1, punto 2). Infine, un'altra sgRNA clonato in PXL può essere posizionato a valle della prima guida in pRubi-Guide1 (Figura 1, fase 3), il targeting un'altra zona del gene e aumentando le possibilità di un knockout via NHEJ. Verifica dei costrutti corretti dovrebbe essere determinata mediante analisi di sequenza. Una volta che questo costrutto è fatto, può essere confezionato in un virus a seguito del "Protocollo per il Retro / lentivirali Produzione- CaPO4 Method". confezionamento di successo è confermata da infezione da virus in cellule HEK293 per poi titolare la vIRU. Se non vi è alcuna espressione fluoroforo allora è probabile un errore durante il confezionamento del virus.
La figura 2 è un risultato rappresentativo di 2 retrovirus, una GFP esprimere e l'altra esprimere mCherry, co-iniettato nel giro dentato topo neonatale (7 giorni di età) e ripreso 21 giorni dopo l'iniezione. Etichettatura di neuroni con mCherry o GFP permette la valutazione morfologica delle varie manipolazioni genetiche in uno stesso tessuto, in cui un virus può esprimere un CRISPR / Cas9 mediata KO e l'altro un virus di controllo, che esprime esclusivamente un fluoroforo. iniezione stereotassica consente precisa selettività anatomica come dimostra l'infezione discreta delle coordinate destinate, giro dentato. Quando si analizzano le sezioni di cervello per l'infezione, è importante mantenere i tessuti circostanti fino a quando non è stato accertato che la regione anatomica corretta è stato infettato. Se non vi è alcun segno di infezione, allora è possibile che l'iniezione avvenuta in una regione di una vicinad può essere identificato nelle sezioni circostanti. Può anche essere utile per individuare il percorso dell'ago per trovare la regione di iniezione esatto. VSVG virus pseudotyped raramente sparsi dei margini del giro dentato quando iniettato in vivo, e tendono a diffondersi lungo le cellule asse infettare rostrali / caudale lungo tutto il giro dentato, come analizzato da ricostruzione 3D (Figura 3).
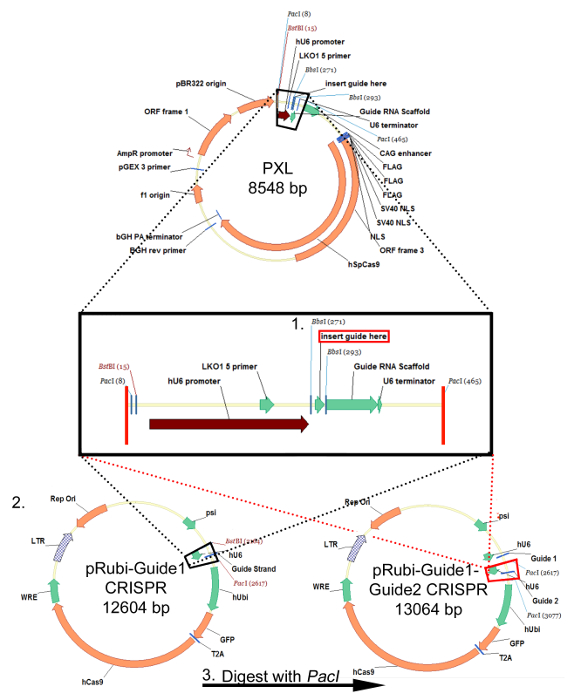
Figura 1:. Clonazione strategia per il plasmide pRubi-Guide1-Guide2-retrovirale CRISPR Questa strategia è identica per i plasmidi lentivirali FU-based. oligo sgRNA sono ricotto e inseriti in PXL utilizzando i siti di clonazione BSBI. Dopo il sequenziamento per garantire che il sgRNA è inserita con successo in PXL, digerire il plasmide con BstBI e Paci. L'inserto che viene abbandonato (scatola nera) viene poi clonato nel virale posteriore buno (il nero linee tratteggiate) pRubi-Guide1 CRISPR. Una seconda sgRNA può essere inserito anche in PXL e digerito utilizzando l'enzima PACI. Questo viene poi clonato nel vettore CRISPR pRubi-Guide1 (rosse linee tratteggiate) utilizzando il sito PACI. Il plasmide risultante contiene quindi entrambi i filamenti di guida, nonché gli elementi virali necessari, promotori, e fluorofori. Clicca qui per vedere una versione più grande di questa figura.
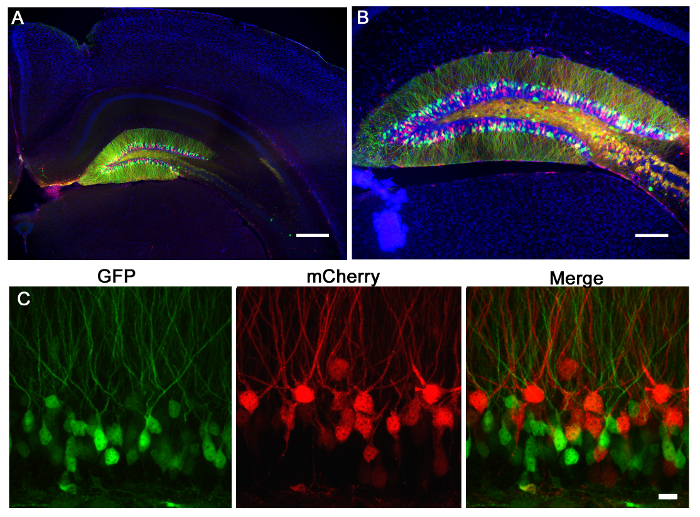
Figura 2:. Iniezione retrovirale del giro dentato del mouse retrovirus che esprimono mCherry (rosso) o GFP (verde) sono stati iniettati nel giro dentato di un mouse p7. 21 giorni dopo, i topi sono stati perfusi e il cervello sezionati e colorati per la GFP e mCherry. (A) Un immagine fluorescente a grande campo 5X mostra la prECISIONE dell'iniezione giro dentato e la specificità di etichettatura dentate neuroni granulari giro. La morfologia dell'ippocampo può essere visto attraverso la (blu) colorazione Dapi. barra della scala misura 200 micron. (B) L'immagine fluorescente 10 volte più grande campo dimostra che questi lentivirus alto titolo infettare un gran numero di cellule la cui morfologia è possibile accedere tramite espressione fluoroforo. barra della scala misura 100 micron. (C) I virus che esprimono GFP o mCherry sono stati co-iniettate in giro dentato. Utilizzando un sistema di retrovirus, si può usare un virus per effettuare una manipolazione genetica segnata da GFP e un'altra manipolazione segnata da mCherry e quindi valutare le variazioni singole o additivi a causa di ogni virus. barra della scala misura 10 micron.
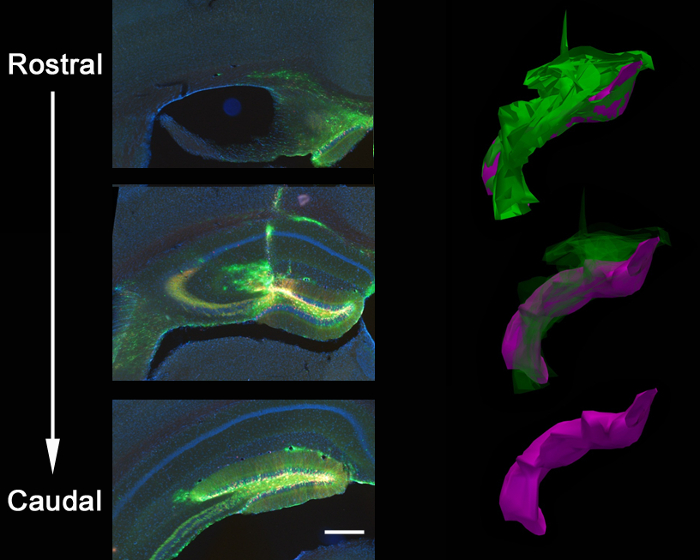
Figura 3:. Diffusione anatomica di iniezione lentivirali coiniezione Stereotassica di un virus GFP-shPten e un virus di controllo mCherry nel cervello di un adulto Pten loxP / + topo ha provocato una diffusione virale lungo tutta la rostrale / asse caudale del giro dentato dell'ippocampo. Questo è mostrato in una ricostruzione 3D della portata di iniezione in cui chiusi contorni della diffusione virale sono stati rintracciati oltre 21 sezioni seriali (Z = 50 um / sezione) utilizzando software di ricostruzione. I tracciati di contorno sono poi stati allineati per generare le immagini 3D per il volume quantificazione. Diffusione virale totale viene visualizzato in verde (volume = 54.730.800 micron 3) e la diffusione dentato-localizzato è mostrato in viola (volume = 27.275.200 micron 3). Il virus si diffonde lungo il percorso dell'ago e il corpo calloso all'intersezione della traccia dell'ago oltre al riempimento all'asse rostrale / caudale del giro dentato. barra della scala misura 200 micron.
les / ftp_upload / 53783 / 53783video1frame.jpg "/>
Supplemental Video 1. Progettazione di sgRNAs per clonare in backbone retrovirali e lentivirali.
In questo esempio di progettazione guida sintetica RNA (sgRNA), la sequenza genomica di CHD8 mouse viene scaricato da NCBI. Il codone di inizio e la struttura esone vengono poi visualizzati in Vector NTI. Questo ci permette di copiare la regione genomica intorno al primo esone codifica ed entriamo in questa sequenza Benchling. Benchling ci permette di visualizzare tutti i potenziali sgRNAs nella regione. Inoltre, dopo che indica la regione genomica che abbiamo in ingresso, Benchling ci mostrerà i punteggi on-target e off-bersaglio per ogni RNA guida. L'utente può quindi selezionare l'RNA guida con i punteggi più alti in bilancio e fuori bersaglio. Clicca qui per visualizzare questo video. (Tasto destro del mouse per scaricare).
Discussione
Ci sono alcuni passaggi critici che sono importanti per il confezionamento virale di successo. salute delle cellule è critico prima e durante la trasfezione, le cellule non sane ridurrà notevolmente la quantità di virus prodotto. Se la trasfezione e confezionamento hanno successo, allora il 100% delle cellule dovrebbe esprimere il fluoroforo e le celle deve formare un sincizio funzionale. Nel passaggio 3.2.4, toccando il tubo è necessaria per alto titolo trasfezione efficiente, e il pH della soluzione salina tamponata con HEPES-deve essere esatta. Il maxi-prep che producono i plasmidi necessari per il confezionamento virale devono essere estremamente puro. A questo punto, è utile etanolo precipitare il DNA eluizione finale e risospendere in tampone Tris-EDTA. È inoltre molto importante per ridurre la quantità di siero a 2% o meno nei media che la caffeina è aggiunta a il giorno 6 (passo 3.4) prima della raccolta virale. Se il siero non viene ridotta, quindi il virus purificato finale contiene una quantità indesiderabile di prot sieroEin. L'uso di polietilene glicole 6000 quando precipitare le particelle virali esclude la necessità di ultracentrifugazione. E 'anche importante notare che i CRISPR contenente virus Cas9 tipicamente hanno un titolo circa 10 volte inferiore a virus contenenti esclusivamente un fluoroforo.
Per la radiochirurgia stereotassica, l'uso di anestesia inalatoria permette un controllo rapido e preciso o coscienza dell'animale rispetto agli anestetici iniettabili e permette anestesia su una gamma di età maggiore. E 'molto importante mantenere gli strumenti chirurgici pulito e sterile, e il targeting riproducibile richiede un posizionamento preciso della testa. Assicurarsi che non vi è alcuna beccheggio o di rollio della testa nello strumento stereotassico e che il cranio si sente saldamente in posizione. Può essere utile per consentire il cranio asciugare per trovare i punti di sutura per determinare le coordinate stereotassica. Inoltre, il tasso e il volume per ogni stereotassica coordinate dovrebbero essere empiricamente determinato.
Questa tecnica è limitante in quanto la diffusione di un lenti- o di retrovirus è limitato, soprattutto se confrontato con i virus adeno-associati (AAV) .Pertanto, questi virus sono utili quando infettare una regione del cervello discreta, ma non per l'infezione complessivo associato con AAV utilizzato per l'analisi del comportamento in animali. L'uso di caffeina in questo protocollo aumenta notevolmente il titolo di questi virus, ma non sono ancora alto come i titoli ottenuti in imballaggi AAV. Inoltre, l'integrazione stabile è solo un vantaggio di espressione fluoroforo, come forma CRISPR / Cas9 stabili modifiche genomiche anche quando transientemente trasfettate ed è possibile che in corso espressione del Cas9 e sgRNA possono eventualmente produrre effetti fuori bersaglio. L'espressione transiente del sistema / Cas9 CRISPR con AAV è sufficiente a produrre cambiamenti genomici che si propagano per tutta divisioni cellulari, tuttavia, espressione fluoroforo non venga mantenuta.
Creazione di lenbrano e retrovirus che utilizzano il sistema CRISPR / Cas9 impartirà la possibilità di indirizzare qualsiasi nuova gene in un'ampia varietà di organismi. L'efficienza di editing gene sembra essere dipendente dalla sequenza del RNA guida mira la scissione Cas9. È stato determinato empiricamente che tra il 10% e il 80% dei cloni contengono indels dopo sequenziamento infettato cellule Neuro2A. E 'attualmente noto se le frequenze INDEL calcolati in cellule Neuro2A sono riflettenti di quelle in neuroni. Guida al software di progettazione RNA come Benchling ora includono un punteggio "on-target", che può essere in grado di predire l'efficacia di una data sequenza bersaglio. Fino a che punto tali punteggi "a bersaglio" sono bisogni affidabili da determinare empiricamente nei neuroni e altri tipi di cellule, come il sistema di CRISPR-Cas9 diventa più ampiamente attuate.
produzione animale transgenico lentivirus-based è stato variamente successo con i rapporti che i transgeni lentivirus-consegnato diventano SIricevitore in presenza 11. CRISPR editing gene mediata del DNA può essere passato attraverso la linea germinale per generare modelli animali interi. Così, stabile l'editing genomico può essere realizzabile, nonostante il silenziamento di fluorofori virali-consegnato e transgeni Cas9. Questo può fornire una piattaforma efficiente per alterazioni genomiche mirati. La consegna virale del sistema / Cas9 CRISPR, pur non richiedendo organismi transgenici, è complementare a quelle tecniche. Ad esempio, l'iniezione di tali particelle virali in un animale transgenico composto che esprime inducibile Cre e Cre opto dipendente o chimico-genetico transgeni dovrebbe facilitare studi complessi sul rapporto tra manipolazioni genetiche e attività neuronale. Un secondo esempio è quello di fornire queste particelle virali Cas9 / sgRNA in un knockout condizionale, nel tentativo di screening per le interazioni gene-gene. Infine, un altro itinerario interessante di questa ricerca è lo screening di fenotipi e composti terapeutici nelle cellule del paziente derivata, che puòessere utilizzato per convalidare e scoprire le reti genetiche che sono interrotti in varie malattie.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dal NINH concedere R01MH097949 e l'autismo parla pilota di Grant 7359 al BWL e il Norris Cotton Cancer Center Optical Imaging Shared Strumentazione di Grant P30CA023108.
Materiali
| Name | Company | Catalog Number | Comments |
| List of Cell Culture Reagents | |||
| 293FT cell line | Life Technologies | R700-07 | For Lentivirus |
| 293GP cell line | Clontech | 631458 | For Retrovirus |
| Iscove's Modification of DMEM (IMDM) | Corning | 10-016-CV | Complete IMDM with 10% FBS, 1% NEAA, 1% L-Gln, and 1% P/S |
| Fetal Bovine Serum (FBS) | Corning | 35-011-CV | |
| MEM Nonessential Amino Acids (NEAA) | Corning | 25-025-CI | |
| L-Glutamine solution, 100x (L-Gln) | Corning | 25-005-CI | |
| Pennicillin/Streptomycin solution, 100x (P/S) | Corning | 30-002-CI | |
| Polystyrene 10 cm plate | USA Scientific | CC7682-3394 | |
| Trypsin EDTA 1x | Corning | 25-053-CI | |
| List of Transfection Reagents | |||
| 5 ml polystyrene tubes | Fisher Scientific | 352054 | |
| Calcium Chloride Dihydrate (CaCl2) | Fisher Scientific | C69-500 | Make a 2.5 M solution in ddH2O |
| Sodium Chloride (NaCl) | Fisher Scientific | S271-3 | |
| HEPES | Fisher Scientific | BP2939-100 | |
| Sodium phosphate dibasic (Na2HPO4) | Fisher Scientific | S369-500 | |
| 2x HEPES Buffered Saline (HBS) | 500 ml: 8.2 g NaCl, 5.95 g HEPES, 0.106 g Na2HPO4, pH 7.01 (exact!) | ||
| Caffeine | Sigma-Aldrich | C0750-5G | |
| 0.22 µM syringe filter unit | EMD Millipore | SLGV033RS | |
| 0.45 µM syringe filter unit | EMD Millipore | SLHP033RS | |
| 60 cc L/L Syringe | Med-Vet International | MV60CCLL | |
| 50 ml Conical Tube | Corning | 352098 | |
| polyethylene glycol 6000 (PEG 6000) | Millipore | 528877 | |
| (10x) Phosphate Buffered Saline (PBS) | National Diagnostics | CL-253 | |
| 0.5 ml microcentrifuge tubes | USA Scientific | 1605-0000 | |
| Matrigel | Fisher Scientific | CB-40230A | |
| 6-well plate | Fisher Scientific | 353046 | |
| Paraformaldehyde | Fisher Scientific | AC41678-5000 | |
| Donor Horse Serum | Cellgro | 35-030-CV | |
| TritonX-100 | Sigma-Aldrich | X100-500ML | |
| 10 ml serological pipette | Fisher Scientific | 357551 | |
| anti-GFP, rabbit, 488 conjugate | Invitrogen | A21311 | |
| Stereotaxic Surgery Reagents | |||
| Vet-Syringe vet use T.B. Syringe only 1 cc Luer slip T.B. 100/bx | Med-Vet International | 1CCVLS | |
| Isoflurane | |||
| Stainless Steel Scalpel Blades, #10, 100-pk | Med-Vet International | JOR580S | |
| artificial tear ointment | Med-Vet International | RXPARALUBE-O | |
| PVP PrepSolution | Med-Vet International | HPIV108208H | |
| normal saline | Med-Vet International | DYND500MLSH | |
| cotton tipped applicators | Med-Vet International | CTA6 | |
| Triple antibiotic ointment | Med-Vet International | RXTRIP-OI15 | |
| MONOJECT® Needles Soft Pack 25 g x 5/8" | Med-Vet International | 25058 | |
| 6-0 silk sutures | Med-Vet International | MV-711 | |
Riferimenti
- Williams, M. R., DeSpenza, T., Li, M., Gulledge, A. T., Luikart, B. W. Hyperactivity of newborn Pten knock-out neurons results from increased excitatory synaptic drive. J Neurosci. 35 (3), 943-959 (2015).
- Luikart, B. W., et al. Pten knockdown in vivo increases excitatory drive onto dentate granule cells. J Neurosci. 31 (11), 4345-4354 (2011).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol. 32 (4), 347-355 (2014).
- Luikart, B. W., et al. miR-132 mediates the integration of newborn neurons into the adult dentate gyrus. PLoS One. 6 (5), e19077 (2011).
- Nasri, M., Karimi, A., Allahbakhshian Farsani, M. Production, purification and titration of a lentivirus-based vector for gene delivery purposes. Cytotechnology. 66 (6), 1031-1038 (2014).
- Ellis, B. L., Potts, P. R., Porteus, M. H. Creating higher titer lentivirus with caffeine. Hum Gene Ther. 22 (1), 93-100 (2011).
- Fricano, C. J., et al. Fatty acids increase neuronal hypertrophy of Pten knockdown neurons. Front Mol Neurosci. 7, 30 (2014).
- Park, F. Lentiviral vectors: are they the future of animal transgenesis?. Physiol Genomics. 31 (2), 159-173 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon