Method Article
Integración de Procesos húmedo y en seco de banco Optimiza Targeted secuenciación de próxima generación de baja calidad y biopsias de tumores de baja cantidad
En este artículo
Resumen
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
Resumen
Todos los procedimientos de secuenciación de próxima generación (NGS) incluyen ensayos realizados en el banco de laboratorio ( "banco húmedo") y los datos de los análisis realizados en el uso de la bioinformática tuberías ( "banco seco"). Ambos elementos son esenciales para producir resultados precisos y fiables, que son especialmente críticos para los laboratorios clínicos. tecnologías NGS dirigidos, han encontrado cada vez más a favor de las aplicaciones de oncología para ayudar a los objetivos de la medicina de precisión de avance, sin embargo, a menudo los métodos implican desconectado y variable de los flujos de trabajo de banco húmeda y seca y juegos de reactivos no coordinadas. En este informe, se describe un método para la secuenciación de muestras de cáncer de desafiar con un panel de 21 genes como un ejemplo de un sistema integral orientado NGS. El sistema integra la cuantificación de ADN funcional y calificación, de un solo tubo de multiplexado de enriquecimiento PCR, y la purificación de la biblioteca y la normalización utilizando verificadas analíticamente, reactivos de una sola fuente con una suite bioinformática independientes.Como resultado, la variante exacta llamadas de baja calidad y fijado en formalina baja cantidad, incluido en parafina (FFPE) y la aspiración con aguja fina (FNA) biopsias tumorales pueden ser alcanzados. El método se puede evaluar de manera rutinaria variantes asociadas con el cáncer desde una entrada de 400 copias de ADN amplificables, y tiene un diseño modular para adaptarse a los nuevos contenidos de genes. Dos tipos diferentes de controles definidos analíticamente proporcionan aseguramiento de la calidad y la precisión de llamadas ayudan a proteger con muestras clínicamente relevantes. Una "etiqueta" etapa de PCR flexibles incrusta adaptadores específicos de la plataforma y los códigos de índice para permitir que los códigos de barras de la muestra y la compatibilidad con los instrumentos de sobremesa común NGS. Es importante destacar que el protocolo es más sencillo y puede producir 24 bancos de secuencias listo en un solo día. Por último, el enfoque vincula los procesos de banco húmedas y secas mediante la incorporación de los resultados de control de calidad de la muestra pre-analítica directamente en la variante llamar algoritmos para mejorar la precisión de la detección de mutaciones y diferenciar falsos negativos y indeterminate llamadas. Este método utiliza NGS dirigida avances tanto en wetware y software para lograr una alta profundidad, la secuenciación y el análisis multiplexado sensible de las muestras de cáncer heterogéneos para aplicaciones de diagnóstico.
Introducción
la medicina de precisión se basa en la individualización de las opciones diagnósticas y terapéuticas para los pacientes. La promesa de tratamientos a medida es una consecuencia directa de una mejor comprensión de las vías de la enfermedad que pueden informar a la vinculación de diagnóstico molecular y terapias dirigidas. Por ejemplo, el uso de terapias molecularmente dirigidas aumentó del 11% al 46% desde 2003 hasta 2013 1, y fármacos contra el cáncer tales como vemurafenib y crizotinib se aprobado por la FDA con las pruebas diagnósticas complementarias. Con su capacidad de recuperar con precisión los objetivos de secuencias de baja abundancia en todos los grupos de muestras altamente multiplexados, secuenciación de próxima generación (NGS) ha emergido como un método de elección para la evaluación de las aberraciones genéticas asociadas con el cáncer y la identificación de dianas moleculares para la medicina de precisión.
Las biopsias de tumores sólidos más comunes para las pruebas moleculares incluyen fijado en formol e incluido en parafina (FFPE) y la aspiración con aguja fina (FNA) uestraens. Estas muestras están llenos de ácidos nucleicos que desafían evaluaciones precisas NGS 2-5 de baja cantidad y / o de baja calidad. Los actuales métodos de NGS comerciales para el análisis de estas muestras se basan en un mosaico de diferentes reactivos, protocolos y herramientas informáticas que representan blancos móviles de mejoras continuas. Por ejemplo, los cambios en la química y / o software de ensayo se produjo cada 1-2 meses para los kits de NGS específicas de uso más común 6. Esta inestabilidad refleja una falta de coherencia en la construcción y la verificación de un sistema unificado para NGS tipos de muestras difíciles, sobre todo para las pruebas de cáncer, y pone una carga excesiva a los laboratorios para desarrollar protocolos cohesivos que están optimizados de la muestra a los resultados. De hecho, una reciente encuesta de usuarios NGS puso de relieve las dificultades de estas "tecnologías que cambian rápidamente", junto con los requisitos establecidos para el contenido, punto de vista médico-procesable, experiencia bioinformática arraigada, una solidified y procedimiento integrado que puede ser implementado rápidamente, y simplificados flujos de trabajo y protocolos simplificados que faciliten en el puesto de trabajo 7. En este artículo, se describe un sistema integral de NGS dirigida que se ocupa de estas lagunas.
La metodología que se presenta integra todos los pasos del procedimiento de pre-analítica a la post-analítica en bancos tanto húmedas como secas para mejorar la precisión, sensibilidad y fiabilidad de la cuantificación de destino y la detección de NGS de loci de genes del cáncer clínicamente relevante. Este enfoque comienza con la cuantificación de ADN "funcional" 4 para evaluar la calidad del ADN, la guía de entrada a la etapa de enriquecimiento PCR, y proteger contra llamadas de falsos positivos que pueden derivarse de la interrogación de copias muy bajos plantilla. Un múltiplex de un solo tubo de PCR a continuación, enriquece el 46 loci en 21 genes del cáncer utilizando sólo 400 copias de ADN amplificables, seguido por la incorporación de secuencias específicas de la plataforma para NGS using escritorio comunes instrumentos de secuenciación. Las bibliotecas se purificaron usando un simple procedimiento de perlas magnéticas y se cuantificaron con una novela, ensayo qPCR libre de calibración. Una suite bioinformática independientes, informado por la muestra de ADN resultados de CC para mejorar el desempeño de la llamada, proporciona análisis de la secuencia siguiente NGS. Se presentan los datos que utilizan este enfoque de sistemas para NGS dirigida a revelar base de mutaciones de sustitución, inserción / deleciones (indeles), y número de copia variantes (CNV) en biopsias de baja calidad y tumorales de baja cantidad como FFPE y FNA muestras, y correr controles.
Protocolo
Nota: Este protocolo describe el procesamiento simultáneo de las muestras usando un sistema de MiSeq NGS pero puede ser adaptado para el instrumento Personal Genome Machine (PGM). Para la entrada de ADN mínima recomendada de 400 copias de la plantilla amplificables, el ensayo es capaz de producir al menos 3,000x cobertura mediana para cada una de 96 muestras por NGS plazo, y la profundidad de cobertura equivalente para 24 muestras utilizando el PGM en un chip 318. El método también requiere el uso de un instrumento de PCR en tiempo real.
1. Cuantificación de ADN funcional y control de calidad (QC)
- reactivos de descongelación: 2x Master Mix, Primer Mezcla de sondas, Inhibición Primer Mezcla de sondas, 6-carboxi-X-rodamina (ROX), diluyente, y los cuatro estándares de la curva de calibración de ADN genómico humano (estándar de ADN (50 ng / l), el ADN estándar (10 ng / l), Standard DNA (2 ng / l), y Estándar de ADN (0,4 ng / l)) (Tabla 1). Vórtex todos los reactivos para 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.Mantener la mezcla maestra 2x en hielo.
- Preparar una cantidad suficiente de mezcla maestra para el número total de muestras a ensayar e incluyen 10% más de volumen para evitar la escasez debido a pipeteado. Preparar la mezcla maestra en un tubo de microcentrífuga usando los siguientes volúmenes por ejemplo: 5 l 2x Master Mix, 0,5 l Primer Mezcla de sondas, 0,5 l Inhibición Primer Mezcla de sondas, 0,05 y 2,95 l ROX diluyente l. Vortex durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Añadir 9 l mezcla maestra en pocillos de una placa de 96 pocillos.
- Añadir 1 l de los patrones de ADN por duplicado para generar una curva de calibración. Mezclar pipeteando arriba y abajo 5 veces.
- Asegúrese de que la muestra de ácido nucleico se mezcla bien antes de su uso. Añadir 1 l de muestra a la mezcla principal y mezclar pipeteando arriba y abajo 5 veces.
- Sellar la placa, vórtice durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Colocar la placa en el Sistema de PCR. Asignar tanto FAM (cuantificación funcional) y VIC (inhibición funcional) detectores para cada muestra de acuerdo con las instrucciones del fabricante. Realizar ciclos de PCR de 10 min a 95 ° C, y 40 ciclos (15 segundos a 95 ° C, 1 min a 60 ° C).
- Analizar los datos qPCR mediante la generación de un gráfico de regresión lineal para cada uno de los patrones de ADN duplicadas utilizando protocolos de software.
- Trazar el registro 10 del número de copias para cada estándar de ADN en el eje X y el correspondiente valor de q FAM C en el eje y.
- Confirmar que los resultados de la caída muestra de ácido nucleico dentro de la gama dinámica de la curva de calibración de patrones de ADN, y luego calcular la concentración del ADN desconocido en el número de copias "funcional" o amplificable por l de su posición correspondiente en la curva patrón de referencia. La Figura 1 muestra ejemplos de curvas de calibración que han pasado y han fracasado.
- Determinar si la amplificación se produjo en cada reacción mediante la comprobación de la presencia del amplicón diana no humano en el canal de VIC.
Nota: Como control positivo, la sonda Mix Inhibición cebador contiene cebadores específicos para una diana exógeno no humana y la plantilla correspondiente. El Primer Mezcla de sondas de inhibición es un componente de la mezcla maestra que se añade a cada reacción, incluyendo el control sin plantilla (NTC). En ausencia de un inhibidor, el producto de PCR para el objetivo no humano siempre debe ser detectada en el canal de VIC. Un q "no detectado" C para una muestra en el canal de VIC indica la presencia de inhibidores de la PCR que pueden beneficiarse de la posterior limpieza de la muestra antes de su procesamiento posterior.
2. Biblioteca Preparación: Gen-específica (SG) PCR
- Preparar una cantidad suficiente de mezcla maestra para el número total de muestras del ensayo e incluyen 10% más de volumen para evitar la escasez debido a pipeteeEn g. Preparar la mezcla maestra GS PCR en un tubo de microcentrífuga usando los siguientes volúmenes por muestra: 5 l 2x de amplificación Master Mix (Tabla 1) y 1 l Panel Primer cáncer Pan (Tabla 1). Mezclar pipeteando arriba y hacia abajo, vórtice durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Alícuota de la 6 l GS PCR master mix en pocillos de una placa de 96 pocillos. Añadir 4 l de cada muestra de ácido nucleico en pocillos individuales. Para otros pozos, añadir 4 l de la FFPE de control (Tabla 1), 4 l del control de múltiples variables (Tabla 1), y 4 l de agua libre de nucleasa para un procedimiento de NTC. Para cada adición, la mezcla pipeteando arriba y abajo 5 veces.
- Sellar la placa, vórtice durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Colocar la placa en el termociclador para los siguientes ciclos de PCR: 5 min a 95 ° C,2 ciclos (15 segundos a 95 ° C, 4 min a 60 ° C), 23 ciclos (15 segundos a 95 ° C, 4 min a 72 ° C), y una extensión final de 10 min a 72 ° C. Mantener a 4 ° C.
Nota: Después de la terminación de la etapa 2.4, se hará referencia a la placa como la placa de PCR GS.
3. Biblioteca Preparación: Etiqueta de PCR
- Descongelar los reactivos: 2x Índice de Master Mix (Tabla 1) y Códigos de Clasificación (Tabla 1). Vortex durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
Nota: Códigos de Clasificación se mezclan previamente para proporcionar un conjunto único de índices pares (códigos de barras) para cada muestra. - En una placa de 96 pocillos, añadir 7,5 l de la mezcla maestra Índice de 2x y 5,5 l de un código de índice para un determinado bien y mezclar con la pipeta hacia arriba y hacia abajo 5 veces.
- Abrir con cuidado la placa de PCR GS, y añadir 2 l de producto de PCR GS a la nueva placa con la mezcla maestra. Mezclar pipeteando arriba y abajo 5 veces. Para cada muestra, registrar el ID de la muestra y los correspondientes Códigos de Clasificación por pares. Sellar la placa, vórtice durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Colocar la placa en el termociclador y PCR durante 5 minutos a 95 ° C, 10 ciclos (30 segundos a 95 ° C, 30 seg a 55 ° C, 1 min a 72 ° C), y una extensión final de 10 min a 72 DO. Mantener a 4 ° C.
Nota: Después de la terminación de la etapa 3.4, se hará referencia a la placa como la placa de PCR Tag.
4. Biblioteca Purificación y Selección del tamaño
- Retire las perlas magnéticas Biblioteca Pure Prep (Tabla 1) y Tampón de Elución (Tabla 1) 2-8 ° C y permitir que se equilibre a temperatura ambiente durante 30 min. Añadir 9,6 ml de etanol al 100% a la solución de lavado (tabla 1) contenedor, tapa y mezcle invirtiendo el frasco varias veces.
- Vortex las perlas magnéticas durante 10 segundos y añadir 11 l en pocillos separados de un 9placa de 6 pocillos.
- Abra la placa de PCR de etiquetas y añadir 10 l de producto de PCR Tag hasta el talón y pipeta de mezcla 5 veces. Incubar la mezcla durante 4 min a TA.
- Coloque la placa de 96 pocillos en el soporte magnético (Tabla 1) durante 4 min. Con la placa de 96 pocillos todavía en el soporte, retirar y desechar el sobrenadante con una pipeta.
- Retire la placa de 96 pocillos del soporte magnético y se añaden 100 que contiene etanol l de tampón de lavado a cada pocillo y mezclar con la pipeta hacia arriba y hacia abajo 5 veces. Incubar durante 2 min.
- Coloque la placa de 96 pocillos en el soporte magnético durante 2 minutos, luego retirar y desechar el sobrenadante con una pipeta.
- Repita el paso 4.5, para un total de 2 lavados con etanol, la eliminación de la solución de lavado lo más posible después del segundo lavado.
- Con la placa de 96 pocillos en el soporte magnético, se secan los granos durante 2 minutos a temperatura ambiente, a continuación, retire la placa de la base.
- Resuspender las perlas mediante la adición de 20 l tampón de elución a cada well y pipetear hacia arriba y hacia abajo 5 veces.
- Incubar durante 2 min a TA.
- Coloque la placa de 96 pocillos de nuevo en el soporte magnético durante 4 minutos, y retirar con cuidado y transferir 18 l del sobrenadante claro para un nuevo pozo.
Nota: El procedimiento se puede detener de forma segura en este paso y las muestras se almacenaron a -15 hasta -30 ° C. Para reiniciar, descongelar las muestras congeladas en el hielo antes de proceder.
5. Cuantificación Biblioteca
- Descongelar la Biblioteca Quant (LQ) Reactivos: 2x LQ Master Mix, LQ cebadores / sondas Mix, LQ estándar, control positivo LQ, LQ diluyente y LQ ROX (Tabla 1). Vortex durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- El uso de productos purificados de la biblioteca, lleve a cabo una serie de dilución de cada muestra individual en LQ diluyente.
- Añadir 2 l (producto purificado biblioteca) a 198 l LQ Diluyente y mezclar hacia arriba y hacia abajo con una pipeta de 10 veces.
- Añadir 2 l (1: 100 dilución) a 198 l LQ Diluyente y mezclar hacia arriba y hacia abajo con una pipeta de 10 veces.
- Preparar una cantidad suficiente de LQ mezcla maestra para el número total de muestras a ensayar e incluyen 10% más de volumen para evitar la escasez debido a pipeteado. Preparar la mezcla de LQ Maestro en un tubo de microcentrífuga usando los siguientes volúmenes por ejemplo: 5 l 2X LQ Master Mix, 2 l LQ Primer / Mezcla de sondas, 0,5 l LQ estándar y 0,5 l LQ ROX. Mezclar pipeteando arriba y hacia abajo, vórtice durante 10 segundos y centrifugar a máxima velocidad durante 10 segundos para recoger contenidos.
- Añadir 8 l maestro de la mezcla LQ a un pocillo de una placa de 96 pocillos óptica.
- En pocillos separados, añadir 2 l biblioteca diluida, 2 l LQ control positivo y 2 l LQ diluyente (NTC) y mezclar pipeteando arriba y abajo 5 veces. Sellar la placa con película adhesiva óptica, vórtice durante 10 segundos y se centrifuga a 400 g durante 10 segundos para recoger contenidos.
- Asignar tanto FAM y VIC Detectors para cada muestra. Realizar amplificación por PCR utilizando las condiciones del ciclo de 5 min a 95 ° C, y 40 ciclos (15 segundos a 95 ° C, 1 min a 60 ° C).
- Determinar la concentración de cada muestra (nM) usando el método C q comparativa. Calcular la diferencia de la conocida LQ Estándar (C q VIC) a la biblioteca desconocida (C q FAM). La concentración de la muestra diluida (PM) se calcula utilizando la siguiente ecuación:
[Lib Conc] nM = 12,5 x 2 Δ Cq
Si se utiliza un factor de dilución que no sea 10.000, calcular la relación entre el factor de dilución objetivo a 10.000, y multiplicar este factor por el resultado de la ecuación en 5.7.
6. Biblioteca de Normalización y la agrupación de la muestra
- Determinar la mediana de la concentración (nM) en todas las muestras (cada uno que contiene un índice único pairwise) para su agrupación.
- Determinar el volum muestra individuale (l) a la piscina multiplicando la concentración media en todas las muestras en un 5, luego dividiendo por su concentración específica (nM). Ronda el valor resultante al entero más cercano. volúmenes redondos con los valores de <2 l a 2 l, y los volúmenes> 15 l a 15 l.
- Añadir el volumen normalizado (l) para cada muestra a un solo tubo de microcentrífuga para crear el conjunto de muestras.
- Calcular la nueva concentración para cada muestra usando valores enteros redondeados y registrar los resultados.
- Para determinar la concentración de la piscina de la muestra, calcular la suma de todas las concentraciones individuales y registrar el valor resultante (nM).
- Diluir el conjunto de muestras a 1,25 nM usando secuenciación Diluyente (Tabla 1).
Nota: El procedimiento se puede detener de forma segura en este paso y las muestras se almacenaron a -15 hasta -30 ° C. Para reiniciar, descongelar las muestras congeladas en el hielo antes de proceder.
7. Secuenciación
- denature el conjunto de muestras en presencia de v3 control PhiX (Tabla 1) mediante la adición de los siguientes: 15 l de 1,25 nM conjunto de muestras, 3 l de 0,5 nM PhiX y 2 l de NaOH 1 N. Vortex seguido brevemente por una breve centrifugación y se incuba durante 5 min a TA.
- Coloque la piscina muestra desnaturalizada en hielo.
- Añadir 8 l de biblioteca desnaturalizado a 992 l de tampón de HT1-Hyb pre-refrigerada a un tubo de microcentrífuga. Vortex brevemente para mezclar, seguido de una breve centrifugación para recoger contenidos. Mantener en hielo.
- Añadir 600 l de la biblioteca desnaturalizado y se diluyó a la posición # 17 del cartucho de reactivo.
- Descongelar Lea 1 Secuenciación Los cebadores (Tabla 1), el índice de lectura de secuenciación cebadores (Tabla 1), y leer 2 cebadores de secuenciación (Tabla 1). En tubos de microcentrífuga, diluir separado 4 l cebadores de secuenciación con 636 l de tampón HT1-Hyb. Nota: tampón HT1-Hyb es proporcionado ingenioh el kit de reactivos de secuenciación (Tabla 1).
- Mezclar mediante agitación durante 10 s y se centrifuga a velocidad máxima durante 10 segundos para recoger contenidos. Añadir 600 l de diluidas Lea 1 Secuenciación cebadores para la posición # 18 del cartucho de reactivos de secuenciación, 600 l de cebadores diluidos de índice leídos a la posición # 19 y 600 l de cebadores de secuenciación Leer 2 diluidas a la posición # 20.
- Cargar los reactivos sobre el instrumento NGS (Tabla 1) y la secuencia de acuerdo con las instrucciones del fabricante. Realizar una gama emparejado 2 x 150 ejecución de secuenciación cíclica.
Análisis de datos 8.
Nota: El software del instrumento NGS convierte las imágenes en racimo a base de las llamadas y sus niveles de calidad, y demultiplexa índices pares para generar FASTQ comprimida con gzip individuo (* .fastq.gz) archivos para cada muestra. Antes de analizar los archivos desmultiplexadas, el lector debe descargar e instalar el software de bioinformática asociada (tabla1). El software puede ser instalado en un grado de consumo de Windows PC y no requiere hardware de computación especializada o una conexión a Internet para realizar el análisis de datos.
- Haga doble clic en el icono del escritorio de software.
- Entrar al sistema con el nombre de usuario y la contraseña proporcionada en el manual del software.
- Abra el panel de control del proyecto y haga clic en "Nuevo proyecto".
- Nombre del proyecto y proporcionar una descripción del proyecto opcional. Seleccione el tipo de panel de NGS NGS objetivo y tipo de instrumento. Haga clic en "Guardar y continuar".
- Sube los archivos FASTQ comprimido para el avance y retroceso lee. No cargue las FASTQs "sin asignar", que contienen lee que fallaron para demultiplexar. Haga clic en "Guardar y continuar".
- Introduzca el número de copias funcionales de entrada utilizados para preparar cada biblioteca como se determina mediante el ensayo de cuantificación de ADN funcional (ver paso 1). añadir manualmente los valores o los valores de copiar y pegar de una propagaciónhoja en la mesa de anotación. Haga clic en "Guardar y continuar".
- Revisar las bibliotecas anotado subidas para el análisis y haga clic en "Enviar el análisis" para iniciar el análisis.
- Controlar el progreso de los análisis que se muestra a través del tablero de instrumentos proyecto.
Nota: Una indicación de estado de finalización análisis se presenta cuando los resultados están listos para su revisión. - Revisar los resultados analizados para la muestra métricas de control de calidad, incluyendo la cobertura total por la biblioteca, lee porcentaje de filtros de paso, profundidad de la cobertura de amplificación y la uniformidad. Revisar la variante de llamadas para cada biblioteca secuenciado con dbSNP, cósmico, 1.000 genomas y otras fuentes de anotación funcional y la población de nivel.
- Exportar los resultados brutos como tablas de resumen de hoja de cálculo, archivos * .bam y archivos * .vcf para el almacenamiento a largo plazo o análisis de aguas abajo con herramientas informáticas complementarias.
Resultados
Un total de 90 muestras (74 único) que representan controles positivos y negativos, líneas celulares previamente caracterizadas, y biopsias tumorales FFPE clínicos residuales se evaluaron para el ADN amplificable, de entrada en multiplex PCR de enriquecimiento, etiquetadas con adaptadores de secuencia, con código de barras, y se analizó en un solo NGS mesa de trabajo de ejecución del instrumento (Figura 2) que produjo 19,1 M lee pasando filtro. la puesta en común de la muestra equimolar resultó en la secuenciación de profundidad alta (3,692x lecturas) y la cobertura uniforme (97,8% de los amplicones cubiertos dentro de 5 veces de la profundidad de lectura mediana). Los valores atípicos compuestos no-plantilla controles, uno de ADN de la línea celular con una gran amplificación del número de copias, y una DNA FFPE que se encuentra en posición de inhibición de la PCR por el ensayo de control de calidad pre-analítica (Figura 3). Uniformidad de la cobertura en los 46 amplicones se mantuvo mediante tres operadores diferentes (Figura 4A), y para diferentes muestras de ADN FFPE de baja calidad ( Figura 4B). Un control de ADN tumoral FFPE, formulada a partir de una mezcla de muestras clínicas residuales para lograr 5% V600E BRAF (cuantificado por gotita PCR digital), se informó de que el objetivo BRAF mutación en abundancias de 3,9, 5,3, y 6,5% en tres operadores usando una entrada de 400 copias amplificables (y por lo tanto sólo 20 copias mutantes) (Figura 4B y "FFPE3" de la tabla del recuadro). Además, una mezcla de 12 plantillas de ADN sintético, cada uno representando un conocido "controlador" base de mutación de sustitución, reveló las mutaciones esperadas en el rango deseado 9 - 17% la frecuencia de alelos media (Tabla 2). La dilución de la línea celular y las muestras de ADN FFPE con amplificaciones número de copias demostró dependencia de la dosis para las variantes en EGFR y KRAS, respectivamente (Figura 5). Es importante destacar que, de entrada de ADN FFPE podría reducirse a tan sólo 50 copias amplificables o 1,2 ng de DNA mayor preservando al mismo tiempo la detección de mutaciones conocidas sin falsos positivosllamadas (Figura 6). Entradas de ADN fueron alojados en un rango de 100 veces hasta al menos 50.000 copias amplificables (Tabla 3). En este y otros experimentos, la variante de llamadas en 22 FFPE y no se reportaron 20 FNA especímenes de acuerdo con métodos independientes con cobertura mutación compartida (Tabla 4).
La sensibilidad y el valor predictivo positivo para el ensayo se determinó a partir de un análisis de 97 muestras, incluyendo FFPE, FNA, fresco congelado, y la línea celular de ADN, y un total de 195 resultados de la secuenciación. Los resultados revelaron 365 verdaderos positivos llamadas variantes, 4 llamadas de falsos negativos y falsos positivos 1 llamada para una sensibilidad del 98,9% (IC del 95%: 97,1-99,7%) y un valor predictivo positivo (VPP) del 99,7% (IC del 95% : 98,2 a 99,99%). Los análisis de indeles se realizaron durante dos variantes comunes de EGFR (p.E746_A750delELREA y p.V769_D770insASV) en 33 muestras-corre, lo que demuestra una sensibilidad del 93,9% (IC del 95%: 78,4 a 98,9%) y un VPP del 100% (IC del 95%: 86,3 a 100%) con variantes detectadas en un rango de 2,4 a 84,8%.
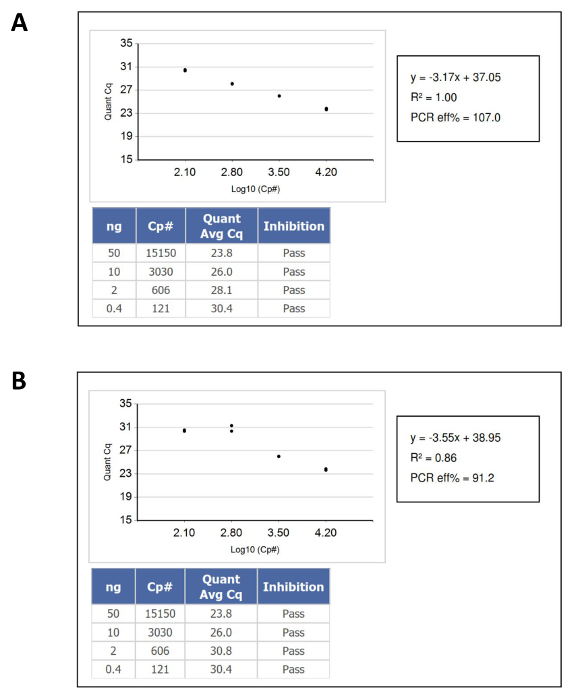
Figura 1: Un ejemplo de curvas de calibración que Cuantificación de ADN de aprobación y rechazo del control de calidad Criterios (A) una curva estándar que pasa.. (B) una curva estándar que falla. En este caso, el fallo fue causado por pipeteo duplicado del patrón de ADN de entrada más bajo. Por favor, haga clic aquí para ver una versión más grande de esta figura.
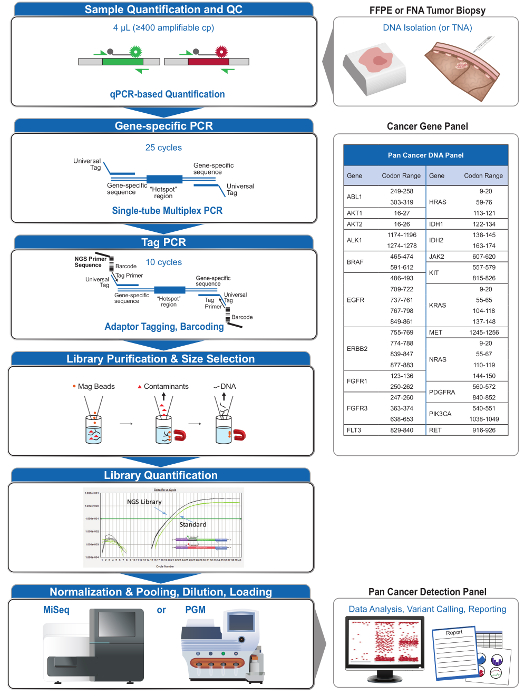
Figura 2: Visión general de un sistema integral dirigida NGS para aplicaciones de oncología que integra preanalíticaLos flujos de trabajo al, analítica y post-analítica. Haga clic aquí para ver una versión más grande de esta figura.
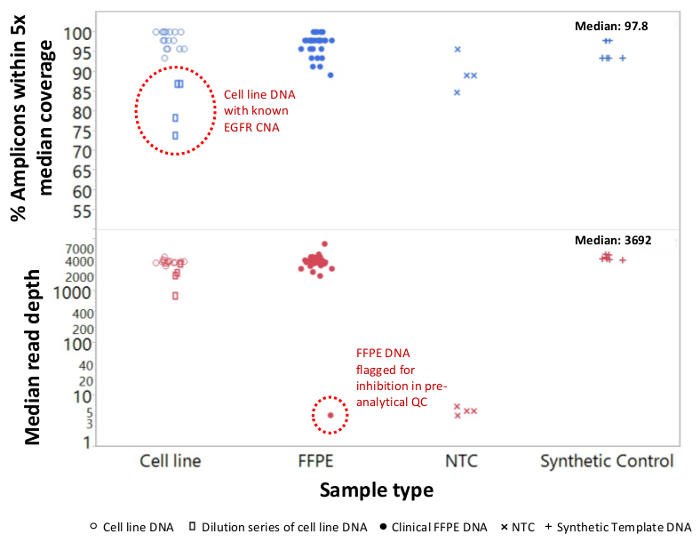
Figura 3: Lee cobertura y uniformidad de NGS dirigida de genes cancerígenos en el ADN FFPE calidad baja en comparación con el ADN y los controles de la línea celular intacto Un total de 90 muestras que incluyeron FFPE clínica residual (círculos cerrados), la línea celular (círculos abiertos. ), y el ADN de molde sintético (más símbolos) se procesaron utilizando un solo tubo, de 21 genes PCR multiplex enriquecimiento. Cada biblioteca de amplificación fue etiquetado con secuencias adaptadoras para el instrumento NGS, con código de barras con un código distinto de doble índice, purificada, cuantificado, y normalizado a una concentración de 2,5 nM. La biblioteca de ADN fue secuenciado y analizado por el software de bioinformática de compañía. Devi muestrasciones incluyen una serie de diluciones de células MDA-MB-468 línea celular de ADN que lleva una amplificación número de copia grande EGFR (arriba, rectángulos abiertos dentro de círculo de puntos) que distorsionaron la uniformidad de cobertura y una muestra de melanoma FFPE que no lograron generar un número apreciable de lecturas debido para el arrastre de los inhibidores de PCR de la extracción de ADN. El fracaso de la muestra de melanoma (abajo, círculo punteado) fue predicho por el ensayo de PCR cuantitativa de ADN de control de calidad preanalítica. NTC, control sin plantilla (x símbolos). Por favor, haga clic aquí para ver una versión más grande de esta figura.
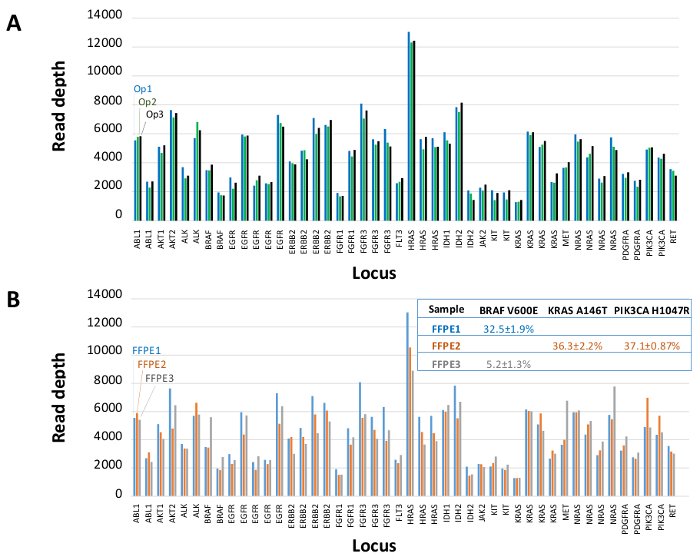
Figura 4: El amplicón por amplicón Lee cobertura, la homogeneidad y la detección de variantes en el ADN tumoral FFPE clínico residual. (A) Leer cobertura en todos los loci enriquecido en una muestra representativa de ADN medido FFPEa través de tres operadores diferentes. Operador 1, Op1 (barras azules); Operador 2, Op2 (barras verdes); Operador 3, Op3 (barras negras). (B) uniformidad de cubrición y variantes llamadas evaluadas utilizando tres muestras tumorales FFPE, incluyendo una mezcla de control (barras grises FFPE3, y el texto) que constan de un conocido 5% BRAF c.1799T> Una mutación. FFPE1 (barras azules y texto), FFPE2 (barras de color naranja y el texto). Haga clic aquí para ver una versión más grande de esta figura.
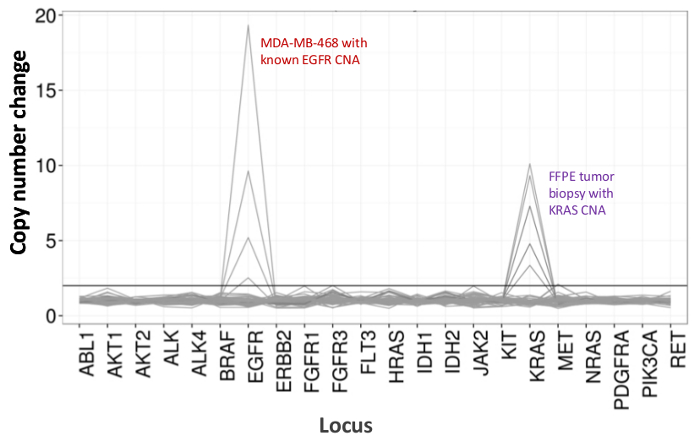
Figura 5:. Detección dependiente de la dosis del número de copias Las variantes en la línea celular y el ADN de la línea celular FFPE ADN MDA-MB-468 con una amplificación del número de copias EGFR bien caracterizado se diluyó progresivamente en un fondo de una celda de referencia no mutado ADN -line para ilustrar la disminución de la copia número cambio como unaen función de la dilución. El porcentaje de cada muestra de ADN de la línea celular se muestra con una línea distinta (0, 12.5, 25, 50, y 100%). La dilución de una muestra de tumor de ovario FFPE con una amplificación del gen KRAS conocida reveló un perfil similar usando la misma serie de titulación pero con repeticiones de ADN FFPE 100% para las dos líneas superiores. Por favor, haga clic aquí para ver una versión más grande de esta figura.
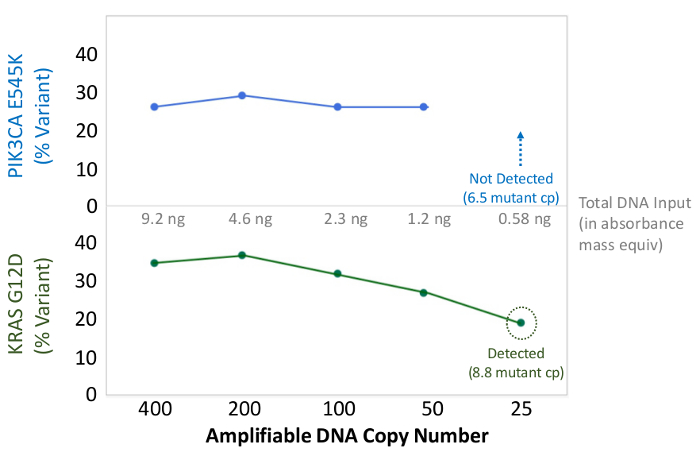
Figura 6: detección precisa de mutación y cuantificación a 50 copias de ADN FFPE amplificable, o 1,2 ng de ADN a granel El número de copias amplificable de un cáncer de colon ADN FFPE se determinó por el ensayo de control de calidad basado en la qPCR, y se diluye de 400 a 25 copias como un. entrada en multiplex PCR de enriquecimiento antes de la secuenciación. El gasoducto bioinformática llama correctamente tanto de la conocida Varianct hasta 50 copias, o el equivalente a ~ 10 plantillas de mutantes. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre del material / material | Empresa | Numero de catalogo |
| 2x Mezcla Maestra Quantidex | Asuragen | 145.345 |
| Quant mezcla de iniciador de la sonda | Asuragen | 145.336 |
| La inhibición mezcla de iniciador de la sonda | Asuragen | 145344 |
| ROX | Asuragen | 145.346 |
| Diluente | Asuragen | 145339 |
| ADN estándar (50) | Asuragen | 145340 |
| ADN estándar (10) | Asuragen | 145.341 |
| ADN estándar (2) | Asuragen | 145.342 |
| ADN estándar (0,4) | Asuragen | 145.343 |
| 2x mezcla de amplificación Maestro | Asuragen | 145.348 |
| Grupo Pan cáncer cartilla | Asuragen | 145.347 |
| Control del movimiento del cáncer FFPE | Asuragen | 145349 |
| Control del movimiento del cáncer de Multi-Variant | Asuragen | 145350 |
| Perlas de Prep Pure biblioteca | Asuragen | 145.351 |
| Tampón de Lavado | Asuragen | 145352 |
| tampón de elución | Asuragen | 145353 |
| 2x Mezcla Maestra LQ | Asuragen | 145.358 |
| LQ diluyente | Asuragen | 145354 |
| LQ positivoControlar | Asuragen | 145.355 |
| LQ Estándar | Asuragen | 145.356 |
| LQ Primer / Mezcla de sondas (ILMN) | Asuragen | 145.357 |
| LQ ROX | Asuragen | 145359 |
| Códigos de Clasificación (ILMN) - Set A | Asuragen | 150004 |
| AIL001 - AIL048 (48) | ||
| Códigos de Clasificación (ILMN) - Grupo B | Asuragen | 150005 |
| AIL049 - AIL096 (48) | ||
| 2x Mix Master Index | Asuragen | 145.361 |
| Leer 1 Secuenciación Los cebadores | Asuragen | 150001 |
| Leer índice de secuenciación cebadores | Asuragen | 150002 |
| Leer 2 Secuenciación Los cebadores | Asuragen | 150003 |
| Diluyente de secuenciación | Asuragen | 145.365 |
| Illumina MiSeq | Illumina | |
| MiSeq Reactivo v3 Kit (600-ciclo) | Illumina | MS-102-3003 |
| Kit Reactivo MiSeq Nano v2 (300-ciclo) | Illumina | MS-103-1001 |
| PhiX control v3 | Illumina | FC-110-3001 |
| Stand-96 magnética (o equivalente) | Ambion | AM10027 |
| Software Reportero Quantidex | Asuragen |
Tabla 1:. Reactivos y Kits con el primer uso de la ROX, almacene el vial a 2-8 ° C. No vuelva a congelar. El software puede ser descargado en www.asuragen.com.
| GRAMOeno | variante COSMIC | Aminoácido COSMIC | Variante% |
| ANR | c.182A> G | p.Q61R | 13.3 |
| ANR | c.35G> A | p.G12D | 15.2 |
| HRAS | c.182A> G | p.Q61R | 17.8 |
| HRAS | c.35G> A | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G> A | p.G12D | 19.1 |
| PIK3CA | c.1633G> A | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| EQUIPO | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14.9 |
| BRAF | c.1799T> A | p.V600E | 17.3 |
Tabla 2: Un sintético de Control de Fondos Comunes está compuesta por 12 "controlador" gen del cáncer de variantes que son cuantificados al 9-17% Abundancia Se evaluó una mezcla de 12 plantillas sintéticos de doble cadena diferentes que llevan 12 mutaciones distintas siguiente secuenciación.. Todas las variantes se denominan correctamente sin falsos positivos.
>% Variant| ID de la muestra | Funcional cps | Gene | variante COSMIC | Aminoácido COSMIC | La mediana de la profundidad de lectura | % Dentro de 5x de la mediana | |
| BCPAP | 400 | BRAF | c.1799T> A | p.V600E | 99.5 | 3289 | 96% |
| BCPAP | 10.000 | BRAF | c.1799T> A | p.V600E | 99.7 | 4040 | 98% |
| BCPAP | 25.000 | BRAF | c.1799T> A | p.V600E | 99.4 | 3687 | 96% |
| BCPAP | 50.000 | BRAF | c.1799T> A | p.V600E | 99.7 | 4611 | 93% |
Tabla 3: Cobertura de llamada y la variante se conservan en un rango de> 100 veces de entrada de ADN. ADN amplificable de una línea celular BCPAP usado como entrada multiplex PCR de enriquecimiento de 400 a 50.000 copias y secuenciado. Leer la profundidad, la uniformidad de la cobertura, la detección de mutaciones, y la precisión de mutación se conservaron en toda la gama de entrada.
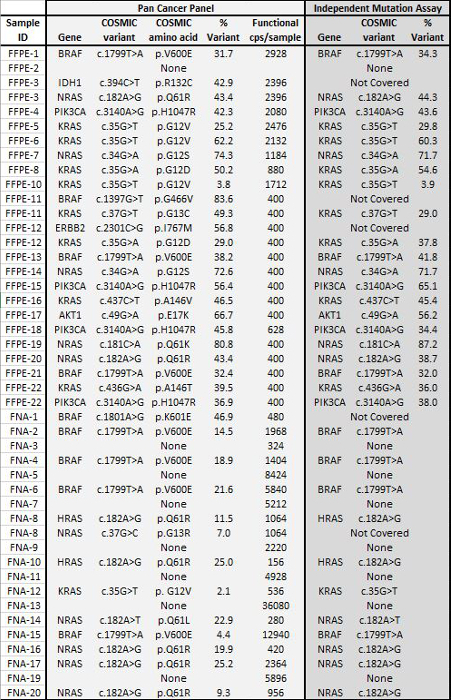
Tabla 4: Variante de llamadas en 22 FFPE y 20 biopsias de tumores FNA De acuerdo con los resultados de Independiente Mutación ensayos, un conjunto de 22 ADN tumoral FFPE con el estado de mutación previamente determinada por ensayos de NGS dirigidas ortogonales era de entrada de 400 a 2.928 copias amplificables en el enriquecimiento de la PCR. paso y se secuenciaron utilizando el panel de cáncer de Pan de 21 genes. Además, una cohorte de 20 muestras de ADN FNA previamente caracteriza el uso de un ensayo de mutación de bolas de serie líquido 8 se amplificó por PCR usando 156 a 36.080 copias de entrada amplificables y secuenciado. Todas las llamadas se superponen entre el panel de Pan cáncer NGS y el referemétodos NCE estaban de acuerdo. Por favor, haga clic aquí para ver una versión más grande de esta figura.
Discusión
Tecnologías NGS han redefinido las expectativas para interrogar a los perfiles moleculares de las biopsias de tumores en el ámbito clínico 9. Una serie de paneles orientados NGS se han desarrollado como tecnologías de investigación, pruebas de laboratorio desarrollado, y los productos disponibles en el mercado y paneles personalizados para la evaluación de múltiples tipos de muestras clínicas 3,5,10-15. Los informes de múltiples estudios han demostrado el valor de NGS como herramienta clínica sensible y específico para la detección de alteraciones genómicas 3,10-12,14. Sin embargo, los estudios también han demostrado el riesgo de artefactos que pueden causar resultados falsos positivos de las biopsias de cáncer desafiantes como FFPE especímenes 2-5,12,14. Además, las publicaciones recientes han puesto de relieve altas tasas de fracaso para NGS utilizando especímenes de oncología y 16 falsos positivos llamadas con paneles NGS dirigidos comerciales que se ven agravadas por el uso de ADN de bajos insumos 12. Como resultado, algunos laboratorios han modificado o añadido pesos y contrapesos adicionales para las tecnologías comercialmente disponibles NGS dirigidos a mejorar el rendimiento, a menudo para asegurar la exactitud o confirmar los hallazgos del análisis bioinformático 5,10,11 tampoco.
El panel de 21 genes (Figura 2) fue desarrollado como el contenido destinado para un sistema integral de NGS para interrogar, mutaciones de acciones concretas basadas en la evidencia en los tipos de muestras complejas, como FFPE y biopsias de tumores FNA. El flujo de trabajo tiene una serie de ventajas: 1) la coherencia proporcionando una cobertura uniforme de amplificación (Figuras 3 y 4, Tabla 3); 2) la facilidad de uso, proporcionando pre-formulado, conjuntos de reactivos optimizados, y la simplificación de los requisitos de la bioinformática; 3) la eficiencia mediante la racionalización del flujo de trabajo y reducir el número de pasos de pipeteado en comparación con otros métodos de NGS comerciales; y 4) la precisión mediante la incorporación de un ensayo de ADN QC para la evaluación de la amplifiel número de copias de ADN capaz de asegurar la diversidad plantilla aceptable y evitar fluctuaciones estocásticas en la detección de la variante 4. La integración de los datos de control de calidad preanalítica con el análisis bioinformática permite entradas de baja de FFPE de ADN. Esto se logró mediante la formación de un algoritmo de árbol de decisiones utilizando diferentes copias funcionales de entrada de ADN a través de 400 muestras FFPE con medidas independientes de la verdad, y la incorporación de este algoritmo en el software bioinformático. Como resultado, la entrada recomendada de 400 copias amplificables, típicamente equivalentes a ~ 5-20 ng de ADN FFPE, se compara favorablemente con otros métodos 10-12, incluido el enriquecimiento basado en la hibridación que se recomienda ~ 250 ng de ADN FFPE 17,18 . Aunque la técnica se describe para su uso en una plataforma MiSeq, se puede modificar mediante el uso de cebadores de PCR de la etiqueta con los adaptadores específicos de instrumentos para permitir el análisis de secuencia en otras plataformas NGS.
Varios pasos son críticos para asegurar el éxitodel procedimiento. El ensayo de control de calidad pre-analítica determina el número de copias del ADN amplificable y los informes de la inhibición funcional. Sin embargo, si se utilizan menos de 400 copias de ADN amplificables en la etapa de enriquecimiento PCR, existe un mayor riesgo de una llamada de falsos negativos de muestras con mutaciones de baja abundancia (Figura 6). Además, se debe tener cuidado durante la purificación biblioteca para evitar el exceso de secado de las perlas magnéticas durante el lavado o elución pasos. Además, el éxito de cuantificación biblioteca depende de la dilución precisa de ADN de biblioteca de alta. Para el mejor resultado, la diferencia resulta de la qPCR para la biblioteca de la muestra (Cq FAM) en comparación con el estándar LQ (Cq VIC) debe ser ≤3.3 Cq. Si la diferencia es mayor que 3,3 Cq, re-dilución y ensayo de la muestra se recomienda. Aunque una correlación excelente se ha observado entre este método qPCR competitivo y kits comerciales que ofrecen cuantificación absoluta utilizando una curva estándar, Un desplazamiento de la entrada de la biblioteca en la etapa de amplificación clonal en relación con otros métodos puede ser necesaria para lograr densidad de siembra óptima.
Algunos especímenes de cáncer son particularmente difíciles de secuenciar debido a que persisten después de inhibidores de aislamiento de ADN. Para identificar estas muestras antes de la preparación de la biblioteca, el ensayo qPCR QC también detecta la inhibición de amplificación mediante la inclusión de una plantilla exógeno que sirve tanto como un control interno y un centinela para la inhibición funcional. Un ejemplo se presenta en la Figura 3 en la que una muestra de ADN melanoma no pudo pasar la inhibición del control de calidad métrica antes de la secuenciación y luego no pudo generar una biblioteca que podrían ser secuenciado. El fracaso fue probablemente una consecuencia de la contaminación por melanina, un inhibidor conocido de PCR, transferidos de la etapa de aislamiento de ADN FFPE. Las muestras que se identifican mediante el ensayo de control de calidad para estar en riesgo de fracaso de amplificación puede ser salvado a través de un extra de limpieza de paso to eliminar inhibidores potenciales.
El panel de 21 genes objetivo se centra en puntos calientes de genes basados en la evidencia y proporciona un sistema completo con reactivos y controles optimizados para el ADN de control de calidad, NGS y la bioinformática software que se informó por resultados de la cuantificación de ADN pre-analíticos "funcionales". El método detecta con precisión mutaciones base de sustitución y indeles a partir del ADN bajo de entrada, y proporciona un ejemplo de un sistema de NGS con la opción de ampliar contenido del panel, para detectar variantes adicionales, tales como CNV y ser adaptado para la secuenciación de ARN objetivo.
Divulgaciones
JH, AH, RZ, BCH, y GJL son empleados y tienen la propiedad de acciones en Asuragen, Inc. RZ, BCH, y GJL son co-inventores en una solicitud de patente para la mejora de la variante llamada utilizando la información de número de copias amplificable determinado para cada muestra.
Agradecimientos
Agradecemos al Dr. Annette Schlageter para la revisión del manuscrito. Este trabajo fue apoyado en parte por CP120017 subvención de la Prevención del Cáncer y el Instituto de Investigación de Texas (PI: GJL).
Materiales
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
Referencias
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados