Method Article
ウェットとドライベンチ・プロセスの統合は、低品質と低量腫瘍生検の標的次世代シーケンシングを最適化
要約
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
要約
すべての次世代シーケンシング(NGS)の手順は、バイオインフォマティクスパイプライン(「ドライベンチ」)を用いて行わ分析(「ウェットベンチ」)とデータを実験台で行うアッセイが含まれます。両方の要素は、臨床検査室にとって特に重要であり、正確で信頼性の高い結果を生成するために必須です。ターゲットを絞ったNGS技術はますます事前精密薬の目標を支援するために腫瘍学のアプリケーションに好意を発見した、まだ方法はしばしば切断され、ウェットとドライベンチワークフローと非協調的な試薬セット変数伴います。本稿では、総合的な目標とNGSシステムの一例として、21個の遺伝子パネルで癌標本に挑戦配列決定のための方法を説明します。システムは、スタンドアロンのバイオインフォマティクススイートで分析的に検証し、シングルソースの試薬を使用して、機能的DNA定量および資格、シングルチューブ多重PCR濃縮、およびライブラリの精製および正規化を統合します。その結果、正確なバリアントからコール低品質、低量のホルマリン固定パラフィン包埋(FFPE)と微細針吸引(FNA)腫瘍生検を得ることができます。この方法は、日常的に400増幅DNAコピーの入力から癌関連変異体を評価し、新しい遺伝子のコンテンツに対応するために設計にモジュラーであることができます。解析的に定義されたコントロールの2種類が品質保証を提供し、臨床的に関連するサンプル精度を呼び出す守るのに役立ちます。柔軟な「タグ」PCR工程は、一般的なベンチトップNGS機器とサンプルのバーコードとの互換性を可能にするためにプラットフォーム固有のアダプタとインデックスコードを埋め込みます。重要なのは、プロトコルが合理化され、一日で24の配列に対応したライブラリーを作製することができます。最後に、このアプローチは、変異検出精度を向上させ、偽陰性とindetermを区別するためのアルゴリズムを呼び出すバリアントに直接事前分析試料の品質管理結果を組み込むことにより、湿式及び乾式ベンチプロセスをリンクinateコール。この目標とNGSの方法は、人間の頭脳と高深度、多重シーケンシングおよび診断用途のための異種の癌試料の高感度分析を実現するソフトウェアの両方の進歩を使用しています。
概要
精密薬は患者のための診断および治療法の選択肢の個別化に依存しています。合わせた治療の約束は、分子診断と標的治療の連携を通知することができる疾患経路理解の向上の直接的な結果です。例えば、分子標的療法の使用は、2013年1への2003から46%に11%から増加し、ベムラフェニブとクリゾチニブなどの抗がん剤は、コンパニオン診断テストでFDA-クリアされます。正確性の高い多重サンプルセットにわたって低存在量配列標的を回復する能力を有する、次世代シークエンシング(NGS)は、癌に関連する遺伝子異常を評価し、高精度の薬のための分子標的を同定するための最適な方法として出現しました。
分子検査のための最も一般的な固形腫瘍生検をホルマリン固定パラフィン包埋(FFPE)と穿刺吸引(FNA)SPECIMを含みますENS。これらのサンプルは、低量および/ または低品質の正確なNGSは2-5を査定挑戦核酸に満ちています。これらの試料を分析するための現在市販されているNGS方法が異なる試薬、プロトコル、および継続的改善の移動標的を表すインフォマティクスツールのパッチワークに基づいています。例えば、分析化学および/ またはソフトウェアの変更は、最も一般的に使用される標的NGSキット6毎に1~2ヶ月を生じました。この不安定性は、特に癌のテストのために、困難な試料の種類の統一NGSシステムを構築し、検証における一貫性の欠如を反映しており、サンプルツー結果から最適化された凝集のプロトコルを開発するために研究室に過度の負担をかけます。確かに、NGSユーザーの最近のある調査では、確立され、医学的に実用的なコンテンツ、強固なバイオインフォマティクスの専門知識、solidifiの要件に沿って、これらの「急速に変化する」技術の難しさを強調しましたエドかつ迅速に実施することができる統合された手順、および合理化されたワークフローとオンザジョブトレーニング7を容易に簡略化されたプロトコル。この記事では、対象とNGSのための包括的なシステムは、これらのギャップに対処することが記載されています。
提示方法は、精度、感度、および臨床的に関連する癌遺伝子座のNGSのための目標の定量化および検出の信頼性を向上させるために、ウェットとドライの両方のベンチで、分析を投稿する前分析からすべての手続きの手順を統合しています。このアプローチは、DNAの品質を評価PCRの濃縮工程への入力を案内し、かつ非常に低いテンプレートコピーの尋問から生じ得る偽陽性の呼び出しを防ぐために「機能的」DNA 4の定量化から始まります。シングルチューブマルチプレックスPCRは、その後、NGSのusinのためのプラットフォーム固有の配列を組み込むことによって、その後、唯一の400増幅DNAコピーを使用して、46座21の癌遺伝子を豊かにグラム一般的なデスクトップシーケンシング器具。ライブラリーは、単純な磁気ビーズの手順を用いて精製し、新規の、キャリブレーションフリーのqPCRアッセイを用いて定量します。コールのパフォーマンスを改善するために、サンプルDNA QC結果によって通知スタンドアロンバイオインフォマティクススイートは、NGS以下の配列解析を提供します。そのようなFFPEとFNAサンプル、および実行などの低品質と低量の腫瘍生検で(CNVを)我々は、ベース置換変異を明らかにすることを目標とNGSのためにこのシステムアプローチを使用してデータを提示し、挿入/欠失(インデル)、およびコピー数変異体コントロール。
プロトコル
注:このプロトコルは、MiSeq NGSシステムを使用して、サンプルの同時処理が記載されているが、個人ゲノムマシン(PGM)の機器に適合させることができます。 400増幅テンプレートコピーの推奨最小DNA入力の場合、アッセイは318チップにPGMを使用して、24のサンプルについてNGSランあたり96サンプルのそれぞれについて、少なくとも3,000x中央値カバレッジ、および同等のカバレッジの深さを生成することができます。この方法は、リアルタイムPCR機器を使用する必要があります。
1. DNA機能の定量化及び品質管理(QC)
- 雪解けの試薬:2×マスターミックス、プライマープローブミックス、阻害プライマープローブミックス、6-カルボキシ-X-ローダミン(ROX)、希釈剤、および4つのヒトゲノムDNAの検量線標準(DNA標準(50 ngの/μl)を、DNAスタンダード(10 ngの/μl)を、DNAスタンダード(2 ngの/μL)、およびDNAスタンダード(0.4 ngの/μL))( 表1)。 10秒間最大速度で10秒間遠心のための全ての試薬は、コンテンツを収集するための渦。氷上で2×マスターミックスをしてください。
- 試験されるサンプルの総数のためのマスターミックスを十分に準備し、ピペッティングによる不足を回避するために、10%より多くの量が含まれます。サンプルごとに、次のボリュームを使用して、マイクロチューブ内のマスターミックスを調製する:5μlの2×マスターミックス、0.5μlのプライマープローブミックス、0.5μlの阻害プライマープローブミックス、0.05μlのROX及び2.95μlの希釈液。コンテンツを収集するために10秒間最大速度で10秒間遠心のための渦。
- 96ウェルプレートのウェルに9μlのマスターミックスを加えます。
- 検量線を作成するために二重にDNA規格の1μLを加えます。アップダウンを5回ピペッティングして混ぜます。
- 核酸試料は、使用前に十分に混合であることを確認してください。マスターミックスに1μlのサンプルを追加し、上下を5回ピペッティングして混ぜます。
- コンテンツを収集するために10秒間最大速度で10秒間遠心分離用プレート、渦をシール。
- PCRシステムにプレートを置きます。 FAM(機能的定量化)およびVIC(機能阻害)を製造者の指示に従って、各サンプル用の検出器の両方を割り当てます。 95℃で10分間のPCRサイクルを実行し、(95℃、60℃で1分、15秒)を40サイクル。
- ソフトウェア・プロトコルを使用して、重複するDNA標準の各々についての線形回帰プロットを生成することによって定量PCRデータを分析します。
- x軸とy軸上の対応するFAM CのQ値の各DNAを、標準のコピー数のログ10をプロットします 。
- DNA標準検量線のダイナミックレンジ内の核酸試料の秋からの結果ことを確認し、参照標準曲線上の対応する位置から1μl当たり「機能」または増幅コピー数の未知のDNAの濃度を計算します。 図1は、渡された、失敗した検量線の例を示します。
- 増幅はVICチャネルにおける非ヒト標的アンプリコンの存在を確認することにより、各反応で発生したかどうかを確認します。
注:ポジティブコントロールとして、阻害プライマープローブミックスは、非ヒト外因性標的および対応するテンプレートに特異的なプライマーが含まれています。阻害プライマープローブミックスは、無鋳型対照(NTC)を含めて、各反応に添加されたマスターミックスの成分です。阻害剤の非存在下では、非ヒト対象のためのPCR産物は、常に、VICチャネルで検出されるべきです。 VICチャネル内の試料のための「未検出」C qはさらなる処理の前に、試料の後続のクリーンアップから利益を得ることができるPCR阻害剤の存在を示します。
2.ライブラリの準備:遺伝子特異的(GS)PCR
- pipettによる不足を回避するために試験されるサンプルの総数のためのマスターミックスの十分な量を調製し、10%以上の体積を含みる。サンプルごとに、次のボリュームを使用して、マイクロチューブ内のGS PCRマスターミックスを調製する:5μlの2倍増幅マスターミックス( 表1)と1μlのパンがんプライマーパネル( 表1)。コンテンツを収集するために10秒間最大速度で10秒間遠心のためのアップダウン、渦をピペットで混ぜます。
- 96ウェルプレートのウェルに6μlのGS PCRマスターミックスを分注し。個々のウェルに各核酸サンプルの4μlを添加します。他のウェルに、FFPEコントロール4μlの( 表1)、マルチバリアント・コントロール( 表1)の4μlの、および手続きNTCのためのヌクレアーゼフリー水4μlを添加します。各添加のために、アップダウンを5回ピペッティングして混ぜます。
- コンテンツを収集するために10秒間最大速度で10秒間遠心分離用プレート、渦をシール。
- 以下のPCRサイクルのためのサーマルサイクラーでプレートを置き:95℃で5分間、2サイクル(95℃で15秒、60℃で4分)、23サイクル(95℃、72℃、4分で15秒)、72℃で10分間の最終伸長。 4℃で保持します。
注:ステップ2.4が完了した後、プレートをGS PCRプレートと呼ぶことにします。
3.ライブラリの準備:タグPCR
- 雪解けの試薬 :2倍指数マスターミックス( 表1)とインデックスコード( 表1)。コンテンツを収集するために10秒間最大速度で10秒間遠心のための渦。
注:インデックスコードは、各サンプルについてのペアワイズ指数(バーコード)の固有のセットを提供するために予混合されます。 - -とダウン最大5回を96ウェルプレートで、よく指定に2倍のインデックスマスターミックスの7.5μlのとインデックスコードの5.5μlを添加し、ピペッティングにより混合します。
- 慎重GS PCRプレートを開き、マスターミックスを使用して新しいプレートに2μLGS PCR産物を追加します。アップダウンを5回ピペッティングで。各サンプルについて、サンプルIDと対応するペアごとのインデックスコードを記録します。コンテンツを収集するために10秒間最大速度で10秒間遠心分離用プレート、渦をシール。
- 10サイクル(95℃、55℃、72℃で1分、30秒で30秒)、及び72で10分間の最終伸長、95℃で5分間、サーモサイクラーおよびPCRにプレートを置き°C。 4℃で保持します。
注:ステップ3.4が完了した後、プレートをタグPCRプレートと呼ぶことにします。
4.図書館精製およびサイズの選択
- 2-8°Cからライブラリピュア準備磁気ビーズ( 表1)および溶出緩衝液( 表1)を取り外し、30分間室温に平衡化することができます。洗浄緩衝液( 表1)容器、キャップに9.6ミリリットルの100%エタノールを加え、ボトルを数回転倒混和します。
- 10秒のための磁気ビーズをボルテックスし、9の別々のウェルに11μLを追加6ウェルプレート。
- タグPCRプレートを開き、ビーズやピペットミックスに5回タグPCR産物の10μlを添加します。 RTで4分間混合物をインキュベートします。
- 4分間磁気スタンド( 表1)に、96ウェルプレートを置きます。まだスタンドに96ウェルプレートで、ピペットで上清を除去し、廃棄します。
- -とダウン最大5回を磁気スタンドから96ウェルプレートを取り外し、各ウェルに100μlのエタノール含有洗浄緩衝液を添加し、ピペッティングにより混合します。 2分間インキュベートします。
- ピペットで上清を除去し、廃棄した後、2分間磁気スタンドに96ウェルプレートを置きます。
- 第二洗浄した後、可能な限り多くの洗浄液を除去する2エタノール洗浄の合計を繰り返しステップ4.5、、。
- 磁気スタンド上で96ウェルプレートを使用すると、その後、室温で2分間ビーズを乾燥スタンドからプレートを取り外します。
- 各WELに20μlの溶出バッファーを添加することにより、ビーズを再懸濁リットルとピペット5回-とダウンアップ。
- 室温で2分間インキュベートします。
- 4分間の磁気スタンドに戻って96ウェルプレートを置き、慎重に新しいウェルに透明な上清の18μLを削除して転送します。
注:手順は安全-15 -30℃で保存し、このステップサンプルで停止することができます。再起動するには、先に進む前に、氷上で凍結サンプルを解凍。
5.図書館定量
- 解凍ライブラリクワント(LQ)の試薬 :2倍LQマスターミックス、LQプライマー/プローブミックス、LQスタンダードLQポジティブコントロール、LQ希釈液、およびLQ ROX( 表1)。コンテンツを収集するために10秒間最大速度で10秒間遠心のための渦。
- 精製されたライブラリ製品を使用して、LQ希釈剤の個々のサンプルの連続希釈を行います。
- 198μlのLQ希釈液に2μlの(ライブラリ精製物)を追加し、アップダウンピペットで10回を混ぜます。
- 1:(100希釈1)2μLを追加98μlのLQ希釈液とは、ピペット10倍で上下を混ぜます。
- 試験されるサンプルの総数のためのLQマスターミックスを十分に準備し、ピペッティングによる不足を回避するために、10%より多くの量が含まれます。サンプルごとに、次のボリュームを使用して、マイクロチューブ内LQマスターミックスを準備:5μlの2X LQマスターミックス、2μlのLQプライマー/プローブミックス、0.5μlのLQ標準と0.5μlのLQ ROX。コンテンツを収集するために10秒間最大速度で10秒間遠心のためのアップダウン、渦をピペットで混ぜます。
- 光学96ウェルプレートのウェルに8μlのLQマスターミックスを追加します。
- 別々のウェルでは、2μlの希釈されたライブラリー、2μlのLQ陽性コントロールと2μlのLQの希釈液(NTC)を追加し、-とダウン最大5回ピペッティングして混ぜます。内容物を収集するために10秒間400×gで10秒間遠心分離するための光学接着フィルム、ボルテックスでプレートを密封します。
- FAMとVIC detectoの両方を割り当て各サンプルのRS。 95℃で5分間のサイクル条件を用いてPCR増幅を行い、(95°C、60°Cで1分、15秒)を40サイクル。
- 比較C qを法を用いて、各サンプル(NM)の濃度を決定します。未知のライブラリ(C Qが FAM)に知られLQ標準(C qをVIC)の違いを計算します。希釈したサンプル(PM)の濃度は、以下の式を用いて計算されます。
[ コンクlibに ] nMで = 12.5×2Δ はCq
万以外の希釈係数を使用している場合は、10,000の目標希釈率の比を計算し、5.7での式の結果によって、この係数を掛けます。
6.図書館正規化とサンプル・プーリング
- プールされるべきすべてのサンプル(それぞれが固有のペアごとのインデックスを含む)全体の中央値濃度(nM)を決定します。
- 個々の試料volumを決定E(μl)を、その個々の濃度(nM)をで割るその後、5によってすべてのサンプル全体の中央値濃度を乗じてプールに。最も近い整数に結果の値ラウンド。ラウンドの値を持つボリューム<2μLに2μlを、およびボリューム> 15μlを15μlの。
- サンプルプールを作成するために、単一のマイクロチューブに各サンプルの正規化されたボリューム(μl)を追加します。
- 丸められた整数値を使用して、各サンプルについての新しい濃度を計算し、結果を記録します。
- サンプルプールの濃度を決定するために、すべての個々の濃度の合計を計算し、その結果の値(nM)を記録します。
- シーケンシング希釈液( 表1)を用いて、1.25 nMのにサンプルプールを希釈します。
注:手順は安全-15 -30℃で保存し、このステップサンプルで停止することができます。再起動するには、先に進む前に、氷上で凍結サンプルを解凍。
7.シーケンシング
- DenatURE以下を追加してPHIXコントロールv3の( 表1)の存在下でのサンプルプール:1.25 nMのサンプルプール15μlの、0.5 nMのPHIXの3μlを1 N NaOHを2μLを。渦が簡単に短時間遠心分離し、室温で5分間インキュベートします。
- 氷上で変性したサンプルプールを配置します。
- マイクロチューブにあらかじめ冷却HT1-hyb緩衝液の992μlに変性したライブラリーの8μlを添加します。ボルテックスは簡潔に内容を収集するために、短時間の遠心分離に続いて、混合します。氷の上に保管してください。
- 試薬カートリッジの#17を配置するために変性し、希釈したライブラリーの600μLを加えます。
- 解凍読む1シーケンシングプライマー( 表1)、インデックス読むシーケンシングプライマー( 表1)、および読み取り2シーケンシングプライマー( 表1)。マイクロ遠心チューブでは、別々に636μlのHT1-hyb緩衝液で4μlの配列決定プライマーを希釈。注:HT1-hyb緩衝液は、ウィットに提供されH配列決定試薬キット( 表1)。
- コンテンツを収集するために10秒間最大速度で10秒間遠心ボルテックスにより混合します。シーケンシング試薬カートリッジの#18を位置決めするための希釈読む1の配列決定プライマーの600μlを添加し、希釈したインデックスを読むプライマー600μlのは#20を配置するために#19、および希釈読む2配列決定プライマーの600μLを位置決めします。
- 製造元の指示に従ってNGS機器( 表1)および配列上の試薬 をロードします。ペアエンド2×150サイクルシークエンスランを実行します。
8.データ解析
注:NGS機器・ソフトウェアは、ベースコールと品質スコアにクラスタの画像を変換し、各サンプルの個々のgzipで圧縮FASTQ(* .fastq.gz)ファイルを生成するために、ペアごとのインデックスを分離します。逆多重化されたファイルを分析する前に、読者は、 表 (関連するバイオインフォマティクスソフトウェアをダウンロードし、インストールする必要があります1)。ソフトウェアは、消費者向けのWindows PCにインストールすることができ、データ分析を実行するために特化したコンピューティングハードウェアまたはインターネット接続を必要としません。
- ソフトウェアのデスクトップアイコンをダブルクリックします。
- ソフトウェアのマニュアルに提供されたユーザ名とパスワードを使用してシステムにログインします。
- プロジェクトダッシュボードを開き、「新規プロジェクト」をクリックします。
- プロジェクトに名前を付け、任意のプロジェクトの説明を提供します。対象とNGSパネルの種類とNGS機器の種類を選択します。 「保存して続行」をクリックします。
- 前進用の圧縮FASTQファイルをアップロードし、読み込み逆。それが逆多重化に失敗した読み込み含まれ、「未割り当て」FASTQsを、アップロードしないでください。 「保存して続行」をクリックします。
- インプットDNAの機能的定量アッセイによって決定される各ライブラリーを調製するために使用される機能的な入力コピー数(上記のステップ1を参照)。手動でスプレッドから値またはコピー&ペーストの値を追加します注釈テーブルにシート。 「保存して続行 "をクリックします。
- 分析のためにアップロードされた注釈付きのライブラリを確認し、分析を開始するには、「分析を送信」をクリックします。
- プロジェクトダッシュボードで表示された分析の進行状況を監視します。
注:結果はレビューのために準備ができているとき、分析、完全な状態表示が提示されます。 - ライブラリーあたりの総カバレッジを含むサンプルのQCメトリックの分析結果を確認し、の割合は、フィルタを通過し、アンプリコンのカバレッジの深さと均一性を読み込みます。 dbSNP、COSMIC、千ゲノムおよび機能と人口レベルの注釈の他のソースと、各配列を決定し、ライブラリのバリアントの呼び出しを確認します。
- 補完的なインフォマティクスツールとの長期保存や下流の分析のためのサマリー表計算表、* .bamファイルと*の.vcfファイルとして生の結果をエクスポートします。
結果
陽性および陰性対照を表す90サンプル(74一意)の合計は、以前に細胞株を特徴付け、そして残留臨床FFPE腫瘍生検は、増幅DNA、配列アダプターでタグ付けされたマルチプレックスPCR濃縮に入力、バーコードについて評価し、そして単一で分析しました。 19.1 Mを生成した卓上NGS機器ラン( 図2)は、フィルタを通過し読み込みます。等モルのサンプルプーリングは、高深度シーケンシング(3,692xの読み取り)と均一なカバレッジ(中央値読み取り深度の5倍以内で覆われたアンプリコンの97.8パーセント)が得られました。外れ値は、分析前QCアッセイ( 図3)によりPCR阻害のためにフラグが付けられた無テンプレートコントロール、大きなコピー数の増幅を1細胞株のDNA、および1 FFPE DNAを含んでいました。 46アンプリコン全体のカバレッジの均一性は、(3異なるオペレータ( 図4A)、および異なる低品質FFPE DNAサンプルのためを使用して維持しました。 図4B)。 5%BRAF V600E(液滴デジタルPCRにより定量化)を達成するために、残留臨床検体の混合物から配合FFPE腫瘍DNAコントロールは、使用して3つの演算子によって5.3、3.9の存在量の目標BRAF変異を有することが報告され、そして6.5%ました400増幅コピー(したがって、唯一の20の変異体コピー)( 図4Bおよびインセットテーブルの「FFPE3」)の入力。 17%平均対立遺伝子頻度( 表2) -さらに、12の合成DNA鋳型、既知の「ドライバー」塩基置換変異を表すそれぞれの混合物は、9の意図された範囲内で予想される変異を明らかにしました。コピー数の増幅を持つ細胞株とFFPE DNAサンプルの希釈は、( 図5)は、それぞれ、EGFRおよびKRASにおける変異体についての用量依存性を示しました。偽陽性なし既知の変異の検出を維持しながら、重要なのは、FFPEのDNAの入力は、わずか50増幅コピーまたはバルクDNAの1.2 ngのを低減することができコール( 図6)。 DNAの入力は少なくとも50,000増幅コピー( 表3)に100倍の範囲までにわたって収容しました。これと関連した実験では、変異体は、22 FFPEに呼び出し、20 FNA検体は共有突然変異カバレッジ( 表4)を持つ独立した方法と一致して報告されました。
アッセイの感度および陽性予測値は、FFPE、FNA、新鮮凍結、および細胞株のDNA、および195配列決定結果の合計を含む97サンプルの分析から決定しました。 99.7%(95%CIのと陽性予測値(PPV):結果は、98.9パーセントの感度(97.1から99.7パーセント95%CI)のために365真陽性バリアントコール、4偽陰性コール、1偽陽性コールを明らかにしました:98.2〜99.99%)。インデルの分析は、93.9パーセントの感受性を示す、33サンプルラン中の2つの一般的なEGFR変異型(p.E746_A750delELREAとp.V769_D770insASV)について実施した(95%CI:78.4から98.9パーセント)と100%(95%CIのPPV:86.3から100パーセント)の変異体では、2.4から84.8パーセントの範囲で検出されました。
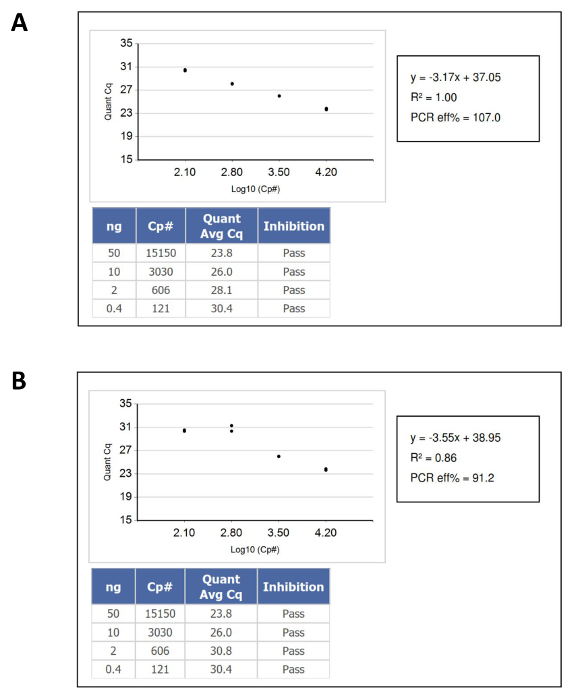
図1:合格と不合格QC基準 (A)を通過する標準曲線DNA定量検量線の例 。 (B)失敗した標準曲線。この場合、障害が最低入力のDNA標準の重複ピペッティングによって引き起こされた。 この図の拡大版をご覧になるにはこちらをクリックしてください。
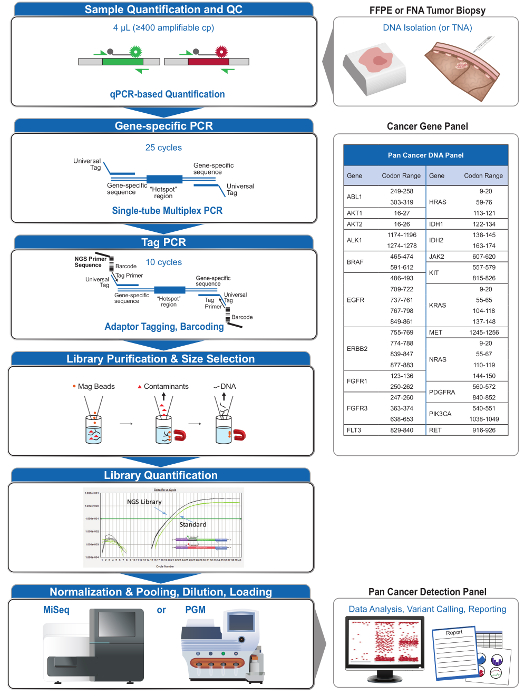
図2:事前分析的統合腫瘍学アプリケーションのための総合的な目標とNGSシステムの概要ら、分析、およびポスト分析ワークフロー。 この図の拡大版をご覧になるにはこちらをクリックしてください。
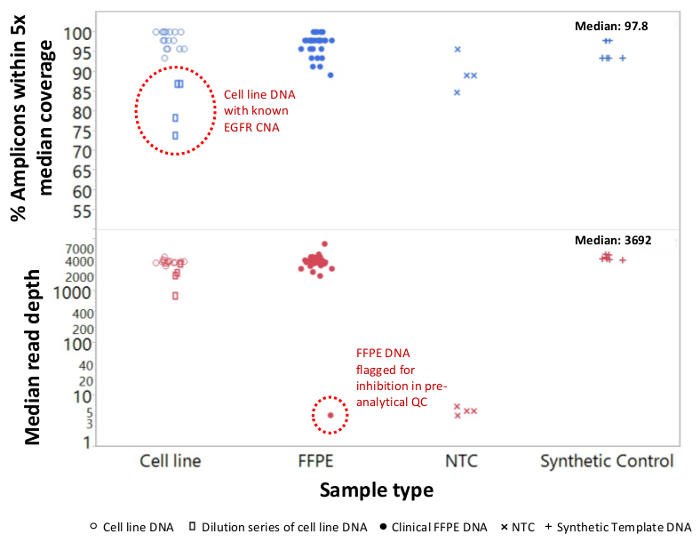
図3:無傷の細胞ラインDNAと対照と比較して低品質FFPE DNAにおける癌遺伝子の標的NGSの読み取り範囲と均一性残留臨床FFPE(黒丸)に含まれる90サンプルの合計、細胞ライン(白丸。 )、および合成鋳型DNA(プラス記号)シングルチューブ、21遺伝子の多重PCR濃縮を用いて処理しました。各アンプリコンライブラリは、NGS機器用のアダプター配列でタグ付けされた個別のデュアルインデックスコードとバーコード、精製し、定量化、および2.5 nMの濃度に標準化しました。 DNAライブラリーは、コンパニオンバイオインフォマティクスソフトウェアにより配列決定し、分析しました。サンプル・デビationsは、大規模なEGFRのコピー数の増幅(トップ、点線円内にオープン長方形)のかなりの数を生成するために失敗したカバレッジ均一性と1メラノーマFFPEサンプルを歪めによる読み込みを保有するMDA-MB-468細胞株DNAの希釈系列を含まDNA抽出からPCR阻害物質のキャリーオーバーします。メラノーマサンプルの失敗(下、点線円)を分析前のqPCR DNA QCアッセイによって予測されました。 NTC、無テンプレート対照(X記号)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
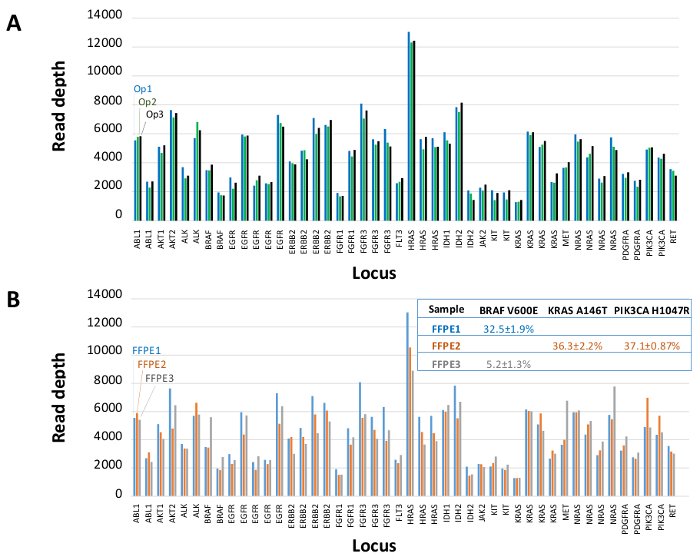
図4:アンプリコンバイアンプリコン読むカバレッジ、均一性と残留臨床FFPE腫瘍DNAにおける変異体の検出 。 (A)を測定した代表FFPE DNAサンプル中のすべての濃縮された遺伝子座全体にカバレッジを読みます3異なるオペレータを横断。オペレータ1、OP1(青棒)。オペレーター2、OP2(緑色のバー)。オペレーター3、OP3(黒棒)。知られている5%BRAF c.1799T>突然変異で構成される対照混合物(FFPE3、灰色のバーやテキスト)を含む3 FFPE腫瘍サンプルを用いて評価(B)カバレッジの均一性およびバリアントの呼び出し、。 FFPE1(青いバーとテキスト)、FFPE2(オレンジ色のバーやテキスト)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
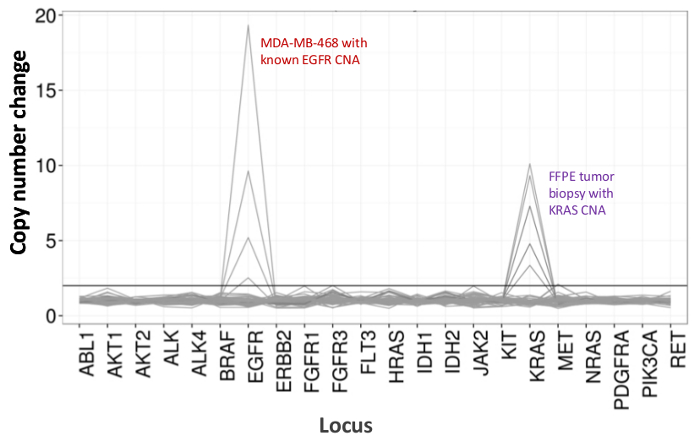
図5は、:。十分に特徴付けられたEGFRのコピー数の増幅を有する細胞株とFFPE DNA MDA-MB-468細胞株DNA におけるコピー数変異体の用量依存検出は次第に参照非突然変異細胞のバックグラウンドで希釈し、 -line DNAは、コピー数の変化の減少を示すために希釈の機能。各細胞株のDNAサンプルのパーセンテージは、異なる行(0、12.5、25、50、100%)で示されています。既知のKRAS遺伝子増幅を伴う卵巣FFPE腫瘍サンプルの希釈は同じ滴定シリーズを使用して同様のプロファイルを明らかにしたが、2つのトップラインのための100%のFFPE DNAの複製と。 この図の拡大版をご覧になるにはこちらをクリックしてください。
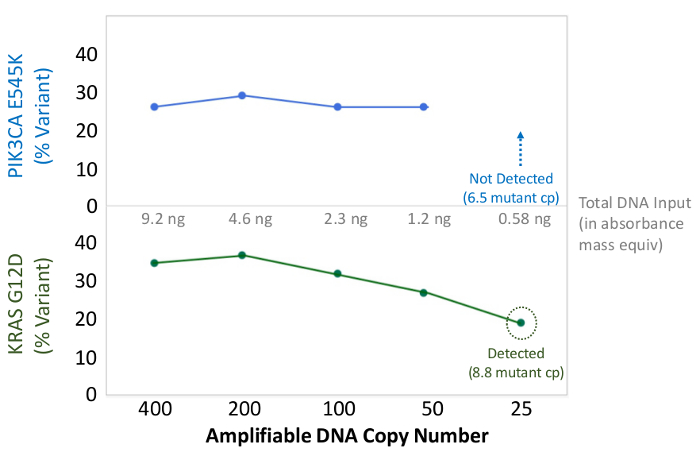
図6:バルクDNAを50ngの増幅可能FFPE DNAのコピーに変異検出および定量正確な、または1.2大腸癌FFPE DNAの増幅コピー数は次のように定量PCRベースのQCアッセイによって決定され、400〜25コピーから希釈しました。配列決定前に、マルチプレックスPCR濃縮に入力。正しく知られているバリアンの両方と呼ばれるバイオインフォマティクスパイプライン50コピー、または〜10変異体テンプレートの同等までTS。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| 素材/材質の名前 | 会社 | カタログ番号 |
| Quantidexマスターミックスを2倍 | Asuragen | 145345 |
| クワントプライマープローブミックス | Asuragen | 145336 |
| 阻害プライマープローブミックス | Asuragen | 145344 |
| ROX | Asuragen | 145346 |
| 希釈剤 | Asuragen | 145339 |
| DNAスタンダード(50) | Asuragen | 145340 |
| DNAスタンダード(10) | Asuragen | 145341 |
| DNAスタンダード(2) | Asuragen | 145342 |
| DNA標準(0.4) | Asuragen | 145343 |
| 2倍増幅マスターミックス | Asuragen | 145348 |
| パンがんプライマーパネル | Asuragen | 145347 |
| パンがんFFPEコントロール | Asuragen | 145349 |
| パンがんマルチバリアントコントロール | Asuragen | 145350 |
| ライブラリピュア準備ビーズ | Asuragen | 145351 |
| 洗浄バッファー | Asuragen | 145352 |
| 溶出バッファー | Asuragen | 145353 |
| LQマスターミックスを2倍 | Asuragen | 145358 |
| LQ希釈液 | Asuragen | 145354 |
| LQ正コントロール | Asuragen | 145355 |
| LQスタンダード | Asuragen | 145356 |
| LQプライマー/プローブミックス(ILMN) | Asuragen | 145357 |
| LQ ROX | Asuragen | 145359 |
| インデックスコード(ILMN) - セットA | Asuragen | 150004 |
| AIL001 - AIL048(48) | ||
| インデックスコード(ILMN) - セットB | Asuragen | 150005 |
| AIL049 - AIL096(48) | ||
| インデックスマスターミックスを2倍 | Asuragen | 145361 |
| 1シーケンシングプライマーを読みます | Asuragen | 150001 |
| インデックス読むシーケンシングプライマー | Asuragen | 150002 |
| 2シーケンシングプライマーを読みます | Asuragen | 150003 |
| シーケンシングの希釈液 | Asuragen | 145365 |
| イルミナMiSeq | イルミナ | |
| MiSeq試薬キットv3の(600サイクル) | イルミナ | MS-102-3003 |
| MiSeq試薬ナノキットv2の(300サイクル) | イルミナ | MS-103-1001 |
| PHIXコントロールv3の | イルミナ | FC-110-3001 |
| マグネットスタンド-96(または同等の装置) | Ambion社 | AM10027 |
| Quantidexレポーターソフトウェア | Asuragen |
表1:試薬およびキット ROXの最初の使用時には、2〜8℃でバイアルを格納します。再凍結しないでください。ソフトウェアはwww.asuragen.comでダウンロードすることができます。
| Gエン | COSMICバリアント | COSMICアミノ酸 | %バリアント |
| NRAS | c.182A> G | p.Q61R | 13.3 |
| NRAS | c.35G> A | p.G12D | 15.2 |
| HRAS | c.182A> G | p.Q61R | 17.8 |
| HRAS | c.35G> A | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G> A | p.G12D | 19.1 |
| PIK3CA | c.1633G> A | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| キット | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14.9 |
| BRAF | c.1799T> A | p.V600E | 17.3 |
表2:プールされた合成コントロールが9から17パーセント豊富で定量されている12 "ドライバ"がん遺伝子の変異体で構成されている 12の異なる変異を保有する12の異なる二本鎖合成鋳型の混合物をシークエンシング以下の評 価を行いました。すべての変異体は正しくありません偽陽性と呼ばれていました。
>%バリアント| 検体ID | 機能的な CPS | 遺伝子 | COSMICバリアント | COSMICアミノ酸 | 中央値の読み取り深度 | 中央値の5倍以内% | |
| BCPAP | 400 | BRAF | c.1799T> A | p.V600E | 99.5 | 3289 | 96% |
| BCPAP | 10000 | BRAF | c.1799T> A | p.V600E | 99.7 | 4040 | 98% |
| BCPAP | 25,000 | BRAF | c.1799T> A | p.V600E | 99.4 | 3687 | 96% |
| BCPAP | 50,000 | BRAF | c.1799T> A | p.V600E | 99.7 | 4611 | 93% |
表3:カバレッジとバリアントの呼び出しは、DNA入力の> 100倍の範囲に渡って保持されます。 BCPAP細胞株から増幅可能なDNAは、400〜50,000コピーし、配列決定におけるマルチプレックスPCR濃縮に入力しました。深さ、被覆の均一性、変異検出、突然変異精度を読み込み、入力範囲にわたって保存しました。
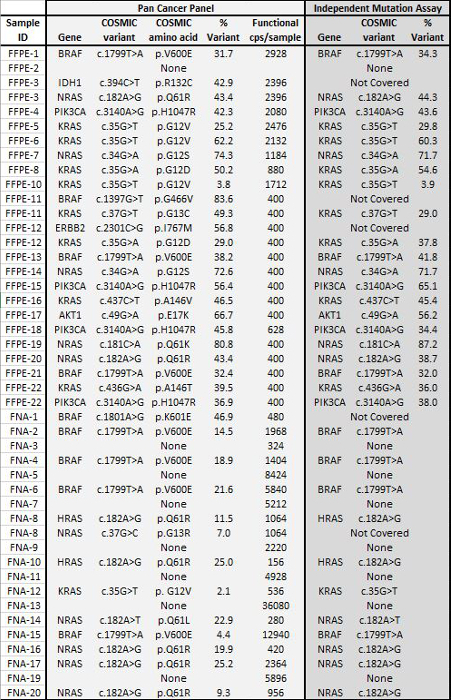
表4:バリアントは22 FFPEに呼び出し、20 FNAの腫瘍生検は、独立した変異アッセイの結果に同意します以前に直交対象とNGSのアッセイによって決定変異状態で22 FFPE腫瘍DNAをPCR濃縮に400 2928まで増幅コピーで入力された一連の。ステップと21-遺伝子パンがんパネルを使用して配列決定しました。 20 FNA DNA試料のコホートは、以前に液体ビーズアレイ突然変異試験8を使用して特徴に加えて、PCRは156 36080に入力増幅コピーを用いて増幅し、配列決定しました。パンがんNGSパネルとREFERE間のすべての重複のコールNCEの方法が一致していた。 この図の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
NGS技術は、臨床現場9に腫瘍生検の分子プロフィールを問い合わせるための期待を再定義しました。標的NGSパネルの数は、臨床検体3,5,10-15複数のタイプを評価するための研究技術研究所が開発試験および市販製品、カスタムパネルとして開発されています。複数の研究からの報告は、ゲノム変化3,10-12,14の検出のための高感度かつ特異的な臨床ツールとしてのNGSの価値が実証されています。しかし、研究はまた、そのようなFFPEが2-5,12,14を標本として挑戦的な癌生検からの偽陽性の結果を引き起こす可能性が成果物の危険性を実証しました。さらに、最近の刊行物は、低入力DNA 12の使用によって悪化している商用の対象とNGSパネル16と偽陽性コールは、腫瘍学を使用してNGSのための高故障率を試験片強調しています。その結果、いくつかのlabora保守党は、市販の標的NGS技術は精度を確保するか、バイオインフォマティクス5,10,11を分析から知見を確認するために、多くの場合、パフォーマンスを改善するためにするために、追加のチェック・アンド・バランスを変更または追加されました。
21-遺伝子パネル( 図2)は 、FFPEおよびFNAの腫瘍生検のような挑戦的な検体の種類に科学的根拠に基づいた、実用的な変異を調べるための包括的なNGSシステムの対象となるコンテンツとして開発されました。ワークフローは、多くの利点があります:均一なアンプリコンカバレッジ( 図3と図 4、 表3)を提供することにより、1)の一貫性を。 2)使いやすさ、最適化された試薬セットを事前に策定提供、およびバイオインフォマティクスの要件を簡素化することにより、ワークフローを合理化し、他の商業NGS方法に比べて、ピペッティングステップの数を減らすことにより、3)効率。 amplifiを評価するためのDNA QC検定を組み込むことによって、および4)精度ことができるDNAのコピー数は、許容可能なテンプレートの多様性を確保し、変異検出の4における確率的変動を回避します。バイオインフォマティクス解析と分析前QCデータの統合は、FFPE DNAの低入力を可能にします。これは真実の独立した対策を400 FFPEサンプル全体のDNA入力の異なる機能のコピーを使用して、意思決定ツリーアルゴリズムを訓練し、バイオインフォマティクスソフトウェアにこのアルゴリズムを組み込むことによって達成されました。その結果、FFPE DNAの〜5-20 ngのに一般的に同等の400増幅コピー、の推奨入力は、FFPE DNAの〜250 ngのは17,18を推奨され、ハイブリダイゼーションベースの濃縮を含む他の方法10月12日 、に匹敵します。技術はMiSeqプラットフォーム上で使用するために記載されているが、他のNGSプラットフォーム上で配列分析を可能にするために機器固有のアダプタでタグPCRプライマーを使用して変更することができます。
いくつかのステップが成功を確実にするために重要です手順の。分析前QCアッセイは、DNAとレポート機能阻害の増幅コピー数を決定します。 400未満の増幅DNAコピーをPCR濃縮工程で使用される場合には、低存在量の突然変異を有する試料からの偽陰性のコール( 図6)の増加した危険性があります。さらに、ケアは、洗浄又は溶出工程の間に磁気ビーズの過乾燥を防止するために、ライブラリ精製時に注意する必要があります。また、成功したライブラリーの定量化は、ライブラリーDNAの正確な希釈に大きく依存しています。最良の結果については、LQスタンダード(CQ VIC)と比較して、サンプル・ライブラリー(CQのFAM)のためのqPCR結果の違いは≤3.3Cqをする必要があります。差はCqを、サンプルの再希釈およびテスト3.3よりも大きい場合には推奨されます。優れた相関は、標準曲線を使用して、この競合的定量PCR法および絶対的定量を提供する市販のキットとの間で観察されたが他の方法に比べクローン増幅工程にライブラリ入力のオフセットを最適な播種密度を達成するために必要であり得ます。
いくつかの癌標本は、特にため、DNA単離後も持続阻害剤の配列に挑戦しています。ライブラリー調製の前にこれらのサンプルを識別するために、定量PCR QCアッセイはまた、内部統制や機能阻害のためのセンチネルの両方として機能する外因性のテンプレートを含めることにより、増幅阻害を検出します。例は、黒色腫のDNA試料を配列決定前にQC阻害メトリックを渡すために失敗した後、配列決定することができるライブラリーを生成するための失敗した図3に示されています。失敗は、おそらくFFPE DNAの単離工程から持ち越さメラニン汚染の結果、既知のPCR阻害剤でした。増幅の障害のためにリスクがあるとQCアッセイにより同定されたサンプルは、余分なクリーンアップステップtを介して救済することができますO潜在的阻害剤を除去します。
対象となる21個の遺伝子パネルは、証拠ベースの遺伝子ホットスポットに焦点を当て、DNA QC、NGSおよび分析前「機能的」DNAの定量結果から通知されたバイオインフォマティクスソフトウェアの最適化された試薬およびコントロールで完全なシステムを提供します。この方法は、正確に、低入力DNAの塩基置換変異及びインデルを検出し、そのようなのCNVのような追加の変異体を検出して、標的RNA配列決定のために適合され、パネルのコンテンツを展開するためのオプションとNGSシステムの例を提供します。
開示事項
JH、AH、RZ、BCH、およびGJLは従業員であり、Asuragen、Inc.のRZ、BCHの株式の所有権を持っている、とGJLは、各サンプルについて決定増幅コピー数の情報を使用して呼び出すバリアントを向上させるための特許出願に共同発明です。
謝辞
私たちは、原稿のレビューのために博士アネットSchlageterに感謝します。この作品は、がん予防テキサスの研究所(:GJL PI)からの助成金CP120017によって部分的にサポートされていました。
資料
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
参考文献
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved