Method Article
Integration von Nass- und Trocken Bench Prozesse werden so optimiert Gezielte Next-Generation Sequenzierung von Low-Qualität und Low-Menge Tumorbiopsien
In diesem Artikel
Zusammenfassung
An integrated system for targeted next-generation sequencing of oncology specimens is described. This cross-platform system is optimized for low-quality and low-quantity tumor biopsies, accommodates low DNA inputs, includes well-characterized multi-variant controls, and features a novel variant caller that is informed by quantitative pre-analytical quality control measures.
Zusammenfassung
Alle der nächsten Generation Sequenzierung (NGS) Verfahren umfassen Tests auf der Laborbank ( "wet bench") durchgeführt und Daten-Analysen der Bioinformatik-Pipelines durchgeführt unter Verwendung von ( "Trockenbank"). Beide Elemente sind für eine genaue und zuverlässige Ergebnisse zu erzeugen, die für klinische Labors besonders kritisch sind. Gezielte NGS-Technologien haben in zunehmendem Maße für die in der Onkologie-Anwendungen gefunden Voraus Präzision Medizin Ziele zu helfen, aber die Methoden beinhalten oft getrennt und variable nassen und trockenen Bank Workflows und unkoordiniert Reagenz-Sets. In diesem Bericht beschreiben wir ein Verfahren zur Sequenzierung herausfordernde Krebsproben mit einem 21-Gen-Panel als Beispiel eines umfassenden gezielten NGS-System. Das System integriert funktionelle DNA-Quantifizierung und Qualifizierung, Einrohr-Multiplex-PCR-Anreicherung und die Bibliothek Reinigung und Normalisierung analytisch verifiziert, Single-Source-Reagenzien mit einem eigenständigen Bioinformatik Suite.Als Ergebnis erfordert eine genaue Variante von geringer Qualität und Low-Menge in Formalin fixierten und in Paraffin eingebetteten (FFPE) und Feinnadel (FNA) Tumorbiopsien erreicht werden kann. Das Verfahren kann bei Krebs vorkommenden Varianten von einem Eingang von 400 amplifizierbare DNA Kopien und ist modular aufgebaut aufzunehmen neues Gen Gehalt routinemßig beurteilen. Zwei verschiedene Arten von analytisch definierten Kontrollen liefern Qualitätssicherung und zum Schutz von Call-Genauigkeit mit klinisch relevanten Proben. Ein flexibles "tag" PCR-Schritt bettet plattformspezifischen Adapter und Indexcodes Probe Strichcode- und Kompatibilität mit gängigen Benchtop NGS Instrumente zu ermöglichen. Wichtig ist, dass das Protokoll rationalisiert und kann 24 Sequenz-ready-Bibliotheken in einem einzigen Tag produzieren. Schließlich verbindet der Ansatz nassen und trockenen Bank Prozesse durch die präanalytische Proben Ergebnisse der Qualitätskontrolle direkt in die Variante enthält Algorithmen Aufruf Mutation Erkennungsgenauigkeit zu verbessern und zu differenzieren falsch-negative und indeterminate Anrufe. Diese gezielte NGS-Methode verwendet Fortschritte sowohl in der Wetware und Software hoch Tiefe, Multiplex-Sequenzierung und empfindliche Analyse von heterogenen Krebsproben für diagnostische Anwendungen zu erreichen.
Einleitung
Präzisions Medizin beruht auf der Individualisierung von diagnostischen und therapeutischen Möglichkeiten für die Patienten. Das Versprechen von maßgeschneiderten Behandlungen ist eine direkte Folge einer verbesserten Verständnis von Krankheitswege, die die Verknüpfung der molekularen Diagnostik und gezielte Therapeutika informieren können. Zum Beispiel erhöht die Verwendung von molekular-zielgerichtete Therapien von 11% auf 46% von 2003 bis 2013 1 und Anti-Krebs - Medikamente wie Vemurafenib und crizotinib sind mit diagnostischen Begleittests der FDA genehmigt. Mit seiner Fähigkeit, Low-Fülle-Sequenz Ziele über hoch Multiplex-Sample-Sets, um genau zu erholen, hat Next-Generation-Sequencing (NGS) als Methode der Wahl entstanden für genetische Abweichungen im Zusammenhang mit Krebs zu bewerten und molekulare Ziele für Präzisionsmedizin zu identifizieren.
Die häufigsten soliden Tumor-Biopsien für molekulare Tests umfassen Formalin-fixierte, in Paraffin eingebettete (FFPE) und Feinnadel (FNA) Specimens. Diese Proben sind behaftet mit niedrigem Menge und / oder minderwertige Nukleinsäuren, die genaue NGS Assessments 2-5 herauszufordern. Aktuelle kommerzielle NGS-Methoden für die Analyse dieser Proben auf einem Flickenteppich von verschiedenen Reagenzien, Protokolle basieren, und Informatik-Tools, die bewegliche Ziele der kontinuierlichen Verbesserung darstellen. Beispielsweise Änderungen in Assay Chemien und / oder Software eingetreten alle 1-2 Monate für die am häufigsten verwendeten gezielte NGS kits 6. Diese Instabilität spiegelt einen Mangel an Kohärenz bei der Konstruktion und ein einheitliches NGS-System für anspruchsvolle Probentypen, insbesondere für Krebs-Tests zu überprüfen und stellt eine unzumutbare Belastung für Laboratorien zusammenhängende Protokolle zu entwickeln, die von der Probe-zu-Ergebnisse optimiert sind. Tatsächlich markiert eine aktuelle Umfrage von NGS-Benutzer, die Schwierigkeiten dieser "sich rasch verändernden" Technologien, zusammen mit den Anforderungen für die etablierten, medizinisch verwertbare Inhalte, verschanzt Bioinformatik Know-how, ein solidified und integriertes Verfahren , die schnell umgesetzt werden können, und optimierte Workflows und vereinfachte Protokolle , die 7 Training on-the-Job erleichtern. In diesem Artikel wird ein umfassendes System für die gezielte NGS beschrieben, die diese Lücken behebt.
Die vorgestellte Methodik integriert alle Verfahrensschritte von präanalytischen zu post-analytische auf nassen und trockenen Bänke, die Genauigkeit, Empfindlichkeit und Zuverlässigkeit der Ziel Quantifizierung und zum Nachweis für NGS von klinisch relevanten Krebs-Gen-Loci zu verbessern. Dieser Ansatz beginnt mit der Quantifizierung der "funktionellen" DNA 4 DNA - Qualität beurteilen zu können , führen Eingang in die PCR - Anreicherungsschritt, und Schutz vor falsch-positiven Anrufe, die von der Abfrage von sehr niedrigen Vorlage Kopien entstehen können. Ein Single-Tube-Multiplex-PCR bereichert dann für 46 Loci in 21 Krebs-Gene nur 400 amplifizierbare DNA-Kopien, gefolgt durch den Einbau von plattformspezifischen Sequenzen für NGS usin mitg gemeinsame Instrumente Desktop-Sequenzierung. Bibliotheken werden gereinigt, um ein einfaches magnetisches Kügelchen Prozedur und mit einem neuen, Kalibrierung freien qPCR-Assay quantifiziert. Eine eigenständige Bioinformatik-Suite, informiert durch DNA-Probe QC Ergebnisse Anruf Leistung zu verbessern, stellt die Sequenzanalyse folgende NGS. Wir präsentieren Daten, die diese Systeme Ansatz für die gezielte NGS mit Basensubstitutionsmutationen zu offenbaren, Insertion / Deletionen (indels), und Nummer des Exemplars Varianten (CNVs) in geringer Qualität und Low-Menge Tumorbiopsien wie FFPE und FNA Proben und Lauf Kontrollen.
Protokoll
Hinweis: Dieses Protokoll die gleichzeitige Verarbeitung von Proben beschreibt ein MiSeq NGS-System verwendet, kann aber für das Personal Genome Machine (PGM) Instrument angepasst werden. Für die empfohlene Mindest DNA Eingabe von 400 amplifizierbare Vorlage kopiert, ist der Test in der Lage mindestens 3,000x mittlere Abdeckung für jeden von 96 Proben pro NGS Lauf und äquivalente Deckungstiefe für 24 Proben, die PGM auf einem 318-Chip. Das Verfahren erfordert auch die Verwendung eines Echtzeit-PCR-Instruments.
1. DNA Funktionelle Quantifizierung und Qualitätskontrolle (QC)
- Auftau Reagenzien: 2x Master Mix, Primer Sondenmischung, Inhibition Primer Sondenmischung, 6-Carboxy-X-Rhodamin (ROX), Diluent und die vier humanen genomischen DNA Kalibrierkurve Standards (DNA Standard (50 ng / ul) DNA-Standard (10 ng / ul) DNA - Standard (2 ng / ul), und DNA - Standard (0,4 ng / & mgr; l)) (Tabelle 1). Vortex alle Reagenzien für 10 sec und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalt zu sammeln.Halten Sie die 2x Master-Mix auf Eis.
- Eine ausreichende Menge von Master-Mix für die Gesamtzahl von Proben getestet werden und umfassen 10% mehr Volumen Mangel aufgrund Pipettieren zu vermeiden. Bereiten Sie den Master-Mix in einem Mikrozentrifugenröhrchen mit den folgenden Volumina pro Probe: 5 ul 2x Master Mix, 0,5 ul Primer Probe Mix, 0,5 ul Hemmung Primer Probe Mix, 0,05 ul ROX und 2,95 ul Verdünnungsmittel. Vortex für 10 sec und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalt zu sammeln.
- In 9 ul Master-Mix in die Vertiefungen einer 96-Well-Platte.
- 1 & mgr; l der DNA-Standards in doppelter Ausführung einer Eichkurve zu erzeugen. Mischen durch Pipettieren von up-and-down 5 mal.
- Stellen Sie sicher, die Nukleinsäureprobe vor dem Gebrauch gut gemischt ist. 1 & mgr; l Probe in den Master-Mix und mischen durch Pipettieren up-and-down 5 mal.
- Verschließen Sie die Platte, Vortex für 10 Sekunden und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalte zu sammeln.
- Die Platte wird in die PCR-System. Weisen beide FAM (funktionelle Quantifizierung) und VIC (funktionelle Hemmung) Detektoren für jede Probe gemäß den Anweisungen des Herstellers. Führen PCR-Zyklen von 10 min bei 95 ° C und 40 Zyklen (15 sec bei 95 ° C, 1 min bei 60 ° C).
- Analysieren Sie die qPCR-Daten durch eine lineare Regression Plot für jede der doppelten DNA-Standards zu erzeugen unter Verwendung von Softwareprotokollen.
- Plotten der Log 10 der Kopienzahl für jede DNA - Standard auf der x-Achse und dem entsprechenden FAM C q Wert auf der y-Achse.
- Bestätigen, dass die Ergebnisse aus der Nukleinsäureprobe fallen in den Dynamikbereich der DNA Standards Kalibrierungskurve und dann auf die Konzentration der unbekannten DNA in "funktionalen" berechnen oder amplifizierbare Kopienzahl pro & mgr; l von seiner entsprechenden Position auf der Referenzstandardkurve. Abbildung 1 zeigt Beispiele von Kalibrierungskurven , die vergangen und fehlgeschlagen.
- Bestimmen, ob eine Amplifikation in jeder Reaktion erfolgte durch die Anwesenheit des nicht-menschlichen Ziel Amplikon in der VIC Kanal überprüfen.
Anmerkung: Als Positivkontrolle wurde die Hemmung Primer Probe Mix Primer enthält der spezifisch für ein nicht-menschliches exogene Ziel und der entsprechenden Vorlage. Die Hemmung Primer Sondenmischung ist eine Komponente der Master-Mix, der zu jeder Reaktion hinzugefügt wird, einschließlich der Leerwert-Kontrolle (NTC). In Abwesenheit eines Inhibitors sollte das PCR-Produkt für die nicht-menschliches Ziel immer im VIC-Kanal detektiert werden. Ein "unerkannt" C q für eine Probe in den Kanal VIC zeigt die Anwesenheit von PCR - Inhibitoren , die aus nachfolgenden Bereinigungs der Probe vor der Weiterverarbeitung profitieren können.
2. Bibliotheks Zubereitung: Genspezifische (GS) PCR
- Eine ausreichende Menge an Master-Mix für die Gesamtzahl der Proben getestet werden und beinhalten 10% mehr Volumen Engpässe aufgrund pipett zu vermeidening. Bereiten Sie die GS - PCR - Master - Mix in einem Mikrozentrifugenröhrchen mit den folgenden Volumina pro Probe: 5 ul 2x Amplification Master Mix (Tabelle 1) und 1 ul Pan Cancer Primer Panel (Tabelle 1). Mischen durch Pipettieren von up-and-down, Vortex für 10 Sekunden und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalte zu sammeln.
- Aliquot der 6 ul GS-PCR-Master-Mix in die Vertiefungen einer 96-Well-Platte. In 4 ul jeder Nukleinsäureprobe in einzelne Wells. Zum anderen Vertiefungen, fügen 4 ul der FFPE Control (Tabelle 1), 4 ul der Multivariantensteuerung (Tabelle 1), und 4 ul Nuklease-freies Wasser für eine Verfahrens NTC. durch Pipettieren von up-and-down 5 mal für jeden zusätzlich mischen.
- Verschließen Sie die Platte, Vortex für 10 Sekunden und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalte zu sammeln.
- Die Platte wird in den Thermocycler für die folgenden PCR-Zyklen: 5 min bei 95 ° C,2 Zyklen (15 sec bei 95 ° C, 4 min bei 60 ° C), 23 Zyklen (15 sec bei 95 ° C, 4 min bei 72 ° C) und eine abschließende Verlängerung von 10 Minuten bei 72 ° C. Halten bei 4 ° C.
Hinweis: Nach Beendigung des Schritts 2.4, wird die Platte als die GS-PCR-Platte bezeichnet.
3. Bibliothek Vorbereitung: Tag PCR
- Thaw Reagenzien: 2x Index Master Mix (Tabelle 1) und Index - Codes (Tabelle 1). Vortex für 10 sec und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalt zu sammeln.
Hinweis: Index-Codes sind vorgemischten einen einzigartigen Satz von paarweise Indizes (Barcodes) für jede Probe zur Verfügung zu stellen. - In einer 96-Well-Platte, fügen 7,5 ul des 2x Index Master Mix und 5,5 ul eines Index-Code auf einen bestimmten gut und durch Pipettieren up-and-down 5 mal mischen.
- Öffnen Sie vorsichtig das GS-PCR-Platte, und mit 2 ul GS-PCR-Produkt auf die neue Platte mit dem Master-Mix. Mischen durch Pipettieren von up-and-down 5 mal. Für jede Probe, notieren Sie die Proben-ID und die entsprechenden paarweise Index-Codes. Verschließen Sie die Platte, Vortex für 10 Sekunden und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalte zu sammeln.
- Die Platte wird in den Thermocycler und PCR für 5 min bei 95 ° C, 10 Zyklen (30 sec bei 95 ° C, 30 sec bei 55 ° C, 1 min bei 72 ° C) und eine abschließende Extension von 10 min bei 72 ° C. Halten bei 4 ° C.
Anmerkung: Nach Beendigung des Schritts 3.4 wird die Platte bezeichnet werden als Tag PCR Platte.
4. Bibliothek Reinigung und Größenauswahl
- Entfernen Sie die Bibliothek Reine Prep magnetische Kügelchen (Tabelle 1) und Elution Buffer (Tabelle 1) von 2-8 ° C und lassen RT für 30 min äquilibrieren. In 9,6 ml 100% Ethanol zum Waschpuffer (Tabelle 1) Behälter, Deckel und mischen durch die Flasche mehrmals umgekehrt wird .
- Vortex die magnetischen Beads für 10 Sekunden und fügen Sie 11 ul in getrennte Vertiefungen einer 96-Well-Platte.
- Öffnen Sie den Tag PCR-Platte und fügen Sie 10 ul Tag PCR-Produkt an den Perlen und pipettieren Mischung 5 mal. Inkubieren der Mischung für 4 min bei RT.
- Legen Sie die 96-Well - Platte auf dem Magnetständer (Tabelle 1) für 4 min. Mit der 96-Well-Platte immer noch auf dem Stand, zu entfernen und den Überstand mit einer Pipette zu verwerfen.
- Entfernen Sie die 96-Well-Platte aus dem magnetischen stehen und mit 100 & mgr; l Ethanol enthaltenden Waschpuffer in jede Vertiefung und mischen durch Pipettieren up-and-down 5 mal. Inkubieren für 2 min.
- Legen Sie die 96-Well-Platte auf dem Magnetständer für 2 Minuten, dann entfernen und den Überstand mit einer Pipette zu verwerfen.
- Wiederholen Sie Schritt 4.5, für insgesamt 2 Ethanol wäscht, so viel Waschlösung möglich nach der zweiten Wäsche zu entfernen.
- Mit der 96-Well-Platte auf dem Magnetständer, trocknen die Kügelchen für 2 min bei RT, dann die Platte aus dem Ständer zu entfernen.
- Resuspendieren der Kügelchen durch Zugabe von 20 & mgr; l Elutionspuffer zu jedem well und pipettieren up-and-down 5 mal.
- Inkubieren für 2 min bei RT.
- Legen Sie die 96-Well-Platte wieder auf die magnetische Halterung für 4 min, und entfernen Sie vorsichtig und Transfer 18 ul des klaren Überstand zu einem gut neu.
Hinweis: Das Verfahren kann sicher bei diesem Schritt und die Proben bei -15 bis -30 ° C gelagert gestoppt werden. Zum Neustart auftauen gefrorenen Proben auf Eis, bevor Sie fortfahren.
5. Bibliothek Quantifizierung
- Thaw Bibliothek Quant (LQ) Reagenzien: 2x LQ Master Mix, LQ Primer / Probe - Mix, LQ Standard LQ Positive Kontrolle, LQ Diluent und LQ ROX (Tabelle 1). Vortex für 10 sec und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalt zu sammeln.
- Unter Verwendung von gereinigter Bibliotheksprodukte, führen Sie eine serielle Verdünnung von jeder einzelnen Probe in LQ Diluent.
- In 2 ul (Bibliothek gereinigtes Produkt) bis 198 & mgr; l LQ Diluent und mischen up-and-down mit einer Pipette 10 mal.
- Fügen Sie 2 ul (1: 100 Verdünnung) auf 1und 98 & mgr; l LQ Diluent mischen up-and-down mit einer Pipette 10 mal.
- Eine ausreichende Menge an LQ Mastermix für die Gesamtzahl von Proben getestet werden und umfassen 10% mehr Volumen Mangel aufgrund Pipettieren zu vermeiden. Bereiten Sie die LQ Master Mix in ein Mikrozentrifugenröhrchen folgende Volumina je Probe: 5 ul 2X LQ Master Mix, 2 ul LQ Primer / Probe-Mischung, 0,5 ul LQ Standard- und 0,5 ul LQ ROX. Mischen durch Pipettieren von up-and-down, Vortex für 10 Sekunden und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalte zu sammeln.
- In 8 ul LQ Master-Mix in eine Vertiefung einer optischen 96-Well-Platte.
- In getrennten Vertiefungen, fügen Sie 2 ul verdünnte Bibliothek, 2 ul LQ Positivkontrolle und 2 ul LQ Diluent (NTC) und mischen durch Pipettieren von up-and-down 5 mal. Verschließen Sie die Platte mit optischen Klebefilm, Vortex für 10 Sekunden und Zentrifuge bei 400 × g für 10 sec Inhalte zu sammeln.
- Weisen beide FAM und VIC Detectors für jede Probe. Führen PCR Amplifikation unter Verwendung von Zyklusbedingungen von 5 min bei 95 ° C und 40 Zyklen (15 sec bei 95 ° C, 1 min bei 60 ° C).
- Bestimmen Sie die Konzentration jeder Probe (nM) die vergleichende C q - Methode. Berechnen Sie die Differenz des bekannten LQ Standard (C q VIC) zu dem unbekannten Bibliothek (C q FAM). Die Konzentration der verdünnten Probe (pM) wird unter Verwendung der folgenden Gleichung berechnet:
[Konz lib] nM = 12,5 x 2 Δ Cq
Wenn ein Verdünnungsfaktor unter Verwendung von anderen als 10.000, wird das Verhältnis des Ziel-Verdünnungsfaktor bis 10.000 und multiplizieren diesen Faktor durch das Ergebnis der Gleichung 5.7 in.
6. Bibliothek Normalisierungs und Sample Pooling
- Bestimmen Sie die mittlere Konzentration (nM) in allen Proben (jeweils eine einzigartige paarweise Index enthält) gebündelt werden.
- Bestimmen Sie die einzelnen Proben volume (ul) durch Multiplikation der mittlere Konzentration in allen Proben von 5 zu bündeln, dann durch seine einzelne Konzentration (nM) dividiert wird. Um den resultierenden Wert auf die nächste ganze Zahl. Rund Volumen mit Werten von <2 ul bis 2 ul und Volumen> 15 & mgr; l bis 15 & mgr; l.
- Fügen Sie den normalisierten Volumen (ul) für jede Probe auf einen einzigen Mikrozentrifugenröhrchen die Probe Pool zu erstellen.
- Berechnen Sie die neue Konzentration für jede Probe mit gerundeten ganzzahligen Werte und die Ergebnisse aufzuzeichnen.
- Um die Konzentration der Probe Pool bestimmen, berechnen die Summe aller einzelnen Konzentrationen und notieren Sie die resultierende Wert (nM).
- Verdünnen Sie die Probe Pool auf 1,25 nM unter Verwendung von Sequenzierungs Diluent (Tabelle 1).
Hinweis: Das Verfahren kann sicher bei diesem Schritt und die Proben bei -15 bis -30 ° C gelagert gestoppt werden. Zum Neustart auftauen gefrorenen Proben auf Eis, bevor Sie fortfahren.
7. Sequenzierung
- Denature die Probe Pool in Gegenwart von PhiX Steuer v3 (Tabelle 1) durch Zugabe der folgenden: 15 & mgr; l von 1,25 nM Sample Pool, 3 ul 0,5 nM PhiX und 2 ul 1 N NaOH. Vortex kurz gefolgt von einer kurzen Zentrifugation und Inkubation für 5 min bei RT.
- Legen Sie die denaturierte Probe Pool auf dem Eis.
- In 8 ul denaturiert Bibliothek bis 992 & mgr; l vorgekühlte HT1-Hyb-Puffer zu einem Reaktionsgefäß. Vortex kurz zu mischen, gefolgt von einer kurzen Zentrifugierung Inhalt zu sammeln. Halten Sie sich auf dem Eis.
- In 600 ul der denaturierten und verdünnt Bibliothek # 17 der Reagenzienkartusche zu positionieren.
- Thaw 1 internationale Sequenzierprimer (Tabelle 1), Index lesen Sequenzierprimer (Tabelle 1) und 2 internationale Sequenzierprimer (Tabelle 1). In Mikrozentrifugenröhrchen, verdünnte separat 4 ul Sequenzierprimer mit 636 & mgr; l HT1-Hyb-Puffer. Hinweis: HT1-Hyb-Puffer ist vorgesehen Witzh die Sequenzierung Reagenzienkit (Tabelle 1).
- Mischen durch Vortexen für 10 sec und Zentrifuge bei maximaler Geschwindigkeit für 10 sec Inhalt zu sammeln. In 600 ul verdünnt 1 internationale Sequenzierprimer bis # 18 der Sequenzierung Reagenzpatrone Lage, 600 & mgr; l des verdünnten Index lesen Grundierungen auf Position # 19, und 600 & mgr; l verdünnt 2 internationale Sequenzierprimer # 20 zu positionieren.
- Laden der Reagenzien auf dem NGS Instrument (Tabelle 1) und die Sequenz entsprechend den Anweisungen des Herstellers. Führen Sie eine Paired-End 2 x 150 Zyklus-Sequenzierungslauf.
8. Datenanalyse
Hinweis: Die NGS Gerätesoftware wandelt Cluster Bilder zu Basis Anrufe und Qualität Partituren, und demultiplext paarweise Indizes einzelne gzip-komprimierte fastq zu erzeugen (* .fastq.gz) Dateien für jede Probe. Bevor die demultiplexten Dateien zu analysieren, muss der Leser herunterladen und die damit verbundenen Bioinformatik - Software (Tabelle installieren1). Die Software kann auf einem Consumer-Windows-PC installiert werden und erfordert keine spezielle Computer-Hardware oder eine Internetverbindung, die Datenanalyse durchzuführen.
- Doppelklicken Sie auf das Desktop-Symbol Software.
- Einloggen Benutzername und Passwort für das System im Software-Handbuch zur Verfügung gestellt werden.
- Öffnen Sie das Projekt-Dashboard, und klicken Sie auf "Neues Projekt".
- Nennen Sie das Projekt und bieten eine optionale Projektbeschreibung. Wählen Sie das gezielte NGS Panel-Typ und NGS Gerätetyp. Klicken Sie auf "Speichern und weiter".
- Laden Sie die komprimierten fastq-Dateien für die Vorwärts- und Rückwärts liest. Sie nicht, die "nicht zugeordneten" FASTQs laden, die enthalten liest, dass zu demultiplexen ist fehlgeschlagen. Klicken Sie auf "Speichern und weiter".
- Geben Sie die Anzahl der Funktionseingang Kopien verwendet, um jede Bibliothek herzustellen, wie durch die DNA-funktionelle Quantifizierung Test bestimmt (siehe Schritt 1 oben). Fügen Sie manuell Werte oder Kopieren und Einfügen von Werten aus einer VerbreitungBlatt in die Annotation-Tabelle. Klicken Sie auf "Speichern und weiter."
- Überprüfen Sie die kommentierten Bibliotheken für die Analyse hochgeladen und klicken Sie auf "Submit-Analyse", um die Analyse zu starten.
- Überwachen Sie den Fortschritt der Analyse durch das Projekt-Dashboard angezeigt.
Hinweis: Eine Analyse vollständige Statusanzeige dargestellt wird, wenn die Ergebnisse zur Überprüfung bereit sind. - Überprüfen Sie die analysierten Ergebnisse für Probe QC-Metriken, einschließlich Gesamtdeckung pro Bibliothek, Prozentsatz der liest vorbei Filter, Amplikon Abdeckung Tiefe und Einheitlichkeit. Überprüfen Sie die Variante für jede sequenziert Bibliothek ruft mit dbSNP, kosmisch, 1000 Genome und andere Quellen von funktionellen und Populationsebene Anmerkung.
- Exportieren Sie die rohen Ergebnisse als Zusammenfassung Tabellenkalkulationstabellen, * .bam Dateien und * VCF-Dateien für die Langzeitlagerung oder Downstream-Analyse mit komplementären Informatik-Tools.
Ergebnisse
Insgesamt 90 Proben (74 einzigartig), die positive und negative Kontrollen, zuvor charakterisierten Zelllinien und Rest klinischen FFPE Tumorbiopsien wurden für amplifizierbare DNA, Eingang in die Multiplex-PCR-Anreicherung bewertet, mit der Sequenz Adapter markiert, mit Barcode versehene und in einem einzigen analysiert Benchtop NGS Instrument Lauf (Abbildung 2) , die 19,1 M erzeugt liest Filter passiert. Equimolare Probe-Pooling in Folge hoher Tiefensequenzierung (3,692x liest) und gleichmäßige Abdeckung (97,8% der Amplicons innerhalb von 5-fache des mittleren Lesetiefe abgedeckt). Ausreißer besteht keine-Template - Kontrollen, eine Zelllinie DNA mit einer großen Kopienzahl Verstärkung und ein FFPE DNA , die für die PCR - Hemmung durch die präanalytische QC - Test (Abbildung 3) markiert war. Coverage Einheitlichkeit in den 46 Amplikons wurde unter Verwendung beibehalten drei verschiedene Operatoren (4A), und für andere minderwertige FFPE DNA - Proben ( 4B). Eine FFPE Tumor DNA Kontrolle, die aus einer Mischung von Rest klinischen Proben formuliert 5% BRAF V600E zu erreichen (quantifiziert durch Tröpfchen digital PCR), berichtet wurde, die Ziel BRAF-Mutation an Abundanzen von 3,9, 5,3 und 6,5% nach drei Betreiber haben unter Verwendung ein Eingang von 400 amplifizierbare Kopien (und damit nur 20 mutierten Kopien) (4B und "FFPE3" ausheben Tabelle). Weiterhin offenbart ein Gemisch aus 12 synthetischen DNA - Matrizen, die jeweils eine bekannte "driver" base-Substitutionsmutation darstellt, die erwarteten Mutationen an den vorgesehenen Bereich von 9-17% mittlere Allelfrequenz (Tabelle 2). Verdünnung der Zelllinie und FFPE DNA - Proben mit Kopienzahl Amplifikationen nachgewiesen Dosisabhängigkeit für Varianten in EGFR und KRAS, bzw. (Abbildung 5). Wichtig könnte, FFPE DNA-Eingang, so wenig wie 50 amplifizierbare Kopien oder 1,2 ng von Bulk-DNA reduziert werden, während die Erkennung von bekannten Mutationen ohne falsch-positive ErhaltungAnrufe (Abbildung 6). DNA Eingänge wurden über einen 100-fachen Bereich von bis zu mindestens 50.000 Kopien amplifizierbaren (Tabelle 3) untergebracht ist . In diesem und ähnlichen Experimenten Variante ruft in 22 FFPE und 20 FNA Proben wurden in Übereinstimmung mit unabhängigen Methoden mit gemeinsamen Mutation Abdeckung (Tabelle 4) berichtet.
Die Sensitivität und positiver prädiktiver Wert für den Test wurde aus einer Analyse von 97 Proben bestimmt, einschließlich FFPE, FNA, frisch eingefroren, und Zell-Linie DNA und insgesamt 195 Sequenzierungsergebnisse. Die Ergebnisse zeigten, 365 echte positive Variante Anrufe, 4 falsch negative Anrufe und 1 falsch positiven Ruf nach einer Sensitivität von 98,9% (95% CI: 97,1-99,7%) und ein positiver prädiktiver Wert (PPV) von 99,7% (95% CI : 98,2 bis 99,99%). Die Analysen von indels wurden zwei gemeinsame EGFR-Varianten (p.E746_A750delELREA und p.V769_D770insASV) durchgeführt in 33 Probeläufe, eine Sensitivität von 93,9% festgestellt (95% CI: 78,4-98,9%) und ein PPV von 100% (95% CI: 86,3 bis 100%) mit Varianten erfasst einen Bereich von 2,4 bis 84,8% gegenüber.
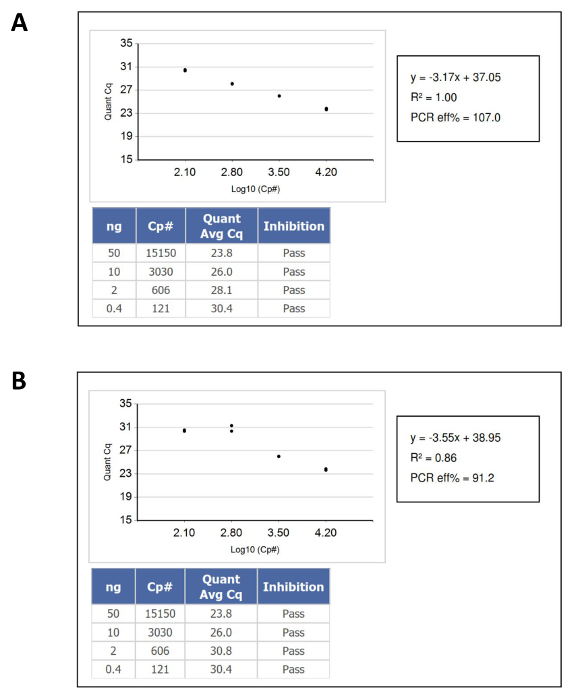
Abbildung 1: Ein Beispiel für DNA - Quantifizierung Kalibrierung Kurven, Pass und Fail - QC Kriterien (A) Eine Weitergabe Standardkurve.. (B) Eine ausbleibende Standardkurve. In diesem Fall wurde der Fehler durch doppelte Pipettieren der niedrigsten Eingangs DNA - Standard verursacht werden . Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
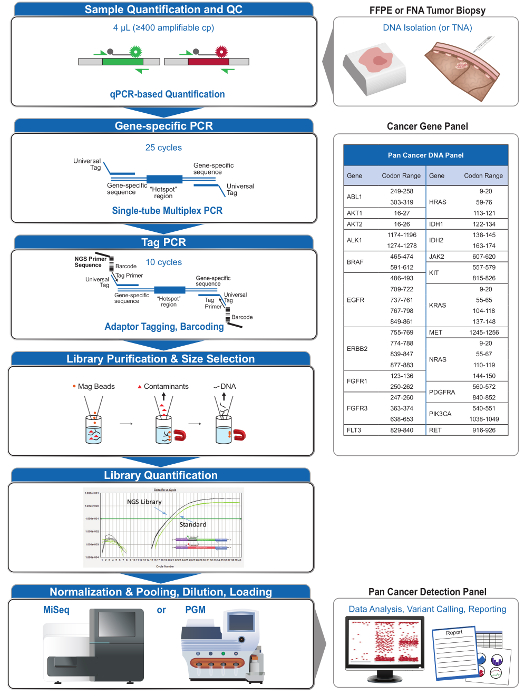
Abbildung 2: Überblick über die Entwicklung einer umfassenden gezielten NGS - System für Onkologie Anwendungen , die Integration voranalytischenal, analytische und Post-analytischen Workflows. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
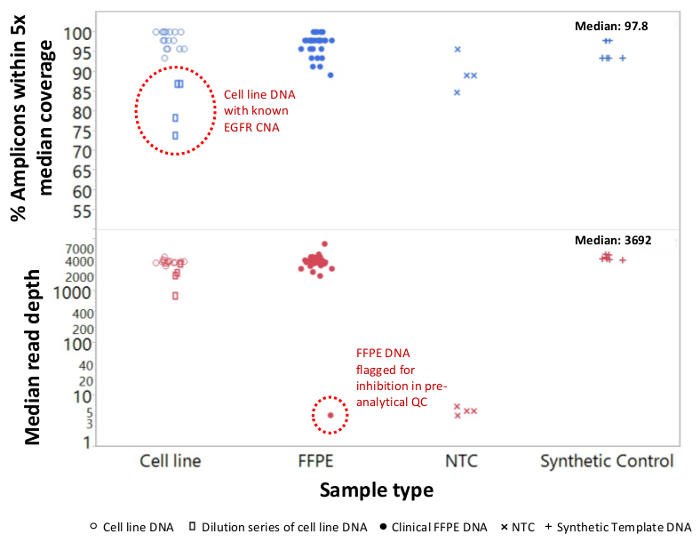
Abbildung 3: Lesen Sie Coverage und Gleichmäßigkeit für Targeted NGS von Krebs - Gene in Low-Qualität FFPE DNA im Vergleich zu Intakte Zelllinien - DNA und Kontrollen Insgesamt 90 Proben , die Rest klinischen FFPE (geschlossene Kreise) enthalten, Zell-Linie (offene Kreise. ) und synthetische Templat-DNA (plus Symbole) wurden unter Verwendung von Single-Tube, 21-Gen multiplex PCR-Anreicherung verarbeitet. Jede Amplikon Bibliothek wurde mit Adaptersequenzen für die NGS Instrument tagged barcodiert mit einem ausgeprägten Doppelindexcode, gereinigt, quantifiziert und normiert auf eine Konzentration von 2,5 nM. Die DNA-Bibliothek wurde von der Begleit-Bioinformatik-Software sequenziert und analysiert. Beispiel Devitionen enthalten eine Verdünnungsreihe von MDA-MB-468-Zelllinie DNA eine große EGFR Kopienzahl Verstärkung Lager (oben, offene Rechtecke innerhalb gepunkteten Kreis), die eine Abdeckung Gleichförmigkeit ein Melanom FFPE Probe, die eine beträchtliche Anzahl von liest aufgrund erzeugen fehlgeschlagen verzerrt und um Verschleppung von PCR-Inhibitoren aus der DNA-Extraktion. Das Melanom Versagen der Probe (unten, gepunkteten Kreis) wurde durch die präanalytische qPCR DNA QC-Test vorhergesagt. NTC, No-Template - Steuerung (x Symbole). Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
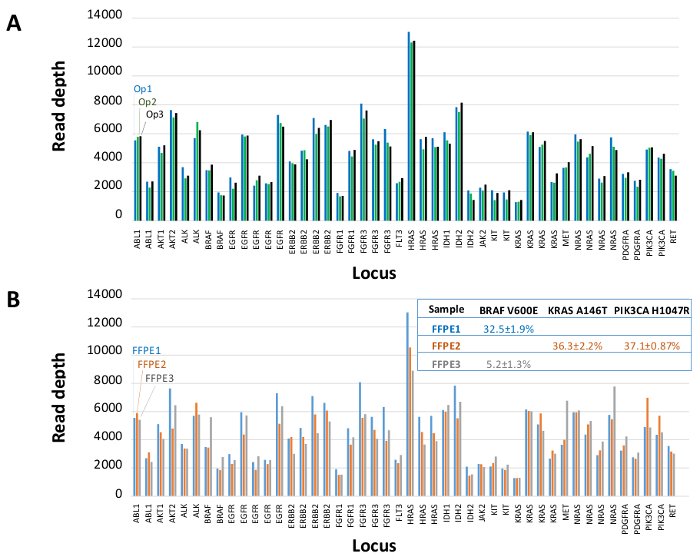
Abbildung 4: Amplicon-by-Amplikon lesen Coverage, Homogenität und Variantenerkennung in Residual Clinical FFPE Tumor - DNA. (A) Lesen Sie Berichterstattung über alle angereicherten Loci in einer repräsentativen DNA - Probe FFPE gemessenin drei verschiedenen Betreibern. Operator 1, Op1 (blaue Balken); Operator 2, Op2- (grüne Balken); Operator 3, Op3 (schwarze Balken). (B) Coverage Einheitlichkeit und Varianten Anrufe ausgewertet unter Verwendung von drei FFPE Tumorproben, einschließlich einer Kontrollmischung (FFPE3, graue Balken und Text) , die aus einer bekannten 5% BRAF c.1799T> Eine Mutation. FFPE1 (blaue Balken und Text), FFPE2 (orange Balken und Text). Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
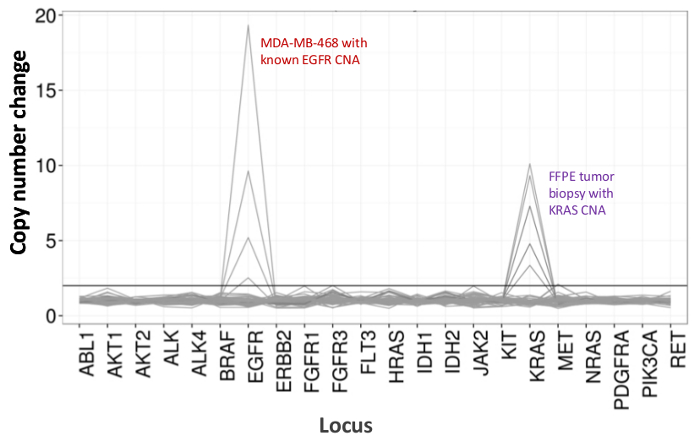
Abb . 5: Dosisabhängige Detektion von Copy Number Varianten in Zell-Linie und FFPE DNA MDA-MB-468 - Zelllinie DNA mit einem gut charakterisierten EGFR Kopienzahl - Amplifikation wurde schrittweise in einem Hintergrund von einer nicht-mutierten Zellbezug verdünnt -Linie DNA, die die Abnahme der Kopienzahl Änderung als zu illustrierenFunktion der Verdünnung. Der Prozentsatz jeder Zelllinie DNA Probe wird mit einer bestimmten Leitung (0, 25 12,5, 50 und 100%) gezeigt. Die Verdünnung eines ovarian FFPE Tumorprobe mit einer bekannten KRAS - Amplifikation ergab ein ähnliches Profil die gleiche Titrationsreihe verwenden , aber mit Replikaten von 100% FFPE DNA für die beiden oberen Zeilen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
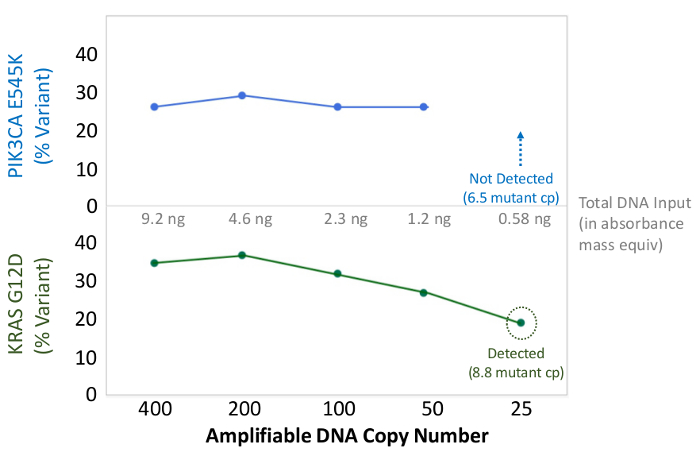
Abbildung 6: Genaue Mutationsnachweis und Quantifizierung bis 50 Amplifizierbare FFPE DNA - Kopien oder 1,2 ng Bulk - DNA Das amplifizierbare Kopienzahl eines Darmkrebs FFPE DNA durch die qPCR-basierte QC - Test bestimmt wurde, und 400 bis 25 Kopien als verdünnt. Eingabe in Multiplex-PCR-Anreicherung vor der Sequenzierung. Die Bioinformatik-Pipeline genannt korrekt sowohl der bekannten Variants bis zu 50 Kopien, oder das Äquivalent von ~ 10 Mutant - Vorlagen. Bitte klicken Sie hier , um eine größere Version dieser Figur zu sehen.
| Name des Materials / Material | Unternehmen | Katalognummer |
| 2x Quantidex Master Mix | Asuragen | 145345 |
| Quant Primer Probe Mix | Asuragen | 145336 |
| Die Hemmung Primer Probe Mix | Asuragen | 145344 |
| ROX | Asuragen | 145346 |
| Diluent | Asuragen | 145339 |
| DNA Standard (50) | Asuragen | 145340 |
| DNA Standard (10) | Asuragen | 145341 |
| DNA Standard (2) | Asuragen | 145342 |
| DNA-Standard (0,4) | Asuragen | 145343 |
| 2x Amplification Master Mix | Asuragen | 145348 |
| Pan Cancer Primer-Panel | Asuragen | 145347 |
| Pan Cancer FFPE Kontrolle | Asuragen | 145349 |
| Pan Cancer Multi-Variante Control | Asuragen | 145350 |
| Bibliothek Reine Prep Beads | Asuragen | 145351 |
| Waschpuffer | Asuragen | 145352 |
| Elution Buffer | Asuragen | 145353 |
| 2x LQ Master Mix | Asuragen | 145358 |
| LQ Diluent | Asuragen | 145354 |
| LQ PositiveSteuern | Asuragen | 145355 |
| LQ Norm | Asuragen | 145356 |
| LQ Primer / Probe Mix (ILMN) | Asuragen | 145357 |
| LQ ROX | Asuragen | 145359 |
| Index-Codes (ILMN) - Set A | Asuragen | 150004 |
| AIL001 - AIL048 (48) | ||
| Index-Codes (ILMN) - Set B | Asuragen | 150005 |
| AIL049 - AIL096 (48) | ||
| 2x Index Master Mix | Asuragen | 145361 |
| Lesen Sie 1 Sequenzierprimer | Asuragen | 150001 |
| Index lesen Sequenzierprimer | Asuragen | 150002 |
| Lesen Sie 2 Sequenzierprimer | Asuragen | 150003 |
| Sequencing Diluent | Asuragen | 145365 |
| Illumina MiSeq | Illumina | |
| MiSeq Reagenz-Kit v3 (600-Zyklus) | Illumina | MS-102-3003 |
| MiSeq Reagenz Nano Kit v2 (300-Zyklus) | Illumina | MS-103-1001 |
| PhiX Steuerung v3 | Illumina | FC-110-3001 |
| Magnetische Stand-96 (oder einer gleichwertigen Einrichtung) | Ambion | AM10027 |
| Quantidex Reporter Software | Asuragen |
Tabelle 1:. Reagenzien und Kits Bei der ersten Verwendung des ROX, speichern Sie die Fläschchen bei 2-8 ° C. Nicht wieder einfrieren. Die Software kann auf www.asuragen.com heruntergeladen werden.
| Gen | COSMIC Variante | COSMIC Aminosäure | % Variant |
| NRB | c.182A> G | p.Q61R | 13.3 |
| NRB | c.35G> A | p.G12D | 15.2 |
| HRAS | c.182A> G | p.Q61R | 17.8 |
| HRAS | c.35G> A | p.G12D | 9.2 |
| KRAS | c.182A> G | p.Q61R | 13.5 |
| KRAS | c.35G> A | p.G12D | 19.1 |
| PIK3CA | c.1633G> A | p.E545K | 9.3 |
| PIK3CA | c.3140A> G | p.H1047R | 9.1 |
| KIT | c.2447A> T | p.D816V | 14.6 |
| EGFR | c.2369C> T | p.T790M | 11.3 |
| EGFR | c.2573T> G | p.L858R | 14.9 |
| BRAF | c.1799T> A | p.V600E | 17.3 |
Tabelle 2: A Pooled Synthetic Steuerung besteht aus 12 "Driver" Krebs - Gen - Varianten , die bei 9-17% Fülle Quantifizierte sind eine Mischung aus 12 verschiedenen doppelsträngigen synthetischen Vorlagen 12 verschiedene Mutationen trägt , wurde folgende Sequenzierung ausgewertet.. Alle Varianten wurden korrekt ohne Fehlalarme bezeichnet.
>% Variant| Proben - ID | Funktions- cps | Gen | COSMIC Variante | COSMIC Aminosäure | Median Lesetiefe | % Innerhalb von 5 - fach des Median | |
| BCPAP | 400 | BRAF | c.1799T> A | p.V600E | 99.5 | 3289 | 96% |
| BCPAP | 10.000 | BRAF | c.1799T> A | p.V600E | 99,7 | 4040 | 98% |
| BCPAP | 25.000 | BRAF | c.1799T> A | p.V600E | 99.4 | 3687 | 96% |
| BCPAP | 50.000 | BRAF | c.1799T> A | p.V600E | 99,7 | 4611 | 93% |
Tabelle 3: Coverage und Variant Gespräche über ein konserviertes> 100-fache Reichweite von DNA - Eingang. Amplifizierbare DNA aus einer BCPAP Zelllinie eingegeben wurde in Multiplex-PCR-Anreicherung bei 400 bis 50.000 Exemplaren und sequenziert. Lesen Tiefe, Abdeckung Einheitlichkeit, Detektion von Mutationen und Mutations Genauigkeit wurden über den Eingangsbereich erhalten.
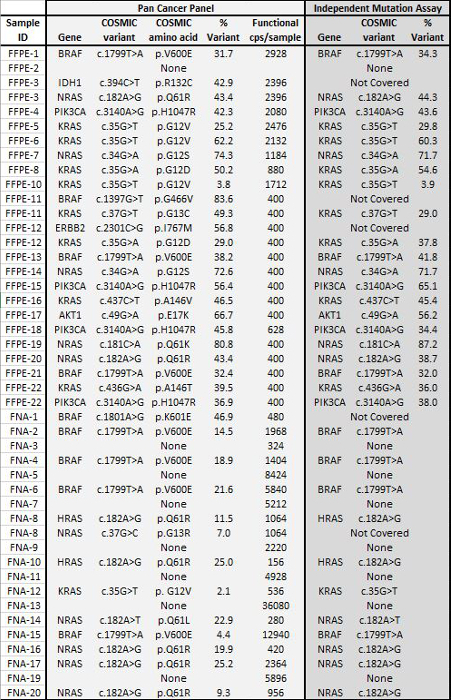
Tabelle 4: Variante Anrufe in 22 FFPE und 20 FNA Tumorbiopsien Vereinbaren Sie mit Ergebnisse von unabhängigen Mutationstests Ein Satz von 22 FFPE Tumor - DNA mit Mutationsstatus zuvor durch orthogonale gezielte NGS - Assays bestimmt eingegeben wurde bei 400 bis 2928 amplifizierbare Kopien in die PCR - Anreicherung. Schritt und sequenziert das 21-Gen Pan Cancer Panel. Darüber hinaus charakterisiert eine Kohorte von 20 FNA DNA - Proben zuvor eine flüssige Perlenanordnung Mutationstest unter Verwendung von 8 wurde PCR amplifizierbaren Kopien und sequenziert unter Verwendung von 156 bis 36.080 Eingang verstärkt. Alle überlappenden Anrufe zwischen dem Pan Cancer NGS-Panel und der reference Methoden waren zustimmend. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
Diskussion
NGS Technologien haben Erwartungen umdefiniert 9 die molekularen Profilen von Tumorbiopsien in klinischen Einrichtungen zur Abfrage. Eine Reihe von gezielten NGS Platten wurden als Forschungstechnologien, Labor entwickelten Tests entwickelt und im Handel erhältliche Produkte und kundenspezifische Panels für 3,5,10-15 mehrere Arten von klinischen Proben zu bewerten. Berichte von mehreren Studien haben den Wert von NGS als empfindliche und spezifische klinisches Werkzeug für den Nachweis von genomischer Veränderungen 3,10-12,14 demonstriert. Noch Studien haben gezeigt , auch das Risiko von Artefakten , die falsch-positive Ergebnisse aus anspruchsKrebsBiopsien wie FFPE - Proben 2-5,12,14 verursachen können. Darüber hinaus haben die jüngsten Publikationen Raten hohe Ausfall für NGS hervorgehoben Onkologie mit Proben 16 und falsch-positive Gespräche mit kommerziellen gezielte NGS Platten , die 12 durch die Verwendung von Low-Input - DNA noch verschärft werden. Als Ergebnis einige laboraTories haben entweder geändert oder zusätzliche Kontrollen und Salden zusätzliche Leistung zu kommerziell erhältlichen gezielte NGS - Technologien zu verbessern, oft die Genauigkeit zu gewährleisten oder die Ergebnisse bestätigen aus der bioinformatischen 5,10,11 analysiert.
Der 21-Gen - Panel (Abbildung 2) wurde als zielgerichtete Inhalte für ein umfassendes NGS System entwickelt , evidenzbasierte, umsetzbare Mutationen in schwierigen Probentypen wie FFPE und FNA Tumorbiopsien zu befragen. Der Workflow hat eine Reihe von Vorteilen: 1) Konsistenz , indem sie einheitliche Amplikon Abdeckung (3 und 4, Tabelle 3); 2) die Einfachheit der Nutzung durch die Bereitstellung vorformulierten, optimierte Reagenzien-Sets, und Vereinfachung der Bioinformatik Anforderungen; 3) Effizienz durch den Workflow zu straffen und die Anzahl der Pipettierschritte reduziert im Vergleich zu anderen kommerziellen NGS-Methoden; und 4) Genauigkeit durch eine DNA-QC-Test für die Beurteilung der Endverstärker EinbeziehungLage , die DNA - Kopienzahl auf akzeptable Vorlage sicherstellen und stochastischen Schwankungen in der Variantenerkennung 4 zu vermeiden. Die Integration der präanalytischen QC-Daten mit der bioinformatischen Auswertung ermöglicht niedrige Eingänge von FFPE DNA. Dies wurde durch die Ausbildung einen Entscheidungsbaum-Algorithmus unter Verwendung von verschiedenen funktionellen Kopien der DNA-Eingabe über 400 FFPE Proben mit unabhängigen Maßnahmen der Wahrheit erreicht, und die Einbeziehung dieses Algorithmus in die bioinformatischen Software. Als Ergebnis vergleicht der empfohlenen Eingang 400 amplifizierbare Kopien typischerweise äquivalent zu ~ 5-20 ng von FFPE DNA, vorteilhaft mit anderen Methoden 10-12, einschließlich Hybridisierungs-Anreicherungs wo ~ 250 ng DNA FFPE 17,18 empfohlen . Obwohl die Technik zur Verwendung auf einer MiSeq Plattform beschrieben wird, kann es durch die Verwendung von PCR-Primern Tag mit gerätespezifischen Adaptern modifiziert werden, um die Sequenzanalyse auf anderen NGS-Plattformen ermöglichen.
Mehrere Schritte sind entscheidend den Erfolg sicherzustellendes Verfahrens. Die präanalytische QC-Test bestimmt die amplifizierbare Kopienzahl von DNA und Berichte funktionellen Hemmung. Wenn jedoch weniger als 400 amplifizierbaren DNA - Kopien in der PCR Anreicherungsschritt verwendet werden, besteht ein erhöhtes Risiko eines falsch-negativen Anruf von Proben mit niedriger Abundanz Mutationen (Abbildung 6). Außerdem muss darauf geachtet werden während der Bibliotheks Reinigung zu verhindern Übertrocknung der magnetischen Beads in den Wasch- oder Elutionsschritte genommen werden. Darüber hinaus ist erfolgreich Bibliothek Quantifizierung über die genaue Verdünnung von Bibliotheks-DNA in hohem Maße abhängig. Für das beste Ergebnis ergibt sich die Differenz der qPCR für die Probe-Bibliothek (CQ FAM) im Vergleich zum LQ Standard (Cq VIC) sollte ≤3.3 Cq sein. Wenn die Differenz größer als 3,3 Cq, Wiederverdünnung und Testen der Probe wird empfohlen. Obwohl eine ausgezeichnete Korrelation zwischen dieser Wettbewerbs qPCR-Methode und kommerzielle Kits, die bieten absolute Quantifizierung unter Verwendung einer Standardkurve beobachtetEine der Bibliothekseintrag in den klonalen Amplifikationsschritt relativ zu anderen Methoden Offset kann notwendig sein, eine optimale seeding Dichte zu erreichen.
Einige Krebsproben sind eine Herausforderung vor allem wegen der Inhibitoren zu sequenzieren, die nach der DNA-Isolierung bestehen. Um diese Proben vor der Bibliothek Vorbereitung identifizieren, erkennt der qPCR QC-Test auch Amplifizierungshemmung durch eine exogene Vorlage einschließlich, die als sowohl eine interne Kontrolle und ein Wächter für die funktionelle Hemmung dient. Ein Beispiel ist in Abbildung 3 , in dem ein Melanom DNA Probe versagt stellte die QC Hemmung metric vor der Sequenzierung zu passieren und dann versagt , eine Bibliothek zu erzeugen , die sequenziert werden konnten. Das Scheitern war wahrscheinlich eine Folge von Melanin Kontamination, einem bekannten PCR-Inhibitor, aus der FFPE DNA-Isolierung Schritt übertragen. Die Proben, die durch den QC-Test identifiziert werden, bei Gefahr für die Amplifikation Versagen sein kann durch einen zusätzlichen Reinigungsschritt t geborgen werdeno entfernen potentielle Inhibitoren.
Die gezielte 21-Gen-Panel konzentriert sich auf evidenzbasierte Gen-Hotspots und bietet ein komplettes System mit optimierten Reagenzien und Kontrollen für die DNA-QC, NGS und Bioinformatik-Software, die von präanalytischen "funktional" DNA-Quantifizierung Ergebnisse informiert wird. Das Verfahren genau erkennt Basensubstitutionsmutationen und indels von low-input DNA und stellt ein Beispiel eines NGS System mit der Option Panel Inhalt zu erweitern, zusätzliche Varianten wie CNV zu erkennen und zur gezielten RNA-Sequenzierung angepasst werden.
Offenlegungen
JH, AH, RZ, BCH und GJL sind Mitarbeiter und haben Aktienbesitz in Asuragen, Inc. RZ, BCH und GJL sind Miterfinder auf einer Patentanmeldung für die Verbesserung der Variante für jede Probe bestimmt unter Verwendung von amplifizierbaren Kopienzahl Informationen aufrufen.
Danksagungen
Wir danken Dr. Annette Schlageter für die Überprüfung des Manuskripts. Diese Arbeit wurde teilweise durch Gewährung CP120017 von der Cancer Prevention Research Institute of Texas (: GJL PI) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 2x Quantidex Master Mix | Asuragen | 145345 | |
| Quant Primer Probe Mix | Asuragen | 145336 | |
| Inhibition Primer Probe Mix | Asuragen | 145344 | |
| ROX | Asuragen | 145346 | |
| Diluent | Asuragen | 145339 | |
| DNA Standard (50) | Asuragen | 145340 | |
| DNA Standard (10) | Asuragen | 145341 | |
| DNA Standard (2) | Asuragen | 145342 | |
| DNA Standard (0.4) | Asuragen | 145343 | |
| 2X Amplification Master Mix | Asuragen | 145348 | |
| Pan Cancer Primer Panel | Asuragen | 145347 | |
| Pan Cancer FFPE Control | Asuragen | 145349 | |
| Pan Cancer Multi-Variant Control | Asuragen | 145350 | |
| Library Pure Prep Beads | Asuragen | 145351 | |
| Wash Buffer | Asuragen | 145352 | |
| Elution Buffer | Asuragen | 145353 | |
| 2X LQ Master Mix | Asuragen | 145358 | |
| LQ Diluent | Asuragen | 145354 | |
| LQ Positive Control | Asuragen | 145355 | |
| LQ Standard | Asuragen | 145356 | |
| LQ Primer/Probe Mix (ILMN) | Asuragen | 145357 | |
| LQ ROX | Asuragen | 145359 | |
| Index Codes (ILMN) - Set A, AIL001 - AIL048 (48) | Asuragen | 150004 | |
| Index Codes (ILMN) - Set B, AIL049 - AIL096(48) | Asuragen | 150005 | |
| 2x Index Master Mix | Asuragen | 145361 | |
| Read 1 Sequencing Primers | Asuragen | 150001 | |
| Index Read Sequencing Primers | Asuragen | 150002 | |
| Read 2 Sequencing Primers | Asuragen | 150003 | |
| Sequencing Diluent | Asuragen | 145365 | |
| Illumina MiSeq | Illumina | ||
| MiSeq Reagent Kit v3 (600-cycle) | Illumina | MS-102-3003 | |
| MiSeq Reagent Nano Kit v2 (300-cycle) | Illumina | MS-103-1001 | |
| PhiX Control v3 | Illumina | FC-110-3001 | |
| Magnetic Stand-96 (Or equivalent device) | Ambion | AM10027 | |
| Quantidex Reporter Software | Asuragen |
Referenzen
- Chen, G., Mosier, S., Gocke, C. D., Lin, M. T., Eshleman, J. R. Cytosine deamination is a major cause of baseline noise in next-generation sequencing. Mol Diagn Ther. 18 (5), 587-593 (2014).
- Choudhary, A., et al. Evaluation of an integrated clinical workflow for targeted next-generation sequencing of low-quality tumor DNA using a 51-gene enrichment panel. BMC Med Genomics. 7, 62 (2014).
- Sah, S., et al. Functional DNA quantification guides accurate next-generation sequencing mutation detection in formalin-fixed, paraffin-embedded tumor biopsies. Genome Med. 5 (8), 77 (2013).
- Zhang, L., et al. Profiling cancer gene mutations in clinical formalin-fixed, paraffin-embedded colorectal tumor specimens using targeted next-generation sequencing. Oncologist. 19 (4), 336-343 (2014).
- Latham, G. J. Next-generation sequencing of formalin-fixed, paraffin-embedded tumor biopsies: navigating the perils of old and new technology to advance cancer diagnosis. Expert Rev Mol Diagn. 13 (8), 769-772 (2013).
- Crawford, J. M., et al. The business of genomic testing: a survey of early adopters. Genet Med. 16 (12), 954-961 (2014).
- Smith, D. L., et al. A multiplex technology platform for the rapid analysis of clinically actionable genetic alterations and validation for BRAF p.V600E detection in 1549 cytologic and histologic specimens. Arch Pathol Lab Med. 138 (3), 371-378 (2014).
- Thomas, F., Desmedt, C., Aftimos, P., Awada, A. Impact of tumor sequencing on the use of anticancer drugs. Curr Opin Oncol. 26 (3), 347-356 (2014).
- Singh, R. R., et al. Clinical validation of a next-generation sequencing screen for mutational hotspots in 46 cancer-related genes. J Mol Diagn. 15 (5), 607-622 (2013).
- Beadling, C., et al. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping. J Mol Diagn. 15 (2), 171-176 (2013).
- McCall, C. M., et al. False positives in multiplex PCR-based next-generation sequencing have unique signatures. J Mol Diagn. 16 (5), 541-549 (2014).
- Schleifman, E. B., et al. Next generation MUT-MAP, a high-sensitivity high-throughput microfluidics chip-based mutation analysis panel. PLoS One. 9 (3), e90761 (2014).
- Wong, S. Q., et al. Sequence artefacts in a prospective series of formalin-fixed tumours tested for mutations in hotspot regions by massively parallel sequencing. BMC Med Genomics. 7, 23 (2014).
- Narayan, A., et al. Ultrasensitive measurement of hotspot mutations in tumor DNA in blood using error-suppressed multiplexed deep sequencing. Cancer Res. 72 (14), 3492-3498 (2012).
- Hagemann, I. S., et al. Clinical next-generation sequencing in patients with non-small cell lung cancer. Cancer. 121 (4), 631-639 (2015).
- Won, H. H., Scott, S. N., Brannon, A. R., Shah, R. H., Berger, M. F. Detecting somatic genetic alterations in tumor specimens by exon capture and massively parallel sequencing. J Vis Exp. (80), e50710 (2013).
- Simen, B. B., et al. Validation of a next-generation-sequencing cancer panel for use in the clinical laboratory. Arch Pathol Lab Med. 139 (4), 508-517 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten