Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Todo el cuerpo de Espectrometría de Masas de imágenes por infrarrojos asistida por matriz de desorción láser de ionización electrospray (IR-MALDESI)
En este artículo
Resumen
A mass spectrometry imaging (MSI) source operated at atmospheric pressure was developed by coupling mid-infrared laser desorption and electrospray post-ionization. Exogenous ice matrix was used as the energy-absorbing matrix to facilitate resonant desorption of tissue-related material. This manuscript provides a step-by-step protocol for performing IR-MALDESI MSI of whole-body neonatal mouse.
Resumen
fuentes de ionización del ambiente para la espectrometría de masas (MS) han sido objeto de gran interés en la última década. Láser asistida por matriz de desorción ionización por electrospray (MALDESI) es un ejemplo de tales métodos, en los que las características de láser de desorción / ionización asistida por matriz (MALDI) (por ejemplo, de la naturaleza pulsante de desorción) y la ionización por electrospray (ESI) (por ejemplo, soft-ionización ) se combinan. Una de las principales ventajas de MALDESI es su versatilidad inherente. En experimentos MALDESI, una luz ultravioleta (UV) o un láser de infrarrojos (IR) se pueden usar para excitar resonantemente una matriz endógena o exógena. La elección de la matriz no es analito dependiente, y depende únicamente de la longitud de onda de láser que se utiliza para la excitación. En los experimentos de IR-MALDESI, una fina capa de hielo se deposita sobre la superficie de la muestra como una matriz de absorción de energía. La geometría de la fuente IR-MALDESI se ha optimizado el uso de diseño estadístico de experimentos (DOE) para el análisis de muestras líquidas, así como biolmuestras de tejido ogical. Además, una fuente de formación de imágenes IR-MALDESI robusto ha sido desarrollado, donde un láser de infrarrojo medio sintonizable está sincronizado con una etapa de traslación XY controlado por ordenador y un espectrómetro de masas de alto poder de resolución. Una interfaz de usuario personalizada gráfica (GUI) el usuario puede seleccionar la frecuencia de repetición del láser, el número de disparos por voxel, el paso de tamaño de la etapa de la muestra, y el retardo entre la desorción y escanear eventos para la fuente. IR-MALDESI se ha utilizado en diversas aplicaciones tales como el análisis forense de fibras y colorantes y MSI de secciones de tejidos biológicos. Distribución de los diferentes analitos que van de metabolitos endógenos a xenobióticos exógenos dentro de secciones de tejido se puede medir y se cuantificó usando esta técnica. El protocolo presentado en este manuscrito describe los principales pasos necesarios para IR-MALDESI MSI de secciones de tejido de todo el cuerpo.
Introducción
formación de imágenes Espectrometría de masas (MSI) en el modo de microsonda implica la desorción de la muestra de una superficie por un haz (láser o iones) en localizaciones discretas sobre la superficie de una muestra. En cada punto de trama, se genera un espectro de masas y los espectros adquiridos, junto con la ubicación espacial de la que se recogieron, se puede utilizar para mapear simultáneamente numerosos analitos dentro de la muestra. Esta forma libre de etiquetas de imágenes acoplado a la sensibilidad y la especificidad de la espectrometría de masas han ayudado MSI convertirse en uno de los campos más rápida evolución en la espectrometría de masas 1,2.
Asistida por matriz de desorción / ionización por láser (MALDI) es el método de ionización más común utilizado para los análisis de MSI. Sin embargo, la necesidad de una matriz orgánica y los requerimientos de vacío de MALDI plantean limitaciones significativas en la reproducibilidad, rendimiento de la muestra, y los tipos de muestras que pueden ser analizadas usando el método. Un número de io presión atmosférica (AP)métodos nización se han desarrollado en los últimos años para eludir estas restricciones 3. Estos métodos de ionización ambiente permiten el análisis de muestras biológicas en un ambiente que está mucho más cerca a su estado natural y simplificar los pasos de preparación de muestras antes del análisis. Asistida por matriz de ionización de desorción láser electrospray (MALDESI) es un ejemplo de un 4,5 tal método de ionización.
En los experimentos de IR-MALDESI, una fina capa de hielo se deposita sobre la superficie del tejido como la matriz de absorción de energía. Un pulso de láser infrarrojo medio es absorbido por la matriz de hielo, y facilita la desorción de materiales neutros de la superficie por resonante excitar el modo de OH de agua estiramiento. La partición neutrales desorbidos en las gotitas cargadas de un electrospray ortogonal y son de post-ionizada de un modo ESI-como 4-6. La adición de la matriz de hielo exógeno es preferible a confiar únicamente en el agua endógena en el tejido, ya que ayuda accontar para variaciones en el contenido de agua en diferentes compartimentos de tejido, y se ha demostrado para mejorar la desorción 6 y mejorar la abundancia de iones por ~ 15 veces 7,8 en los experimentos de formación de imágenes de tejidos.
En este trabajo, utilizamos IR-MALDESI MSI para provocar la distribución de metabolitos a través de diferentes órganos en todo el cuerpo del ratón neonatal. Se da una visión general de los parámetros ajustables de la fuente de IR-MALDESI, y los pasos necesarios para la formación de imágenes con éxito de las secciones de tejido se ponen de manifiesto.
Protocolo
Nota: El siguiente protocolo describe todos los pasos necesarios para llevar a cabo experimentos IR-MALDESI MSI. En profundidad los detalles sobre la geometría optimizada de la fuente de IR-MALDESI y su sincronización con el láser, el estadio y espectrómetro de masas se pueden encontrar en otros lugares 5,6. muestras de tejido de los animales utilizados en este protocolo se obtuvieron de Cuidado de Animales institucional y el empleo Comisión (IACUC) y los reglamentos de la Universidad Estatal de Carolina del Norte.
1. Preparación del tejido
- Preparar un baño de hielo isopentano / seco mediante la colocación de ~ 200 ml de isopentano en un vaso de precipitados limpio dentro de un recipiente secundario de hielo seco en una campana de humos. Usar guantes y gafas de seguridad en todo momento al manipular el isopentano baño / hielo seco y el tejido.
- La eutanasia a toda 2 días de edad, las crías de ratón neonatal por sobredosis de Avertin (7,5 mg / g de peso corporal), y luego congelar el tejido en el isopentano baño de hielo / seco para preservar la estructura de los tejidos. Use un pre-limpioed par de pinzas para colocar y retirar la cría de ratón en criomolde del isopentano baño de hielo / seco. Almacenar el tejido congelado instantáneamente a -80 ° C hasta su análisis.
- El uso de guantes de protección, aplicar una capa de temperatura óptima de corte (OCT) medio de montaje a un disco de criostato de muestras 40 mm. Con cuidado, coloque el ratón entero congelado en el soporte de la muestra recubierta octubre de adherirse ratón para el disco en la orientación deseada para el corte.
Nota: OCT se utiliza únicamente para adherir el tejido a la platina para seccionar. No tejido completamente incrustar en OCT y evitar el exceso como PTU sabe que tienen efectos perjudiciales sobre el análisis de MSI. Otros medios, como la gelatina, se pueden utilizar para la crio-incrustar el tejido antes de cortar que 9. - Coloque el disco (con sección de tejido PTU y) en un soporte de Peltier en criostato y pulse el botón de encendido Peltier. Espere 10 minutos para la muestra para llegar a un equilibrio térmico.
- Coloque el disco de especímenes (con el tejido montado en) del interior de la bandeja de CD, y se enfrentan a los tejidos hasta el plano deseado para su análisis. Cortar el tejido en secciones de espesor deseado a -20 ° C utilizando el micrótomo rotatorio alojado en el interior del criostato 10.
Nota: Para los análisis de todo el cuerpo, cortar el tejido en secciones de 25 micras de espesor para mantener la integridad de los tejidos. Para las regiones más pequeñas (por ejemplo, hígado, cerebro, riñón), cortar el tejido en secciones de 10 micras de espesor.- Utilice la placa de vidrio estabilizadora para prevenir la sección de tejido en rodajas ruede. Utilice la manguera de vacío criostato y cepillos para eliminar los restos de tejido no deseado. Una vez se secciona el tejido, utilice el protector de la hoja para cubrir la cuchilla de microtomo afilado para evitar lesiones personales.
- Sección ratón Orient en la placa de muestra y descongelar de montaje sobre un vidrio portaobjetos de microscopio limpiado previamente por lo que el tobogán lo más cerca posible a la sección de tejido, sin tocarlo 10.
Nota: Para cuantitativa expe MSItos, capa la diapositiva con un patrón interno usando un pulverizador neumático automatizado antes de montar el tejido, y el deshielo de montaje en la sección de tejido en el portaobjetos recubierto 11. - Retire la hoja microtomo utilizado para seccionar el tejido utilizando el eyector hoja, y desechar de forma segura la hoja en el contenedor "de residuos aguda" apropiado.
- Mantenga el portaobjetos de vidrio en el interior del criostato hasta el paso 3,2 para mantener la crioconservación del tejido.
2. IR-MALDESI Preparación / Calibración
- Encienda el láser infrarrojo medio y poner en marcha la aplicación de control de láser en el equipo. Elige la longitud de onda deseada con la aplicación de control de láser suministrado por el fabricante. experimentos IR-MALDESI se realizan típicamente a 2940 nm.
Nota: Los experimentos recientes han investigado la dependencia de las longitudes de onda de láser infrarrojo medio y la matriz de hielo con diferencias observadas que parecen ser analito 8 específico. - Encienda el ciervoControlador e, poner en marcha el programa de control de usuario personalizado (RASTIR), y calibrar la posición de fase usando el botón de inicio.
- Preparar la solución de electrospray. Típicamente, utilizar una mezcla 50:50 (v / v) de metanol / agua con ácido fórmico 0,2% para el disolvente electrospray en modo de ion positivo. Seleccione la composición del disolvente electrospray de acuerdo con el experimento. Por ejemplo, el uso de hidróxido de amonio 5 mM como modificador de electrospray para aplicaciones de imagen en el modo de ion negativo.
Nota: Para cambiar la polaridad de IR-MALDESI MSI, use 1 mM de ácido acético como modificador. Este modificador se encontró óptimo para la obtención de señal estable y reproducible tanto en el modo por positivo y negativo de ion-12. - Llene una jeringa de 1 ml con disolvente electrospray, y enjuague capilar de sílice con solvente nuevo.
- Alinear ESI emisor en el eje de entrada con MS utilizando los parámetros de la fuente tales como la distancia ESI-punto, punto-entrada de la distancia, altura de la muestra, y el caudal de disolvente que se han optimizado el uso de la DO estadística E para el análisis de secciones de tejido 6. Nota: Véase la Figura 1 para un esquema que representa a estos parámetros y valores optimizados para MSI de secciones de tejido.
- Iniciar el electrospray y evaluar su estabilidad (> 10 min) mediante el control de la corriente de iones total (TIC) que, en este punto, consiste únicamente de compuestos ambientales. Típicamente, una variación TIC de <10% durante 10 a 15 min es una buena indicación de la estabilidad.
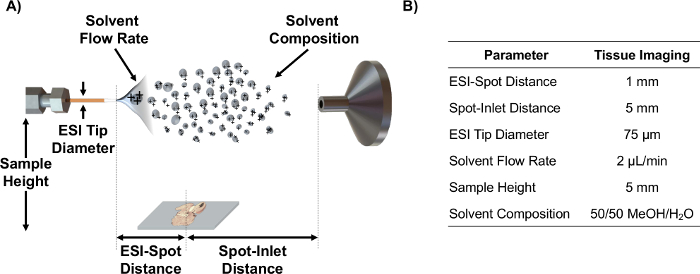
Figura 1. IR-MALDESI esquemática y parámetros. (A) Representación esquemática de la configuración fuente IR-MALDESI (no a escala) y los parámetros ajustables. (B) los valores de parámetros optimizados para obtener imágenes de cortes de tejido. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Precaución: Apague el electrospray antes de colocar la muestra en el escenario.
- Coloque el tejido deshielo montado sobre la placa de muestra de IR-MALDESI dentro de la vista de la cámara.
- Asegúrese de que las manos estén alejadas del emisor ESI y reinicie el electrospray.
- Cerrar la puerta de acceso de la fuente y de purga recinto IR-MALDESI con nitrógeno seco para evitar la condensación de agua en la superficie del tejido. Una vez que la humedad relativa dentro de los confines del recinto <3%, encender la fuente de alimentación de CC (~ 12 V) a la placa Peltier, que se enfríe la placa de muestra. Nota: El proceso de enfriamiento en la etapa de enfriado con Peltier se discute en detalle en otra parte 13.
- Se enfría la etapa a -9 ° C, y permitir que el tejido montado en el equilibrio térmico (~ 5-10 minutos).
- Deja de purga de nitrógeno y exponer el tejido a la humedad relativa del ambiente mediante la apertura de la puerta de acceso de origen. Una fina capa de hielo se deposita sobre la cosección de tejido de superficie ld de la etapa de Peltier y montado debido a la desublimación de agua del aire.
Nota: Si la humedad relativa en el laboratorio está por debajo de 10 a 15%, coloque un vaso de precipitados de agua caliente dentro de la caja para facilitar la formación de la capa de matriz de hielo. - Después de que se formó la capa de matriz de hielo, cierre la puerta de acceso y reiniciar el flujo de nitrógeno seco para reducir la humedad relativa de 10 ± 2%. Ajustar la velocidad de flujo de nitrógeno para mantener la humedad relativa a este nivel, que se encuentra empíricamente ser el equilibrio de la formación de hielo y la sublimación para mantener una capa constante de hielo durante todo el experimento 6.
4. La espectrometría de masa de imágenes Adquisición de Datos
- Uso de la interfaz gráfica de usuario RASTIR (Figura 2), mover la etapa de análisis de la posición haciendo clic en el botón "posición del láser". Utilice el diodo de ajuste del láser para garantizar que se realiza la ablación de una región fuera de los tejidos. Disparar un disparo de láser en una re fuera de tejidoregión para calibrar el desplazamiento con respecto a la vista de la cámara láser. Haga clic en el botón "Prueba de fuego láser" para actualizar la posición del láser.
- Volver a "la posición CAMERA" y actualizar la posición del láser en RASTIR mediante la colocación de la alineación del retículo láser en el punto de ablación por láser (Figura 2-1).
- Haga clic en el botón Región de interés (ROI) y ajustar el tamaño y la posición de retorno de la inversión para la sección de tejido que está siendo analizado (Figura 2-2). Entrada parámetros experimentales apropiados, tales como el tamaño del paso en las direcciones X e Y (en mm) (Figura 2-3), y el nombre del archivo MSI en el cuadro "Nombre del proyecto" (Figura 2-4).
- Al dibujar un retorno de la inversión, incluir una parte de fuera de tejido que sirve como control. Utilice esta sección fuera de tejido para la recolección de pico (Paso 5.4).
Nota: El diámetro de desorción (tamaño del punto) del láser sobre el tejido es de aproximadamente 150 micras; Sin embargo, las resoluciones espaciales más altas se pueden obtener por USIng el método de sobremuestreo 14,15.
- Al dibujar un retorno de la inversión, incluir una parte de fuera de tejido que sirve como control. Utilice esta sección fuera de tejido para la recolección de pico (Paso 5.4).
- Seleccione la frecuencia del láser adecuada en el cuadro desplegable, el número de pulsos láser por voxel (valores enteros), y el retraso entre el disparo de láser y adquisición de señales espectrómetro de masas (Figura 2-5). Típicamente utilizar dos pulsos de láser por voxel a 20 Hz para desorber completamente material en un experimentos IR-MALDESI MSI, con un tiempo de retardo de 10 mseg para permitir que los iones generados para alcanzar el analizador de masas para la medición de 5.
Nota: Elija la tasa de repetición más alto que el láser es capaz de funcionar a. Recientemente se ha mostrado que el uso de más alta tasa de repetición mejorará detectabilidad analito 16.
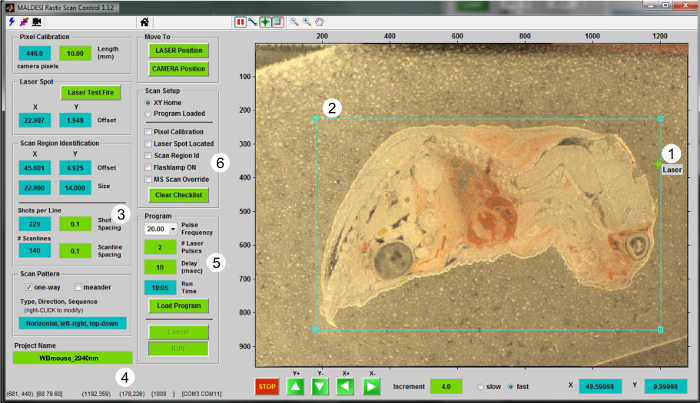
Se presenta la Figura 2. Interfaz de usuario para la operación IR-MALDESI MSI. Captura de pantalla del programa de control de Escaneo RASTIR. Los pasos para performing un experimento de MSI son (1) la localización del punto de láser, (2) la elaboración de un retorno de la inversión, (3) la elección del tamaño del paso de fase (en mm), (4) dar un nombre al archivo, (5) elegir el número correcto de impulsos por voxel, junto con la velocidad deseada repetición, y (6) la verificación de la lista para la imagen y la configuración de la EM. Haga clic aquí para ver una versión más grande de esta figura.
- Elija parámetros del espectrómetro de masas, tales como el modo de ionización, la tensión de electrospray, la tasa de flujo de disolvente, temperatura del capilar, y el tiempo de inyección en el software de espectrómetro de masas. Consulte la Tabla 1 para un ejemplo de los parámetros utilizados en todo el cuerpo IR-MALDESI MSI.
Nota: Debido a la complejidad de las muestras biológicas y la falta de métodos de separación en los análisis de MSI, la fuente IR-MALDESI está acoplado a un alto poder de resolución y alta precisión instrumento de masas. Un método de adquisición se puede cargar para analyses como la vigilancia de reacciones paralelas (PRM) 7 o la polaridad de conmutación 12 MSI. - Una vez que se han elegido todos los parámetros (láser, el escenario, y espectrómetro de masas), coloque la MS en el modo de "apretón de manos" para la sincronización e inicie la adquisición de MS.
- El uso de software de imágenes RASTIR, verificar todos los pasos en la lista de control (Figura 2-6) se completan mediante la comprobación de las cajas, y cargar el programa. Una vez que el programa ha sido cargado, el botón "Ejecutar" estará disponible. Presione Ejecutar para iniciar la adquisición de señales de MSI.
- Al finalizar el experimento de formación de imágenes, detener la adquisición espectrómetro de masas y colocar el instrumento en el modo de espera. Desactive purga de nitrógeno a la caja, apague la fuente de alimentación a la placa Peltier, y apague el controlador de platina y el láser.
- Abra la puerta de acceso, quite la punta emisor electrospray desde el interior del recinto de la fuente, y quitar la entrada de MS metal calentado extendido el uso de guantes de protección. Precaución: El prolongado MS metálica de entrada estará muy caliente.
- El uso de guantes y gafas de seguridad, limpiar la entrada del capilar de metal extendido por el baño de ultrasonidos en ácido nítrico al 15% durante 10 minutos, luego en agua grado HPLC durante 10 minutos, y finalmente en metanol grado HPLC durante 10 minutos. Se seca la entrada del capilar de metal bajo una corriente de nitrógeno, y vuelva a insertar en la EM. Nota: Eliminar el disolvente en los contenedores de residuos adecuados después.
| Parámetro | Valor |
| Modo de ionización | Positivo |
| Tensión por electrospray | |
| Velocidad de flujo de disolvente | 2 l / min |
| Temperatura capilar | 275 ° C |
| Tamaño de escaneado | m / z 250-1.000 |
| Tipo de escaneo | Análisis completo |
| Tiempo de inyección | 110 ms |
| Poder de resolución |
Tabla 1. Parámetros de instrumento utilizado en todo el cuerpo IR-MALDESI MSI.
Análisis 5. Datos
- Convertir los archivos de datos brutos generados por el instrumento de formatos de datos tales como mzXML 17 o 18 imzML utilizando software libre como MSConvert 17 y el convertidor 19 imzML.
- Comience MSiReader v1.0, un software de código abierto desarrollado para el análisis de alto poder de resolución de datos MSI 20, en un equipo de procesamiento dedicado. Cargar el archivo de imagen en MSiReader. Para la interfaz de usuario de MSiReader y algunas de sus características incorporadas, véase la Figura 3.
- Generar mapas de iones de analitos de interés mediante la introducción de su m / z y elija una ventana de m / z adecuada en partes por millón (ppm) o Thomson (Th) (Figura 3-1). Para los instrumentos de alto poder de resolución (RP)elegir una ventana en el rango de bajo ppm.
Nota: La ventana m / z apropiada depende de la RP y la precisión de medición de masa (MMA) del instrumento que la fuente de IR-MALDESI está acoplado a. - Además de interpretar los datos utilizando las funciones integradas, tales como la interpolación (Figura 3-2), la normalización (Figura 3-3), superposición de imagen óptica (Figura 3-4), y el pico de la cosecha (Figura 3-5) 20.
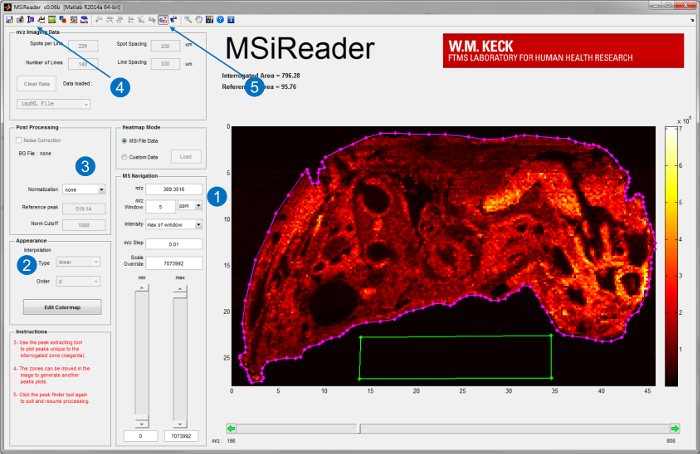
Figura 3. Interfaz de usuario de MSiReader; v1.0 20. Una vez que un archivo se carga en el software, mapas de iones de analitos de interés se muestran por (1) la introducción de la m / z y la tolerancia en ppm o Th. Un análisis más detallado tal como (2) de interpolación o (3) normalización también se puede realizar. Una imagen óptica del tejido también se puede importar y superpone conlos mapas de iones (4) para una mejor visualización. Para no directo analiza el pico de la función (5) picking se puede utilizar para extraer picos específicos de tejido por la elección de la zona de tejido (línea magenta) y una zona de referencia fuera de tejido (caja verde). Haga clic aquí para ver una versión más grande de esta figura.
- Utilice la función de pico recogiendo para generar una lista de valores de m / z específicos de tejido utilizando criterios definidos por el usuario. Nota: El algoritmo utiliza PeakFinder diferencias en el espectro de masa media de las dos regiones. Estos valores m / z continuación, se pueden buscar en bases de datos de metabolitos tales como METLIN 21 o lípido MAPS. 22
- Consulte la Figura 3 para un ejemplo de la elección de una región de tejido interrogado (magenta polígono ROI) y la región de referencia fuera de tejido (polígono verde ROI).
Resultados
Las imágenes presentadas en la Figura 4 muestran la distribución espacial de los metabolitos en diferentes órganos en la sección de tejido de todo el cuerpo. Valores únicos m / z a regiones específicas del cuerpo se encontraron usando MSiReader PeakFinder, seguido por el procesamiento por lotes para la generación de imagen. La herramienta de superposición de imágenes (Figura 3-4) se utiliza para alinear la imagen óptica tomada antes de...
Discusión
El protocolo anterior se describen los pasos principales para la realización de un experimento IR-MALDESI MSI. El proceso de aplicación de la matriz (Sección 3) dura aproximadamente 20 min, que es similar a un proceso de solicitud de matriz típico para experimentos MALDI MSI por sublimación o por pulverización el revestimiento utilizando un pulverizador robótico. Por otra parte, IR-MALDESI no se basa en la partición de los analitos en los cristales de la matriz 6, y la matriz de hielo se puede utiliza...
Divulgaciones
The authors declare no competing financial interests.
Agradecimientos
The authors thank Professor H. Troy Ghashghaei from NCSU Department of Molecular Biomedical Sciences for providing the whole mouse tissue. The authors also gratefully acknowledge the financial assistance received from National Institutes of Health (R01GM087964), the W.M. Keck foundation, and North Carolina State University.
Materiales
| Name | Company | Catalog Number | Comments |
| IR-MALDESI Source | Custom-made | N/A | Please refer to references 4 and 12 for an in-depth discussion of IR-MALDESI source development. |
| Q Exactive Plus | Thermo Scientific | Q Exactive Plus Hybrid Quadrupole-Orbitrap Mass Spectrometer | |
| Water, HPLC Grade | Burdick & Jackson | AH365-4 | |
| Methanol, HPLC Grade | Burdick & Jackson | AH230-4 | |
| Formic Acid | Sigma Aldrich | 56302 | |
| Tunable mid-IR Laser | Opotek Inc. | IR Opolette | Tunable 2,700-3,100 nm IR OPO laser |
| Nitrogen Gas | Arc3 Gases | AG S-NI300-5.0 | Grade 5.0 high purity nitrogen gas cylinder (300) |
| Cryostat | Leica Biosystems | CM 1950 | Cryomicrotome |
| High Profile Microtome Blades | Leica Biosystems | 3802123 | Leica DB80HS |
| Mounting Medium (OCT) | Leica Biosystems | 3801480 | Surgipath FSC 22 mounting medium |
| Cryostat Specimen Disc | Leica Biosystems | 14047740045 | 40 mm diameter |
| Glass Microscope Slides | VWR | 48312-003 | Frosted, selected, pre-cleaned |
Referencias
- Mcdonnell, L. A., Heeren, R. M. A. Imaging Mass Spectrometry. Mass Spectrom. Rev. 26, 606-643 (2007).
- Chughtai, K., Heeren, R. M. A. Mass spectrometric imaging for biomedical tissue analysis. Chem. Rev. 110 (5), 3237-3277 (2010).
- Robichaud, G., Barry, J. A., Muddiman, D. C. Atmospheric Pressure Mass Spectrometry Imaging. Encycl. Anal. Chem. , (2014).
- Sampson, J. S., Hawkridge, A. M., Muddiman, D. C. Generation and detection of multiply-charged peptides and proteins by matrix-assisted laser desorption electrospray ionization (MALDESI) Fourier transform ion cyclotron resonance mass spectrometry. J. Am. Soc. Mass Spectrom. 17 (12), 1712-1716 (2006).
- Robichaud, G., Barry, J. A., Garrard, K. P., Muddiman, D. C. Infrared matrix-assisted laser desorption electrospray ionization (IR-MALDESI) imaging source coupled to a FT-ICR mass spectrometer. J. Am. Soc. Mass Spectrom. 24 (1), 92-100 (2013).
- Robichaud, G., Barry, J. A., Muddiman, D. C. IR-MALDESI Mass Spectrometry Imaging of Biological Tissue Sections Using Ice as a Matrix. J. Am. Soc. Mass Spectrom. 25 (3), 319-328 (2014).
- Barry, J. A., et al. Mapping Antiretroviral Drugs in Tissue by IR-MALDESI MSI Coupled to the Q Exactive and Comparison with LC-MS/MS SRM Assay. J. Am. Soc. Mass Spectrom. 25 (12), 2038-2047 (2014).
- Rosen, E. P., Bokhart, M. T., Ghashghaei, H. T., Muddiman, D. C. Influence of Desorption Conditions on Analyte Sensitivity and Internal Energy in Discrete Tissue or Whole Body Imaging by IR-MALDESI. J. Am. Soc. Mass Spectrom. 26, 899-910 (2015).
- Nelson, K. A., Daniels, G. J., Fournie, J. W., Hemmer, M. J. Optimization of whole-body zebrafish sectioning methods for mass spectrometry imaging. J. Biomol. Tech. 24 (3), 119-127 (2013).
- Park, J. J., Cunningham, M. G. Thin sectioning of slice preparations for immunohistochemistry. J. Vis. Exp. (3), e194 (2007).
- Bokhart, M. T., Rosen, E., Thompson, C., Sykes, C., Kashuba, A. D. M., Muddiman, D. C. Quantitative mass spectrometry imaging of emtricitabine in cervical tissue model using infrared matrix-assisted laser desorption electrospray ionization. Anal. Bioanal. Chem. 407 (8), 2073-2084 (2015).
- Nazari, M., Muddiman, D. C. Polarity Switching Mass Spectrometry Imaging of Healthy and Cancerous Hen Ovarian Tissue Sections by Infrared Matrix-Assisted Laser Desorption Electrospray Ionization (IR-MALDESI). Analyst. 141, 595-605 (2016).
- Hsu, C. C., et al. Design and Application of a Low-Temperature Peltier-Cooling Microscope. J. Pharm. Sci. 85 (1), 70-74 (1996).
- Jurchen, J. C., Rubakhin, S. S., Sweedler, J. V. MALDI-MS imaging of features smaller than the size of the laser beam. J. Am. Soc.Mass Spectrom. 16 (10), 1654-1659 (2005).
- Nazari, M., Muddiman, D. C. Cellular-level mass spectrometry imaging using infrared matrix-assisted laser desorption electrospray ionization (IR-MALDESI) by oversampling. Anal. Bioanal. Chem. 407 (8), 2265-2271 (2015).
- Rosen, E. P., Bokhart, M. T., Nazari, M., Muddiman, D. C. Influence of C-Trap Ion Accumulation Time on the Detectability of Analytes in IR-MALDESI MSI. Anal. Chem. 87, 10483-10490 (2015).
- Kessner, D., Chambers, M., Burke, R., Agus, D., Mallick, P. ProteoWizard: open source software for rapid proteomics tools development. Bioinformatics. 24 (21), 2534-2536 (2008).
- Schramm, T., et al. ImzML - A common data format for the flexible exchange and processing of mass spectrometry imaging data. J. Proteomics. 75 (16), 5106-5110 (2012).
- Race, A. M., Styles, I. B., Bunch, J. Inclusive sharing of mass spectrometry imaging data requires a converter for all. J. Proteomics. 75 (16), 5111-5112 (2012).
- Robichaud, G., Garrard, K. P., Barry, J. A., Muddiman, D. C. MSiReader: an open-source interface to view and analyze high resolving power MS imaging files on Matlab platform. J. Am. Soc. Mass Spectrom. 24 (5), 718-721 (2013).
- Smith, C. A., O'Maille, G., et al. METLIN: a metabolite mass spectral database. Ther. Drug. Monit. 27 (6), 747-751 (2005).
- Sud, M., et al. LMSD: LIPID MAPS structure database. Nucleic Acids Res. 35, D527-D532 (2007).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. J. Mass Spectrom. 38 (7), 699-708 (2003).
- Takai, N., Tanaka, Y., Inazawa, K., Saji, H. Quantitative analysis of pharmaceutical drug distribution in multiple organs by imaging mass spectrometry. Rapid Commun. Mass Spectrom. 26 (13), 1549-1556 (2012).
- Liu, J., Gingras, J., Ganley, K. P., Vismeh, R., Teffera, Y., Zhao, Z. Whole-body tissue distribution study of drugs in neonate mice using desorption electrospray ionization mass spectrometry imaging. Rapid Commun. Mass Spectrom. 28 (2), 185-190 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados