Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Verificación rápida de Terminators Uso de la PGR-Blue plásmido y montaje de puerta de oro
En este artículo
Resumen
This protocol utilizes Golden Gate Assembly and the plasmid pGR-blue to rapidly quantify the strength of terminators found in silico.
Resumen
The goal of this protocol is to allow for the rapid verification of bioinformatically identified terminators. Further, the plasmid (pGR-Blue) is designed specifically for this protocol and allows for the quantification of terminator efficiency. As a proof of concept, six terminators were bioinformatically identified in the mycobacteriophage Bernal13. Once identified, terminators were then made as oligonucleotides with the appropriate sticky ends and annealed together. Using Golden Gate Assembly (GGA), terminators were then cloned into pGR-Blue. Under visible light, false positive colonies appear blue and positively transformed colonies are white/yellow. After induction of an arabinose inducible promoter (pBad) with arabinose, colony strength can be determined by measuring the ratio of green fluorescent protein (GFP) produced to red fluorescent protein (RFP) produced. With pGR-Blue, the protocol can be completed in as little as three days and is ideal in an educational setting. Additionally, results show that this protocol is useful as a means for understanding in silico predictions of terminator efficiency related to the regulation of transcription.
Introducción
Large synthetic biology projects necessitate the use of highly effective transcription terminators to help regulate gene expression. Identification of novel terminators requires bioinformatic analysis of novel genomes. However, as increasing amounts of bioinformatic software are developed, each with a unique algorithm utilized for prediction, more discrepancy between putative results occurs. Because this process is somewhat subjective and is done in silico, these predictions need biological confirmation.1 Additionally, the volume of putative terminators identified through in-silico analysis requires the use of cloning strategies that can be completed in a relatively short time frame.
The PGR-Blue plasmid is a modification of the PGR plasmid that has been redesigned to use Golden Gate Assembly (GGA) to simplify the cloning procedure by allowing for all reaction steps to be simultaneously performed in one micro-centrifuge tube.2,3 Color selection was incorporated into the plasmid to increase the ease of identifying positive colonies. A successful ligation should be white/yellow in visible light and fluoresce green under blue (450 nm) or ultraviolet (UV) light when grown on plates containing arabinose. Because uncut pGR-blue contains a blue chromo protein (amilCP), colonies containing an unmodified plasmid are blue under visible light. This simplification along with the streamlined protocol allows researchers to proceed from bioinformatic identification to biological confirmation in three to four days. The design nature of this system can be beneficial both in the research lab and in educational settings.
The pGR-Blue plasmid allows for quantification of terminator strength.4 A single arabinose inducible promoter is used to produce green fluorescent protein (GFP) and red fluorescent protein (RFP). The terminator is cloned into the plasmid after the GFP sequence but before the RFP sequence, thus stopping the transcription of the RFP protein. The strength of the terminator is determined by the ratio of GFP produced to RFP produced.
The Vision and Change5 report suggested that Science, Technology, Engineering and Math (STEM) education incorporate research based experiences into the classroom.6 However, this requires the development of protocols that can be done by students with limited skill sets in a defined time frame. While the protocol can be accomplished in as little as three days, it was also designed so that each major step could be accomplished in a separate weekly (2-3 hr) lab period to create a Course Research Experience (CRE). When used in this manner, the procedure will take between three and six weeks and is appropriate for both introductory and advanced courses in Genetics, Cell Biology or Bioinformatics.
Protocolo
1. Diseñar y oligonucleótidos pedidos con las apropiadas pegajosas Final
- Identificar posibles terminadores rho-independientes a través del análisis genómico utilizando programas que están disponibles gratuitamente en línea. 7
- Cuando se trabaja con ADN de doble cadena, determinar la orientación del terminador a ensayar. 7 El plásmido PGR-azul sólo verifica terminadores ligados en el 5 'a 3' en la parte superior (capítulo adelante).
- Convertir una cadena de terminador inferior (hacia atrás) con su complemento invertido para reorientar la secuencia para realizar pruebas en PGR-Azul uso de software libre en línea, por ejemplo, mono.
- Después de la orientación se determina que es correcta, añadir el extremo cohesivo "5'-CGAC-3 '" al extremo 5' de la cadena superior. Añadir el extremo pegajoso "5'-CCGC-3 '" al extremo 5' de la cadena inferior. 8 Esto asegurará que inserto se ligó se insertará en la apropiadaorientación usando GGA.
- Una vez que se determinan las secuencias, la orden como oligonucleótidos de cadena simple vista comercial.
2. El recocido oligonucleótidos (sólo se aplica a liofilizada ADN)
- Vuelva a suspender los oligonucleótidos individuales en agua libre de nucleasa a una concentración de 100 mM.
- Hacer 10x tampón de hibridación: 1 M NaCl y Tris-HCl pH 7,4 100. tampón de reasociación se puede almacenar a 4 ° C durante varias semanas. Una vez que se hace tampón de reasociación y los oligonucleótidos se resuspendió, comenzar el recocido 8.
- Para cada terminador a ensayar, añadir 16 l de agua ultra pura a un tubo de microcentrífuga de 1,5 ml.
- Añadir 2 l de 10x tampón de reasociación a cada tubo de microcentrífuga.
- Añadir 1 l de oligonucleótido cadena superior (100 m) desde el terminador deseada.
- Añadir 1 l de oligonucleótido cadena inferior (100 mM) de el terminador deseado.
- Crear un punto de ebullición de agua baTH utilizando un vaso de precipitados de 1000 ml que contenía aproximadamente 400 ml de agua del grifo y colocar en un plato caliente.
- Coloque los tubos de microcentrífuga de la etapa 2.6 en un flotador y hervir durante 4 minutos. Después de 4 minutos, apagar la placa de calor, pero dejar el tubo y flotar en el baño de agua fría para O / N (18 h) 8 lentamente. Almacenar los terminadores recocidas a -20 ° C.
3. Montaje de Golden Gate (Standard Mix - Costo Eficiente)
- Hacer una dilución 40 nM de los terminadores recocidos. Añadir 124 l de agua libre de nucleasa a 1 l de recocido mezcla terminadores (100 M).
- Centrifugar las muestras durante 30 segundos a 10.000 xg y almacenar en hielo.
- Siendo utilizado en un nuevo tubo apropiado para el termociclador, añadir 6 l de agua libre de nucleasa.
- Añadir 1 l de ADN comercial Ligase Buffer [300 mM Tris-HCl (pH 7.7 hasta 7.8), 100 mM MgCl 2 mM, DTT 100 mM, y 10 ATP]. Realizar todos los pasos subsiguientes en el hielo.
- Añadir1 l de PGR-azul (35-50 ng / l) plásmido destino al tubo de microcentrífuga.
- Añadir 1 l de los 40 nm terminadores recocidos al tubo de microcentrífuga.
- Añadir 0,5 l de endonucleasa de restricción comercial de alta fidelidad BsaI al tubo de microcentrífuga.
- Añadir 0,5 l de ADN ligasa comercial al tubo de microcentrífuga.
- Centrifugar la mezcla de reacción a 10 000 xg durante 120 seg. A continuación, coloque el tubo directamente en el termociclador.
- Ejecutar el programa del termociclador: 20 ciclos de 1 min a 37 ° C, seguido de 1 min a 16 ° C, seguido de 1 ciclo de 15 minutos a 37 ° C. Use muestras inmediatamente o congelación y la conservación a -20 ° C. 8
4. Asamblea Golden Gate (Master Mix Comercial - Tiempo Eficiente)
- Añadir 124 l de agua libre de nucleasa a 1 l de los 100 M terminadores creado en el paso 2. Esto diluye la concentración de ADN terminador de 40 nM recocidos.Realice este paso en una nueva, marcada 1,5 ml tubo de microcentrífuga.
- En un nuevo tubo apropiado para el termociclador, añadir 14 l de agua libre de nucleasa. Asegúrese de que el nuevo tubo está etiquetado en consecuencia.
- Añadir 2 l de Tampón Comercial Master Mix al tubo de microcentrífuga.
- Añadir 1 l de PGR-azul plásmido (35-50 ng / l) al tubo de microcentrífuga.
- Añadir 2 l de los 40 nm terminadores recocidos al tubo de microcentrífuga.
- Añadir 1 l de Master Mix Comercial al tubo de microcentrífuga.
- Centrifugar el tubo de 40 nM a 10.000 xg durante 120 seg. A continuación, coloque el tubo directamente en el termociclador.
- Ejecutar el programa del termociclador: 1 ciclo de 60 minutos a 37 ° C seguido de 1 ciclo de 15 min a 55 ° C. Use muestras inmediatamente o congelación y la conservación a -20 ° C.
5. Transformación y selección de colonias
- Preparar la base de Luria (LB) placas de Petri que contenían agar concentrat 1 mMion de ampicilina (Amp) y una concentración 10 mM de arabinosa (ARB). Preparar al menos 10 ml de 1 M L solución de reserva -arabinose porque esta solución madre se usará en pasos posteriores.
- Utilizar cualquier de alta eficiencia (10 8 - 10 9 transformantes / mg) E. químicamente competentes coli (JM109) las células para la transformación. 9 Para obtener los mejores resultados, agregue 3-5 l de la reacción de ligación a 50 l de células competentes. Al utilizar células competentes químicos, siga el protocolo de transformación específica a las células que se utilizan.
- Extienda la mitad (25 l) de las células transformadas en LB precalentado (Amp / ARB) placas de agar utilizando un "palo de hockey" en forma de L estéril y se incuba O / N a 37 ° C. Debido a la rápida velocidad de descomposición de la ampicilina, no se incuba a 37 ° C durante 24 hr.
- Realizar selección de colonias basada en el color mediante inspección visual. Una ligadura exitosa debe ser de color blanco / amarillo a la luz visible y fluorescencia verde under azul (450 nm) o la luz UV. Una ligadura sin éxito producirá una colonia que es de color azul bajo luz visible después de 18 - 20 h.
6. Verificación y cuantificación de Terminator
- Preparar tubos de 5 ml de caldo de LB-ampicilina estériles con 1 mM y 10 mM arabinosa. El número de tubos necesarios depende de la cantidad de terminadores que se están probando más controles (células con resistencia a la ampicilina que no emiten fluorescencia y las células que contienen el plásmido PGR-azul sin cortar).
- Bajo la luz visible recoger 2-3 colonias de color blanco / amarillo (paso 5.4) utilizando un asa estéril. Permitir que las muestras de incubar en caldos en un agitador a 37 ° C y 160 rpm durante 16 a 18 hr. No incubar las muestras durante más de 24 horas.
- Opcionalmente, utilizar un espectrofotómetro para determinar la densidad celular óptica a 600 nm (OD 600) para asegurar el crecimiento celular adecuado. En general, una DO 600 de ~ 0,8-1,0 se observa después de 16-18 h de crecimiento.
- Utilizaruna placa de 96 pocillos estéril para los pasos restantes. Llevar a cabo estos pasos en un ambiente aséptico y por triplicado. Añadir 199 l de caldo estéril LB + Amp / arabinosa (ampicilina 1 mM / arabinosa 10 mM) a cada pocillo. Incluir piezas en la placa por tres controles separados.
- Utilice los tres controles siguientes: solamente un pozo (1) que contiene el caldo / arabinosa LB + Amp y un pozo (2) para células competentes transformadas (en blanco) para servir como un espacio en blanco. Para la cuantificación (3) de la resistencia de terminación, las células que contienen pGR original, sin cortar o contiene PGR-azul se ligó con una secuencia no de terminación deben ser incluidos (control de referencia para cuantificación). Véase la Figura 1 para el diseño lector de placas.
- Pipetear 1 l de la solución de células de la O / N caldos en pocillos individuales de la placa de 96 pocillos.
- placas de sellado con una cubierta transpirable y agitar durante 18-20 horas a 37 ° C y 160 rpm.
- El uso de un lector de placas, determinar OD 600 mediante la lecturala absorbancia a 600 nm. Medir la fluorescencia de GFP de la siguiente manera: 395 de excitación y emisión de fluorescencia de RFP 509. Medida de la siguiente manera: 575 de excitación y emisión 605. Después de 18-20 horas, el OD 600 debe ser de aproximadamente 0,5.
- Para normalizar la variación en el crecimiento, utilizar la ecuación:
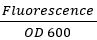 tanto para GFP y RFP.
tanto para GFP y RFP. - Después de la normalización, determinar la resistencia de terminación relativa usando la ecuación
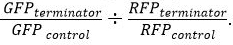
Resultados
Este protocolo va a producir células que contienen PGR-azul con un terminador ligaron entre GFP y RFP usando oro conjunto de corredera (Figura 2). Las colonias positivas que contienen inserto se ligó se pueden seleccionar sobre la base de color. En luz visible colonias positivas serán de color blanco / amarillo y falsos positivos producirán colonias azules después de 18-20 horas de incubación a 37 ° C (Figura 3).
Discusión
El paso más importante en este protocolo es adecuado diseño de oligonucleótidos antes de ordenar. Los oligonucleótidos deben tener los extremos cohesivos apropiados añadidos a los extremos 5 'de la parte superior e hilos de fondo para asegurar que GGA incorporación es posible. Además, es importante para cambiar la orientación de los interruptores de frente a la izquierda (Terminator que impiden la transcripción de la cadena inferior) a la de la derecha frente (termina la transcripción en la cadena superior...
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Los autores desean reconocer Malcolm Campbell y Todd Eckdahl con el Consorcio del Genoma de la enseñanza activa (GCAT) y la Alianza de Educación HHMI-Ciencia - Programa de cazadores de fagos que subieron Genómica Evolutiva y Ciencia (SEA-FAGOS).
Este proyecto fue apoyado por becas de la National Center for Research Resources (P20RR016460) y el Instituto Nacional de Ciencias Médicas Generales (P20GM103429) de los Institutos Nacionales de Salud. Esta investigación fue financiada en parte por la National Science Foundation bajo la subvención # IIA-1457888. (Ouachita Baptist University) los fondos institucionales, además, se proporcionan a través de la JD Patterson verano beca de investigación.
Materiales
| Name | Company | Catalog Number | Comments |
| pGR-Blue Plasmid | Addgene | 68374 | |
| pGR-Plasmid | Addgene | 46002 | |
| AeraSeal-(Sterile Sheets) | Excel Scientific | BS-25 | Sterile Sheets only |
| 10X T4 DNA ligase Buffer | NEB | ||
| BsaI-HF | NEB | R3535S | The non-HF enzyme will work but is less heat stable. |
| NEB Golden Gate Assembly Mix | NEB | E1600S | Commerial Master Mix refered to in the protocol. |
| T4 DNA ligase | NEB | M0202S | |
| Round Microcentrifuge Floating Rack | Nova Tech International | F18875-6401 | |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | |
| L-(+)-Arabinose | Sigma Aldrich | A-3256 | D-Arabinose will not induce the pBAD promoter |
| Luria Base (LB) - Broth, Miller | Sigma Aldrich | L1900 | |
| Luria Base (LB) - Agar , Miller | Sigma Aldrich | L2025 | |
| Tecan-Infinite M200 Plate Reader | Tecan | ||
| Mix & Go Competent Cells - Strain JM109 | Zymo Research | T3005 | Use company recommended transformation protocol |
| ApE: A plasmid editor-software | http://biologylabs.utah.edu/jorgensen/wayned/ape/ | ||
| Tris-HCl, Molecular Grade | Promega | H5121 | |
| Sodium Chloride (Crystalline/Biological, Certified) | Fisher Chemical | S671 | |
| Comercial Oligonucleotide synthesis | Integrated DNA Technologies (IDT) | http://www.idtdna.com/site | |
| Microtest Tissue Culture Plates- 96 well (Sterile) | Falcon | 35-3072 | |
| mycobacteriophage "Bernal13" | Genebank | KJ510413 | |
| Nuclease Free Water | Integrated DNA Technologies (IDT) | IDT004 | |
| Sterile, L-shaped Hockey-Stick Cell | Life Science Products | 6444-S1 | |
| Nano-Drop 2000c UV-Vis Spectrometer | Thermo Scientific | 2000c | |
| ARNold: a web tool for the prediction of Rho-independent transcription terminators. | http://rna.igmors.u-psud.fr/ |
Referencias
- Li, J., Zhang, Y. Relationship between promoter sequence and its strength in gene expression. Eur Phys J E Soft Matter. 37 (9), (2014).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Lampropoulos, A., et al. GreenGate---a novel, versatile, and efficient cloning system for plant transgenesis. PLoS One. 8 (12), e83043 (2013).
- Chen, Y. J., et al. Characterization of 582 natural and synthetic terminators and quantification of their design constraints. Nat Methods. 10 (7), 659-664 (2013).
- Woodin, T., Carter, V. C., Fletcher, L. Vision and change in biology undergraduate education, a call for action--initial responses. CBE Life Sci Educ. 9 (2), 71-73 (2010).
- Vasaly, H. L., Feser, J., Lettrich, M. D., Correa, K., Denniston, K. J. Vision and change in the biology community: snapshots of change. CBE Life Sci Educ. 13 (1), 16-20 (2014).
- Naville, M., Ghuillot-Gaudeffroy, A., Marchais, A., Gautheret, D. ARNold: a web tool for the prediction of Rho-independent transcription terminators. RNA Biol. 8 (1), 11-13 (2011).
- Campbell, A. M., et al. pClone: Synthetic Biology Tool Makes Promoter Research Accessible to Beginning Biology Students. CBE Life Sci Educ. 13 (2), 285-296 (2014).
- Rhee, J. I., et al. Influence of the medium composition and plasmid combination on the growth of recombinant Escherichia coli JM109 and on the production of the fusion protein EcoRI::SPA. J Biotechnol. 55 (2), 69-83 (1997).
- Dirla, S., Chien, J. Y., Schleif, R. Constitutive mutations in the Escherichia coli AraC protein. J Bacteriol. 191 (8), 2668-2674 (2009).
- Schleif, R. Regulation of the L-arabinose operon of Escherichia coli. Trends Genet. 16 (12), 559-565 (2000).
- Kosuri, S., et al. Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proc Natl Acad Sci U S A. 110 (34), 14024-14029 (2013).
- Sharon, E., et al. Inferring gene regulatory logic from high-throughput measurements of thousands of systematically designed promoters. Nat Biotechnol. 30 (6), 521-530 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados