È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rapida verifica di Terminator Uso del PGR-blu plasmidi e Golden Gate Assembly
In questo articolo
Riepilogo
This protocol utilizes Golden Gate Assembly and the plasmid pGR-blue to rapidly quantify the strength of terminators found in silico.
Abstract
The goal of this protocol is to allow for the rapid verification of bioinformatically identified terminators. Further, the plasmid (pGR-Blue) is designed specifically for this protocol and allows for the quantification of terminator efficiency. As a proof of concept, six terminators were bioinformatically identified in the mycobacteriophage Bernal13. Once identified, terminators were then made as oligonucleotides with the appropriate sticky ends and annealed together. Using Golden Gate Assembly (GGA), terminators were then cloned into pGR-Blue. Under visible light, false positive colonies appear blue and positively transformed colonies are white/yellow. After induction of an arabinose inducible promoter (pBad) with arabinose, colony strength can be determined by measuring the ratio of green fluorescent protein (GFP) produced to red fluorescent protein (RFP) produced. With pGR-Blue, the protocol can be completed in as little as three days and is ideal in an educational setting. Additionally, results show that this protocol is useful as a means for understanding in silico predictions of terminator efficiency related to the regulation of transcription.
Introduzione
Large synthetic biology projects necessitate the use of highly effective transcription terminators to help regulate gene expression. Identification of novel terminators requires bioinformatic analysis of novel genomes. However, as increasing amounts of bioinformatic software are developed, each with a unique algorithm utilized for prediction, more discrepancy between putative results occurs. Because this process is somewhat subjective and is done in silico, these predictions need biological confirmation.1 Additionally, the volume of putative terminators identified through in-silico analysis requires the use of cloning strategies that can be completed in a relatively short time frame.
The PGR-Blue plasmid is a modification of the PGR plasmid that has been redesigned to use Golden Gate Assembly (GGA) to simplify the cloning procedure by allowing for all reaction steps to be simultaneously performed in one micro-centrifuge tube.2,3 Color selection was incorporated into the plasmid to increase the ease of identifying positive colonies. A successful ligation should be white/yellow in visible light and fluoresce green under blue (450 nm) or ultraviolet (UV) light when grown on plates containing arabinose. Because uncut pGR-blue contains a blue chromo protein (amilCP), colonies containing an unmodified plasmid are blue under visible light. This simplification along with the streamlined protocol allows researchers to proceed from bioinformatic identification to biological confirmation in three to four days. The design nature of this system can be beneficial both in the research lab and in educational settings.
The pGR-Blue plasmid allows for quantification of terminator strength.4 A single arabinose inducible promoter is used to produce green fluorescent protein (GFP) and red fluorescent protein (RFP). The terminator is cloned into the plasmid after the GFP sequence but before the RFP sequence, thus stopping the transcription of the RFP protein. The strength of the terminator is determined by the ratio of GFP produced to RFP produced.
The Vision and Change5 report suggested that Science, Technology, Engineering and Math (STEM) education incorporate research based experiences into the classroom.6 However, this requires the development of protocols that can be done by students with limited skill sets in a defined time frame. While the protocol can be accomplished in as little as three days, it was also designed so that each major step could be accomplished in a separate weekly (2-3 hr) lab period to create a Course Research Experience (CRE). When used in this manner, the procedure will take between three and six weeks and is appropriate for both introductory and advanced courses in Genetics, Cell Biology or Bioinformatics.
Protocollo
1. Progettazione e ordinare oligonucleotidi con le appropriate Sticky Ends
- Identificare i potenziali terminatori Rho-indipendente attraverso l'analisi genomica utilizzando programmi che sono liberamente disponibili online. 7
- Quando si lavora con DNA a doppio filamento, determinare l'orientamento del terminatore da testare. 7 Il plasmide PGR-Blue verifica solo terminatori legatura in 5 'a 3' sulla parte superiore (filamento in avanti).
- Convertire un fondo (reverse) Strand terminatore al suo complemento invertita per riorientare la sequenza di test in PGR-blu utilizzando il software gratuito on-line, ad esempio, scimmia.
- Dopo l'orientamento è determinato per essere corretta, aggiungere alla fine appiccicoso "5'-CGAC-3 '" al 5' estremità del filo superiore. Aggiungere fine appiccicoso "5'-CCGC-3 '" al 5' estremità del filo inferiore. 8 Questo assicurerà che inserto legatura sarà inserito nell'appositoorientamento utilizzando GGA.
- Una volta che le sequenze sono determinati, ordinare come singoli oligonucleotidi bloccati commercialmente.
2. ricottura oligonucleotidi (si applica solo a liofilizzato DNA)
- Risospendere singoli oligonucleotidi in acqua priva di nucleasi ad una concentrazione di 100 pM.
- Fare 10x tampone ricottura: 1 M NaCl e 100 mM Tris-HCl pH 7,4. buffer di ricottura può essere conservato a 4 ° C per diverse settimane. Una volta buffer di ricottura è fatto e oligonucleotidi sono ri-sospesi, iniziare la ricottura 8.
- Per ogni terminazione da testare, aggiungere 16 ml di acqua ultra-pura in una provetta da 1,5 ml microcentrifuga.
- Aggiungere 2 ml di 10x tampone di ricottura ad ogni provetta.
- Aggiungere 1 ml di oligonucleotide filo superiore (100 micron) dal terminatore desiderato.
- Aggiungere 1 ml di oligonucleotide fondo filamento (100 micron) dal terminatore desiderato.
- Creare un bollente ba acquaesimo con un bicchiere di 1.000 ml contenente circa 400 ml di acqua di rubinetto e posto su un piatto caldo.
- Mettere le provette da microcentrifuga dal punto 2.6 in un galleggiante e far bollire per 4 minuti. Dopo 4 minuti, spegnere il calore a piastre, ma lasciare il tubo e galleggiare nel bagno d'acqua per raffreddare lentamente O / N (18 ore) 8. Conservare le terminazioni ricotto a -20 ° C.
3. Golden Gate Assembly (Mix Standard - Costo efficiente)
- Fare un nM diluizione dei terminatori ricotto 40. Aggiungere 124 ml di acqua priva di nucleasi di 1 ml di Terminator mix ricotto (100 micron).
- campioni di centrifugazione per 30 secondi a 10.000 xg e memorizzare sul ghiaccio.
- In un nuovo tubo appropriata per il termociclatore in uso, aggiungere 6 ml di acqua priva di nucleasi.
- Aggiungere 1 ml di commerciale DNA Ligase Buffer [300 mM Tris-HCl (pH 7,7-7,8), 100 mM MgCl 2, 100 mM DTT, e 10 mm ATP]. Eseguire tutti i passaggi successivi su ghiaccio.
- Aggiungere1 ml di PGR-blu (35-50 ng / ml) plasmide destinazione alla provetta.
- Aggiungere 1 ml di i 40 nm terminatori ricotto alla provetta.
- Aggiungere 0,5 microlitri commerciale ad alta fedeltà BsaI endonucleasi di restrizione alla provetta.
- Aggiungere 0,5 microlitri di DNA ligasi commerciale alla provetta.
- Centrifugare la miscela di reazione a 10.000 xg per 120 sec. Quindi inserire il tubo direttamente nel termociclatore.
- Eseguire il programma termociclatore: 20 cicli di 1 minuto a 37 ° C seguita da 1 min a 16 ° C, seguiti da 1 ciclo di 15 min a 37 ° C. Utilizzare i campioni immediatamente o congelare e conservare a -20 ° C. 8
4. Golden Gate Assembly (Commercial Master Mix - tempo efficiente)
- Aggiungere 124 ml di acqua priva di nucleasi a 1 l di 100 micron terminazioni creato nel passaggio 2. Questo diluisce la concentrazione di DNA di terminazione a 40 nm ricottura.Eseguire questo passaggio in una nuova, etichettato 1,5 ml provetta.
- In un nuovo tubo appropriata per il termociclatore, aggiungere 14 ml di acqua priva di nucleasi. Assicurarsi che il nuovo tubo è etichettato di conseguenza.
- Aggiungere 2 ml di Commercial Master Mix Buffer alla provetta.
- Aggiungere 1 ml di PGR-Blue plasmide (35-50 ng / mL) alla provetta.
- Aggiungere 2 ml di i 40 nm terminatori ricotto alla provetta.
- Aggiungere 1 ml di Commercial Master Mix alla provetta.
- Centrifugare il tubo di 40 Nm a 10.000 xg per 120 sec. Quindi inserire il tubo direttamente nel termociclatore.
- Eseguire il programma termociclatore: 1 ciclo di 60 min a 37 ° C seguita da 1 ciclo di 15 min a 55 ° C. Utilizzare i campioni immediatamente o congelare e conservare a -20 ° C.
5. Trasformazione e selezione Colony
- Preparare Luria Base (LB) piastre di agar Petri contenenti 1 mM concentratione di ampicillina (Amp) e una concentrazione di 10 mM di arabinosio (ARB). Preparare almeno 10 ml di 1 M L -arabinose soluzione madre perché questa soluzione madre verrà utilizzato nei passaggi successivi.
- Utilizzare qualsiasi alta efficienza (10 8 - 10 9 trasformanti / mg) E. chimicamente competente coli (JM109), le cellule per la trasformazione. 9 Per ottenere i migliori risultati, aggiungere 3-5 ml della reazione legatura a 50 ml di cellule competenti. Quando si usano le cellule chimici competenti, seguire il protocollo di trasformazione specifico per le cellule utilizzate.
- Stendere la metà (25 ml) delle cellule trasformate sul pre-riscaldato LB (Amp / ARB) piastre di agar utilizzando una sterile forma di L "bastone da hockey" e incubare O / N a 37 ° C. A causa del rapido tasso ampicillina di decomposizione, non incubare a 37 ° C superiore a 24 ore.
- Eseguire la selezione delle colonie in base al colore mediante ispezione visiva. Una legatura di successo deve essere di colore bianco / giallo in luce visibile e fluorescenza verde undeR blu (450 nm) o la luce UV. Una legatura successo produrrà una colonia che è di colore blu sotto la luce visibile dopo 18 - 20 ore.
6. Verifica e quantificazione dei Terminator
- Preparare 5 ml sterili tubi LB-brodo con 1 mM ampicillina e 10 mm arabinosio. Il numero di tubi necessari dipende dal numero di terminatori essere testati più controlli (cellule con resistenza all'ampicillina che non fluorescenti e cellule contenenti l'uncut plasmide PGR-blu).
- Sotto la luce visibile raccogliere 2-3 colonie bianche / gialle (passo 5,4) utilizzando un'ansa sterile. Permettere ai campioni di incubare in brodi su un agitatore a 37 ° C e 160 rpm per 16-18 ore. Non incubare questi campioni per più di 24 ore.
- Facoltativamente, utilizzare uno spettrofotometro per determinare la densità cellulare ottica a 600 nm (OD 600) per garantire un'adeguata crescita cellulare. In generale, un diametro esterno 600 di ~ 0,8-1,0 si osserva dopo 16-18 ore di crescita.
- Usouna piastra a 96 pozzetti sterili per i passaggi rimanenti. Eseguire questi passaggi in un ambiente asettico e in triplicato. Aggiungere 199 ml di brodo sterile LB + Amp / arabinosio (1 mM ampicillina / 10 mM arabinosio) in ciascun pozzetto. Includere camera sul piatto per tre comandi separati.
- Utilizzare i seguenti tre controlli: solo un bene (1) contenente brodo / arabinosio LB + Amp e un pozzo (2) per le cellule competenti non trasformate (vuote) per servire come un vuoto. Per la quantificazione (3) della forza terminatore, cellule contenenti non tagliati PGR originale o contenenti PGR-blu legatura con un non-terminazione sequenza dovrebbero essere incluse (controllo di riferimento per la quantificazione). Vedere la Figura 1 per il layout lettore di piastre.
- Pipettare 1 ml di soluzione di cella dal O / N brodi in singoli pozzetti della piastra a 96 pozzetti.
- piastre di tenuta con una copertura traspirante e agitare per 18-20 ore a 37 ° C e 160 giri al minuto.
- Utilizzando un lettore di piastre, determinare OD 600 con la letturaassorbanza a 600 nm. Misurare la fluorescenza GFP come segue: eccitazione 395 ed emissione 509. Misura RFP fluorescenza come segue: eccitazione 575 ed emissione 605. Dopo 18-20 ore, l'OD 600 deve essere di circa 0,5.
- Per normalizzare le variazioni nella crescita, utilizzare l'equazione:
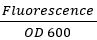 sia per la GFP e RFP.
sia per la GFP e RFP. - Dopo la normalizzazione, determinare la resistenza di terminazione relativa utilizzando l'equazione
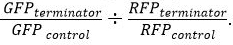
Risultati
Questo protocollo produrrà cellule contenenti PGR-blu con un terminatore legatura tra la GFP e RFP utilizzando il Golden Gate di montaggio (Figura 2). colonie positive contenenti inserto legatura possono essere selezionati in base al colore. Alla luce visibile colonie positive saranno bianco / giallo e falsi positivi produrranno colonie blu dopo 18-20 ore di incubazione a 37 ° C (Figura 3).
...
Discussione
Il passo più importante in questo protocollo è corretta progettazione oligonucleotide prima di ordinare. Gli oligonucleotidi devono avere le estremità coesive opportune aggiunte alla estremità 5 'della parte superiore e inferiore trefoli per garantire che GGA incorporazione è possibile. Inoltre, è importante cambiare l'orientamento delle terminazioni rivolto verso sinistra (terminatori che fermano la trascrizione sul filamento basso) a quella di destra di fronte (termina la trascrizione sul filo alto) term...
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
Gli autori vorrebbero riconoscere Malcolm Campbell e Todd Eckdahl con il Genome Consortium per l'insegnamento attivo (GCAT) e il HHMI-Science Education Alliance - Cacciatori fagi Avanzando Genomica e Evolutionary Science programma (SEA-fagi).
Questo progetto è stato sostenuto da sovvenzioni dal National Center for Research Resources (P20RR016460) e l'Istituto Nazionale di General Medical Sciences (P20GM103429) dal National Institutes of Health. Questa ricerca è stata sostenuta in parte dalla National Science Foundation sotto concessione # IIA-1.457.888. (Ouachita Baptist University) fondi Inoltre istituzionali sono stati forniti attraverso il JD Patterson Estate Research Fellowship.
Materiali
| Name | Company | Catalog Number | Comments |
| pGR-Blue Plasmid | Addgene | 68374 | |
| pGR-Plasmid | Addgene | 46002 | |
| AeraSeal-(Sterile Sheets) | Excel Scientific | BS-25 | Sterile Sheets only |
| 10X T4 DNA ligase Buffer | NEB | ||
| BsaI-HF | NEB | R3535S | The non-HF enzyme will work but is less heat stable. |
| NEB Golden Gate Assembly Mix | NEB | E1600S | Commerial Master Mix refered to in the protocol. |
| T4 DNA ligase | NEB | M0202S | |
| Round Microcentrifuge Floating Rack | Nova Tech International | F18875-6401 | |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | |
| L-(+)-Arabinose | Sigma Aldrich | A-3256 | D-Arabinose will not induce the pBAD promoter |
| Luria Base (LB) - Broth, Miller | Sigma Aldrich | L1900 | |
| Luria Base (LB) - Agar , Miller | Sigma Aldrich | L2025 | |
| Tecan-Infinite M200 Plate Reader | Tecan | ||
| Mix & Go Competent Cells - Strain JM109 | Zymo Research | T3005 | Use company recommended transformation protocol |
| ApE: A plasmid editor-software | http://biologylabs.utah.edu/jorgensen/wayned/ape/ | ||
| Tris-HCl, Molecular Grade | Promega | H5121 | |
| Sodium Chloride (Crystalline/Biological, Certified) | Fisher Chemical | S671 | |
| Comercial Oligonucleotide synthesis | Integrated DNA Technologies (IDT) | http://www.idtdna.com/site | |
| Microtest Tissue Culture Plates- 96 well (Sterile) | Falcon | 35-3072 | |
| mycobacteriophage "Bernal13" | Genebank | KJ510413 | |
| Nuclease Free Water | Integrated DNA Technologies (IDT) | IDT004 | |
| Sterile, L-shaped Hockey-Stick Cell | Life Science Products | 6444-S1 | |
| Nano-Drop 2000c UV-Vis Spectrometer | Thermo Scientific | 2000c | |
| ARNold: a web tool for the prediction of Rho-independent transcription terminators. | http://rna.igmors.u-psud.fr/ |
Riferimenti
- Li, J., Zhang, Y. Relationship between promoter sequence and its strength in gene expression. Eur Phys J E Soft Matter. 37 (9), (2014).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Lampropoulos, A., et al. GreenGate---a novel, versatile, and efficient cloning system for plant transgenesis. PLoS One. 8 (12), e83043 (2013).
- Chen, Y. J., et al. Characterization of 582 natural and synthetic terminators and quantification of their design constraints. Nat Methods. 10 (7), 659-664 (2013).
- Woodin, T., Carter, V. C., Fletcher, L. Vision and change in biology undergraduate education, a call for action--initial responses. CBE Life Sci Educ. 9 (2), 71-73 (2010).
- Vasaly, H. L., Feser, J., Lettrich, M. D., Correa, K., Denniston, K. J. Vision and change in the biology community: snapshots of change. CBE Life Sci Educ. 13 (1), 16-20 (2014).
- Naville, M., Ghuillot-Gaudeffroy, A., Marchais, A., Gautheret, D. ARNold: a web tool for the prediction of Rho-independent transcription terminators. RNA Biol. 8 (1), 11-13 (2011).
- Campbell, A. M., et al. pClone: Synthetic Biology Tool Makes Promoter Research Accessible to Beginning Biology Students. CBE Life Sci Educ. 13 (2), 285-296 (2014).
- Rhee, J. I., et al. Influence of the medium composition and plasmid combination on the growth of recombinant Escherichia coli JM109 and on the production of the fusion protein EcoRI::SPA. J Biotechnol. 55 (2), 69-83 (1997).
- Dirla, S., Chien, J. Y., Schleif, R. Constitutive mutations in the Escherichia coli AraC protein. J Bacteriol. 191 (8), 2668-2674 (2009).
- Schleif, R. Regulation of the L-arabinose operon of Escherichia coli. Trends Genet. 16 (12), 559-565 (2000).
- Kosuri, S., et al. Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proc Natl Acad Sci U S A. 110 (34), 14024-14029 (2013).
- Sharon, E., et al. Inferring gene regulatory logic from high-throughput measurements of thousands of systematically designed promoters. Nat Biotechnol. 30 (6), 521-530 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon