このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
PGR-ブループラスミドとゴールデンゲートアセンブリを使用してターミネータの迅速な検証
要約
This protocol utilizes Golden Gate Assembly and the plasmid pGR-blue to rapidly quantify the strength of terminators found in silico.
要約
The goal of this protocol is to allow for the rapid verification of bioinformatically identified terminators. Further, the plasmid (pGR-Blue) is designed specifically for this protocol and allows for the quantification of terminator efficiency. As a proof of concept, six terminators were bioinformatically identified in the mycobacteriophage Bernal13. Once identified, terminators were then made as oligonucleotides with the appropriate sticky ends and annealed together. Using Golden Gate Assembly (GGA), terminators were then cloned into pGR-Blue. Under visible light, false positive colonies appear blue and positively transformed colonies are white/yellow. After induction of an arabinose inducible promoter (pBad) with arabinose, colony strength can be determined by measuring the ratio of green fluorescent protein (GFP) produced to red fluorescent protein (RFP) produced. With pGR-Blue, the protocol can be completed in as little as three days and is ideal in an educational setting. Additionally, results show that this protocol is useful as a means for understanding in silico predictions of terminator efficiency related to the regulation of transcription.
概要
Large synthetic biology projects necessitate the use of highly effective transcription terminators to help regulate gene expression. Identification of novel terminators requires bioinformatic analysis of novel genomes. However, as increasing amounts of bioinformatic software are developed, each with a unique algorithm utilized for prediction, more discrepancy between putative results occurs. Because this process is somewhat subjective and is done in silico, these predictions need biological confirmation.1 Additionally, the volume of putative terminators identified through in-silico analysis requires the use of cloning strategies that can be completed in a relatively short time frame.
The PGR-Blue plasmid is a modification of the PGR plasmid that has been redesigned to use Golden Gate Assembly (GGA) to simplify the cloning procedure by allowing for all reaction steps to be simultaneously performed in one micro-centrifuge tube.2,3 Color selection was incorporated into the plasmid to increase the ease of identifying positive colonies. A successful ligation should be white/yellow in visible light and fluoresce green under blue (450 nm) or ultraviolet (UV) light when grown on plates containing arabinose. Because uncut pGR-blue contains a blue chromo protein (amilCP), colonies containing an unmodified plasmid are blue under visible light. This simplification along with the streamlined protocol allows researchers to proceed from bioinformatic identification to biological confirmation in three to four days. The design nature of this system can be beneficial both in the research lab and in educational settings.
The pGR-Blue plasmid allows for quantification of terminator strength.4 A single arabinose inducible promoter is used to produce green fluorescent protein (GFP) and red fluorescent protein (RFP). The terminator is cloned into the plasmid after the GFP sequence but before the RFP sequence, thus stopping the transcription of the RFP protein. The strength of the terminator is determined by the ratio of GFP produced to RFP produced.
The Vision and Change5 report suggested that Science, Technology, Engineering and Math (STEM) education incorporate research based experiences into the classroom.6 However, this requires the development of protocols that can be done by students with limited skill sets in a defined time frame. While the protocol can be accomplished in as little as three days, it was also designed so that each major step could be accomplished in a separate weekly (2-3 hr) lab period to create a Course Research Experience (CRE). When used in this manner, the procedure will take between three and six weeks and is appropriate for both introductory and advanced courses in Genetics, Cell Biology or Bioinformatics.
プロトコル
1.適切な粘着末端を有するオリゴヌクレオチドを設計し、注文
- オンラインで自由に利用可能なプログラムを用いてゲノム解析を通じて潜在的なローに依存しないターミネータを識別します。7
- 二本鎖DNAを操作する場合は、テストされるターミネーターの方向を決定する。7 PGR-ブループラスミドは上部のみ(フォワード鎖)の5 'から3'方向に連結したターミネータを検証します。
- 底に変換し、無料のオンラインソフトウェア、 例えば 、APEを使用して、PGR-ブルーでテストするためのシーケンスを再配向するために、その逆相補体に鎖ターミネーター(リバース)。
- 向きが正しいと判断された後、上部の鎖の5 '末端に粘着末端「5'-CGAC-3 "'を追加します。下の鎖の5 '末端に粘着末端「5'-CCGC-3 "'を追加。8これが連結したインサートが適切に挿入されることを保証しますGGAを使用して向き。
- 配列が決定されると、商業的に一本鎖オリゴヌクレオチドとして注文します。
2.アニーリングオリゴヌクレオチドは、(凍結乾燥するDNAをのみ適用されます)
- 再懸濁、100μMの濃度にヌクレアーゼフリー水で個々のオリゴヌクレオチド。
- 1 M NaClおよび100 mMトリス - 塩酸pHが7.4:10×アニーリング緩衝液を作ります。アニーリング緩衝液は、数週間4℃で保存することができます。アニーリングバッファーが行われ、オリゴヌクレオチドが再懸濁された後、アニール8を開始します。
- 各終端抵抗をテストするためには、1.5ミリリットルのマイクロチューブに超純水16μlを添加します。
- 各マイクロチューブに10倍アニーリング緩衝液2μLを加えます。
- 希望ターミネータから上の鎖オリゴヌクレオチドの1μlの(100μM)を追加します。
- 希望ターミネータからボトム鎖オリゴヌクレオチド(100μM)の1μLを加えます。
- 沸騰したお湯のBAを作成します。ホットプレート上で水道水と場所の約400ミリリットルを含む千ミリリットルのビーカーを使用して目。
- フロートのステップ2.6からマイクロ遠心チューブを置き、4分間沸騰。 4分後、熱板をオフにするが、チューブを残し、水浴にゆっくりとクールなO / N(18時間)8に浮きます。 -20℃でアニールターミネータを保管してください。
3.ゴールデンゲートアセンブリ(標準ミックス - コスト効率のよいです)
- アニールターミネーターの40 nMの希釈を行います。アニールターミネーターミックス(100μM)の1μlにヌクレアーゼフリー水124μlを添加します。
- 氷上で30万×gで秒、店舗用の遠心分離機サンプル。
- 使用されているサーモサイクラーに適した新たなチューブに、ヌクレアーゼを含まない水の6μlを添加します。
- 市販のDNAリガーゼバッファー[300mMのトリス塩酸(pHが7.7から7.8)、100 mMのMgCl 2を、100mMのDTT、および10mM ATP]の1μLを加えます。氷の上で後続のすべての手順を実行します。
- 加えますPGR-ブルー(35-50 ngの/μl)をマイクロ遠心チューブに先プラスミドの1μlの。
- マイクロチューブに40 nMの焼鈍ターミネーターの1μLを加えます。
- マイクロチューブに0.5μlの市販の高忠実度のBsaI制限エンドヌクレアーゼを追加します。
- マイクロ遠心チューブに市販のDNAリガーゼの0.5μlを添加します。
- 120秒間万×gで反応混合物を遠心分離します。そして、サーマルサイクラーに直接チューブを配置します。
- サーモサイクラープログラムを実行する:37℃で1分間の20サイクル、37℃で15分間の1サイクル、続いて16℃で1分間続きます。使用サンプル直ちにまたは-20℃で凍結保存する。8
4.ゴールデンゲートアセンブリ(商業マスターミックス - 時間効率的な)
- これは、40 nmのターミネーターDNA濃度を希釈するステップ2で作成したターミネータをアニールし、100μMの1μlにヌクレアーゼフリー水124μlを添加します。新たに、このステップを実行し、1.5ミリリットルのマイクロチューブは、標識されました。
- サーモサイクラー用の新しいチューブの適切に、ヌクレアーゼフリー水14μlを添加します。新しいチューブはそれに応じてラベル付けされていることを確認します。
- マイクロチューブに商業マスターミックス緩衝液の2μlを添加します。
- マイクロチューブにPGR-ブループラスミド(35-50 ngの/μL)の1μLを加えます。
- マイクロチューブに40 nMの焼鈍ターミネーターの2μLを加えます。
- マイクロチューブに商業マスターミックスの1μLを加えます。
- 120秒間万×gで40 nMのチューブを遠心。そして、サーマルサイクラーに直接チューブを配置します。
- 55℃で15分間の1サイクル、続いて37℃で60分間の1サイクル:サーモサイクラープログラムを実行します。使用してすぐにサンプルまたは-20℃で凍結して保存。
5.形質転換およびコロニーの選択
- 準備の1mM concentratを含むペトリ皿の寒天ルリアベース(LB)アンピシリンのイオン(アンプ)とアラビノースの10 mMの濃度(ARB)。このストック溶液は、後の工程で使用されるため、1 MのL -arabinose原液の少なくとも10 mlで調製します。
- 任意の高効率使用(8月10日から9月10日までの形質転換体/μgのを)化学的コンピテントE.変換のための大腸菌 (JM109)細胞。最良の結果を得るために9、コンピテント細胞を50μlに連結反応の3-5μlを添加します。化学的コンピテント細胞を使用する場合、細胞が使用されている固有の形質転換プロトコルに従います。
- 無菌のL字型の「ホッケースティック」を使用して寒天プレート予熱したLB(アンプ/ ARB)に形質転換された細胞の半分(25μl)を広げ、37℃でO / Nインキュベートします。分解のアンピシリンの急速なレートに、24時間にわたって37℃でインキュベートしません。
- 目視による色に基づいて、コロニーの選択を行います。成功したライゲーションは、可視光で黄色/白と緑のアンクルを蛍光を発する必要がありますr個の青(450 nmの)またはUV光。 20時間 - 失敗したライゲーションは18の後に、可視光下での色は青であるコロニーを生成します。
6.検証とターミネーターの定量
- 1 mMのアンピシリンおよびアラビノースの10mMで5ミリリットル滅菌LBブロスチューブを準備します。必要な管の数は、試験されるターミネータープラスコントロール(細胞アンピシリン耐性を持つ蛍光を発しないとノーカットPGR-ブループラスミドを含む細胞)の数に依存しています。
- 可視光下で滅菌ループを使用して2-3の黄色/白色コロニー(ステップ5.4)を選択します。試料は37℃、16-18時間、160rpmでシェーカー上ブロスでインキュベートすることを可能にします。以上24時間これらのサンプルをインキュベートしないでください。
- 必要に応じて、適切な細胞の成長を確実にするために、600nmの(OD 600)での光学セル密度を決定するために分光光度計を使用します。一般的に、〜0.8〜1.0のOD 600は、成長の16-18時間後に観察されます。
- つかいます残りのステップのための滅菌96ウェルプレート。無菌環境と三重にこれらのステップを実施します。各ウェルに滅菌LB +アンプ/アラビノースブロス(1 mMのアンピシリン/ 10 mMのアラビノース)の199μLを加えます。 3つの別々のコントロールのプレート上でルームなどがあります。
- 次の3つのコントロールを使用します。ウェル(1)を含むLB +アンプ/アラビノースブロスのみとウェル(2)(空白)非形質転換コンピテントセルは空白として機能します。定量化のために(3)ターミネータ強度の、非終結配列と連結したノーカットオリジナルPGRを含むまたはPGR-ブルーを含む細胞は、(定量化のための基準制御)に含まれるべきです。プレートリーダーのレイアウトについては、 図1を参照してください。
- 96ウェルプレートの個々のウェルにO / N培養液から細胞溶液のピペット1μlの。
- 通気性のカバーとシールプレートとは、37℃で18-20時間、および160 rpmで振とうします。
- プレートリーダーを用いて読み取ることにより、OD 600を決定します600 nmの吸光度。次のようにGFP蛍光を測定する:励起395及び発光509メジャーRFP蛍光を次のように:励起575および発光605を18から20時間後、OD 600が約0.5であるべきです。
- 成長の変化を標準化するために、式を使用します。
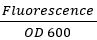 GFPとRFPの両方のために。
GFPとRFPの両方のために。 - 正規化の後、式を用いて相対的なターミネーターの強度を決定します
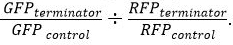
結果
このプロトコルは、ゴールデンゲートアセンブリ( 図2)を使用して、GFPとRFPの間に連結ターミネータでPGR-ブルーを含む細胞を生成します。ライゲーションされた挿入物を含む陽性コロニーの色に基づいて選択することができます。可視光では陽性コロニーは白/黄色になり、偽陽性は37°C( 図3)でのインキュベーションの18から20時間後に青...
ディスカッション
このプロトコルの中で最も重要なステップは、発注前に、適切なオリゴヌクレオチドの設計です。オリゴヌクレオチドは、適切な粘着末端は、GGAの取り込みが可能であることを保証するために、両方の上部と下部の鎖の5 '末端に付加されている必要があります。 GFPおよびRFP式が(トップ鎖に向かって右にあるのでさらに、右向きのものに左向きターミネーター(下の鎖上で転写を停?...
開示事項
The authors have nothing to disclose.
謝辞
ゲノミクスと進化科学を推進するファージハンター(SEA-ファージ)プログラム - 著者は、Active教育のためのゲノムコンソーシアム(GCAT)とHHMI-科学教育連盟とマルコム・キャンベルとトッドEckdahlを承認したいと思います。
このプロジェクトは、研究資源のための国立センター(P20RR016460)と国立衛生研究所から一般医科学研究所(P20GM103429)からの助成金によってサポートされていました。この研究は、助成金#のIIA-1457888の下で国立科学財団によって部分的にサポートされていました。さらに制度(ウォシタ・バプティスト大学)資金はJDパターソン夏の特別研究員を介して提供されました。
資料
| Name | Company | Catalog Number | Comments |
| pGR-Blue Plasmid | Addgene | 68374 | |
| pGR-Plasmid | Addgene | 46002 | |
| AeraSeal-(Sterile Sheets) | Excel Scientific | BS-25 | Sterile Sheets only |
| 10X T4 DNA ligase Buffer | NEB | ||
| BsaI-HF | NEB | R3535S | The non-HF enzyme will work but is less heat stable. |
| NEB Golden Gate Assembly Mix | NEB | E1600S | Commerial Master Mix refered to in the protocol. |
| T4 DNA ligase | NEB | M0202S | |
| Round Microcentrifuge Floating Rack | Nova Tech International | F18875-6401 | |
| Ampicillin sodium salt | Sigma Aldrich | A9518 | |
| L-(+)-Arabinose | Sigma Aldrich | A-3256 | D-Arabinose will not induce the pBAD promoter |
| Luria Base (LB) - Broth, Miller | Sigma Aldrich | L1900 | |
| Luria Base (LB) - Agar , Miller | Sigma Aldrich | L2025 | |
| Tecan-Infinite M200 Plate Reader | Tecan | ||
| Mix & Go Competent Cells - Strain JM109 | Zymo Research | T3005 | Use company recommended transformation protocol |
| ApE: A plasmid editor-software | http://biologylabs.utah.edu/jorgensen/wayned/ape/ | ||
| Tris-HCl, Molecular Grade | Promega | H5121 | |
| Sodium Chloride (Crystalline/Biological, Certified) | Fisher Chemical | S671 | |
| Comercial Oligonucleotide synthesis | Integrated DNA Technologies (IDT) | http://www.idtdna.com/site | |
| Microtest Tissue Culture Plates- 96 well (Sterile) | Falcon | 35-3072 | |
| mycobacteriophage "Bernal13" | Genebank | KJ510413 | |
| Nuclease Free Water | Integrated DNA Technologies (IDT) | IDT004 | |
| Sterile, L-shaped Hockey-Stick Cell | Life Science Products | 6444-S1 | |
| Nano-Drop 2000c UV-Vis Spectrometer | Thermo Scientific | 2000c | |
| ARNold: a web tool for the prediction of Rho-independent transcription terminators. | http://rna.igmors.u-psud.fr/ |
参考文献
- Li, J., Zhang, Y. Relationship between promoter sequence and its strength in gene expression. Eur Phys J E Soft Matter. 37 (9), (2014).
- Engler, C., Kandzia, R., Marillonnet, S. A one pot, one step, precision cloning method with high throughput capability. PLoS One. 3 (11), e3647 (2008).
- Lampropoulos, A., et al. GreenGate---a novel, versatile, and efficient cloning system for plant transgenesis. PLoS One. 8 (12), e83043 (2013).
- Chen, Y. J., et al. Characterization of 582 natural and synthetic terminators and quantification of their design constraints. Nat Methods. 10 (7), 659-664 (2013).
- Woodin, T., Carter, V. C., Fletcher, L. Vision and change in biology undergraduate education, a call for action--initial responses. CBE Life Sci Educ. 9 (2), 71-73 (2010).
- Vasaly, H. L., Feser, J., Lettrich, M. D., Correa, K., Denniston, K. J. Vision and change in the biology community: snapshots of change. CBE Life Sci Educ. 13 (1), 16-20 (2014).
- Naville, M., Ghuillot-Gaudeffroy, A., Marchais, A., Gautheret, D. ARNold: a web tool for the prediction of Rho-independent transcription terminators. RNA Biol. 8 (1), 11-13 (2011).
- Campbell, A. M., et al. pClone: Synthetic Biology Tool Makes Promoter Research Accessible to Beginning Biology Students. CBE Life Sci Educ. 13 (2), 285-296 (2014).
- Rhee, J. I., et al. Influence of the medium composition and plasmid combination on the growth of recombinant Escherichia coli JM109 and on the production of the fusion protein EcoRI::SPA. J Biotechnol. 55 (2), 69-83 (1997).
- Dirla, S., Chien, J. Y., Schleif, R. Constitutive mutations in the Escherichia coli AraC protein. J Bacteriol. 191 (8), 2668-2674 (2009).
- Schleif, R. Regulation of the L-arabinose operon of Escherichia coli. Trends Genet. 16 (12), 559-565 (2000).
- Kosuri, S., et al. Composability of regulatory sequences controlling transcription and translation in Escherichia coli. Proc Natl Acad Sci U S A. 110 (34), 14024-14029 (2013).
- Sharon, E., et al. Inferring gene regulatory logic from high-throughput measurements of thousands of systematically designed promoters. Nat Biotechnol. 30 (6), 521-530 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved