Method Article
Visualización interior de la membrana bacteriana para la selección de una biblioteca de fragmentos de anticuerpos
En este artículo
Resumen
We provide a method to simultaneously screen a library of antibody fragments for binding affinity and cytoplasmic solubility by using the Escherichia coli twin-arginine translocation pathway, which has an inherent quality control mechanism for intracellular protein folding, to display the antibody fragments on the inner membrane.
Resumen
Antibodies engineered for intracellular function must not only have affinity for their target antigen, but must also be soluble and correctly folded in the cytoplasm. Commonly used methods for the display and screening of recombinant antibody libraries do not incorporate intracellular protein folding quality control, and, thus, the antigen-binding capability and cytoplasmic folding and solubility of antibodies engineered using these methods often must be engineered separately. Here, we describe a protocol to screen a recombinant library of single-chain variable fragment (scFv) antibodies for antigen-binding and proper cytoplasmic folding simultaneously. The method harnesses the intrinsic intracellular folding quality control mechanism of the Escherichia coli twin-arginine translocation (Tat) pathway to display an scFv library on the E. coli inner membrane. The Tat pathway ensures that only soluble, well-folded proteins are transported out of the cytoplasm and displayed on the inner membrane, thereby eliminating poorly folded scFvs prior to interrogation for antigen-binding. Following removal of the outer membrane, the scFvs displayed on the inner membrane are panned against a target antigen immobilized on magnetic beads to isolate scFvs that bind to the target antigen. An enzyme-linked immunosorbent assay (ELISA)-based secondary screen is used to identify the most promising scFvs for additional characterization. Antigen-binding and cytoplasmic solubility can be improved with subsequent rounds of mutagenesis and screening to engineer antibodies with high affinity and high cytoplasmic solubility for intracellular applications.
Introducción
Anticuerpos capaces de doblar y funcionando en el entorno intracelular son herramientas prometedoras tanto para aplicaciones terapéuticas y de investigación. Tienen la capacidad de modular la actividad de la proteína mediante la unión a una proteína diana dentro de las células para evitar interacciones proteína-proteína, interrumpir las interacciones proteína-ácido nucleico, o impedir el acceso de sustrato a las enzimas 1-5.
Aunque los anticuerpos tienen un gran potencial para aplicaciones intracelulares, ingeniería ellos para el plegamiento adecuado y la solubilidad en el medio ambiente intracelular, mientras que se mantiene la capacidad de unirse a un antígeno diana es un reto. El entorno citoplásmico reducir evita la formación de los enlaces disulfuro que normalmente se requieren para el plegado estable de anticuerpos de longitud completa y fragmentos de anticuerpos, incluyendo anticuerpo de cadena sencilla (scFv) anticuerpos 6,7. Una serie de enfoques de evolución dirigida se han empleado para diseñar anticuerpos con hafinidades IGH para encauzar antígenos 8-10. Estos enfoques utilizan comúnmente presentación de fagos, presentación de superficie de levadura, o presentación de superficie bacteriana para cribar grandes bibliotecas de anticuerpos 11-13. Estos métodos son de gran alcance y eficaz para la identificación de anticuerpos que se unen a objetivos, sin embargo, dependen de la vía secretora para el transporte de las proteínas que se mostrarán 14-16. La vía secretora transloca proteínas desplegadas desde el citoplasma reducción en el lumen del retículo endoplasmático en la levadura o en el periplasma en bacterias. Las proteínas se pliegan a continuación en condiciones oxidantes y se muestran en la superficie celular o empaquetados en partículas de fago para la detección de la afinidad de unión a 17,18. Como resultado, los anticuerpos aislados usando estas técnicas no necesariamente plegar bien en el citoplasma, y la solubilidad intracelular a menudo deben ser diseñados por separado si se utilizan los anticuerpos en aplicaciones intracelulares.
Mejorarla eficacia de los anticuerpos de ingeniería que están bien plegadas en el citoplasma, se informó anteriormente el éxito de MAD-TRAP (pantalla anclada a la membrana para el reconocimiento basado en Tat de proteínas que asocian), un método para el cribado de una biblioteca de anticuerpos scFv utilizando Escherichia coli inervación pantalla membrana 19. visualización del centro de la membrana bacteriana se basa en la vía de doble arginina translocación (Tat) para el transporte de los anticuerpos que se muestran, en contraste con otros métodos de visualización común que utilizan la vía secretora. La vía de Tat contiene un mecanismo de control de calidad que sólo permite, proteínas correctamente plegadas solubles para ser transportados de la E. citoplasma coli, a través de la membrana interna, y en el periplasma 20,21. Sustratos sobreexpresa Tat (es decir., Proteínas dirigidas a la vía de Tat con una fusión N-terminal con el péptido señal Tat ssTorA) que son bien doblado en el citoplasma forman una translocación de larga vida intermedio con el N-terminal in el citoplasma y el C-terminal en el periplasma 19. Esto permite la visualización de Tat sustratos correctamente plegadas, incluyendo fragmentos de anticuerpos, en la cara periplásmico de la E. membrana interna coli. Después de retirar la membrana externa mediante digestión enzimática para generar esferoplastos, los anticuerpos están expuestos al espacio extracelular (Figura 1). Esto permite Tat sustratos que se muestran en la membrana interna que se proyectarán por la unión a un objetivo específico. Es importante destacar que, el aprovechamiento de la vía de Tat para la visualización de la superficie celular asegura que sólo los anticuerpos de la biblioteca que son bien plegadas en el citoplasma serán interrogados por la unión, lo que permite la ingeniería simultánea de afinidad y plegado intracelular de unión. En este protocolo, se describe cómo mostrar una biblioteca de scFv en el E. membrana interna coli, Pan la biblioteca contra un antígeno diana, y llevar a cabo una pantalla secundaria para identificar los componentes más prometedores de la biblioteca. Mientras nos centramos el protocolo de scFv, el método podría ser aplicado a la ingeniería de cualquier proteína cuya aplicación requiere de plegado y unión intracelular.
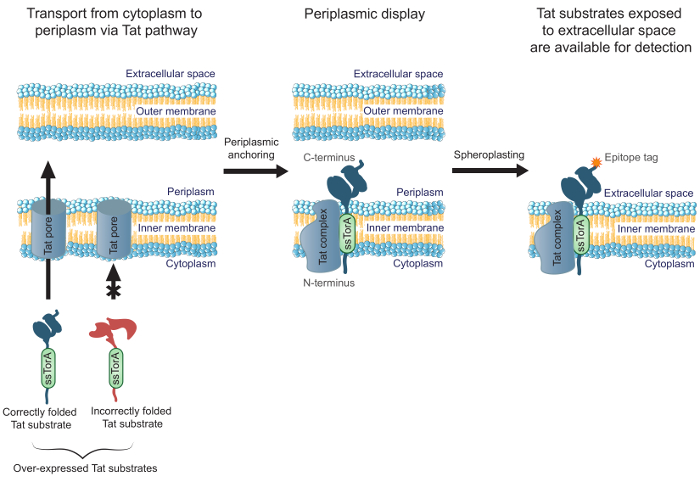
Figura 1. Pantalla-Tat membrana interna. En E. coli, los anticuerpos scFv que se expresan como una fusión con la secuencia señal ssTorA y correctamente plegadas en el citoplasma son transportados a través de la membrana interna. Una translocación formas intermedias, donde los scFv están anclados en la membrana interna con el extremo N-terminal en el citoplasma y el C-terminal en el periplasma. El E. membrana externa coli se digiere enzimáticamente para formar esferoplastos, exponiendo de este modo los anticuerpos anclados al espacio extracelular y ponerlos a disposición para la detección mediante el uso de un anticuerpo que se une a la etiqueta epítopo fusionado C-terminal en el anticuerpo visualizada.carga / 54583 / 54583fig1large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
1. Preparar la biblioteca de scFv como una fusión con la secuencia señal ssTorA
- Obtener una biblioteca de ácido desoxirribonucleico (ADN) que contiene variantes de un gen de scFv.
NOTA: La biblioteca también puede construirse usando cualquier modo apropiado para generar diversidad en todo el gen scFv o dominios específicos 22 (por ejemplo, la tercera regiones determinantes de complementariedad, CDR3.). - Insertar la biblioteca de ADN en el plásmido PIMD (Figura 2), utilizando métodos de clonación molecular estándar 23.
NOTA: Este plásmido expresa scFv como una fusión genética de la secuencia de señal ssTorA (N-terminal de scFv) y la etiqueta de epítopo FLAG (C-terminal a la scFv). El diseño del plásmido para la visualización de la membrana interna se ha descrito previamente 19. El plásmido PIMD está disponible de los autores.
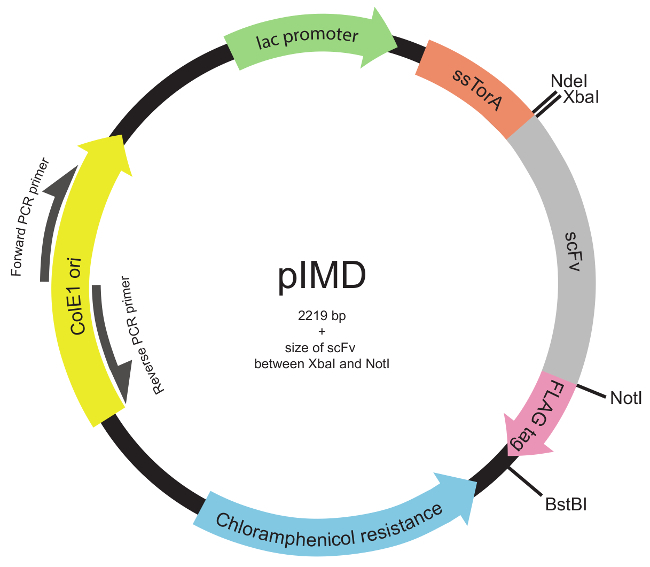
Figura 2. Pantalla plásmido-membrana interna (PIMD) mapa (pasos 1.2 a 1.3). Este plásmido contiene un promotor lac, el origen ColE1 de replicación, y un gen de resistencia a cloranfenicol. El gen scFv insertado se fusiona con la secuencia señal ssTorA para apuntar el scFv a la vía de Tat y a una etiqueta de epítopo FLAG, con los tres en el mismo marco de lectura. sitios de enzimas de restricción se indican. Para una biblioteca insertado entre los sitios de enzimas de restricción XbaI y NotI, el tamaño del plásmido es 2219 pb más el tamaño del scFv. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Transformar el ADN plásmido que contiene la biblioteca en MC4100 de E. 23 células de E. coli. Recuperarse y crecer esta forma bacteriana de la biblioteca. Centrifugar a 4000 xg durante 15 min a TA para recoger las células. Eliminar el sobrenadante, y resuspender las células recogidasen 25% de glicerol en Luria-Bertani medios (LB). alícuotas almacenar a -80 ° C hasta que se necesite, o continúe con el paso 2.
NOTA: El protocolo ha sido verificado con células MC4100, aunque otras E. También se espera que las cepas de E. coli para ser compatible con el protocolo. La electroporación es el método preferido para la transformación, debido a su alta eficiencia de transformación. La biblioteca típicamente debe consistir en al menos 10 9 scFv variantes en esta etapa, y cada alícuota debe contener suficientes células de manera que la biblioteca está cubierta 100 veces.
2. Expresar la Biblioteca y Preparar esferoplastos
- Descongelar una alícuota de la biblioteca bacteriana (del Paso 1.3) a temperatura ambiente, y añadir la alícuota a un matraz que contenía 100 ml de medio LB con 20 mg / ml de cloranfenicol (Cm). Crecer durante 3 horas a 37 ° C y 225 rpm en un agitador se incubaron.
- Después de 3 horas, retirar el matraz del agitador se incubaron 37 ° C. Permitir la expresión de la biblioteca de scFv aproceder O / N para 15 a 22 horas a 20 ° C y 225 rpm en un agitador se incubaron.
NOTA: No se necesita inductor cuando se utiliza el plásmido PIMD, como el promotor tiene fugas. Tenga en cuenta que las células MC4100 no sobreexpresan el represor Lac (y LacI no se encuentra en el plásmido).

Figura 3. células de E. coli y esferoplastos. (A) E. coli células son de forma cilíndrica. (B) Después de formación de esferoplastos usando EDTA y lisozima, la membrana exterior de la E. coli células se rompen, y los esferoplastos resultantes son de forma esférica. De contraste de interferencia diferencial (DIC) Imágenes de microscopía se obtuvieron utilizando un objetivo de 100X en un microscopio invertido. Por favor, clamer aquí para ver una versión más grande de esta figura.
- Preparar los esferoplastos biblioteca.
NOTA: Los esferoplastos se forman por la ruptura de la membrana externa de E. coli y son de forma esférica (figura 3).- Preparar los tampones necesarios.
NOTA: Todos los tampones deben ser estériles.- Preparar 1 × solución salina tamponada con fosfato (PBS; pH 7,4) disolviendo 8 g NaCl, 0,2 g KCl, 1,44 g de Na 2 HPO 4, y 0,24 g KH 2 PO 4 en H 2 O destilada a un volumen final de 1.000 ml. Mantener en hielo.
- Preparar PBS con 0,1% (w / v) de suero de albúmina bovina (BSA) por disolución de 0,2 g de BSA en 200 ml de 1 x PBS. Mantener en hielo.
- Preparar el tampón de fraccionamiento (FB) mezclando 7,5 ml de H 1 M de sacarosa, 1 ml de tampón de Tris 1 M (pH 8,0), y 1,5 ml filtrada estéril destilada 2 O. Mantener en hielo.
- Preparar 1 mM de ácido etilendiaminotetraacético (EDTA) mediante la adición de 30 l of EDTA 0,5 M a 14.97 ml H2O destilada
- Preparar 0,5 M MgCl2 disolviendo 4,76 g de MgCl 2 en 100 ml de H2O destilada Mantener en hielo.
- Retirar el matraz del agitador, y medir la densidad óptica (DO) a 600 nm utilizando un espectrofotómetro para determinar la densidad celular. Calcular el volumen de cultivo inducido sea necesario de manera que cada muestra para formación de esferoplastos tiene 1 × 10 10 células.
NOTA: La aproximación de una DO 600 de 1 que indica una concentración de 10 9 células / ml para E. coli se puede utilizar 24. - Centrifugar el volumen calculado de cultivo inducido en un tubo de microcentrífuga de 1,5 ml a 12.000 × g a TA durante 5 min. Preparar al menos dos muestras en el caso de que se plantee una cuestión de preparación de la muestra.
- Eliminar el sobrenadante de los cultivos se centrifugaron y resuspender cada sedimento celular en 100 l de FB enfriado con hielo. Centrifugar a 12.000 ×g a TA durante 1 min, y luego eliminar el sobrenadante mediante pipeteo. Resuspender cada sedimento en 350 l de FB enfriado en hielo suplementado con 3,5 l de 10 mg / ml de lisozima.
- vórtice lentamente cada tubo mientras se añade, gota a gota, a 700 l de EDTA 1 mM, y luego se incuban los tubos a TA durante 20 min mientras que gira lentamente en un mezclador de tubo para mezclar las muestras. Retire los tubos del agitador, se añaden 50 l de helado 0,5 M MgCl 2 a cada tubo, y se incuba en hielo durante 10 min. Centrifugar los tubos a 11.000 × g a 4 ° C durante 10 min.
- Aislar el pellet de esferoplastos.
- Utilice una micropipeta con una punta de 1 ml para extraer lentamente hasta parte de pellets. Mientras sostiene el tubo en un ángulo con la abertura directamente encima de un nuevo tubo de 1,5 ml, levante lentamente la punta de pipeta fuera del sobrenadante y deslice el pellet en el nuevo tubo.
- Si un volumen significativo de sobrenadante se transfiere al nuevo tubo, y eliminar con la pipeta. Si la pastilla no es firme enouf para transferir, re-centrifugar a 11.000 xg durante 2 min y el aislamiento pellet intento de nuevo.
- Resuspender el pellet de esferoplastos en cada tubo en 1 ml de helado 1 × PBS. Alternan entre la pipeta y el vórtex lentamente en un gancho o agitar hasta que el sedimento se volvió a suspender totalmente. No mantener las muestras fuera del hielo durante más de 2 min a la vez, y volver a la hielo durante al menos 5 min antes de sacarlo del hielo de nuevo. Mantenga los esferoplastos a 4 ° C (para un máximo de 2 días) hasta que se usaron para la expansión en el paso 4.
- Preparar los tampones necesarios.
3. Se debe inmovilizar el antígeno diana en perlas magnéticas
- Biotinilar el antígeno diana in vivo durante la producción recombinante en E. células de E. coli. Como alternativa, utilice la conjugación química 25 o antígeno objetivo de compra que ya ha sido biotinilado, y vaya al paso 3.2.
- Añadir 816 g bicina a 50 ml de agua para hacer 10 × tampón bicina. Se diluye la buffer a 1 × en H2O destilada y se calienta a 50 ° C. Añadir 14,7 mg de biotina a 12 ml de la climatizada tampón 1 × bicina para hacer una solución de biotina que es biotina 5 mM en 10 mM de tampón bicina. Almacenar a -20 ° C hasta que se necesite.
- Expresar y biotinilar la proteína diana utilizando el plásmido pAK400cb-BCCP 26, que permite la producción del antígeno diana como una fusión con la proteína portadora de carboxilo biotina (BCCP).
NOTA: E. células de E. coli nativa biotinilar BCCP, eliminando la necesidad de purificar y químicamente biotinilar la proteína diana antes de la inmovilización sobre perlas recubiertas con estreptavidina. El E. nativo coli biotina ligasa BirA es suficiente para biotinilar la proteína de fusión.- Crecer E. coli que contiene el plásmido de biotinilación (con el antígeno diana insertado como una fusión a la N-terminal de BCCP) O / N durante 15 a 18 horas en 5 ml de medio LB suplementado con 20 g / ml Cm a 37 ° C con agitación a 225 rpm.
- Medir la DO a 600 nm usando un espectrofotómetro y se calcula el volumen de cultivo necesario (V ADD) a subcultivo en un OD a partir de 0.05 en 25 ml de medio LB fresco con 20 mg / ml Cm usando la ecuación: V añadir = (0,05 × 25 ml) / (OD 600 a 0,05), donde OD 600 es la densidad óptica de la cultura O / N y V añadir es el volumen del cultivo O / N para añadir a la LB. fresco Subcultivo y crecer hasta una DO de 0,5 a 0,8 en un agitador se incubaron a 37 ° C y 225 rpm.
- Añadir isopropilo β-D-1-tiogalactopiranósido a una concentración final de 100 mM y biotina a una concentración final de 5 mM. Inducir la expresión en un agitador se incubaron durante 15-22 horas a 20 ° C y 225 rpm.
- bacterias de la cosecha por centrifugación a 4000 × g a 4 ° C durante 10 min. Eliminar el sobrenadante. Almacenar el sedimento a -20 ° C hasta que esté listo para su uso.
- Añadir1 ml de un detergente de lisis de células por 0,2 g de sedimento celular. Resuspender mediante pipeteo y rotar suavemente durante 20 min para lisar las células. Después de la lisis, se centrifuga a 16.000 x g y 4 ° C durante 20 min. Pipetear el lisado soluble (sobrenadante) en un nuevo tubo de 1,5 ml.
- Utilice una columna peso molecular de corte de 3 kDa para eliminar la biotina no unido. Pipetear el lisado en la columna, y se centrifuga a 20 ° C de acuerdo con las instrucciones del fabricante. Lavar con 1 x PBS hasta que la biotina en el lisado se ha diluido 100 veces y el volumen del lisado de lavado es igual al volumen original del lisado. Transferir el lisado a un tubo nuevo.
- Inmovilizar el antígeno diana biotinilado sobre perlas magnéticas recubiertas con estreptavidina.
- Preparar 1 × PBS y 1 × PBS con 0,1% de BSA (v / w) como se describe en el paso 2.3.1.
- Preparar las perlas magnéticas.
NOTA: Esto requiere el uso de una rejilla de separación magnética.- streptav ResuspenderIdin-perlas magnéticas recubiertas en su envase original. Cualquiera de vórtice durante al menos 30 seg o girar durante 5 min.
- Transferencia 7-10 × 10 9 perlas a un tubo de 1,5 ml.
NOTA: El volumen requerido dependerá de la concentración de talón suministrado por el fabricante. - Se coloca el tubo que contiene las perlas en el estante imán para 2 min para recoger las perlas en el lado del tubo. Con el tubo todavía en el imán, retirar con cuidado el sobrenadante con la pipeta sin interrumpir las perlas.
- Para lavar, quitar el tubo del imán, y volver a suspender las perlas en 1 ml de PBS 1X con la pipeta sin generar burbujas. Vuelva a colocar el tubo al imán durante 2 minutos para recoger las perlas, y retirar con cuidado el sobrenadante con la pipeta. Repetir el proceso dos veces más para un total de tres lavados. Asegúrese de que el líquido no se deja en el tubo después del lavado final.
- Añadir el lisado que contiene el antígeno biotinilado a la bea magnéticads.
- Retire el tubo del imán y resuspender las perlas en 1 ml de lisado (de la etapa 3.1.5). Incubar a RT durante 30 min mientras se gira suavemente.
- Se coloca el tubo en el imán durante 3 min para recoger las perlas recubiertas de antígeno. Lavar las perlas recubiertas cinco veces con 1 x PBS con 0,1% de BSA en la misma manera como se describe en los pasos 3.2.2.3 a 3.2.2.4. Después del lavado final, resuspender las perlas en 1 × PBS con 0,1% de BSA hasta el mismo volumen utilizado en el paso 3.2.2.2.
- Si el antígeno diana inmovilizada es estable a 4 ° C, almacenar las perlas recubiertas a 4 ° C hasta que sea necesario para la expansión. De lo contrario, continúe con el paso 4.
4. Pantalla de la biblioteca de scFv mediante los movimientos horizontales contra el antígeno diana (Figura 4)
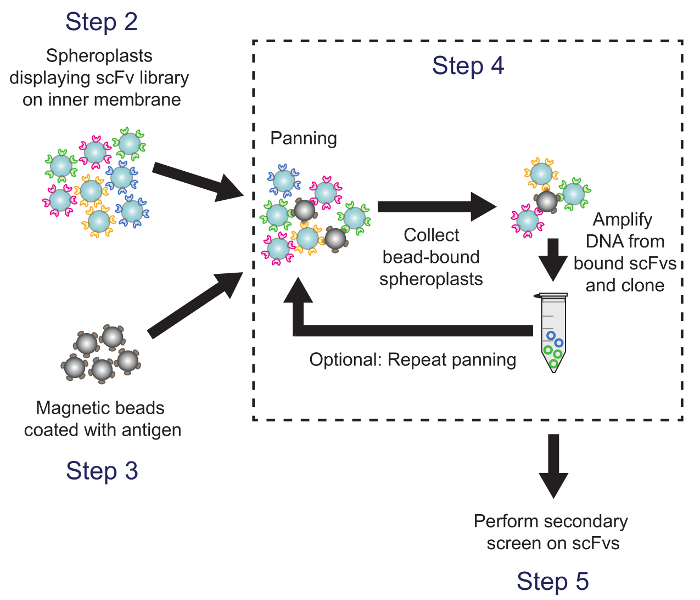
Figura 4. Toma panorámica (Paso 4). Recubiertas de antígeno perlas magnéticas are incubó con esferoplastos que expresan variantes de la biblioteca de anticuerpos. El plásmido ADN de esferoplastos de talón unido se recupera y se utiliza para generar una sub-biblioteca, que se analiza usando la pantalla secundaria basado en ELISA. Correspondientes pasos del protocolo se indique lo contrario. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Se incuban las perlas recubiertas con esferoplastos.
- Utilice una de esferoplastos a talón relación de aproximadamente 5: 1. Añadir 4 × 10 9 esferoplastos y 8 × 10 8 perlas a un tubo estéril de 15 ml.
NOTA: Se supone que no hay células se perdieron durante el proceso de formación de esferoplastos, por lo que la concentración es todavía 1 × 10 10 esferoplastos / ml. - Añadir 1 × PBS con 0,1% de BSA para llevar el volumen total a 4 ml. Alícuota en cuatro tubos de 1,5 ml con 1 ml cada uno. Se incuban las reacciones a 4 ° C durante 5 horas mientras se gira suavemente.
- Utilice una de esferoplastos a talón relación de aproximadamente 5: 1. Añadir 4 × 10 9 esferoplastos y 8 × 10 8 perlas a un tubo estéril de 15 ml.
- Prepare los esferoplastos talón determinada para la reacción en cadena de la polimerasa (PCR).
- Colocar los tubos de reacción paneo en el imán durante 3 min. Eliminar el sobrenadante por pipeteo, y lavar los esferoplastos de talón unido a cuatro veces con hielo frío 1 × PBS con 0,1% de BSA en la misma manera como se describe en los pasos 3.2.2.3 a 3.2.2.4. Volver a suspender los esferoplastos talón de ruedas en cada tubo en 25 l de H2O destilada Almacenar los granos a -20 ° C o continúe en el paso 4.3.
- Realizar todo el plásmido PCR en esferoplastos talón de ruedas para amplificar los plásmidos que contienen los genes de scFv talón de ruedas.
- Obtener cebadores con las siguientes secuencias: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(cebador directo) y 5'TAGCTCTTGATCCGGCAAACAAA3' (cebador inverso).
NOTA: Estos se unirá de extremo a extremo de las cadenas opuestas del plásmido PIMD (Figura 2) y están diseñados para hibridar con una característica común de PIMD, por lo que la amplificación se producirá con independencia de lasecuencia variante scFv. - Fosforilar los cebadores.
Nota: Sin fosforilación, no se producirá re-ligación. Los cebadores también pueden pedirse con 5'-fosforilación, en lugar de utilizar este método fosforilación en este protocolo.- En un tubo de 0,5 ml, configure una reacción de fosforilación para el cebador de PCR directo como se describe en la Tabla 1. Repita este proceso para el cebador inverso.
- Se incuban las reacciones a 37 ° C durante 1 hora. Entonces incubarlos a 65 ° C durante 20 minutos para desactivar el polinucleótido quinasa de T4 (PNK). Almacenar los cebadores fosforilados a -20 ° C.
- Realizar la PCR.
- En un tubo de PCR, preparar la reacción de PCR como se describe en la Tabla 2.
NOTA: reacciones múltiples pueden ser preparados para un mayor rendimiento. Los esferoplastos de talón de ruedas no utilizados se pueden almacenar a -20 ° C. - Calentar las reacciones de PCR a 98 ° C durante 15 min en un ciclador térmico para asegurar la lisis completa de los esferoplastos. Retire los tubos del termociclador, y añadir 0,5 l de una polimerasa de alta fidelidad de cada uno. Volver tubos para el termociclador y ejecute utilizando el programa detallado en la Tabla 3.
- Agrupar los productos de la PCR, según proceda. Almacenar a -20 ° C o continúe en el paso 4.4.
- En un tubo de PCR, preparar la reacción de PCR como se describe en la Tabla 2.
- Obtener cebadores con las siguientes secuencias: 5'CCAACTCTTTTTCCGAAGGTAACTG3 '(cebador directo) y 5'TAGCTCTTGATCCGGCAAACAAA3' (cebador inverso).
Tabla 1. reacción de fosforilación PNK (Paso 4.3.2.1).
| Reactivo | Volumen (l) |
| H2O destilada | 15 |
| tampón de reacción de ligasa de ADN T4 10x | 2 |
| imprimación 100 M | 2 |
| T4 polinucleótido quinasa (PNK) | 1 |
| Reactivo | Volumen (l) |
| H2O destilada | 28.5 |
| 5x tampón de alta fidelidad de la polimerasa | 10 |
| cebador directo 10 M fosforilada | 2.5 |
| cebador inverso 10 M fosforilada | 2.5 |
| 40 mM dNTP mix (10 mM de cada dNTP) | 1 |
| esferoplastos talón de ruedas | 5 |
Tabla 3. programa de PCR (Paso 4.3.3.2).
| Paso | Temperatura (° C) | Tiempo (min: seg) | Número de ciclos |
| desnaturalización inicial | 98 | doce y media | 1 |
| Desnaturalizar | 98 | doce y diez | 35 |
| Recocido | 69 | doce y media | |
| Extensión | 72 | Doce y treinta por kb | |
| La extensión final | 72 | 06:00 | 1 |
| Sostener | 12 | Infinito | 1 |
- Re-distribuir circulares enteros plásmido los productos de PCR, y utilizar el producto ligado para transformar E. MC4100 células de E. coli.
- Se purifica el producto de PCR mediante la ejecución de la reacción de PCR en un gel de agarosa al 23, manchando el ADN en el gel 23, y usando unakit de limpieza en gel para purificar el plásmido linealizado siguiendo las instrucciones proporcionadas por el fabricante. Medir la concentración usando un espectrofotómetro a 260 nm. Almacenar el fragmento purificado a -20 ° C hasta que sea necesario, o continuar con el paso 4.4.2.
- Re-circularizar plásmido a partir del producto PCR.
- Para evitar la ligación intermolecular del producto de PCR, llevar a cabo la reacción de ligación con una baja concentración de 27 1 ng / l de producto de PCR. Calcular el volumen necesario para la preparación de una reacción de ligación 800 l a esta concentración.
- Preparar la reacción de ligación en hielo. En un tubo, añadir el volumen calculado en el paso 4.4.2.1 del producto de PCR, 80 l de tampón ligasa 10 × ADN, y H 2 O destilada hasta 800 l. Añadir 4 l de ADN ligasa de T4, e inmediatamente colocar los tubos a 16 ° C en un baño de agua o el termociclador. Se incuba a 16 ° CO / N de 14 a 18 h. Almacenar las reacciones de ligación completados a -20 ° Chasta que sea necesario, o continúe en el paso 4.4.3.
- Coloque la reacción de ligación en un bloque de calor a 65 ° C durante 15 min para calentar-inactivar la ADN ligasa. A continuación, utilice un kit de limpieza de membrana de microdiálisis o ADN de de-sal del ADN ligado. Almacenar a 20 ° C o continúe en el paso 4.4.4.
- Utilizar todo el, producto de ligación de-salada inactivado por calor para transformar MC4100 de E. 23 células de E. coli. Preparar las existencias de glicerol, como se describe en el paso 1.3, de las células que contienen el sub-biblioteca criticada resultante, y alícuotas almacenar a -80 ° C.
- Repita el paso 4 en su totalidad utilizando una parte alícuota de la Etapa 4.4.4 hacer una segunda toma panorámica en la sub-biblioteca.
NOTA: Una segunda paneo ayuda a enriquecer la biblioteca de componentes que se unen así al antígeno diana 19.
5. Realizar una pantalla secundaria utilizando un método de ensayo de inmunoabsorción ligado a enzimas para identificar clones prometedores para una caracterización adicional (Figura 5)
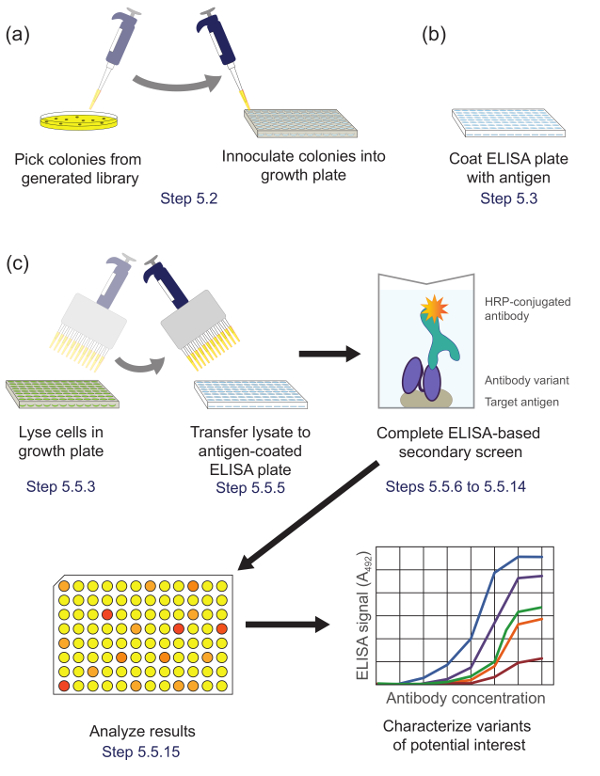
Cribado basado en ELISA Figura 5. secundaria (Paso 5). Variantes (A) Biblioteca de la sub-biblioteca enriquecida durante panning se inoculan en pocillos individuales de una placa de cultivo para el crecimiento y expresión. (B) Un plato de ELISA se recubre con antígeno diana. (C) Las variantes de la biblioteca se seleccionan utilizando la pantalla secundaria basado en ELISA descrito en el protocolo. Tras el análisis de los datos obtenidos a partir de la pantalla secundaria, las variantes de interés se seleccionan y se caracterizan además. Correspondientes pasos del protocolo se indique lo contrario. Por favor, haga clic aquí para ver una versión más grande de esta figura.
- Descongelar un tubo de la sub-biblioteca criticada (de la etapa 4.4.4) y la placa en placas de agar LB. Placa varias dilucionesen concentraciones lo suficientemente bajas como para asegurar colonias individuales (por ejemplo, 10 2 - 10 6 diluciones -fold). Las placas se incuban durante 15 a 18 horas a 37 ° C. Almacenar las placas a 4 ° C o continúe en el paso 5.2.
- Cultura e inducir colonias de la sub-biblioteca criticada. Realizar todas las etapas en condiciones estériles. Utilice una pipeta multicanal para los pasos que implican placas de 96 pocillos.
- Añadir 200 l de LB con 20 mg / ml Cm en cada pocillo de una placa de cultivo de fondo redondo de 96 pocillos.
- Escoja una colonia individual de la placa de agar con una punta de pipeta, coloque la punta en el primer pocillo de la placa de 96 pocillos y agitar suavemente para inocular. Utilizar una punta para cada pozo. Inocular una colonia en cada pocillo. Como control, incluir al menos un control de esterilidad bien con ninguna colonia inoculado.
- Repita los pasos 5.2.1 y 5.2.2 para inocular varias placas de 96 pocillos.
- Colocar las placas de 96 pocillos en un agitador de microplacas a 310 rpm. Incubar a 376; C durante 20 a 24 horas para expresar los scFv.
- Para cada placa de cultivo preparado en el Paso 5.2, capa uno de 96 pocillos de placas de ELISA con el antígeno diana.
- Diluir el antígeno diana purificada hasta una concentración apropiada (por ejemplo, 1 mg / ml a 4 mg / ml) en 1 x PBS para hacer la solución de revestimiento. Hacer 5 ml de solución de recubrimiento para cada placa de 96 pocillos.
NOTA: La concentración apropiada depende del antígeno específico que se utiliza y puede ser necesario ajustar. - Añadir 50 l de la solución de revestimiento a cada pocillo de una placa de poliestireno ELISA claro de 96 pocillos de alta unión. Golpear suavemente la placa sobre la superficie de mesa para asegurarse de que toda la superficie de cada pocillo se recubre. Repita este procedimiento para cada placa. Se incuban las placas a 4 ° CO / N.
- Diluir el antígeno diana purificada hasta una concentración apropiada (por ejemplo, 1 mg / ml a 4 mg / ml) en 1 x PBS para hacer la solución de revestimiento. Hacer 5 ml de solución de recubrimiento para cada placa de 96 pocillos.
- Replicar las colonias de las placas de cultivo de 96 pocillos en placas de agar.
- Coloque un replicador de poliestireno estéril en los pocillos de una placa de cultivo para recoger una pequeña cantidad de jabón lcarné de identidad. Levante con cuidado el replicador y transfieren a una placa de agar LB 15 cm de tal manera que todas las puntas estén en contacto con la placa. Una vez que el líquido se haya transferido, levante el replicador hacia arriba. Repita este procedimiento para cada placa de cultivo.
- Etiqueta de la placa de agar con la orientación correcta de manera que los resultados de la pantalla secundaria en la placa de 96 pocillos se pueden combinar con la colonia replicado correcta en la placa, si se desea una caracterización adicional. Crecer a 37 ° C durante 15 a 18 horas, y luego se almacena a 4 ° C hasta que se necesite.
- Realizar la pantalla secundaria ELISA.
- Preparar la solución de bloqueo al hacer 2% (w / v) de leche en polvo en 1 × PBS. Vaciar la solución de recubrimiento de las placas de ELISA. Añadir 100 l de la solución de bloqueo a cada pocillo. Se incuba a temperatura ambiente durante al menos 2 horas, o bloque de O / N a 4 ° C.
- Preparar el tampón de lavado mediante la adición de polisorbato 20 a una concentración final de 0,05% en 1 x PBS. Hacer 250 ml por placa de ELISA.
- Añadir 20 l de unaconcentrada lisis celular detergente a cada pocillo de la placa de cultivo de fondo redondo, y se incuba la placa de cultivo en un agitador de microplacas a temperatura ambiente durante 15-20 min. Comience la lisis al mismo tiempo que el bloqueo de las placas de ELISA se completa de manera que la lisis y el lavado Paso 5.5.4 se pueden realizar simultáneamente.
- Vaciar la solución de bloqueo de las placas de ELISA. Se lavan las placas de ELISA bloqueados cuatro veces con 200 l de tampón de lavado por pocillo por lavado. Vaciar el buffer de lavado de los pocillos.
- Transferencia de 50 l de cada pocillo de la placa de lisis celular al pocillo correspondiente de la placa de ELISA, utilizando una punta nueva para cada pocillo. Se incuba la placa ELISA a temperatura ambiente durante 1-2 h.
- Preparar la solución de anticuerpos para detectar scFv unidos.
- Utilice una peroxidasa de rábano picante (HRP) conjugado con anticuerpo primario que se une al epítopo etiqueta FLAG fusionada a los scFv de la biblioteca.
- Diluir el anticuerpo a la dilución apropiada para usar en un ELISA (véase el proveedor & #39; s recomendaciones) en 2% (w / v) de leche en polvo en el 0,05% de polisorbato 20 en 1 × PBS. Preparar 5 ml de cada plato.
- Lavar las placas ELISA cuatro veces con tampón de lavado como se describe en el paso 5.5.4.
- Añadir 50 l de la solución de anticuerpo a cada pocillo de la placa de ELISA. Incubar durante 1 a 2 horas a temperatura ambiente.
- Preparar el sustrato de HRP disolviendo o-fenilendiamina diclorhidrato de tabletas (OPD) en H 2 O destilada por el protocolo del fabricante, evitando la luz. Preparar 20 ml por placa de ELISA.
- Preparar 3 MH 2 SO 4 diluyendo H 2 SO 4 concentrado con H 2 O destilada si es necesario. Preparar 5 ml por placa de ELISA.
Precaución: H 2 SO 4 es un ácido fuerte. Asegúrese de usar el equipo de protección personal adecuado. - Lavar las placas ELISA cuatro veces con tampón de lavado, como se describe en el paso 5.5.4.
- Incubar las placas de ELISA con el Substra HRPte.
- Añadir 200 l de sustrato HRP a cada pocillo. Para minimizar la exposición a la luz, añadir el sustrato para una placa de ELISA en un momento, y se envuelve con papel de aluminio antes de proceder a la siguiente placa. Incubar las placas durante 30 a 60 min a TA en la oscuridad.
- Después de la primera 30 min, comprobar las placas para el oscurecimiento del sustrato, y se incuba durante más tiempo si es necesario para visualizar el desarrollo de color.
- Añadir 50 l de 3 MH 2 SO 4 a cada pocillo para detener la reacción. Utilizando una punta diferente para cada pocillo, se mezcla la solución en los pocillos pipeteando suavemente hacia arriba y hacia abajo sin formación de espuma. Por razones de coherencia y para evitar la saturación, añadir el H 2 SO 4 rápidamente y con cuidado para todas las placas de ELISA antes de la mezcla la solución para cada placa.
- Medir la absorbancia de la solución en los pocillos de cada placa a 492 nm usando un lector de placas.
- Analizar los datos de absorbancia para identificar variantes de scFv que exhibprometiendo que las señales de unión y caracterizar estos scFv prometedores. Seleccionar los scFv que exhiben señales de absorbancia superior a la señal de fondo y superior a la media de la señal en cada plato.
NOTA: El nivel de absorbancia dependerá de las propiedades del antígeno y el anticuerpo anti-FLAG utilizadas, junto con la fuerza de las variantes scFv que fueron aislados en el cribado.
Resultados
El plegamiento de la proteína mecanismo de control de calidad intracelular de la vía de Tat en E. coli limita el transporte a través de la membrana interna de la célula a las proteínas que son bien plegadas en el ambiente citoplasmático de reducción. Por la sobreexpresión de una fusión de un scFv a la secuencia señal ssTorA (la secuencia señal de la proteína de TORA, que es, naturalmente, transportado por la vía de Tat 20), la translocación está estancado, lo que resulta en la pantalla de los scFv en la membrana interna 19. Después de la interrupción enzimática de la membrana externa, los anticuerpos que se muestran se hacen disponibles para el cribado para la actividad de unión al antígeno. La capacidad de tomar ventaja de la vía de Tat para la visualización de scFv se demostró por Karlsson et al. 19 (Figura 6). Los anticuerpos scFv y scFv13 scFv13.R4 se fusionaron a ya sea la secuencia de ssTorA nativo o un ssTorA modificada que carece de la par de residuos de arginina-arginina reconocido porla vía de Tat. scFv13.R4 fue diseñado por Martineau et al. de scFv13 a través de cuatro rondas de evolución dirigida y es conocido para plegar bien en el citoplasma 9. Este scFv se muestra en la membrana interna, pero sólo cuando se expresa como una fusión con la secuencia señal nativa ssTorA (Figura 6). Por el contrario, scFv13 no está bien doblada citoplásmicamente 9, por lo que no se muestra bien en la membrana interna, independientemente de la secuencia señal a la que se fusiona. Además, si los scFv se expresaron en células que carecían de la proteína tatC, un componente vital de la maquinaria Tat 20,28, no se observó la pantalla, mostrando la importancia del vínculo entre la pantalla del centro de la membrana y la vía Tat. Estos resultados demuestran que sólo las proteínas que contienen el péptido señal Tat y que se pliegan correctamente en el citoplasma se muestran en la membrana interna, que permite el transporte a través de la vía de Tat para funcionar como una pantalla para fol intracelulartimbre.
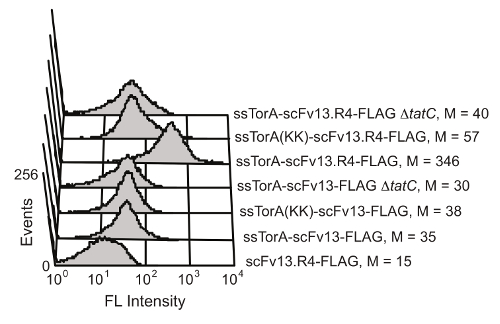
Figura 6. Detección de scFv que se muestran en la membrana interna. La citometría de flujo se realizó un análisis para detectar la pantalla de mal plegada scFv13 y bien plegada scFv13.R4 en la membrana interna. scFv se fusionaron a ssTorA nativo o ssTorA (KK), donde el par Arg-Arg en la secuencia ssTorA se modificó para Lys-Lys. Las etiquetas de epítopo FLAG C-terminal de los scFv se detectaron con un isotiocianato de fluoresceína (FITC) conjugado con anticuerpo anti-FLAG. Las células sin la proteína tatC (ΔtatC) y ssTorA-scFv13 sin la etiqueta FLAG se probaron como controles. M indica el valor mediana de la fluorescencia. Reproducido de la referencia 19 con permiso. Haga clic aquí para ver una versión más grande de esta figura.
pantalla interna de la membrana puede aislar con éxito anticuerpos scFv con altos niveles de afinidad para una proteína diana y los altos niveles de solubilidad citoplasmática. Además, las siguientes rondas de evolución dirigida utilizando visualización membrana interna mejoran las características de anticuerpos 19. Para demostrar esto, una biblioteca de PCR propensa a error sobre la base de scFv13, que tiene un bajo nivel de afinidad de unión para β-galactosidasa, se panorámica contra el antígeno diana β-galactosidasa utilizando la pantalla y paneo método descrito en el protocolo. scFv 1-4 fue aislado después de una ronda de mutagénesis y la panorámica, y exhibió mayor afinidad de unión a beta-galactosidasa de scFv13 (Figura 7A) y un mayor nivel de solubilidad citoplásmica (Figura 7B).
Una nueva biblioteca, basado en scFv 1-4, se hizo usando PCR propensa a error, y la panorámica de esta biblioteca de segunda generación contraβ-galactosidasa se realizó utilizando una modificación del protocolo descrito. El paneo contra la β-galactosidasa para la segunda ronda de la evolución se llevó a cabo en presencia de purificado, scFv soluble 14 como un competidor para mejorar la probabilidad de aislar clones con mayor afinidad que scFv 1-4. Después de esta segunda ronda de mutagénesis y la panorámica, scFv y scFv 2-1 2-3 se aislaron utilizando la selección secundaria basados en ELISA. Estos scFv no sólo exhiben mayor afinidad de unión para la β-galactosidasa de scFv13, pero también mostraron una mejor unión de la primera ronda del clon de scFv 1-4. scFv 2-1 exhibió β-galactosidasa de unión comparable a la de scFv13.R4 (Figura 7A). scFv 2-3 también muestra un aumento adicional de la solubilidad citoplásmica en comparación con scFv 14, destacando la ingeniería simultánea de solubilidad y de unión al antígeno. Desde afinidad y la expresión soluble de los scFv son examinados para detectar simultáneamente, es posible que un scFv seleccionado ha modsolubilidad eRate pero de alta versa vinculante o vicio. Por ejemplo, scFv 2-1 tiene soluble expresión más baja que scFv 2-3, pero exhibe una mayor afinidad de unión a la ß-galactosidasa.

Figura 7. Objetivo de unión y la expresión citoplasmática de scFv variantes aislado utilizando la pantalla del centro de la membrana. ScFv (A) se expresaron en el citoplasma de E. células de E. coli (por ejemplo., sin la secuencia señal ssTorA) con una hexahistidina tag (6 x His) y se purificó usando ácido spin-columnas de níquel-nitrilotriacético. La unión de los scFv purificados a ß-galactosidasa se midió con un ELISA. scFv purificados se cargaron en placas de ELISA de β-galactosidasa-revestido, y se detectaron los scFv unidos con un anticuerpo anti-6 x His. Los datos son un promedio de seis réplicas, y la barra de error muestra el error estándar de la media.(B) Las fracciones soluble e insoluble de los lisados celulares de células que expresan los scFv citoplásmicamente fueron analizados por una transferencia Western sondada con un anticuerpo anti-6 x His. La concentración total de proteína se utilizó para normalizar la carga de las muestras. Reproducido (A) y adaptado (B) de la referencia 19 con permiso. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Ingeniería de anticuerpos para la actividad citoplasmática es una tarea difícil debido al medio reductor del citoplasma, lo que impide la formación de enlaces disulfuro estabilizador 6,7. Esto hace que la mayoría de los anticuerpos para ser citoplásmicamente inactiva a no ser que se han diseñado para la estabilidad y la solubilidad en el citoplasma, además de estar diseñado para la afinidad de unión. Los métodos existentes de presentación de fagos, presentación de superficie bacteriana, y métodos de presentación en superficie de levadura todo el uso de la vía secretora 14-16 para la visualización de anticuerpos manipulados, pero estos métodos no tienen medios para diseñar plegado intracelular. Los anticuerpos manipulados utilizando la pantalla-membrana interna han mejorado la estabilidad y la solubilidad citoplásmica debido a que el control de calidad de plegado de la vía Tat evita la translocación de anticuerpos que están mal plegadas e inestables en el citoplasma. Este método simplifica el proceso iterativo de anticuerpos intracelulares de ingeniería para la afinidad de unand solubilidad, ya que las dos propiedades están diseñados en un solo paso. Aunque este método fue diseñado para la ingeniería de anticuerpos con una solubilidad en el entorno intracelular reductor, también se podría aplicar a los anticuerpos de ingeniería para funcionar en condiciones no reductoras, ya que las proteínas por ingeniería genética usando este método mantienen su plegado en el ambiente oxidante del periplasma.
Aunque esta técnica simplifica el proceso de ingeniería de anticuerpos con alta afinidad y alta solubilidad citoplasmática, varias limitaciones son importantes a considerar cuando se utiliza este protocolo. Al analizar las señales de ELISA pantalla secundaria para identificar variantes de scFv prometedor, el umbral para discernir entre variantes potencialmente interesantes y los que no pueden exhibir adecuada de unión al antígeno no es probable que sea evidente hasta después de varios clones se han caracterizado más. Es importante buscar unión mejorada sobre el anticuerpo parental; sin embargo,una señal anormalmente alta podría ser indicativo de avidez 29 o los efectos de agregación 30, un reto que no es única para el enfoque de detección de visualización de la membrana interior. Una limitación clave a recordar cuando se utiliza este protocolo es la incapacidad para recuperar esferoplastos después del encuadre, ya que son inviables (datos no publicados). Esto requiere las etapas de amplificación de ADN y de transformación para recuperar los plásmidos que codifican el anticuerpo.
Varios pasos críticos del protocolo permiten la ingeniería simultánea de plegado y unión de los anticuerpos. Para el cribado para tener éxito, la biblioteca de scFv se proyectará debe expresarse como una fusión con el péptido señal ssTorA. Sin esta secuencia, los anticuerpos no serán dirigidos a la vía Tat y por lo tanto no se translocan al periplasma 19. Además, es imperativo que una etiqueta de epítopo C-terminal se fusiona a los anticuerpos para permitir la detección de los anticuerpos que se muestran en el binding ensayos. Claramente, la E. coli cepa utilizada para expresar los scFv además, debe tener la maquinaria vía Tat es necesario, pero esto es cierto de la E. comúnmente utilizado cepas de E. coli.
Las modificaciones de este protocolo son posibles para mejorar su potencial para aislar anticuerpos con las características deseadas. Un paso panorámica de sustracción puede ser completado antes de la toma panorámica contra el antígeno diana para agotar la biblioteca de scFv de constituyentes no deseados. Los esferoplastos de la biblioteca se pueden incubar con perlas magnéticas recubiertas con BCCP solo o revestido con una proteína no deseada, y de los esferoplastos que se unen a las perlas pueden ser descartadas antes de cribado de los esferoplastos no unidos restantes para la unión a la diana deseada. Como se mencionó en los resultados representativos, un método para mejorar la afinidad de un scFv aislado es incluir un competidor soluble en la reacción de panning para competir con los scFv que se muestran en los esferoplastos. Debido a que el borrador solublesetitor es una proteína purificada, sin ADN se amplificó a partir de ella, de modo que sólo las secuencias de los scFv que se muestran en los esferoplastos se recuperarán en la reacción de PCR. Además, este método podría extenderse a la ingeniería de otros tipos de anticuerpos o a proteínas de unión que no son anticuerpos.
E. pantalla del centro de la membrana coli es una potente plataforma de ingeniería anticuerpos con alta afinidad y altos niveles de solubilidad intracelular. Este método es particularmente adecuado para la ingeniería eficiente de anticuerpos diseñados para funcionar en el medio ambiente intracelular. Estos anticuerpos intracelulares que ya se están estudiando como agentes terapéuticos potenciales en varios campos, incluyendo las enfermedades neurodegenerativas, cáncer y las infecciones virales 31. Esta técnica permitirá un uso más generalizado de anticuerpos intracelulares como herramientas para la investigación y la medicina en estos campos y cualquier otro campo en el que se desea estudiar es un objetivo proteína in situ.
Divulgaciones
The authors declare that they have no competing financial interests.
Agradecimientos
We thank Tomer Zohar for work on scFv screening and characterization assays. A portion of this work was supported by award number F32CA150622 from the National Cancer Institute (to AJK). The content is solely the responsibility of the authors and does not necessarily represent the official views of the National Cancer Institute or the National Institutes of Health.
Materiales
| Name | Company | Catalog Number | Comments |
| scFv library | Varies | A suitable scFv library should be obtained from a commercial or academic source. | |
| MC4100 E. coli cells | Coli Genetic Stock Center | 6152 | Cells need to be chemically competent or electrocompetent, depending on the selected transformation method. |
| Glycerol | Fisher Scientific | BP229-4 | |
| Difco dehydrated culture media LB Broth, Miller (Luria-Bertani) | BD | 244610 | |
| Chloramphenicol (Cm) | Fisher Scientific | BP904-100 | |
| Sodium chloride (NaCl) | Fisher Scientific | BP358-1 | |
| Potassium chloride (KCl) | Fisher Scientific | BP366-500 | |
| Sodium phosphate, dibasic (Na2HPO4) | Fisher Scientific | BP332-500 | |
| Potassium phosphate, monobasic (KH2PO4) | Fisher Scientific | BP362-500 | |
| Bovine serum albumin (BSA) | Fisher Scientific | BP9706-100 | |
| Sucrose | Fisher Scientific | BP220-1 | |
| Tris base | Fisher Scientific | BP1521 | |
| Ethylenediaminetetraacetic acid (EDTA), 0.5 M | Fisher Scientific | BP2482-500 | |
| Magnesium chloride (MgCl2) | Fisher Scientific | BP214-500 | |
| Lysozyme | Sigma Aldrich | L3790-10X1ML | |
| Vortex mixer | VWR | 97043-564 | |
| Bicine | Fisher Scientific | BP2646100 | |
| D-Biotin | Fisher Scientific | BP232-1 | |
| Isopropyl β-D-1-thiogalactopyranoside | Fisher Scientific | BP1755-1 | |
| BugBuster Master Mix (cell lysis detergent) | EMD Millipore | 71456 | |
| Vivaspin 2 MWCO, 3,000 daltons | GE Healthcare Sciences | 28932240 | |
| Target antigen | Varies | N/A | Purified target antigen may be purchased or produced/purified. |
| Dynabeads MyOne Streptavidin T1 | Invitrogen | 65601 | |
| Dynamag-2 magnet | Invitrogen | 12321D | |
| Tube rotator | VWR | 13916-822 | |
| PCR primers | IDT | N/A | Primer sequences are as described in the protocol. |
| 10x T4 DNA ligase reaction buffer | New England BioLabs | B0202S | |
| T4 Polynucelotide kinase (PNK) | New England BioLabs | M0201S | Make sure the T4 ligase buffer used in the primer phosphorylation reaction contains 1 mM ATP. |
| 5x Phusion HF buffer pack | New England BioLabs | B0518S | |
| Deoxynucleotide (dNTP) solution mix, 10 mM each dNTP | New England BioLabs | N0447L | |
| Phusion DNA polymerase | New England BioLabs | M0530S | Other high-fidelity polymerases may be used as an alternative, but the annealing temperature in Table 3 must be adjusted. |
| C1000 Touch thermal cycler with dual 48/48 fast reaction module | Bio-Rad | 185-1148 | |
| Agarose | Promega | V3121 | |
| SYBR Safe DNA gel stain | Invitrogen | S33102 | |
| Wizard SV gel and PCR clean-up system | Promega | A9281 | |
| T4 DNA ligase | New England BioLabs | M0202S | |
| Microdialysis membrane filter | EMD Millipore | VSWP04700 | |
| Agar | BD | 214030 | |
| 96-well polystyrene round-bottom cell culture plates | VWR | 10062-902 | |
| Costar general polystyrene assay plate lids | Corning | 3931 | |
| Microtitre plate shaker | VWR | 12620-926 | |
| Costar 96 well EIA/RIA Easy Wash clear flat bottom polystyrene high bind microplate | Corning | 3369 | |
| Bel-blotter polycarbonate 96-well replicating tool | Bel-Art Products | 378760002 | |
| Instant nonfat dry milk | Quality Biological | A614-1000 | |
| Tween 20 (polysorbate 20) | Fisher Scientific | BP337-500 | |
| PopCulture reagent (concentrated cell lysis detergent) | EMD Millipore | 71092-3 | |
| Monoclonal ANTI-FLAG M2-Peroxidase(HRP) antibody produced in mouse | Sigma Aldrich | A8592 | |
| SigmaFast OPD | Sigma Aldrich | P9187-50SET | |
| Sulfuric acid (H2SO4), 10 N solution | Fisher Scientific | SA200-1 | |
| Reynolds Wrap aluminum foil | VWR | 89079-075 | |
| BioTek Epoch microplate spectrophotometer | Fisher Scientific | 11120570 |
Referencias
- Biocca, S., Pierandrei-Amaldi, P., Campioni, N., Cattaneo, A. Intracellular immunization with cytosolic recombinant antibodies. Biotechnology (NY). 12 (4), 396-399 (1994).
- Chen, S. Y., Bagley, J., Marasco, W. A. Intracellular antibodies as a new class of therapeutic molecules for gene-therapy. Hum. Gene Ther. 5 (5), 595-601 (2008).
- Gargano, N., Biocca, S., Bradbury, A., Cattaneo, A. Human recombinant antibody fragments neutralizing human immunodeficiency virus type 1 reverse transcriptase provide an experimental basis for the structural classification of the DNA polymerase family. J Virol. 70 (11), 7706-7712 (1996).
- Mhashilkar, A. M., et al. Inhibition of HIV-1 Tat-mediated LTR transactivation and HIV-1 infection by anti-Tat single chain intrabodies. Embo J. 14 (7), 1542-1551 (1995).
- Strube, R. W., Chen, S. Y. Characterization of anti-cyclin E single-chain Fv antibodies and intrabodies in breast cancer cells: enhanced intracellular stability of novel sFv-F-c intrabodies. J. Immunol. Meth. 263 (1-2), 149-197 (2002).
- Mössner, E., Koch, H., Plückthun, A. Fast selection of antibodies without antigen purification: adaptation of the protein fragment complementation assay to select antigen-antibody pairs. J. Mol. Biol. 308 (2), 115-122 (2001).
- Wörn, A., et al. Correlation between in vitro stability and in vivo performance of anti-GCN4 intrabodies as cytoplasmic inhibitors. J Biol Chem. 275 (4), 2795-2803 (2000).
- Knappik, A., Plückthun, A. Engineered turns of a recombinant antibody improve its in vivo folding. Protein Eng. 8 (1), 81-89 (1995).
- Martineau, P., Jones, P., Winter, G. Expression of an antibody fragment at high levels in the bacterial cytoplasm. J Mol Biol. 280 (1), 117-127 (1998).
- Steipe, B., Schiller, B., Plückthun, A., Steinbacher, S. Sequence statistics reliably predict stabilizing mutations in a protein domain. J. Mol. Biol. 240 (3), 188-192 (1994).
- Daugherty, P. S. Protein engineering with bacterial display. Curr Opin Struct Biol. 17 (4), 474-480 (2007).
- Lener, M., et al. Diverting a protein from its cellular location by intracellular antibodies. Eur. J. Biochem. 267 (4), 1196-1205 (2000).
- Lynch, S. M., Zhou, C., Messer, A. An scFv intrabody against the nonamyloid component of α-synuclein reduces intracellular aggregation and toxicity. J. Mol. Biol. 377 (1), 136-147 (2008).
- Gai, S. A., Wittrup, K. D. Yeast surface display for protein engineering and characterization. Curr. Opin. Struc. Biol. 17 (4), 467-473 (2007).
- Kieke, M. C., et al. Selection of functional T cell receptor mutants from a yeast surface-display library. Proc. Natl. Acad. Sci. USA. 96 (10), 5651-5656 (1999).
- Steiner, D., Forrer, P., Stumpp, M. T., Pluckthun, A. Signal sequences directing cotranslational translocation expand the range of proteins amenable to phage display. Nat. Biotechnol. 24, 823-831 (2006).
- Pugsley, A. P. The complete general secretory pathway in gram-negative bacteria. Microbiol. Rev. 57 (1), 50-108 (1993).
- Rapoza, M. P., Webster, R. E. The filamentous bacteriophage assembly proteins require the bacterial SecA protein for correct localization to the membrane. J. Bacteriol. 175 (6), 1856-1859 (1993).
- Karlsson, A. J., et al. Engineering antibody fitness and function using membrane-anchored display of correctly folded proteins. J. Molec. Biol. 416 (1), 94-107 (2012).
- DeLisa, M. P., Tullman, D., Georgiou, G. Folding quality control in the export of proteins by the bacterial twin-arginine translocation pathway. Proc Natl Acad Sci U S A. 100 (10), 6115-6120 (2003).
- Fisher, A. C., Kim, W., DeLisa, M. P. Genetic selection for protein solubility enabled by the folding quality control feature of the twin-arginine translocation pathway. Protein Sci. 15 (3), 449-458 (2006).
- Maynard, J., Georgiou, G. Antibody engineering. Annu Rev Biomed Eng. 2, 339-376 (2000).
- Green, M. R., Sambrook, J. . Molecular Cloning: A Laboratory Manual. 1, (2012).
- Milo, R., Jorgensen, P., Moran, U., Weber, G., Springer, M. BioNumbers--the database of key numbers in molecular and cell biology. Nucleic Acids Res. 38, D750-D753 (2010).
- Hermanson, G. T. . Bioconjugate Techniques. , (2013).
- Tayapiwatana, C., Chotpadiwetkul, R., Kasinrerk, W. A novel approach using streptavidin magnetic bead-sorted in vivo biotinylated survivin for monoclonal antibody production. J Immunol Methods. 317 (1-2), 1-11 (2006).
- Zhu, G., Song, L., Lippard, S. J. Visualizing inhibition of nucleosome mobility and transcription by cisplatin-DNA interstrand crosslinks in live mammalian cells. Cancer Res. 73 (14), 4451-4460 (2013).
- Bogsch, E. G., et al. An essential component of a novel bacterial protein export system with homologues in plastids and mitochondria. J. Biol. Chem. 273, 18003-18006 (1998).
- Julian, M. C., et al. Co-evolution of affinity and stability of grafted amyloid-motif domain antibodies. Protein Eng. Des. Sel. 28 (10), 339-350 (2015).
- Garber, K. Bispecific antibodies rise again. Nat. Rev. Drug. Discov. 13 (11), 799-801 (2014).
- Marschall, A. L., Dübel, S., Böldicke, T. Specific in vivo knockdown of protein function by intrabodies. MAbs. 7 (6), 1010-1035 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados