Method Article
Ordenamiento temporal de los datos de expresión dinámica de la expresión espacial detallada Mapas
En este artículo
Resumen
The segmentation clock drives oscillatory gene expression across the pre-somitic mesoderm (PSM). Dynamic Notch activity is key to this process. We use imaging and computational analyses to extract temporal dynamics from spatial expression data to demonstrate that Delta ligand and Notch receptor expression oscillate in the vertebrate PSM.
Resumen
During somitogenesis, pairs of epithelial somites form in a progressive manner, budding off from the anterior end of the pre-somitic mesoderm (PSM) with a strict species-specific periodicity. The periodicity of the process is regulated by a molecular oscillator, known as the "segmentation clock," acting in the PSM cells. This clock drives the oscillatory patterns of gene expression across the PSM in a posterior-anterior direction. These so-called clock genes are key components of three signaling pathways: Wnt, Notch, and fibroblast growth factor (FGF). In addition, Notch signaling is essential for synchronizing intracellular oscillations in neighboring cells. We recently gained insight into how this may be mechanistically regulated. Upon ligand activation, the Notch receptor is cleaved, releasing the intracellular domain (NICD), which moves to the nucleus and regulates gene expression. NICD is highly labile, and its phosphorylation-dependent turnover acts to restrict Notch signaling. The profile of NICD production (and degradation) in the PSM is known to be oscillatory and to resemble that of a clock gene. We recently reported that both the Notch receptor and the Delta ligand, which mediate intercellular coupling, themselves exhibit dynamic expression at both the mRNA and protein levels. In this article, we describe the sensitive detection methods and detailed image analysis tools that we used, in combination with the computational modeling that we designed, to extract and overlay expression data from distinct points in the expression cycle. This allowed us to construct a spatio-temporal picture of the dynamic expression profile for the receptor, the ligand, and the Notch target clock genes throughout an oscillation cycle. Here, we describe the protocols used to generate and culture the PSM explants, as well as the procedure to stain for the mRNA or protein. We also explain how the confocal images were subsequently analyzed and temporally ordered computationally to generate ordered sequences of clock expression snapshots, hereafter defined as "kymographs," for the visualization of the spatiotemporal expression of Delta-like1 (Dll1) and Notch1 throughout the PSM.
Introducción
Somitas son los primeros segmentos formados en el eje del cuerpo alargándose en el desarrollo de especies de vertebrados y son los precursores de la columna vertebral, las costillas, y dermis tejido, así como de los músculos y células endoteliales. Durante somitogenesis, somitas epiteliales forman a partir del mesodermo no segmentado presomitic (PSM) (revisado en referencia 1). Este proceso es regulado por el "reloj segmentación", que consiste en una red de genes oscilatorios y proteínas, la mayoría pertenece a la ruta de señalización Notch. El reloj de la segmentación consiste en varios circuitos de retroalimentación negativa, que permiten la producción pulsátil de la actividad de Notch dentro de una sola célula 2 (revisado en las referencias 3 - 6). Mientras que el método intracelular de oscilación está bien caracterizado, todavía es en gran medida desconocido cómo estas oscilaciones se coordinan a través del tejido PSM. Se ha demostrado recientemente, tanto a través de estudios experimentales y teóricos, que estas oscilaciones son essential al proceso de somitogenesis y que la vía de Notch desempeña un papel crucial en el proceso de tanto la segmentación y la expresión de genes oscilatorio 7, 8. Sin embargo, se ha informado ampliamente de que receptor Notch 1 (Notch1) y el ligando Delta-como (DLL) -1 tener gradientes estáticos en el PSM 9, 10, 11.
La hipótesis de que las oscilaciones Notch dependientes del reloj PSM segmentación dependen de la activación periódica del principal receptor de vía de Notch y ligando, Notch 1 y Dll1, respectivamente, a través del PSM de ratón. Las conclusiones de los estudios anteriores que informaron un gradiente rostral-caudal estática de estas proteínas se debieron, predecimos, a una falta de sensibilidad en las técnicas de inmunotinción. Eran, por tanto, incapaces de detectar las fluctuaciones de bajo nivel de Dll1 y Notch1 en el PSM caudal.
have ideó un método para examinar más de cerca estos factores, la combinación de datos experimentales con modelos matemáticos para predecir un mecanismo por el cual las oscilaciones de las proteínas de los componentes del reloj se coordinan a través del PSM 12.
El objetivo general de este método consiste en detectar y cuantificar de bajo nivel, la expresión de proteínas dinámica del PSM y trazar los perfiles de expresión de proteínas de interés de acuerdo con la expresión del gen reloj conocido, lunáticos (Lfng). Desde un ciclo del reloj de la segmentación en el embrión de ratón toma 2 h para completar, se requieren varias muestras para construir un perfil de espacio-temporal completa de la expresión de proteínas Dll1 y Notch1 durante una oscilación Lfng en el PSM. Así, hemos desarrollado este protocolo para permitir la detección de alto rendimiento de la expresión de proteínas de bajo nivel en todo el montaje, los explantes PSM contralateral. Sin embargo, esta técnica también puede ser útil para estudios tsombrero objetivo de caracterizar la dinámica de proteínas de bajo nivel dentro de cualquier tejido embrionario que se puede dividir en mitades contralateral.
Protocolo
Todos los experimentos se llevaron a cabo dentro del proyecto número de licencia 6004219 en estricta adhesión a los de 1986 y los Códigos del Ministerio del Interior del Reino Unido de prácticas para la utilización de animales en procedimientos científicos Animales (Procedimientos Científicos).
1. PSM explante Disección
- Obtener el tejido de la cola a partir de embriones producidos por el apareamiento temporizada de tipo salvaje (CD1) 13 ratones. En resumen, en el día embrionario (E) 10,5, la eutanasia el ratón donante embarazada en una cámara de dióxido de carbono. Se recoge el cuerno uterino y colocarlo en 1 x solución salina estéril tamponada con fosfato (PBS), de conformidad con los procedimientos de licencia del Ministerio del Interior o normas locales equivalentes. Transferir el cuerno uterino de una placa de cultivo de tejido que contenía fresca, PBS estéril. Realizar todos los pasos posteriores de disección en esta solución.
- Bajo un microscopio estereoscópico, cortar la membrana muscular gruesa de la trompa uterina con tijeras curvas y extraer cada embrión cuidadosamente usando pinzas finas. Cuídatepara asegurar que el tejido de la cola no está dañado en este proceso. Usando tijeras curvas y unas pinzas finas, diseccionar el saco amniótico de cada embrión, teniendo cuidado de no dañar el embrión.
- Utilice una aguja quirúrgica o tijeras curvas para cosechar el tejido de la cola de cada embrión mediante la reducción de la parte posterior del embrión a las yemas de las extremidades traseras.
- Equilibrar el ventral del lado del tejido de la cola hacia abajo con ambas pinzas y una aguja. Generar pares de explantes PSM de cada cola del embrión mediante la disección del tejido de la cola en dos mitades a lo largo de la línea media; realizar un movimiento de balanceo suave con una aguja. Asegúrese de que el tubo, notocorda y el tejido neuronal PSM se dividen por igual entre los dos explantes.
- Pipetear cada explante PSM contralateral en la parte inferior de un cultivo de plástico tapa de la placa de 35 mm en un pequeño volumen de pre-calentado (37 ° C) medio de cultivo (DMEM-F12 + 0,1% sustituto de L-glutamina suplementado con suero de ternera fetal al 10% , 10 nM bFGF humano, y 1% de penicilina / estreptomicina).
- Coloque el plato en la parte superior de la tapa y rápidamente invertir de modo que el tejido PSM se suspende de la tapa en una "gota colgante" de medio. La cultura de los explantes de PSM en una cámara húmeda a 37 ° C durante 1 - 2 h.
- pares de transferencia de explantes PSM a los pocillos individuales de una placa de cultivo tisular de 24 pocillos. Incubar en 4% de paraformaldehído en PBS durante 1 h a temperatura ambiente (RT) o 4 ° C durante la noche (O / N). PRECAUCIÓN: El paraformaldehído es tóxico, y las medidas de seguridad apropiadas se debe tomar cuando se trabaja con esta solución.
NOTA: Realizar todos los pasos de lavado e incubación posterior en una placa de cultivo de tejidos de 24 pocillos. - Lavar los pocillos de muestras en PBS a temperatura ambiente en una plataforma oscilante, usando una pipeta fina de plástico Pasteur para el intercambio de la solución de PBS de las muestras de PBS fresco 3 - 4 veces. Procesar un explante PSM de cada par mediante inmunohistoquímica (paso 2) y el otro usando hibridación fluorescente in situ para un gen conocido reloj (step 3).
2. Inmunohistoquímica de PSM explantes
- Lavar un explante PSM de cada par de embriones generados en el paso 1 en el 2% de Triton X-100 en PBS durante 1 hora a temperatura ambiente en una plataforma oscilante, y luego enjuague muestras brevemente en PBS. Sustituir el PBS en las muestras con solución de bloqueo (albúmina de 2% de suero bovino (BSA) y 10% de suero normal de cabra (NGS) en PBS + 0,1 Tween-20%) y se incuba O / N a 4 ° C en una plataforma oscilante.
NOTA: Todos los lavados posteriores y las etapas de incubación de esta sección deben llevarse a cabo a temperatura ambiente en una plataforma oscilante, a menos que se indique lo contrario. soluciones de lavado se pueden cambiar fácilmente con un plástico o vidrio pipeta Pasteur de punta fina. - Diluir el anticuerpo / anticuerpos primarios deseados en tampón de trabajo (0,1% de BSA, 0,3% NGS, y 0,2% de Triton X-100 en PBS). En este ejemplo, diluir los anticuerpos Dll1 y Notch1 1:25 en tampón de trabajo.
Se requerirá de optimización para determinar el factor de dilución apropiado requerido en thi: NOTAs paso si se utilizan anticuerpos alternativos. - Incubar los explantes en la solución de anticuerpo durante 3 - 5 días a 4 ° C en una plataforma oscilante. Asegúrese de incluir algunas muestras con tampón de trabajo que contenía ningún anticuerpo primario para actuar como controles de anticuerpos secundarios.
- Recuperar la solución de anticuerpo primario en un tubo de almacenamiento de 1,5 ml con una pipeta y almacenarlo a 4 ° C.
NOTA: Recuperado anticuerpo primario se puede utilizar varias veces, dependiendo del anticuerpo utilizado. - Realizar 2 lavados de las muestras de 5 - 10 min cada uno en PBS, seguido de 3 lavados de 10 min cada uno en 2% de Triton X-100 en PBS a RT en una plataforma oscilante.
- Diluir anticuerpo / anticuerpos secundarios marcados con fluorescencia (epítopo corresponde con el anticuerpo primario / anticuerpos utilizados) en tampón de trabajo. Opcionalmente, añadir 20 g / ml de Hoechst 33342 a esta solución para contrateñir los núcleos.
NOTA: Optimización puede ser necesaria para determinar el factor de dilución apropiado requerido en este paso. En este correojemplo, un factor de dilución de 1: se utiliza típicamente 400. - Centrifugar la solución de anticuerpo secundario durante 10 min a 16 xg para evitar la formación de agregados de anticuerpos. Añadir 250 - 500 l de la solución de anticuerpo secundario a cada pocillo de muestra, teniendo cuidado de no utilizar los últimos microlitros de la solución, que puede contener agregados de anticuerpos.
- Cubrir la placa de la muestra con papel de aluminio para minimizar la exposición a la luz e incubar las muestras en la solución de anticuerpo secundario durante 3 - 5 días a 4 ° C en la oscuridad.
- Antes de muestrear de montaje, lavar las muestras dos veces durante 10 min cada uno en 0,1% de Tween-20 en PBS (PBST) y una vez por 5 min en PBS a RT en una plataforma oscilante (ver paso 4).
3. La hibridación in situ fluorescente (FISH) de PSM explantes
- Si se almacena en un recipiente alternativa, la transferencia de los restantes explantes PSM contralaterales a los pocillos individuales de una placa de cultivo de tejidos de 24 pocillos.
- Se lavan las muestras para10 min en etanol al 50% en PBST, y luego realizar 2 lavados de 10 minutos cada una en etanol al 100% en una plataforma oscilante a temperatura ambiente para deshidratar el tejido.
NOTA: Todos los lavados posteriores y las etapas de incubación de esta sección deben llevarse a cabo a temperatura ambiente en una plataforma oscilante, a menos que se indique lo contrario. - Rehidratar el tejido mediante el lavado de 10 min en etanol al 50% en PBST, seguido de lavado dos veces durante 5 min cada uno en PBST.
NOTA: Los pasos 3.2 y 3.3 son medidas de fijación necesarios que se requieren para este protocolo y no se puede omitir. - Se incuban las muestras con 10 mg / ml de proteinasa K en 0,1% de Tween-20 en PBS (PBST) durante 5 min sin agitación. eliminar rápidamente la proteinasa K y enjuagar las muestras brevemente con PBST antes de post-fijación del tejido durante 30 minutos en 4% de formaldehído + 0,1% de glutaraldehído en PBST. PRECAUCIÓN: Tanto el formaldehído y el glutaraldehído son tóxicos, y las medidas de seguridad apropiadas se deben tomar cuando se trabaja con estas soluciones.
NOTA: La siguiente lavadoy que involucran las etapas de incubación de 50% y 100% mezclas de hibridación (pasos 3.6 a 3.9) deben llevarse a cabo sin agitación. - Después de lavar las muestras dos veces durante 10 min cada uno en PBST, lavar las muestras de una vez en 50% de mezcla de hibridación (adecuado para sondas intrónicas: 50% de formamida, 5x citrato de solución salina de sodio (SSC), EDTA 5 mM, 50 mg / ml tRNA, 0,2% de Tween-20, 0,1% de SDS, y 100 mg / ml de heparina) en PBST preparan a TA. Se incuban las muestras en esta solución durante 10 min a 65 ° C sin agitación.
- Enjuagar las muestras dos veces con pre-calentado (65 ° C) mezcla de hibridación antes de la incubación de las muestras en mezcla de hibridación durante ≥ 2 h (hasta 48 h) a 65 ° C (tiempos de incubación más largos mejoran el contraste de señal a ruido resultante) . Retire la mezcla de hibridación de la etapa anterior, y reemplazarlo con 0,25 - 0,5 mL de pre-calentado (65 ° C) mezcla de hibridación que contiene un digoxigenina (DIG) marcado con sonda de ARN antisentido contra un componente de reloj segmentación conocido.
NOE: Por ejemplo, una franja loca intrónica (Lfng (i)) de la sonda se utilizó a una concentración de 20 l / ml para detectar mRNA Lfng naciente. La dilución utilizado en esta etapa es la sonda dependiente y requerirá la optimización. - Sellar la placa con cinta adhesiva para evitar la evaporación e incubar las muestras en la solución de la sonda durante dos noches a 65 ° C.
- Utilizando una pipeta Pasteur de plástico de punta fina, recuperar la sonda para su reutilización y almacenarlo a 20 ° C. Enjuagar las muestras dos veces con pre-calentado (65 ° C) mezcla post-hibridación (50% formamida, 0,2% de Tween-20, y 1x SSC) antes de lavar las muestras dos veces más durante 20 minutos cada uno a 65 ° C en pre- calentado mezcla de post-hibridación.
- Lavar las muestras durante 15 minutos a 65 ° C en mezcla de hibridación precalentada 50% en 0,1% de Tween-20 en solución salina tamponada con Tris (TBST). Enjuagar las muestras dos veces con TBST antes de lavar durante 30 minutos a RT en TBST en una plataforma oscilante.
- Preincubarlos los explantes en blosolución cking (TBST + 2% de reactivo de bloqueo tampón (BBR) + suero de cabra tratada térmicamente 20%) durante un mínimo de 2 h. Reemplazar esta solución con solución de bloqueo fresco que contiene una dilución 1: 200 de peroxidasa de rábano (HRP) conjugado con anticuerpo anti-digoxigenina. Se incuban las muestras de O / N a 4 ° C.
- Después de la incubación del anticuerpo, enjuagar las muestras 3 veces con TBST a temperatura ambiente y transferirlos a pocillos individuales de una nueva placa de cultivo de tejidos de 24 pocillos. Lavar los explantes con TBST 3 veces durante 1 hora cada uno.
- En este punto, la transferencia de las muestras en tubos de 0.5 ml de almacenamiento o los pocillos individuales de una placa de cultivo tisular de 48 pocillos para reducir el volumen requerido de los reactivos de detección de amplificación de señal de tiramida (TSA) en los pasos siguientes.
- Incubar las muestras en tampón de amplificación TSA (véase la lista de reactivos) a TA durante 1 min sin agitación usando un volumen lo más pequeño posible, lo que garantiza que las muestras están totalmente sumergidos en la solución.
- Añadir el reactivo TSA (véase elLista de reactivos) al tampón de amplificación de la muestra a una dilución de 1:50. mezclar rápidamente la solución hasta que el reactivo de TSA se distribuye uniformemente, cubrir la placa o los tubos en papel de aluminio, y se incuban las muestras durante 60 - 90 min en la oscuridad.
- Eliminar la solución de amplificación TSA y lavar las muestras en TBST 3 veces durante 5 minutos cada uno. La transferencia de los explantes de nuevo a una placa de cultivo tisular de 24 pocillos para incrementar el volumen de lavado e incubar las muestras en peróxido de hidrógeno al 1% en TBST durante 1 h. Lavar las muestras con TBST 3 veces durante 5 min cada uno, y luego dos veces durante 5 minutos cada uno con PBST antes de la muestra de montaje (ver paso 4).
4. Preparación de muestras para Imaging
- Preparar un portaobjetos de vidrio cargado de adhesión para cada par explante mediante la adición de espaciadores de imagen de espesor 0,12 mm, que evitan que las muestras de ser aplastado por la adición de un cubreobjetos. Retirar el revestimiento adhesivo de una superficie de un espaciador y colocarlo del lado adhesivo hacia abajo sobre un portaobjetos de vidrio, presionando firmly para sellar el espaciador a la diapositiva.
NOTA: Para los pasos restantes, esforzarse por mantener las muestras en condiciones de poca luz o en la oscuridad para evitar photobleaching. pares de explantes de pipeta en un portaobjetos preparado usando una pipeta Pasteur de vidrio en el centro del espaciador, lo que garantiza que el lado diseccionado del explante se enfrenta a la corredera. Organizar pares contralateral de lado a lado explantes. - Eliminar la mayor cantidad de líquido posible de la diapositiva utilizando una pipeta Pasteur de vidrio y absorber toda la humedad residual que rodea las muestras usando un pedazo de papel de seda bajo pelusa plegada.
- Permitir que las muestras se adhieran a la corredera durante 45 - 60 s, hasta que el tejido empieza a aparecer pegajoso y translúcido. Durante este tiempo, retirar el forro adhesivo restante del espaciador con unas pinzas. No permita que las muestras se sequen.
- Añadir una gran gota de medio de montaje de doble función y la solución de limpieza (0,5% de p-fenilendiamina y Tris 20 mM, pH 8,8, en 90% de glicerol) a las muestras dentro del centrodel espaciador. NOTA: Esta solución se vuelve marrón / negro cuando está permitido para oxidar.
- Colocar con cuidado un cubreobjetos circular (no. 1.5) a través de las muestras, asegurando que el medio de montaje se distribuye uniformemente y que todos los bordes del cubreobjetos hacer contacto con el espaciador. Coloque la tapa deslizante-deslizado al revés sobre un poco de papel de tejido bajo pelusa.
- Presione firmemente hacia abajo para asegurarse de que el cubreobjetos se adhiere totalmente al separador y que se elimina cualquier exceso de medio de montaje. Repetir hasta que no hay más manchas de montaje que el papel.
- Limpia y la etiqueta de la diapositiva (s) apropiada, y almacenarlos en la oscuridad hasta la imagen, a corto plazo a -20 ° C o largo plazo a -80 ° C. Después de eliminar las diapositivas de almacenamiento, permiten que se descongelen completamente antes de exponer.
- Imagen de las muestras montadas utilizando un microscopio confocal con la adquisición de baldosas y un objetivo de alta magnificación. La imagen de los pares de explantes utilizando un objetivo de inmersión en aceite de 40X a 4 micras-z intervalos utilizando 488 nm, 568 nm y 647 nm láser de lines para excitar los fluoróforos verde, rojo, y rojo lejano, respectivamente, empleadas para la proteína y la detección de mRNA en este estudio 12.
NOTA: baldosas imágenes fueron cosidos posterior a la adquisición para formar una sola imagen para su análisis.
5. posteriores a la adquisición de Análisis de Imágenes
- Utilice el software de análisis de imágenes para definir una región de interés dentro del PSM de cada muestra experimental.
- Para cuantificar los niveles de expresión, fondo de resta y las imágenes de umbral para el nivel de una muestra de control no-primario antes de la cuantificación subsiguiente. Definir un origen, un eje, y una unidad de longitud para cada muestra.
- Calcular la intensidad de fluorescencia como una función de posición a lo largo del eje rostro-caudal normalizado para cada una de las M muestras 12. Después de la normalización de las parcelas de intensidad, coloque el lado perfiles de intensidad a lado y obtener una matriz de intensidad de f (i, j) que describe la intensidad en el i ª posición espacial en la j-ésima muestra.
6. ordenación temporal de las Muestras
- Para inferir ordenamiento temporal de un componente de reloj conocida, definir su matriz de intensidad. Entonces, reorganizar las columnas de la matriz de intensidad a fin de obtener un patrón temporal periódica. Para ello, definir la función
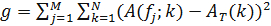
donde A (j f; k) representa la función de autocorrelación de la j-ésima columna de f y A T es una función de autocorrelación objetivo, elegido para hacer cumplir la periodicidad temporal del patrón, dada por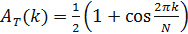
- Utilice el Metropolis-Hastings (u otro algoritmo de minimización) 12 para identificar el orden de las muestras que minimizan la función g. Por lo tanto, determinar el orden de laM muestras que maximiza la periodicidad temporal de un componente de reloj conocido.
- Utilizando el ordenamiento temporal inferido de las muestras M, construir un quimógrafo ordenada por el patrón de expresión en el canal 12 con pareja.
Resultados
Este protocolo permite la visualización del perfil de espacio-temporal de una proteína de interés junto con la transcripción de genes reloj en el PSM 12 del ratón. Por ejemplo, Dll1 (Figura 1A-C) y expresión de la proteína Notch 1 (Figura 1D-F) se muestran a oscilar fuera de sincronía con la transcripción naciente del gen Notch reloj segmentación reguladas Lfng. La cuantificación de Dll1, Notch1, y Lfng (i) la intensidad de señal en relación con el eje antero-posterior (AP) de la PSM (Figura 1G) revela claras dinámica de la expresión oscilatorios para estos objetivos (Figura 1H-J). El perfil de espacio-temporal de la expresión de la proteína Notch 1 Dll1 y durante todo el ciclo de reloj se visualiza claramente y se cuantificó usando este protocolo a través del análisis de imagen posterior a la adquisición de datos de alta resolución de imagen de tejido fijadas.
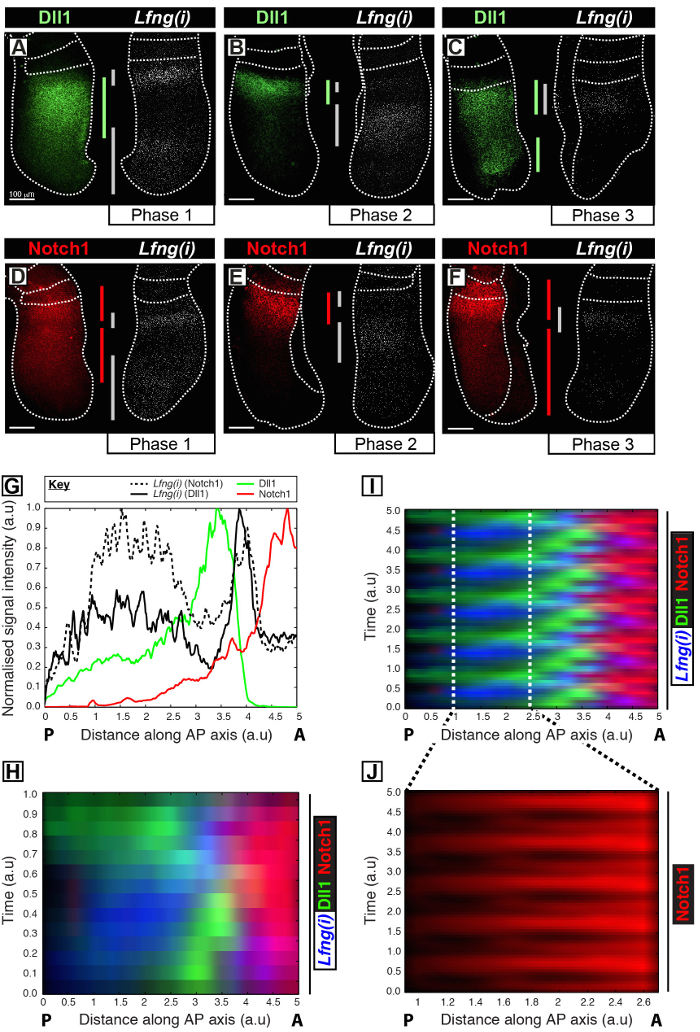
Figura 1: Visualización espacio-temporal y cuantificación de Dll1 y Notch1 expresión de la proteína Dynamics. (AF) pares de explantes de seis E10.5 embriones (AF) que muestra la distribución espacial de la proteína Dll1 (AC) o proteína Notch1 (DF) en un medio junto a la detección de Lfng pre-mRNA (Lfng (i)) en el correspondiendo la mitad contralateral de cada par. Los paneles están dispuestos de acuerdo con la Fase 1 (A y D), la Fase 2 (B y E), y la Fase 3 (C y F) del ciclo de reloj de segmentación, como se determina por el perfil espacial de Lfng (i) expresión. La extensión de los dominios de expresión para Dll1 (verde), Notch1 (rojo), y Lfng (i) (gris) a lo largo del eje antero-posterior de la PSM tiene been demarcada por barras de colores. Las líneas de puntos demarcan las posiciones de la somite más recientemente formado (s), los bordes exteriores de la PSM, y el tejido neural adyacente (C y E). Las barras de escala (abajo a la izquierda de cada panel, AF) representar a 100 micras. (G) Un ejemplo parcela de intensidad que representa la variación axial en la intensidad de la señal a través del PSM. Los datos se representan a partir de dos pares de explantes que muestran Lfng pre-mRNA (línea hash negro) en un explante comparación con la proteína Notch1 (rojo) en el explante contralateral (Embryo 1), así como Lfng pre-mRNA (línea de color negro sólido) en otro explante en comparación con la proteína Dll1 (verde) en el explante contralateral (Embryo 2). intensidad de la señal medida (eje Y) se representa frente a la posición axial (eje x; antero PSM [A] a la derecha y posterior PSM [P] a la izquierda). (H) A quimógrafo que muestra la distribución espacial de Dll1, Notch1, y Lfng (i) a través denumerosas las MEP. Cada fila de la quimógrafo representa la intensidad de señal de un explante individuo PSM. Las filas están dispuestas en secuencia temporal de acuerdo con la distribución espacio-temporal de Lfng pre-mRNA (i) la distribución espacio-temporal de Dll1, Notch1, y Lfng (i) a través de múltiples oscilaciones de reloj se simula mediante la extensión periódica de los datos mostrados en (H) , destacando el carácter oscilatorio de Dll1 y Notch1 dinámica de la expresión. La expresión de proteínas (J) pulsátil Notch1 en el caudal PSM se pone de relieve por magnificación de la región demarcada en el quimógrafo virtual se muestra en (I). Modificado de la referencia 12. Por favor, haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Cuantificación de la dinámica espacio-temporal de Dll1 y Notch1 expresión de la proteína. (A) Una parcela ejemplo intensidad representa la variación axial en la intensidad de la señal a través del PSM. Datos representados a partir de dos pares de explantes que muestra Lfng pre-mRNA (línea hash negro) en un explante comparación con la proteína Notch1 (rojo) en el medio de explante contralateral, así como Lfng pre-mRNA (línea continua negro) en un medio de explante a partir de una segunda cola en comparación con la proteína Dll1 (verde) en la mitad contralateral explante de la segunda cola. intensidades medidas (eje y) se representan frente a la posición axial (eje x; rostral [A] a la derecha y caudal [P] a la izquierda). (BH) Kymographs muestran la distribución espacial de Notch1, Dll1, INET, y Lfng (i) a través de numerosos MEP. (B y C) NICD (B) y Dll1 (C) expresión en secciones de PSM; (D y E) Lfng (i) (D) y Dll1 ( E) por la mitad de explantes contralateral; (F y G) Lfng (i) (F) y Notch1 (G) en mitades de explantes contralaterales. A partir de Referencia 12. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los pasos críticos dentro del Protocolo
El presente protocolo describe un método sensible para realizar el análisis cuantitativo de la expresión de proteínas y oscilatorias dinámicas de bajo nivel en explantes E10.5 ratón PSM. Un protocolo robusto tanto para la inmunohistoquímica y la hibridación fluorescente in situ (FISH) es seguido por de alta resolución de todo el montaje de formación de imágenes confocal, y luego por análisis de imagen y la segmentación temporal de kymographs para generar un mapa espacio-temporal de la expresión de proteínas a través de la PSM. Una alta relación de señal a ruido en la detección de proteínas y mRNA es esencial para garantizar el éxito de esta técnica. Se debe tener cuidado para intercambiar a fondo todas las soluciones de manera efectiva durante las etapas de lavado y para mantener la temperatura de los 65 ° C lavados en las etapas pertinentes del paso 3. Es más ventajoso para tomar el tiempo a la fuente de anticuerpos eficaces y sondas de ARN contra el dianas de interés y para poner a prueba estos reactivosa fondo en muestras de todo el montaje antes de comenzar con este protocolo.
Modificaciones y solución de problemas
Los principales problemas que pueden surgir cuando se realiza este protocolo se deben a una pobre resistencia de detección de señal y calidad. Esto depende en gran medida de la eficacia de los anticuerpos o sondas de ARN utilizados para las etapas de inmunohistoquímica o pescar en el protocolo, respectivamente. Un número de diferentes pasos puede requerir la optimización antes de que se logra la detección de señal adecuada. Una causa común de la mala detección de la señal es la fijación inadecuada; es imprescindible que sea fresco o PFA PFA almacenadas a 4 ° C durante no más de una semana se utiliza para fijar las muestras. La longitud de la fijación puede también requerir la optimización, dependiendo de la sonda de anticuerpo o RNA utilizado. Para los anticuerpos, se recomienda seguir las recomendaciones del fabricante cuando sea posible, mientras que para las sondas de ARN, se aconseja la consulta de la literatura publicada.
En este estudio, se utilizó una sonda de ARN que detecta específicamente el pre-ARNm del gen reloj Lfng. Debido a su relativa falta de abundancia, la detección de Lfng pre-mRNA requiere un largo periodo de incubación con la sonda en la mezcla de hibridación que contenía citrato sódico salino-5x (SSC) para una buena detección de la señal. Las mismas condiciones se pueden aplicar a otras sondas que detectan ARNm expresados débilmente, pero en nuestra experiencia, la detección de mRNA objetivos más estables pueden requerir un paso más corto sonda de hibridación y menores concentraciones de SSC en la mezcla de hibridación (por ejemplo, SSC 1,3x). Por tanto inmunohistoquímica y FISH, el primer protocolo debe ser optimizado en embriones en su conjunto, y la concentración óptima de anticuerpo o sonda debe ser determinada empíricamente.
Limitaciones de la Técnica
Como se mencionó anteriormente, el éxito de esta técnica es altamente dependiente de la calidad de la proteína y la detección de mRNA. We han descrito varias sugerencias en cuanto a cómo la proteína y la detección de ARNm se pueden mejorar, pero en ausencia de detección de la señal fluorescente de alta calidad, no hay forma en que el experimento puede proceder. El número de dianas proteicas que pueden ser analizados en cada muestra de tejido está limitada por la resolución espectral del microscopio confocal y por los epítopos de los anticuerpos utilizados. En este estudio, hemos sido capaces de utilizar hasta tres epítopos para la detección de proteínas junto con una mancha de ADN en cada muestra 12. Este protocolo sólo permite la detección de un objetivo ARNm, aunque se podrían emplear otros métodos actuales para aumentar esta cifra hasta un máximo de tres objetivos 14.
Importancia de la Técnica en Materia de Métodos Alternativos / Existentes
El método descrito aquí proporciona una técnica sensible para detectar fluctuaciones de proteínas de bajo nivel en explantes de todo el montaje de PSM. La cuantificación de estas dinámicas esposible mediante la realización de FISH para un reloj de genes conocidos en explantes contralateral correspondiente. Una biblioteca de kymographs se genera que se puede organizar más de un ciclo de reloj segmentación, destacando la expresión dinámica espacio-temporal de un objetivo de interés dentro de este marco de tiempo. Una diferencia clave en esta técnica sobre los demás es el uso de la automatización de cálculo para ordenar temporalmente grandes conjuntos de datos, que permite la expresión para ser analizados de una manera imparcial dinámica espacio-temporal de los nuevos componentes de reloj. Por ejemplo, esta técnica proporciona una idea de cómo Dll1 y Notch1 proteínas y sus oscilaciones son co-regulada en todo el PSM. Los métodos alternativos en este contexto también se han basado en la inmunotinción, pero no detectar las pequeñas fluctuaciones en Dll1 y Notch1 niveles de proteína en el PSM caudal que fueron evidentes usando este método. En su lugar, se informó de un gradiente estable de expresión que es más fuerte en la región rostral 9 , 10, 11. Esto podría ser debido al hecho de que este protocolo tiene un período más largo anticuerpo primario de incubación (3 - 5 días, a diferencia de durante la noche), que pueden ser necesarios para detectar los niveles más bajos de proteína. A medida que los niveles de expresión de Notch1 Dll1 y son relativamente altos en la rostral PSM, esto puede haber influido en los autores a la imagen de las muestras en un ambiente que no sea menor exposición serían necesarios para detectar la expresión de la proteína caudal. Una discrepancia potencial adicional surge de la utilización de tejido no fijado en el estudio de Chapman et al. , En el que la expresión transitoria de Dll1 y Notch1 en el caudal PSM puede haber sido menos bien conservado 9.
Las aplicaciones futuras o llegar después de dominar la técnica
Una vez que este protocolo ha sido dominado, el análisis de expresión de alto rendimiento se puede realizar para cualquier proteína de interés en el PSM.explantes PSM generados a partir de varias camadas de ratones pueden ser procesados a la vez para generar el número alto de la muestra necesaria para el análisis. Aunque sólo hemos utilizado embriones de tipo salvaje en estos estudios, es posible llevar a cabo este análisis utilizando embriones modificados genéticamente con el fin de evaluar la importancia de uno o más factores en la dinámica de expresión de proteínas. Más allá del PSM, este protocolo se puede adaptar a otros sistemas que se componen de dos mitades contralaterales y se pueden utilizar para detectar con sensibilidad de bajo nivel de expresión de proteínas y oscilatorias dinámicas. Un ejemplo de lo que se podría adaptar este protocolo es el estudio de la expresión de la proteína dinámica en el tubo neural del ratón, desde mitades contralateral podrían ser generados y culta, y la actividad de Notch se ha demostrado tanto su presente e importante para el patrón 15. Alentamos a otros grupos de adaptar este protocolo a otros sistemas y proporcionar información para mejorar el futuro.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
Este trabajo fue apoyado por una beca de MRC a RAB, una beca MRC a CSLB, y una subvención de proyectos WT para JKD (WT089357MA). El trabajo también fue apoyado por un premio de la confianza estratégica de bienvenida (097945 / Z / 11 / Z). Agradecemos al Dr. E. Kremmer para la especie de regalo del anticuerpo Dll1 y el Dr. O. Pourquie para la sonda de ARN Lfng.
Materiales
| Name | Company | Catalog Number | Comments |
| DMEM-F12 | Gibco (ThermoFisher Scientific) | 11320033 | |
| GlutaMAX™-1 (100x) | Gibco (ThermoFisher Scientific) | 35050 | |
| Fetal Bovine Serum, qualified, E.U.-approved, South America origin | Gibco (ThermoFisher Scientific) | 10270106 | |
| Recombinant Human FGF-basic (154 a.a.) | Peprotech | 100-18B | |
| Penicillin/Streptomycin | Gibco (ThermoFisher Scientific) | 15140122 | |
| anti-mouse monoclonal Notch1 antibody^ | BD Pharmingen | 552466 | |
| anti-rat polyclonal Dll1 antibody^* | N/A | N/A | |
| Lfng intronic anti-sense RNA probe^* | N/A | N/A | |
| 16% paraformaldehyde | Pierce (ThermoFischer Scientific) | PI28908 | |
| Proteinase K, recombinant, PCR grade | Roche (Sigma-Aldrich) | 31158 | |
| Phosphate buffered saline (PBS), pH 7.4 | Made in house | N/A | |
| Triton-X 100 | Sigma-Aldrich | T8787 | |
| Bovine serum albumin (BSA) | Sigma-Aldrich | 5470 | |
| Normal goat serum (NGS) (heat-treated) | Gibco (ThermoFisher Scientific) | 16210072 | |
| Hoechst 33342 | ThermoFischer Scientific | H3570 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| Ethanol | Sigma-Aldrich | 46139 | |
| Glutaraldehyde | Sigma-Aldrich | 340855 | |
| Formamide | Sigma-Aldrich | F9037 | |
| Saline-sodium citrate (SSC) | Sigma-Aldrich | 93017 | |
| EDTA | Sigma-Aldrich | 798681 | |
| tRNA | Roche (Sigma-Aldrich) | 101095 | |
| Heparin | Sigma-Aldrich | H3149 | |
| Tris-buffered saline (TBS) | Made in house | N/A | |
| Blocking Buffer Reagent | Roche (Sigma-Aldrich) | 11096176001 | |
| anti-DIG horseradish peroxidase (HRP) conjugated antibody | Roche (Sigma-Aldrich) | 11207733910 | |
| Tyramide signal amplification (TSA) kit | Perkin Elmer | NEL744001KT | |
| *The Dll1 antibody and RNA probe used in this study are not commercially available. Please see acknowledgements for sources. | |||
| ^Antibodies/RNA probes should be sourced which are applicable to the research interests of the reader. | |||
Referencias
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), 625-639 (2012).
- Krol, A. J., Roellig, D., et al. Evolutionary plasticity of segmentation clock networks. Development. 138 (13), 2783-2792 (2011).
- Dequéant, M. -. L., Ahnert, S., et al. Comparison of Pattern Detection Methods in Microarray Time Series of the Segmentation Clock. PLoS ONE. 3 (8), 2856 (2008).
- Bailey, C., Dale, K. . Somitogenesis in Vertebrate Development. , 1-15 (2015).
- Maroto, M., Bone, R. A., Somitogenesis Dale, J. K. Somitogenesis. Development. 139 (14), 2453-2456 (2012).
- Kageyama, R., Masamizu, Y., Niwa, Y. Oscillator mechanism of notch pathway in the segmentation clock. Developmental Dynamics. 236 (6), 1403-1409 (2007).
- Ferjentsik, Z., Hayashi, S., et al. Notch Is a Critical Component of the Mouse Somitogenesis Oscillator and Is Essential for the Formation of the Somites. PLoS Genetics. 5 (9), 1000662 (2009).
- Wiedermann, G., Bone, R. A., Silva, J. C., Bjorklund, M., Murray, P. J., Dale, J. K. A balance of positive and negative regulators determines the pace of the segmentation clock. eLife. 4, 05842 (2015).
- Chapman, G., Sparrow, D. B., Kremmer, E., Dunwoodie, S. L. Notch inhibition by the ligand DELTA-LIKE 3 defines the mechanism of abnormal vertebral segmentation in spondylocostal dysostosis. Human Molecular Genetics. 20 (5), 905-916 (2011).
- Sparrow, D. B., Chapman, G., et al. A Mechanism for Gene-Environment Interaction in the Etiology of Congenital Scoliosis. Cell. 149 (2), 295-306 (2012).
- Okubo, Y., Sugawara, T., Abe-Koduka, N., Kanno, J., Kimura, A., Saga, Y. Lfng regulates the synchronized oscillation of the mouse segmentation clock via trans-repression of Notch signalling. Nature communications. 3, 1141 (2012).
- Bone, R. A., Bailey, C. S. L., et al. Spatiotemporal oscillations of Notch1, Dll1 and NICD are coordinated across the mouse PSM. Development. 141 (24), 4806-4816 (2014).
- Shea, K., Geijsen, N. Dissection of 6.5 dpc mouse embryos. Journal of Visualized Experiments. (2), 160 (2007).
- Denkers, N., García-Villalba, P., Rodesch, C. K., Nielson, K. R., Mauch, T. J. FISHing for chick genes: Triple-label whole-mount fluorescence in situ hybridization detects simultaneous and overlapping gene expression in avian embryos. Developmental Dynamics. 229 (3), 651-657 (2004).
- Stasiulewicz, M., Gray, S. D., et al. A conserved role for Notch signaling in priming the cellular response to Shh through ciliary localisation of the key Shh transducer Smo. Development. 142 (13), 2291-2303 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados