Method Article
Commande temporelle des données d'expression dynamique d'expression spatiale détaillée Plans
Dans cet article
Résumé
The segmentation clock drives oscillatory gene expression across the pre-somitic mesoderm (PSM). Dynamic Notch activity is key to this process. We use imaging and computational analyses to extract temporal dynamics from spatial expression data to demonstrate that Delta ligand and Notch receptor expression oscillate in the vertebrate PSM.
Résumé
During somitogenesis, pairs of epithelial somites form in a progressive manner, budding off from the anterior end of the pre-somitic mesoderm (PSM) with a strict species-specific periodicity. The periodicity of the process is regulated by a molecular oscillator, known as the "segmentation clock," acting in the PSM cells. This clock drives the oscillatory patterns of gene expression across the PSM in a posterior-anterior direction. These so-called clock genes are key components of three signaling pathways: Wnt, Notch, and fibroblast growth factor (FGF). In addition, Notch signaling is essential for synchronizing intracellular oscillations in neighboring cells. We recently gained insight into how this may be mechanistically regulated. Upon ligand activation, the Notch receptor is cleaved, releasing the intracellular domain (NICD), which moves to the nucleus and regulates gene expression. NICD is highly labile, and its phosphorylation-dependent turnover acts to restrict Notch signaling. The profile of NICD production (and degradation) in the PSM is known to be oscillatory and to resemble that of a clock gene. We recently reported that both the Notch receptor and the Delta ligand, which mediate intercellular coupling, themselves exhibit dynamic expression at both the mRNA and protein levels. In this article, we describe the sensitive detection methods and detailed image analysis tools that we used, in combination with the computational modeling that we designed, to extract and overlay expression data from distinct points in the expression cycle. This allowed us to construct a spatio-temporal picture of the dynamic expression profile for the receptor, the ligand, and the Notch target clock genes throughout an oscillation cycle. Here, we describe the protocols used to generate and culture the PSM explants, as well as the procedure to stain for the mRNA or protein. We also explain how the confocal images were subsequently analyzed and temporally ordered computationally to generate ordered sequences of clock expression snapshots, hereafter defined as "kymographs," for the visualization of the spatiotemporal expression of Delta-like1 (Dll1) and Notch1 throughout the PSM.
Introduction
Somites sont les premiers segments formés dans l'axe du corps d'allongement dans le développement des espèces de vertébrés et sont les précurseurs de la colonne vertébrale, les côtes et le derme du tissu, ainsi que des muscles et des cellules endothéliales. Pendant somitogenèse, somites épithéliales se forment à partir du mésoderme non segmenté présomitique (PSM) (examiné dans la référence 1). Ce processus est régi par la «horloge de segmentation", qui se compose d'un réseau de gènes oscillatoires et des protéines, la plupart appartenant à la voie de signalisation Notch. L'horloge de segmentation se compose de différentes boucles de rétroaction négative, qui permettent la production pulsatile de l' activité de Notch dans une seule cellule 2 (revue dans les références 3-6). Bien que la méthode intracellulaire d'oscillation est bien caractérisée, il est encore largement inconnu comment ces oscillations sont coordonnées à travers le tissu de PSM. Il a été récemment montré par les études expérimentales et théoriques, que ces oscillations sont Yessential au processus de somitogenèse et que la voie Notch joue un rôle crucial dans le processus de segmentation à la fois et l' expression génique oscillatoire 7, 8. Cependant, il a été largement rapporté que le récepteur Notch 1 (Notch1) et le ligand Delta-like (Dll) -1 ont des gradients statiques dans le PSM 9, 10, 11.
Nous émettons l'hypothèse que les oscillations de Notch dépendant de l'horloge du PSM de segmentation dépendent de l'activation périodique du récepteur principal de la voie Notch et le ligand, et Notch1 DLL1, respectivement, dans le PSM de la souris. Les conclusions des études précédentes qui ont rapporté un gradient de rostrale-caudale statique de ces protéines étaient dues, nous prévoyons, à un manque de sensibilité dans les techniques de immunocoloration. Ils étaient donc incapables de détecter des fluctuations de faible niveau de DLL1 et Notch1 dans le PSM caudale.
Nous avonse a conçu une méthode pour examiner de plus près ces facteurs, combinant des données expérimentales avec la modélisation mathématique pour prédire un mécanisme par lequel les oscillations des protéines des composants d'horloge sont coordonnées à travers le PSM 12.
L'objectif global de cette méthode est de détecter et de quantifier bas niveau, l' expression dynamique des protéines dans le PSM et de cartographier les profils d'expression des protéines d'intérêt selon l'expression du gène d'horloge connu, frange Lunatic (Lfng). Depuis un cycle de l'horloge de segmentation dans l'embryon de souris prend 2 h pour terminer, divers échantillons sont nécessaires pour construire un profil complet de spatiotemporelle DLL1 et Notch1 expression de la protéine lors d' une oscillation Lfng dans le PSM. Nous avons donc mis au point ce protocole pour permettre la détection à haut débit de l'expression des protéines de bas niveau dans l'ensemble du montage, explants PSM controlatéral. Cependant, cette technique peut également être utile pour les études de tchapeau visent à caractériser la dynamique des protéines de bas niveau au sein de tout tissu embryonnaire qui peut être divisé en deux moitiés controlatéral.
Protocole
Toutes les expériences ont été réalisées au titre du projet numéro de licence 6,004,219 dans le strict respect des animaux (Scientific Procedures) Act de 1986 et les codes UK Home Office de pratique pour l'utilisation d'animaux dans les procédures scientifiques.
1. PSM Explantation Dissection
- Obtenir le tissu de la queue à partir d' embryons produits par l'accouplement temporisée de type sauvage (CD1) souris 13. Brièvement, au jour embryonnaire (E) 10,5, euthanize la souris donneuse enceinte dans une chambre de dioxyde de carbone. Récolter la corne utérine et le placer dans 1x solution saline tamponnée au phosphate stérile (PBS) en conformité avec Home Office procédures de licence ou des règles locales équivalentes. Transférer la corne utérine à une boîte de culture de tissu contenant frais, PBS stérile. Effectuez toutes les étapes de dissection ultérieures dans cette solution.
- En vertu d'un stéréomicroscope, couper la membrane musculaire épaisse de la corne utérine en utilisant des ciseaux courbes et extraire chaque embryon avec soin en utilisant une pince fine. Prends soinfaire en sorte que le tissu de la queue ne soit pas endommagé dans ce processus. Avec des ciseaux courbes et une pince fine, disséquer le sac amniotique de chaque embryon, en prenant soin de ne pas endommager l'embryon.
- Utiliser une aiguille chirurgicale ou ciseaux courbes pour récolter la queue tissu de chaque embryon en coupant la partie postérieure de l'embryon aux bourgeons des membres arrière.
- Équilibrer le tissu de la queue ventrale vers le bas en utilisant les deux pinces et une aiguille. Générer des paires de explants PSM de chaque queue embryonnaire en disséquant le tissu de la queue en deux moitiés le long de la ligne médiane; effectuer un mouvement de balancement doux avec une aiguille. Veiller à ce que le tube, notochorde, et le tissu neural PSM sont répartis également entre les deux explants.
- Pipeter chaque PSM expiant controlatéral sur la face inférieure d'une 35 mm de culture en plastique plat couvercle dans un petit volume de pré-chauffé (37 ° C) du milieu de culture (DMEM-F12 + 0,1% de L-glutamine substitut supplémenté avec 10% de sérum de veau foetal , 10 nM bFGF humain et 1% de pénicilline / streptomycine).
- Placez le plat sur le dessus du couvercle et rapidement inverser afin que le tissu PSM est suspendu à partir du couvercle dans une "goutte suspendue" du milieu. La culture des explants PSM dans une chambre humidifiée à 37 ° C pendant 1 - 2 h.
- des paires de transfert d'explants PSM dans les puits individuels d'une plaque de culture tissulaire à 24 puits. Incuber à 4% de paraformaldehyde dans du PBS pendant 1 h à température ambiante (TA) ou à 4 ° C pendant une nuit (O / N). ATTENTION: paraformaldéhyde est toxique, et des mesures de sécurité appropriées doivent être prises lorsque l'on travaille avec cette solution.
REMARQUE: Effectuer toutes lavage et d'incubation des étapes ultérieures dans une plaque de culture tissulaire de 24 puits. - Laver les puits d'échantillon dans PBS à la température ambiante sur une plate-forme à bascule, en utilisant une pipette fine plastique Pasteur pour échanger la solution PBS sur les échantillons pour PBS frais 3 - 4 fois. Traiter une explantation du PSM de chaque paire en utilisant une immunohistochimie (étape 2) et l'autre en utilisant une hybridation in situ fluorescente pour un gène d'horloge connue (step 3).
2. immunohistochimie de PSM explants
- Laver un explant PSM de chaque paire embryonnaire généré à l'étape 1 à 2% de Triton X-100 dans du PBS pendant 1 h à température ambiante sur une plate-forme à bascule, puis rincer les échantillons brièvement dans PBS. Remplacer le tampon PBS sur les échantillons avec une solution de blocage (2% d'albumine de sérum bovin (BSA) et 10% de sérum de chèvre normal (NGS) dans du PBS + 0,1 Tween-20%) et on incube O / N à 4 ° C sur une plate-forme à bascule.
REMARQUE: Tous les lavages ultérieurs et les étapes d'incubation de cette section doivent être effectuées à la température ambiante sur une plate-forme à bascule, sauf indication contraire. Les solutions de lavage peuvent être facilement modifiés en utilisant une matière plastique ou en verre pipette à pointe fine Pasteur. - Diluer l'anticorps / anticorps primaires souhaités dans un tampon de travail (0,1% de BSA, 0,3% de NGS et 0,2% de Triton X-100 dans du PBS). Dans cet exemple, diluer les anticorps DLL1 et Notch1 1:25 dans un tampon de travail.
NOTE: L'optimisation sera nécessaire pour déterminer le facteur de dilution approprié requis dans this étape si des anticorps de remplacement sont utilisés. - Incuber explants dans la solution d'anticorps pendant 3 - 5 jours à 4 ° C sur une plate-forme à bascule. Assurez-vous d'inclure des échantillons avec un tampon de travail ne contenant pas d'anticorps primaire à agir en tant que témoins d'anticorps secondaires.
- Récupérer la solution d'anticorps primaire dans un tube de stockage de 1,5 ml à l'aide d'une pipette et le stocker à 4 ° C.
REMARQUE: Récupéré anticorps primaire peut être utilisé plusieurs fois, en fonction de l'anticorps utilisé. - Effectuer 2 lavages des échantillons pendant 5 - 10 minutes à chaque fois dans du PBS, puis 3 lavages pendant 10 minutes à chaque fois dans 2% de Triton X-100 dans du PBS à la température ambiante sur une plate-forme à bascule.
- Diluer anticorps / anticorps secondaires marqués par fluorescence (épitope adaptée à l'anticorps primaire / anticorps utilisés) dans un tampon de travail. Éventuellement, ajouter 20 pg / ml de Hoechst 33342 à cette solution à contre-colorer les noyaux.
NOTE: L'optimisation peut être nécessaire pour déterminer le facteur de dilution approprié nécessaire à cette étape. Dans cet exemple, un facteur de dilution de 1: 400 a été généralement utilisé. - Centrifuger la solution d'anticorps secondaire pendant 10 min à 16 xg pour empêcher la formation d'agrégats d'anticorps. Ajouter 250 - 500 ul de la solution d'anticorps secondaire à chaque échantillon, en prenant soin de ne pas utiliser ces dernières microlitres de la solution, qui peut contenir des agrégats d'anticorps.
- Couvrir la plaque d'échantillon avec une feuille d'étain pour minimiser l'exposition de lumière et incuber les échantillons dans la solution d'anticorps secondaire pendant 3 - 5 jours à 4 ° C dans l'obscurité.
- Avant de goûter le montage, se laver les échantillons deux fois pendant 10 min chacune dans 0,1% de Tween-20 dans PBS (PBST) et une fois pendant 5 min dans du PBS à la température ambiante sur une plate-forme à bascule (voir l'étape 4).
3. Fluorescent In Situ Hybridation (FISH) de PSM explants
- Si elles sont stockées dans un récipient alternatif, transférer les autres explants PSM controlatérale dans les puits individuels d'une plaque de culture tissulaire de 24 puits.
- Laver les échantillons pour10 minutes dans l'éthanol à 50% dans du PBST, puis effectuer 2 lavages pendant 10 minutes à chaque fois dans 100% d'éthanol sur une plate-forme à bascule à la température ambiante pour déshydrater le tissu.
REMARQUE: Tous les lavages ultérieurs et les étapes d'incubation de cette section doivent être effectuées à la température ambiante sur une plate-forme à bascule, sauf indication contraire. - Réhydrater le tissu par un lavage pendant 10 min dans de l'éthanol à 50% dans du PBST, suivi par un lavage deux fois pendant 5 min à chaque fois dans du PBST.
REMARQUE: les étapes 3.2 et 3.3 sont des étapes de fixation nécessaires pour ce protocole et ne peut pas être omis. - Incuber les échantillons avec 10 pg / ml de proteinase K dans 0,1% de Tween-20 dans du PBS (PBST) pendant 5 minutes sans agitation. Retirez rapidement la proteinase K et rincer les échantillons brièvement avec PBST avant post-fixation du tissu pendant 30 min dans 4% de formaldéhyde + 0,1% de glutaraldéhyde dans du PBST. ATTENTION: Les deux formaldéhyde et le glutaraldéhyde sont toxiques, et des mesures de sécurité appropriées doivent être prises lorsque l'on travaille avec ces solutions.
NOTE: Le lavage suivantet les étapes d'incubation impliquant 50% et 100% des mélanges d'hybridation (étapes 03.06 à 03.09) doivent être effectuées sans agitation. - Après avoir lavé les échantillons deux fois pendant 10 min chacune dans PBST, laver les échantillons une fois dans 50% mélange d'hybridation (adapté pour les sondes introniques: 50% de formamide, 5x citrate salin-sodium (SSC), EDTA 5 mM, 50 pg / ml d'ARNt, 0,2% de Tween-20, 0,1% de SDS et 100 pg / ml d'héparine) dans le PBST préparé à la température ambiante. Incuber les échantillons dans cette solution pendant 10 min à 65 ° C sans agitation.
- Rincer les échantillons deux fois avec préchauffé (65 ° C) mélange d'hybridation avant l'incubation des échantillons mélange d'hybridation pendant ≥ 2 h (jusqu'à 48 h) à 65 ° C (plus longues durées d'incubation d'améliorer le contraste résultant signal sur bruit) . Retirer le mélange d'hybridation de l'étape précédente, et le remplacer par de 0,25 à 0,5 ml de pré-chauffé (65 ° C) mélange d'hybridation contenant une digoxigénine (DIG) marqué à la sonde d'ARN anti-sens contre un composant connu segmentation d'horloge.
NE PASE: Par exemple, une frange Lunatic intronique (Lfng (i)) sonde a été utilisée à une concentration de 20 ul / ml pour détecter l' ARNm naissant Lfng. La dilution utilisée dans cette étape est dépendante de la sonde et nécessitera l'optimisation. - Sceller la plaque en utilisant du ruban adhésif pour empêcher l'évaporation et incuber les échantillons dans la solution de sonde pour deux nuits à 65 ° C.
- En utilisant une pipette de Pasteur en plastique à pointe fine, récupérer la sonde pour la réutilisation et le stocker à 20 ° C. Rincer les échantillons deux fois avec préchauffé (65 ° C) post-mélange d'hybridation (50% de formamide, 0,2% de Tween-20, et 1x SSC) avant de se laver les échantillons deux fois pour 20 minutes chacun à 65 ° C en pré- réchauffé post-mélange d'hybridation.
- Laver les échantillons pendant 15 min à 65 ° C dans un mélange d'hybridation préchauffé à 50% dans 0,1% de Tween-20 dans une solution saline tamponnée au Tris (TBST). Rincer les échantillons deux fois avec du TBST avant lavage pendant 30 min à température ambiante dans du TBST sur une plate-forme à bascule.
- Pré-incuber les explants dans blosolution tain (TBST + 2% de réactif de blocage du tampon (BBR) + 20% de sérum de chèvre traité à la chaleur) pendant un minimum de 2 heures. Remplacer cette solution avec une solution de blocage frais contenant une dilution 1: 200 de peroxydase de raifort (HRP) conjugué à un anticorps anti-digoxigénine. Incuber les échantillons O / N à 4 ° C.
- Après l'incubation d'anticorps, rincer les échantillons 3 fois avec du TBST à température ambiante et de les transférer à des puits individuels d'une nouvelle plaque de 24 puits de culture tissulaire. Laver les explants avec TBST 3 fois pendant 1 h chacun.
- À ce stade, transférer les échantillons dans 0,5 mL tubes de stockage ou les puits individuels d'une plaque de culture de tissus à 48 puits pour réduire le volume nécessaire des tyramide amplification du signal (TSA) des réactifs de détection dans les étapes suivantes.
- Incuber les échantillons dans un tampon d'amplification TSA (voir la liste des réactifs) à température ambiante pendant 1 min sans agitation en utilisant un volume aussi faible que possible, en veillant à ce que les échantillons sont entièrement immergés dans la solution.
- Ajouter réactif TSA (voir leListe des réactifs) dans le tampon d'amplification de l'échantillon à une dilution de 1:50. mélanger rapidement la solution jusqu'à ce que le réactif TSA est répartie uniformément, couvrir la plaque ou les tubes dans une feuille d'étain, et incuber les échantillons pendant 60 - 90 min dans l'obscurité.
- Retirer la solution d'amplification TSA et laver les échantillons dans TBST 3 fois pendant 5 minutes chacun. Transférer les explants en arrière sur une plaque de culture tissulaire à 24 puits pour augmenter le volume de lavage, et on incube les échantillons à 1% de peroxyde d'hydrogène dans du TBST pendant 1 h. Laver les échantillons avec TBST 3 fois pendant 5 min chacun, puis deux fois pendant 5 min chacun avec PBST avant d'échantillonner de montage (voir étape 4).
4. Préparation de l'échantillon pour l'imagerie
- Préparer une lame de verre chargé d'adhérence pour chaque paire d'expiant par l'addition de 0,12 mm d'épaisseur des entretoises de formation d'image, ce qui empêche les échantillons d'être écrasées par l'addition d'une lamelle. Enlever le revêtement adhésif d'une surface d'une pièce d'écartement et placer l'adhésif vers le bas sur une lame de verre, une pression firmly pour sceller l'élément d'espacement sur la lame.
NOTE: Pour les étapes restantes, s'efforcer de conserver les échantillons en basse lumière ou dans l'obscurité pour éviter photoblanchiment. Pipette explants paires sur une lame préparée en utilisant une pipette Pasteur en verre dans le centre de l'entretoise, assurant que le côté disséqué de l'expiant fait face à la diapositive. Arrangez paires controlatérale de explants côte à côte. - Retirez autant de liquide que possible de la diapositive à l'aide d'une pipette Pasteur en verre et évacuer toute humidité résiduelle entourant les échantillons en utilisant un morceau de papier de soie peu pelucheux plié.
- Laisser les échantillons adhèrent à la diapositive 45 - 60 s, jusqu'à ce que le tissu commence à apparaître collante et translucide. Pendant ce temps, retirez le revêtement adhésif restant de l'entretoise en utilisant une pince. Ne pas laisser les échantillons sécher.
- Ajouter une grosse goutte de milieu de montage à double fonction et d'une solution de compensation (0,5% de p-phénylènediamine et du Tris 20 mM, pH 8,8, dans 90% de glycerol) aux échantillons dans le centrede l'entretoise. NOTE: Cette solution vire au brun / noir lorsque autorisé à s'oxyder.
- Placer délicatement une lamelle couvre-objet circulaire (n. 1,5) pour les échantillons, en veillant à ce que le milieu de montage est uniformément répartie et que tous les bords de la lamelle en contact avec la pièce d'écartement. Placez la glissière de couverture-glissé à l'envers sur un papier de soie peu pelucheux.
- Presser fermement pour faire en sorte que la lamelle adhère parfaitement à la pièce d'écartement, et que tout excès de milieu de montage est retirée. Répétez jusqu'à ce que aucun blots plus Mountant le papier.
- Nettoyer et étiqueter la diapositive (s) de manière appropriée, et de les stocker dans l'obscurité jusqu'à l'imagerie, à court terme à -20 ° C ou à long terme à -80 ° C. Après avoir retiré les diapositives de stockage, de leur permettre de dégeler complètement avant l'imagerie.
- Image les échantillons montés en utilisant un microscope confocal avec l'acquisition carrelée et un objectif à fort grossissement. Image les paires d'explants en utilisant un objectif à immersion d'huile 40X à 4 um z intervalles en utilisant 488 nm, 568 nm et 647 nm li lasernes pour exciter les fluorophores verts, rouges, et bien-rouge, respectivement, utilisées pour les protéines et la détection de l' ARNm dans cette étude 12.
NOTE: carrelée images ont été cousues post-acquisition pour former une seule image pour l'analyse.
5. Post-acquisition Analyse d'image
- Utilisez un logiciel d'analyse d'image pour définir une région d'intérêt dans le PSM de chaque échantillon expérimental.
- Pour quantifier les niveaux d'expression, fond soustraction et images de seuil au niveau de non-primaire échantillon de contrôle avant la quantification ultérieure. Définir une origine, un axe et une longueur unitaire pour chaque échantillon.
- Calculer l'intensité de fluorescence en fonction de la position le long de l'axe rostro-caudale normalisée pour chacun des M échantillons 12. Après la normalisation des parcelles d'intensité, placez le côté des profils d'intensité à côte et obtenir une matrice d'intensité f (i, j) qui décrit l'intensité à l'i e position spatiale dans la j ième échantillon.
6. Commande temporelle des échantillons
- Pour déduire l'ordre temporel d'un composant connu d'horloge, de définir sa matrice d'intensité. Ensuite, réorganiser les colonnes de la matrice d'intensité de façon à obtenir un motif périodique dans le temps. Pour ce faire, définir la fonction
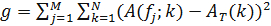
où A (f j, k) représente la fonction d'auto - corrélation de la j - ième colonne de f et A T est une fonction d'auto - corrélation cible, choisi pour respecter la périodicité temporelle du motif donné par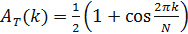
- Utilisez le Metropolis-Hastings (ou un autre algorithme de minimisation) 12 pour identifier l'ordre des échantillons qui minimisent la fonction g. Ainsi, déterminer l'ordre duM échantillons qui maximise la périodicité temporelle d'un composant connu d'horloge.
- Utilisation de l'ordre temporel inférée des M échantillons, construire un kymographe commandé pour le motif d'expression dans le canal 12 en partenariat.
Résultats
Ce protocole permet de visualiser le profil spatio - temporel d'une protéine d'intérêt le long de la transcription du gène d'horloge dans le PSM 12 souris. Par exemple, DLL1 (figure 1A-C) et Notch1 (figure 1D-F) expression de la protéine sont montrées à osciller sur synchronie avec la transcription naissante du gène Notch segmentation horloge régulée Lfng. Quantification de la DLL1, Notch1 et Lfng (i) d'intensité du signal par rapport à l'axe antéro-postérieur (AP) de la MSP (figure 1G) révèle la dynamique d'expression oscillatoires claires pour ces cibles (Figure 1 H-J). Le profil spatiotemporelle de DLL1 et Notch1 expression de la protéine tout au long du cycle d'horloge sont clairement visualisé et quantifié en utilisant ce protocole par le post-acquisition analyse d'image des données d'image fixe tissu à haute résolution.
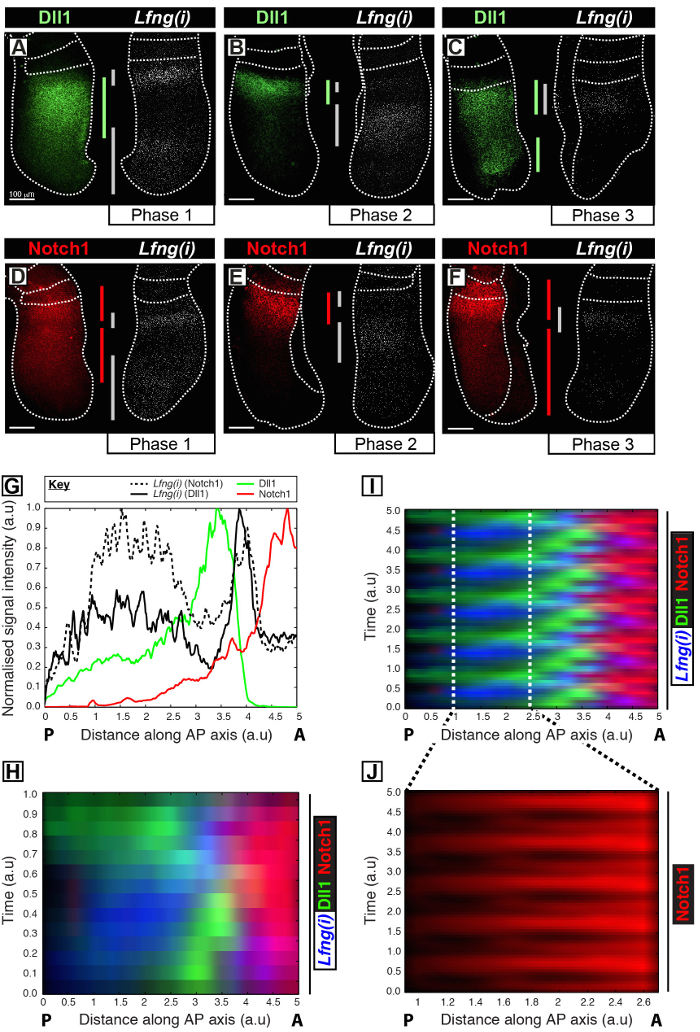
Figure 1: Visualisation spatio-temporelle et la quantification des DLL1 et Notch1 Protein Expression Dynamics. (AF) paires d'explants de six embryons E10.5 (AF) montrant la distribution spatiale des protéines DLL1 (AC) ou d'une protéine Notch1 (DF) dans une moitié le long de la détection de Lfng pré-ARNm (Lfng (i)) dans la la moitié controlatérale de chaque paire correspondante. Les panneaux sont disposés suivant la Phase 1 (A et D), la phase 2 (B et E) et la phase 3 (C et F) du cycle de segmentation d'horloge, telle que déterminée par le profil spatial de Lfng : (i) l' expression. L'étendue des domaines d'expression pour DLL1 (vert), Notch1 (rouge), et Lfng (i) (gris) le long de l'axe antéro-postérieur du PSM ont been délimité par des barres de couleur. Les lignes en pointillés délimitent les positions des somites plus récemment formé (s), les bords extérieurs du PSM et le tissu nerveux adjacent (C et E). Les barres d'échelle ( en bas à gauche de chaque panneau, AF) représentent 100 um. (G) Une parcelle d'intensité exemple illustrant la variation axiale de l'intensité du signal à travers le PSM. Les données sont tracées à partir de deux paires explants montrant Lfng pré-ARNm (ligne hachée noir) dans un explant par rapport à la protéine Notch1 (rouge) dans l'expiant controlatéral (Embryo 1), ainsi que Lfng pré-ARNm (noir ligne continue) en une autre explant par rapport à la protéine DLL1 (vert) dans l'expiant controlatéral (Embryo 2). intensité du signal mesuré (axe y) est représenté en fonction position axiale (axe x; antérieur PSM [A] à droite et postérieure PSM [P] à gauche). (H) un kymographe montrant la répartition spatiale des DLL1, Notch1 et Lfng (i) à traversde nombreux PSMs. Chaque ligne de la kymographe représente l'intensité de signal d'un explant PSM individuel. Les rangées sont disposées en séquence temporelle selon la répartition spatio - temporelle des Lfng pré-ARNm (I) La répartition spatio - temporelle des DLL1, Notch1 et Lfng (i) à travers de multiples oscillations d'horloge est simulée par l'extension périodique des données présentées dans (H) , mettant en évidence la nature oscillatoire de DLL1 et Notch1 dynamique d'expression. (J) pulsatile Notch1 expression de la protéine dans le PSM caudale est mis en évidence par grossissement de la région délimitée dans le kymographe virtuel représenté en (I). Modifié de Référence 12. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.

Figure 2: Quantification de la dynamique spatio-temporelle de DLL1 et Notch1 Protein Expression. (A) Une parcelle par exemple d'intensité représente la variation axiale de l'intensité du signal à travers le PSM. Les données tracées à partir de deux paires explants montrant Lfng pré-ARNm (ligne hachée noir) dans un explant par rapport à la protéine Notch1 (rouge) dans la moitié des explants controlatéral, ainsi que Lfng pré-ARNm (noir ligne continue) dans un demi - explant d'un deuxième queue par rapport à la protéine DLL1 (vert) dans la moitié des explants controlatéral de la seconde queue. intensités mesurées (y-axe) sont tracées en fonction position axiale (axe x; rostrale [A] à droite et caudale [P] à gauche). (BH) Kymographs montrent la distribution spatiale des Notch1, DLL1, NICD et Lfng (i) à travers de nombreux PSMs. (B et C) NICD (B) et DLL1 (C) expression dans les sections PSM; (D et E) Lfng (i) (D) et DLL1 ( E) dans les moitiés d'explants controlatérale; (F et G) Lfng (i) (F) et Notch1 (G) dans les moitiés d'explants controlatéral. De Référence 12. S'il vous plaît cliquer ici pour voir une version plus grande de cette figure.
Discussion
Étapes critiques dans le Protocole
Le présent protocole décrit une méthode sensible pour effectuer l'analyse quantitative de l'expression de la protéine et la dynamique oscillatoire de bas niveau dans les explants E10.5 souris PSM. Un protocole robuste pour les deux immunohistochimie et hybridation in situ fluorescente (FISH) est suivie par haute résolution toute monture imagerie confocale, puis par analyse d'image et de la segmentation temporelle de kymographs pour générer une carte spatiotemporelle d'expression de protéines à travers le PSM. Un rapport signal-bruit élevé dans la détection de la protéine et l'ARNm est essentiel pour assurer le succès de cette technique. Des précautions doivent être prises pour échanger complètement toutes les solutions de manière efficace au cours des étapes de lavage et de maintenir la température de 65 ° C lavages C dans les phases pertinentes de l'étape 3. Il est particulièrement avantageux de prendre le temps de se procurer des anticorps efficaces et des sondes d'ARN contre la cibles d'intérêt et de tester ces réactifsà fond sur des échantillons entiers monture avant de commencer ce protocole.
Modifications et dépannage
Les principaux problèmes qui peuvent être rencontrés lors de l'exécution de ce protocole proviennent de faible résistance de détection de signal et de qualité. Cela dépend en grande partie de l'efficacité des anticorps ou des sondes d'ARN utilisées pour les immunohistochimie ou FISH étapes du protocole, respectivement. Un certain nombre de différentes étapes peut nécessiter une optimisation avant la détection de signal adéquate est obtenue. Une cause commune pour la détection du signal est faible fixation inadéquate; il est impératif que soit PFA frais ou PFA conservés à 4 ° C pendant plus d'une semaine est utilisé pour fixer les échantillons. La longueur de fixation peut également nécessiter une optimisation, en fonction de la sonde de l'anticorps ou de l'ARN utilisé. Pour les anticorps, il est conseillé de suivre les instructions du fabricant lorsque cela est possible, alors que pour des sondes d'ARN, nous vous conseillons la consultation de la littérature publiée.
Dans la présente étude, nous avons utilisé une sonde d'ARN qui détecte spécifiquement le pré-ARNm du gène d'horloge Lfng. En raison de son manque relatif de l' abondance, la détection de Lfng pré-ARNm nécessite une longue période d'incubation avec la sonde dans le mélange d'hybridation contenant du citrate de sodium salin 5x (SSC) pour une bonne détection de signal. Les mêmes conditions peuvent appliquer à d' autres sondes qui détectent les ARNm faiblement exprimées, mais dans notre expérience, la détection de cibles d'ARNm plus stables peuvent nécessiter une étape plus courte sonde d'hybridation et des concentrations plus faibles de la SSC dans le mélange d'hybridation (par exemple, 1,3x SSC). Pour les deux immunohistochimie et FISH, le protocole doit d'abord être optimisé sur des embryons entiers, et la concentration optimale d'anticorps ou de la sonde doit être déterminée de manière empirique.
Limites de la technique
Comme mentionné ci-dessus, le succès de cette technique dépend fortement de la qualité de la protéine et la détection de l'ARNm. We ont présenté plusieurs suggestions quant à la façon dont les protéines et la détection de l'ARNm peut être améliorée, mais en l'absence de détection de signal fluorescent de haute qualité, il n'y a aucun moyen de l'expérience peut se poursuivre. Le nombre de cibles protéiques qui peuvent être analysées dans chaque échantillon de tissu est limité par la résolution spectrale du microscope confocal et par les épitopes des anticorps utilisés. Dans cette étude, nous avons pu utiliser jusqu'à trois épitopes pour la détection de la protéine à côté d' une tache d'ADN sur chaque échantillon 12. Ce protocole ne permet la détection d'un ARNm cible, bien que les méthodes alternatives actuelles pourraient être employées pour augmenter ce jusqu'à trois cibles 14.
Importance de la technique par rapport aux méthodes existantes / alternatives
La méthode décrite ici fournit une technique sensible pour détecter les fluctuations de protéines de bas niveau dans des explants entiers de montage PSM. La quantification de ces dynamiques estpossible en effectuant FISH pour un gène d'horloge connu dans explants controlatérale correspondant. Une bibliothèque de kymographs est généré qui peut être organisée sur un cycle d'horloge de segmentation, mettant en évidence la dynamique d'expression spatio-temporelle d'une cible d'intérêt dans ce laps de temps. Une différence essentielle de cette technique par rapport aux autres est l'utilisation de l'automatisation informatique pour commander temporellement grands ensembles de données, ce qui permet la dynamique d'expression spatio-temporelle des nouveaux composants d'horloge à analyser d'une manière impartiale. Par exemple, cette technique a donné un aperçu de la façon dont DLL1 et Notch1 protéines et leurs oscillations sont co-régulé sur l'ensemble du PSM. Des méthodes alternatives dans ce contexte ont également invoqué immunocoloration, mais ils ne pas détecter les petites fluctuations dans DLL1 et Notch1 niveaux de protéines dans le PSM caudale qui étaient évidentes en utilisant cette méthode. Au lieu de cela, ils ont rapporté un gradient constant d'expression qui est la plus forte dans la région rostrale 9 , 10, 11. Cela pourrait être dû au fait que ce protocole a une période plus longue de l'anticorps primaire d'incubation (3 - 5 jours, par opposition à une nuit), ce qui peut être nécessaire pour détecter des niveaux inférieurs de protéine. Comme les niveaux de DLL1 et d'expression Notch1 sont relativement élevés dans le PSM rostrale, cela peut avoir influencé les auteurs à l'image des échantillons à une exposition plus faible réglage que seraient nécessaires pour détecter l'expression de la protéine caudale. Une autre différence de potentiel provient de l'utilisation d' un tissu non fixé dans l'étude de Chapman et al. , Dans lequel l'expression transitoire de DLL1 et Notch1 dans le PSM caudale peut avoir été moins bien conservé 9.
Applications futures ou les directions après Maîtriser la technique
Une fois que ce protocole a été maîtrisé, l'analyse de l'expression à haut débit peut être effectuée pour toute protéine d'intérêt dans le PSM.explants PSM générés à partir de plusieurs portées de souris peuvent être traitées à la fois pour générer le nombre nécessaire pour l'analyse de l'échantillon élevé. Bien que nous n'avons utilisé des embryons de type sauvage dans ces études, il est possible d'effectuer cette analyse en utilisant des embryons génétiquement modifiés afin d'évaluer l'importance d'un ou plusieurs facteurs sur la dynamique d'expression de protéines. Au-delà du PSM, ce protocole peut être adapté à d'autres systèmes qui se composent de deux moitiés controlatéral et peuvent être utilisés pour détecter avec sensibilité et expression de la protéine oscillatoires dynamique de bas niveau. Un exemple pour lequel ce protocole peut être adapté est l'étude de l' expression des protéines dynamique dans le tube neural de la souris, puisque les moitiés controlatérale ont pu être générées et mises en culture et l' activité de Notch a été démontré à la fois présent et important pour motif 15. Nous encourageons d'autres groupes afin d'adapter ce protocole à d'autres systèmes et de fournir une rétroaction pour l'amélioration future.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Ce travail a été soutenu par une studentship MRC à RAB, un studentship MRC à CSLB, et une subvention de projet de WT à JKD (WT089357MA). Le travail a également été soutenu par un prix stratégique Welcome Trust (097.945 / Z / 11 / Z). Nous remercions le Dr E. Kremmer pour le genre don de l'anticorps DLL1 et le Dr O. Pourquie pour la sonde Lfng ARN.
matériels
| Name | Company | Catalog Number | Comments |
| DMEM-F12 | Gibco (ThermoFisher Scientific) | 11320033 | |
| GlutaMAX™-1 (100x) | Gibco (ThermoFisher Scientific) | 35050 | |
| Fetal Bovine Serum, qualified, E.U.-approved, South America origin | Gibco (ThermoFisher Scientific) | 10270106 | |
| Recombinant Human FGF-basic (154 a.a.) | Peprotech | 100-18B | |
| Penicillin/Streptomycin | Gibco (ThermoFisher Scientific) | 15140122 | |
| anti-mouse monoclonal Notch1 antibody^ | BD Pharmingen | 552466 | |
| anti-rat polyclonal Dll1 antibody^* | N/A | N/A | |
| Lfng intronic anti-sense RNA probe^* | N/A | N/A | |
| 16% paraformaldehyde | Pierce (ThermoFischer Scientific) | PI28908 | |
| Proteinase K, recombinant, PCR grade | Roche (Sigma-Aldrich) | 31158 | |
| Phosphate buffered saline (PBS), pH 7.4 | Made in house | N/A | |
| Triton-X 100 | Sigma-Aldrich | T8787 | |
| Bovine serum albumin (BSA) | Sigma-Aldrich | 5470 | |
| Normal goat serum (NGS) (heat-treated) | Gibco (ThermoFisher Scientific) | 16210072 | |
| Hoechst 33342 | ThermoFischer Scientific | H3570 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| Ethanol | Sigma-Aldrich | 46139 | |
| Glutaraldehyde | Sigma-Aldrich | 340855 | |
| Formamide | Sigma-Aldrich | F9037 | |
| Saline-sodium citrate (SSC) | Sigma-Aldrich | 93017 | |
| EDTA | Sigma-Aldrich | 798681 | |
| tRNA | Roche (Sigma-Aldrich) | 101095 | |
| Heparin | Sigma-Aldrich | H3149 | |
| Tris-buffered saline (TBS) | Made in house | N/A | |
| Blocking Buffer Reagent | Roche (Sigma-Aldrich) | 11096176001 | |
| anti-DIG horseradish peroxidase (HRP) conjugated antibody | Roche (Sigma-Aldrich) | 11207733910 | |
| Tyramide signal amplification (TSA) kit | Perkin Elmer | NEL744001KT | |
| *The Dll1 antibody and RNA probe used in this study are not commercially available. Please see acknowledgements for sources. | |||
| ^Antibodies/RNA probes should be sourced which are applicable to the research interests of the reader. | |||
Références
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), 625-639 (2012).
- Krol, A. J., Roellig, D., et al. Evolutionary plasticity of segmentation clock networks. Development. 138 (13), 2783-2792 (2011).
- Dequéant, M. -. L., Ahnert, S., et al. Comparison of Pattern Detection Methods in Microarray Time Series of the Segmentation Clock. PLoS ONE. 3 (8), 2856 (2008).
- Bailey, C., Dale, K. . Somitogenesis in Vertebrate Development. , 1-15 (2015).
- Maroto, M., Bone, R. A., Somitogenesis Dale, J. K. Somitogenesis. Development. 139 (14), 2453-2456 (2012).
- Kageyama, R., Masamizu, Y., Niwa, Y. Oscillator mechanism of notch pathway in the segmentation clock. Developmental Dynamics. 236 (6), 1403-1409 (2007).
- Ferjentsik, Z., Hayashi, S., et al. Notch Is a Critical Component of the Mouse Somitogenesis Oscillator and Is Essential for the Formation of the Somites. PLoS Genetics. 5 (9), 1000662 (2009).
- Wiedermann, G., Bone, R. A., Silva, J. C., Bjorklund, M., Murray, P. J., Dale, J. K. A balance of positive and negative regulators determines the pace of the segmentation clock. eLife. 4, 05842 (2015).
- Chapman, G., Sparrow, D. B., Kremmer, E., Dunwoodie, S. L. Notch inhibition by the ligand DELTA-LIKE 3 defines the mechanism of abnormal vertebral segmentation in spondylocostal dysostosis. Human Molecular Genetics. 20 (5), 905-916 (2011).
- Sparrow, D. B., Chapman, G., et al. A Mechanism for Gene-Environment Interaction in the Etiology of Congenital Scoliosis. Cell. 149 (2), 295-306 (2012).
- Okubo, Y., Sugawara, T., Abe-Koduka, N., Kanno, J., Kimura, A., Saga, Y. Lfng regulates the synchronized oscillation of the mouse segmentation clock via trans-repression of Notch signalling. Nature communications. 3, 1141 (2012).
- Bone, R. A., Bailey, C. S. L., et al. Spatiotemporal oscillations of Notch1, Dll1 and NICD are coordinated across the mouse PSM. Development. 141 (24), 4806-4816 (2014).
- Shea, K., Geijsen, N. Dissection of 6.5 dpc mouse embryos. Journal of Visualized Experiments. (2), 160 (2007).
- Denkers, N., García-Villalba, P., Rodesch, C. K., Nielson, K. R., Mauch, T. J. FISHing for chick genes: Triple-label whole-mount fluorescence in situ hybridization detects simultaneous and overlapping gene expression in avian embryos. Developmental Dynamics. 229 (3), 651-657 (2004).
- Stasiulewicz, M., Gray, S. D., et al. A conserved role for Notch signaling in priming the cellular response to Shh through ciliary localisation of the key Shh transducer Smo. Development. 142 (13), 2291-2303 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon