Method Article
자세한 공간 식지도에서 동적 식 데이터의 시간적 순서
요약
The segmentation clock drives oscillatory gene expression across the pre-somitic mesoderm (PSM). Dynamic Notch activity is key to this process. We use imaging and computational analyses to extract temporal dynamics from spatial expression data to demonstrate that Delta ligand and Notch receptor expression oscillate in the vertebrate PSM.
초록
During somitogenesis, pairs of epithelial somites form in a progressive manner, budding off from the anterior end of the pre-somitic mesoderm (PSM) with a strict species-specific periodicity. The periodicity of the process is regulated by a molecular oscillator, known as the "segmentation clock," acting in the PSM cells. This clock drives the oscillatory patterns of gene expression across the PSM in a posterior-anterior direction. These so-called clock genes are key components of three signaling pathways: Wnt, Notch, and fibroblast growth factor (FGF). In addition, Notch signaling is essential for synchronizing intracellular oscillations in neighboring cells. We recently gained insight into how this may be mechanistically regulated. Upon ligand activation, the Notch receptor is cleaved, releasing the intracellular domain (NICD), which moves to the nucleus and regulates gene expression. NICD is highly labile, and its phosphorylation-dependent turnover acts to restrict Notch signaling. The profile of NICD production (and degradation) in the PSM is known to be oscillatory and to resemble that of a clock gene. We recently reported that both the Notch receptor and the Delta ligand, which mediate intercellular coupling, themselves exhibit dynamic expression at both the mRNA and protein levels. In this article, we describe the sensitive detection methods and detailed image analysis tools that we used, in combination with the computational modeling that we designed, to extract and overlay expression data from distinct points in the expression cycle. This allowed us to construct a spatio-temporal picture of the dynamic expression profile for the receptor, the ligand, and the Notch target clock genes throughout an oscillation cycle. Here, we describe the protocols used to generate and culture the PSM explants, as well as the procedure to stain for the mRNA or protein. We also explain how the confocal images were subsequently analyzed and temporally ordered computationally to generate ordered sequences of clock expression snapshots, hereafter defined as "kymographs," for the visualization of the spatiotemporal expression of Delta-like1 (Dll1) and Notch1 throughout the PSM.
서문
Somites은 척추 동물 종을 개발 연신 몸통 축에 형성된 제 세그먼트이며, 척추, 늑골의 전구체이며, 진피 조직뿐만 아니라 근육 및 혈관 내피 세포. somitogenesis 동안, 상피 somites는 (참고 1에서 검토)에 비분 presomitic 중배엽 (PSM)에서 형성한다. 이 프로세스는 주로 노치 신호 전달 경로에 속하는 진동 유전자 및 단백질의 네트워크 구성은 "분할 시계"에 의해 조절된다. 분할 클록이 단일 셀 (2) 내에서 노치 활동의 박동성 생산 있도록 다양한 네거티브 피드백 루프로 구성 (참고 문헌에서 검토 3-6). 진동의 세포 내 방법은 잘 특징이 있지만, 이들 진동이 PSM 조직에 걸쳐 조정되는 방법을 여전히 대부분 알려져 있지 않다. 또한, 최근 이러한 진동이 된 essentia 있다는 모두 실험적 및 이론적 연구를 통해 밝혀졌다노치 경로 somitogenesis의 공정에 해당 L은 분할 및 진동 유전자 발현 (7, 8) 둘 다의 공정에서 중요한 역할을한다. 그러나, 광범위하게 해당 노치 수용체 1 (Notch1) 및보고 된 델타 형 리간드 (DLL) -1 PSM 9, 10, 11의 static 구배를 갖는다.
우리는 PSM 분할 클록의 노치 의존 진동 마우스 PSM 걸쳐 각각 메인 통로 노치 수용체 및 리간드와 Notch1 DLL1의 활성주기에 의존한다는 가설을 세웠다. 이들 단백질의 정적 입쪽 - 꼬리 구배를보고 이전 연구의 결론은 우리는 면역 염색 기법의 감도 부족, 예측 예정이었다. 그들은 꼬리 PSM에 DLL1과 Notch1의 낮은 수준의 변동을 검출하는 것이 없습니다.
우리는 근래전자 시계 성분의 단백질의 진동이 PSM (12)를 통해 조정하는 메커니즘을 예측하는 수학적 모델링 실험 데이터를 결합하여 더 자세히 요인을 조사하는 방법을 고안했다.
이 방법의 전반적인 목표 감지 및 PSM에서 낮은 수준의 동적 단백질 발현을 정량화 알려진 시계 유전자의 발현에 따라 관심의 단백질의 발현 프로파일을 매핑하는 것입니다, 미치광이 프린지 (Lfng). 마우스 배아의 분할 클럭의 한 사이클을 완료하는 데 2 시간을 소요하기 때문에, 다양한 샘플은 PSM 한 Lfng 진동 동안 DLL1과 Notch1 단백질 발현의 전체 시공간 프로필을 구축해야합니다. 따라서 우리는 전체 산 낮은 수준의 단백질 발현, 반대측 PSM 이식편의 높은 처리량의 검출이 가능하도록이 프로토콜을 개발했다. 그러나,이 기술은 또한 연구 t에 유용모자는 반대측 반으로 분할 될 수있는 배아 조직 내에서 낮은 수준의 단백질 역학의 특성을 목표로하고 있습니다.
프로토콜
모든 실험은 동물 (과학적인 절차) 1986 년 법 과학적 절차에 동물의 사용을위한 연습의 영국 홈 오피스 코드에 대한 엄격한 준수에 프로젝트 허가 번호 6004219에서 수행 하였다.
1. PSM 이식편 해부
- 야생형 (CD1) 마우스 (13)의 시간 제한 결합에 의해 생성 된 배아의 꼬리 조직을 얻습니다. 간단히, 배아 일 (E) 10.5, 이산화탄소 챔버 내의 도너 임신 마우스를 안락사. 자궁 뿔을 수확 및 홈 오피스 라이센스 절차 또는 이에 상응하는 현지 규정에 따라 1 배 멸균 인산 완충 생리 식염수 (PBS) 용액에 넣습니다. 신선한, 멸균 PBS를 포함하는 조직 배양 접시에 자궁 뿔을 전송합니다. 이 솔루션의 모든 후속 해부 단계를 수행합니다.
- 실체 현미경에서 곡선 가위를 사용하여 자궁의 두꺼운 근육 막 잘라 조심스럽게 미세 집게를 사용하여 각 배아를 추출합니다. 주의꼬리 조직이이 과정에서 손상되지 않았는지 확인합니다. 배아를 손상하지 않도록주의하면서 각 배아에서 양막 낭을 멀리 해부, 곡선 가위 및 미세 집게를 사용.
- 후방 사지 꽃 봉오리에 배아 후방을 절단하여 각 배아의 꼬리 조직을 수확하는 수술 바늘 또는 곡선 가위를 사용합니다.
- 집게와 바늘을 모두 사용하여 꼬리 조직 복부 측을 균형. 중간 선을 따라 두 개의 반으로 꼬리 조직을 해부하여 각 배아의 꼬리에서 PSM 외식의 쌍을 생성; 바늘로 부드럽게 흔들어서을 수행합니다. 신경 튜브, 척색 및 PSM 조직이 동일하게 두 개의 외식 사이에서 분할되어 있는지 확인합니다.
- + 0.1 %의 L 글루타민 대신 10 % 우태 혈청으로 보충 된 예비 가온 된 (37 ℃) 배양 배지 (DMEM-F12의 작은 부피로 35 mm 플라스틱 배양 접시 뚜껑의 밑면 상에 각각 반대측 PSM 이식편 피펫 10 nM의 인간 bFGF를, 1 % 페니실린 / 스트렙토 마이신). <리>는 뚜껑의 상단에 접시를 놓고 빠르게 PSM 조직이 매체의 "매달려 드롭"의 뚜껑에 매달려 있도록 반전. 배양 한 37 ℃에서 가습 실에서 PSM 이식편 - 2 시간.
- 24 웰 조직 배양 플레이트의 각 웰에 PSM 이식편의 전송 쌍. 하룻밤 실온 (RT) 또는 4 ° C에서 1 시간 (O / N)에 대한 PBS에서 4 % 파라 포름 알데히드에 품어. 주의 : 파라 포름 알데히드는 독성이 있으며,이 솔루션 작업을 할 때 적절한 안전 조치가 취해 져야한다.
참고 : 24 잘 조직 배양 접시에 모든 후속 세척 및 배양 단계를 수행합니다. - 4 회 - 신선한 PBS 3 샘플의 PBS 용액을 교환하는 좋은 플라스틱 파스퇴르 피펫을 사용하여 락 플랫폼에서 실온에서 PBS에서 샘플 우물을 씻으십시오. (면역 조직 화학 염색을 (2 단계) 및 알려진 시계 유전자에 대한 현장 하이브리드의 다른 이용하여 형광을 사용하여 일을 각 쌍에서 하나의 PSM 이식편을 처리EP 3).
PSM의 Explants 2. 면역 조직 화학
- 락 플랫폼에서 실온에서 1 시간 동안 PBS에 2 % 트리톤 X-100에서 1 단계에서 생성 된 각 배아 쌍에서 하나의 PSM 이식편을 세척 한 후 PBS에서 간단히 샘플을 씻어. 요동 플랫폼에서 4 ℃에서 배양한다 O 차단 용액 (PBS + 0.1 % 트윈 20 2 % 소 혈청 알부민 (BSA)와 10 % 정상 염소 혈청 (NGS)) / N과 샘플의 PBS를 교체.
참고 : 달리 명시하지 않는 한이 섹션의 모든 후속 세척 및 배양 단계는, 락 플랫폼에서 실온에서 수행해야합니다. 세척 솔루션은 쉽게 끝이 뾰족한 플라스틱이나 유리 파스퇴르 피펫을 사용하여 변경할 수 있습니다. - 작업 버퍼에 원하는 차 항체 / 항체를 희석 (0.1 % BSA, 0.3 % NGS 및 PBS에서 0.2 % 트리톤 X-100). 이 예에서, 버퍼 작업에 DLL1과 Notch1 항체 1:25 희석.
주 : 생 최적화에 필요한 적절한 희석 인자를 결정해야 할 것이다다른 항체를 사용하는 경우의 단계를 포함한다. - 락 플랫폼에서 4 ° C에서 5 일 - 3에 대한 항체 용액에서 외식을 품어. 차 항체 제어 역할을 할 차 항체를 함유하지 않은 버퍼를 작동하는 몇 가지 샘플을 포함해야합니다.
- 피펫을 사용하여 1.5 mL의 저장 튜브의 기본 항체 솔루션을 복구하고 4 ℃에서 보관합니다.
주 : 복구 차 항체가 사용 된 항체에 따라 여러 번 사용될 수있다. - 락 플랫폼에서 실온에서 PBS에 2 % 트리톤 X-100에 10 분마다 3 회 세척 한 다음 PBS에서 10 분 각각 - 5 샘플 2 세척을 수행합니다.
- 형광 표지 된 이차 항체 / 항체를 희석 버퍼 작업에 (차 항체에 항원이 일치 / 항체 사용). 선택적으로, 핵을 Counterstain과이 용액에 20 μg의 / mL의 훽스트 33342를 추가합니다.
주 : 최적화를이 단계에서 필요한 적절한 희석 인자를 결정하기 위해 필요할 수있다. 이 전자에xample 1의 희석 비율 : 400이 일반적으로 사용되었다. - 항체 응집체의 형성을 방지하기 위해 16 XG에서 10 분 동안 차 항체 용액을 원심 분리기. 항체 응집체를 함유 할 수있는 용액의 마지막 몇 마이크로 리터를 사용하지 않도록주의하면서, 각 웰 샘플 이차 항체 용액 500 μL - 250을 추가한다.
- 어둠 속에서 4 ° C에서 오일 - 빛의 노출을 최소화하고 3 차 항체 용액에 샘플을 배양 주석 호일로 샘플 플레이트를 커버.
- 장착 샘플 PBS에서 0.1 % 트윈 20에서 10 분 각 (PBST)과 락을 플랫폼에서 RT에서 PBS에 한 번에 5 분 동안 두 번 샘플을 세척하기 전에 (4 단계 참조).
PSM의 Explants의 제자리 교잡 (FISH) 3. 형광등
- 다른 용기에 저장하면, 24 웰 조직 배양 플레이트의 각각의 웰에 남아있는 반대측 PSM 이식편 옮긴다.
- 대한 샘플을 씻으십시오PBST 10 50 % 에탄올 분 후는 조직을 탈수 RT에서 요동 플랫폼상에서 10 분 동안 100 % 에탄올에서 각각 2 회 세척을 수행한다.
참고 : 달리 명시하지 않는 한이 섹션의 모든 후속 세척 및 배양 단계는, 락 플랫폼에서 실온에서 수행해야합니다. - PBST에서 5 분마다 두 번 세척 한 다음, PBST 50 % 에탄올에서 10 분 동안 세척하여 조직을 재수.
참고 : 3.2 및 3.3이 프로토콜에 필요한 필요한 고정 단계는 생략 할 수없는 단계. - 동요없이 5 분 동안 PBS (PBST)에서 0.1 % 트윈 20에서 10 μg의 / ML의 단백질 분해 효소 K와 샘플을 품어. 빠르게 단백질 분해 효소 K를 제거하고 PBST에 4 % 포름 알데히드 + 0.1 % 글루 타르 알데히드에서 30 분 동안 조직을 후 고정하기 전에 잠시 PBST로 샘플을 씻어. 주의 : 포름 알데히드와 글루 타르 알데히드 모두 독성이 있으며, 이러한 솔루션 작업을 할 때 적절한 안전 조치가 취해 져야한다.
참고 : 다음 세척50 %와 100 %의 혼성화 믹스 (3.6 단계 - 3.9)을 포함하는 배양 단계는 교반없이 실시한다. - PBST 10 분마다 두 샘플을 세척 한 후, 인트론 프로브 (50 %의 혼성화 믹스 한번 적합 샘플을 세척 : 50 % 포름 아미드, 5 배 식염수 구연산 나트륨 (SSC), 5 mM의 EDTA, 50 μg의 / ㎖ tRNA는, 0.2 % 트윈 20, 0.1 % SDS 및 100 μg의 PBST에서 / ㎖ 헤파린) RT에서 제조. 교반없이 65 ° C에서 10 분 동안이 솔루션에 샘플을 품어.
- 65 ℃에서 (48 시간까지) ≥ 2 시간 동안 혼성화 믹스 샘플을 배양하기 전에 예열 (65 ° C) 혼성화 믹스 회 샘플 린스 (긴 배양 시간은 생성 된 신호 - 대 - 잡음 대비를 향상) . 이전 단계의 혼성화 믹스를 제거하고 0.25로 교체 - 예열 된 0.5 ㎖ (65 °의 C)을 제닌 (DIG)은 공지 된 분할 클럭 성분에 대해 안티센스 RNA 프로브 - 표지 함유 혼성화 믹스.
아니E : 예를 들면, 인트론 과격파 (Lfng (I)) 프로브 Lfng 초기의 mRNA를 검출하기 위해 20 μL / ㎖의 농도로 사용 하였다. 이 단계에 사용되는 희석 프로브 의존 및 최적화를 필요로한다. - 증발을 방지하고, 65 ℃에서 이틀 밤에 대한 프로브 용액 샘플을 배양 접착 테이프를 사용하여 플레이트를 밀봉.
- 뾰족한 플라스틱 파스퇴르 피펫을 사용하여 재사용을 위해 프로브를 복구하고 20 ℃에서 보관합니다. 미리 65 ℃에서 20 분마다 샘플을 두 번 더 세척 전에 예열 된 (65 ℃) 후 혼성화 믹스 (50 % 포름 아미드, 0.2 % 트윈 -20, 및 1X SSC)로 두 번 샘플 린스 이후 하이브리드 믹스를 따뜻하게.
- TBS 버퍼 용액 (TBST) 중 0.1 % 트윈 -20로 미리 예열 해 50 %의 혼성화 믹스를 65 ℃에서 15 분 동안 샘플을 세척한다. 락 플랫폼에서 TBST에 RT에서 30 분 동안 세척하기 전에 TBST로 두 번 샘플을 씻어.
- BLO에서 외식을 사전 품어요리하는 용액 (TBST + 2 % 완충 시약 (BBR) + 20 % 열 - 처리 염소 혈청 차단)을 2 시간 이상 동안. 1 포함 신선한 차단 솔루션이 솔루션 교체 : 고추 냉이 퍼 옥시 다제 (HRP)의 200 희석하는 항 디곡시 제닌 항체를 π 공역 계. 4 ° C에서 샘플 O / N을 품어.
- 항체 배양 후, RT에서 시료를 TBST로 3 회 헹구 새로운 24 웰 조직 배양 플레이트의 개별 웰에 전송할. 1 시간마다 TBST로 3 회 외식을 씻으십시오.
- 이 때, 다음 단계에 Tyramide 신호 증폭 (TSA) 검출 시약의 필요한 부피를 줄이기 위해 0.5 mL의 기억 튜브 또는 48- 웰 조직 배양 플레이트의 각 웰에 샘플을 옮긴다.
- 교반이 가능한 한 작은 부피를 사용하여 샘플이 완전히 용액에 침지되는 것을 보장하지 않고 1 분 동안 RT에서 TSA 증폭 샘플 버퍼 (시약 목록 참조) 부화.
- 참조 (TSA 시약을 추가1시 50분의 희석 시료 증폭 버퍼에 시약 목록). 신속 TSA 시약이 균일하게 분포 될 때까지 용액을 혼합 주석 호일 접시 또는 튜브를 포함하고, 60 샘플을 부화 - 어둠 속에서 90 분.
- TSA는 증폭 용액을 제거하고, 5 분마다 TBST로 3 회 샘플을 세척한다. 1 시간 동안 TBST에 1 % 과산화수소 수의 샘플들을 세척 량을 증가시키고 배양 24 웰 조직 배양 플레이트에 다시 전송 이식편. 5 분마다 TBST로 3 번 샘플을 세척 한 다음 두 번 5 분 각각의 PBST에 사전 설치 샘플 (4 단계 참조).
이미징 4. 샘플 준비
- 커버 슬립의 첨가에 의해 분쇄되는 것을 방지 할 샘플을 0.12 mm 두께의 촬상 스페이서들을 추가하여 이식편 각 쌍에 대해 하나의 청구 접착 유리 슬라이드를 준비한다. F를 누르면, 스페이서의 한쪽면으로부터 접착제 라이너를 제거하고 유리 슬라이드 상에 아래로 접착면을 배치irmly 슬라이드에 스페이서를 밀봉합니다.
참고 : 나머지 단계는 광표백을 피하기 위해 낮은 빛 또는 어둠 속에서 샘플을 유지하기 위해 노력하고 있습니다. 이식편의 절개면이 슬라이드를 향하도록 보장 스페이서의 중심 내의 유리 파스퇴르 피펫을 사용하여 제조 된 슬라이드 상에 피펫 이식편 쌍. 나란히 외식 측의 반대측 쌍을 정렬합니다. - 유리 파스퇴르 피펫을 사용하여 슬라이드 극력 액체를 제거하고 접힌 낮은 린트 티슈 종이를 사용하여 표본을 둘러싼 모든 잔류 수분을 심지.
- 조직 스티커 반투명 나타나기 시작 때까지 60 초 - 샘플 (45)의 슬라이드에 부착 할 수 있습니다. 이 시간 동안 집게를 사용 스페이서에 남아있는 접착제 라이너를 제거합니다. 샘플이 건조하는 것을 허용하지 않습니다.
- 센터 내의 샘플들 (90 % 글리세롤 0.5 % -p- 페닐 렌 디아민, 20 mM 트리스, pH를 8.8) 이중 기능 mountant 및 청산 액의 큰 방울을 추가스페이서. 참고 : 산화 허용 할 때이 솔루션은 블랙 / 갈색으로 변합니다.
- 조심스럽게 mountant가 고르게 분포되어 있고 커버 슬립의 모든 에지 스페이서와의 접촉을 보장, 샘플에 걸쳐 원형 커버 슬립 (NO. 15)를 배치합니다. 거꾸로 일부 낮은 린트 티슈 페이퍼 상에 커버 미끄러 슬라이드를 놓습니다.
- 커버 슬립이 완전히 스페이서에 부착 있고 초과 mountant이 제거되도록 단단히 누르십시오. 더 이상 mountant 오 용지까지 반복합니다.
- 깨끗하고 적절하게 슬라이드 (들) 라벨, 및 이미징 -20 ° C에서 단기 또는 -80 ° C에서 장기까지 어둠 속에서 그들을 저장합니다. 저장소에서 슬라이드를 제거한 후, 그들을 완전히 이미징 전에 해동 할 수 있습니다.
- 타일 이미지 획득 및 고배율 대물와 공 촛점 현미경을 사용하여 샘플 마운트. 이미지 488 나노, 568 나노 미터와 647 나노 레이저 리를 사용하여 4 μm의 z를-간격으로 40X 기름 침지 목표를 사용하여 이식편 쌍NES는 녹색, 빨간색, 멀리 붉은 형광, 각각이 연구 (12)에서 단백질과 mRNA의 검출을 위해 고용을 자극합니다.
참고 : 바둑판 식 이미지 분석을위한 하나의 이미지를 형성하기 위해 취득 이후 스티치했다.
5. 후 인수 이미지 분석
- 각각의 실험 샘플의 PSM 내의 관심 영역을 정의하는 이미지 분석 소프트웨어를 사용한다.
- 후속 정량하기 전에 노 주 조용 샘플의 수준으로 발현 빼기 배경 및 임계 이미지를 정량화한다. 원점, 축, 각각의 샘플에 대한 단위 길이를 정의한다.
- M 개의 샘플 (12)의 각각에 대한 정규화 rostro - 꼬리 축을 따라 위치의 함수로서 형광 강도를 계산한다. 세기의 플롯을 정규화 한 후, I의 강도를 설명하는 강도 행렬 F (I, J)를 측면으로 강도 프로파일 측에 배치하고 수득 j 번째의 공간적 위치 일>입니다.
샘플 6. 임시 주문
- 알려진 클럭 성분의 시간적 순서를 추론하는, 그 강도 행렬을 정의합니다. 시간적으로주기 패턴을 얻을 수 있도록 그리고, 세기 매트릭스의 열을 재 배열. 이를 위해, 함수를 정의
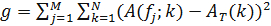
A (F, J, K)는 여기서 F 및 T의 열 j 번째의 자기 상관 함수를 나타낸다 주어진 패턴의 시간적 주기성을 강화하도록 선택 대상 자기 상관 함수이며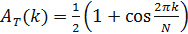
- 함수 g을 최소화 샘플들의 순서를 식별 메트로폴리스 - 스팅 (또는 다른 최소화 알고리즘) (12)를 사용한다. 따라서, 상기의 순서를 결정공지의 클럭 성분의 시간주기를 최대화 M 샘플.
- M 개의 샘플들의 추정 시간적 순서를 사용 트너 채널 (12)의 발현 양상에 대한 지시를 생성 kymograph.
결과
이 프로토콜은 마우스 PSM 12 클록 유전자 전사와 함께 목적 단백질의 시공간 프로파일의 시각화를 허용한다. 예를 들어, DLL1 (그림 1A-C)와 Notch1 (그림 1D-F) 단백질 발현은 노치 규제 분할 시계 유전자 Lfng의 초기 전사와 동시성에서 진동 표시됩니다. DLL1은 Notch1 및 Lfng (ⅰ) PSM (그림 1G)의 안테 - 후부 (AP) 축과 관련하여 신호 강도가 이러한 목표 (그림 1H-J)에 대한 명확한 진동 식의 역 동성을 보여 정량. 클럭 사이클 동안 DLL1 Notch1 및 단백질 발현의 시공간 프로필 분명히 가시화 고해상도 고정 조직 화상 데이터의 취득 후 이미지 분석을 통해이 프로토콜을 사용하여 정량된다.
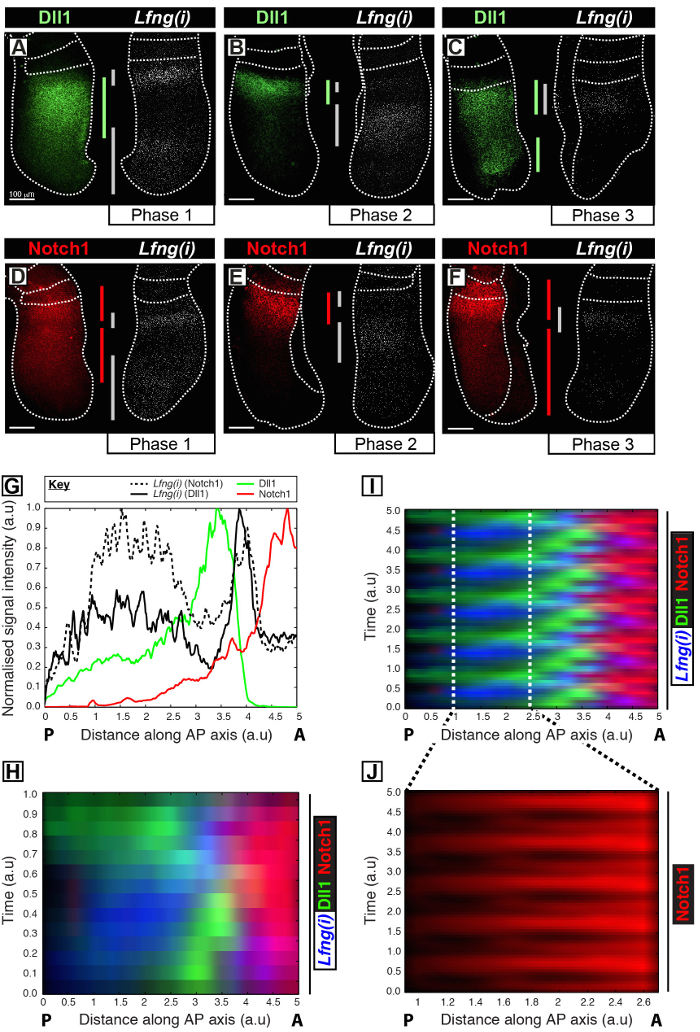
그림 1 : 공간 - 시간 시각화 및 DLL1의 정량 및 Notch1 단백질 발현 역학. (AF)를에서의 검출과 함께 반에서 DLL1 단백질 (AC) 또는 Notch1 단백질 (DF)의 공간적 분포를 나타내는 여섯 E10.5 배아 (AF) Lfng 사전의 mRNA (Lfng (I))에서 외식의 쌍 각 쌍 반대측의 절반에 해당. Lfng (I) 식의 공간적 프로파일에 의해 결정되는 패널은 1 상 (A 및 D), 2 단계 (B 및 E) 및 분할 클록 사이클의 단계 3 (C 및 F)에 따라 배열된다. PSM의 안테 - 후방 축 방향 DLL1 (녹색), Notch1 (적색) 및 Lfng (I) (회색)에 대해 식 영역의 범위는 B가색으로 구분 막대로 경계가 EEN. 점선은 가장 최근에 형성된 체절 (S)는 PSM의 바깥 쪽 가장자리와 인접하는 신경 조직 (C 및 E)의 위치를 구별. 스케일 바 (바닥, 각 패널의 AF 왼쪽) 100 μm의를 나타냅니다. (G)이 PSM에 걸쳐 신호 강도의 축 변화를 나타낸 일례 강도 그래프. 데이터는 반대측 이식편에서 Notch1 단백질 (빨간색) (배아 1)뿐만 아니라 Lfng 사전의 mRNA (검은 색 실선)에 비해 한 이식편에 Lfng 사전의 mRNA (블랙 해시 라인)를 나타내는 두 개의 이식편 쌍에서 도시된다 다른 이식편는 반대측 이식편 (배아 2)에 DLL1 단백질 (녹색)에 비해. 측정 된 신호 강도는 (Y 축)의 축 위치에 대해 도시되어있다 (X 축 [A] 왼쪽 오른쪽 후방 PSM [P]에 PSM 전치). 전역 (H) DLL1, Notch1 및 Lfng (I)의 공간 분포를 나타내는 kymograph수많은 된 PSM. kymograph 각 행의 개별 PSM 이식편의 신호 강도를 나타낸다. 행 Lfng 프리 mRNA의 (I)에 도시 된 데이터의 주기적 확장하여 시뮬레이션 다중 클록 진동 해 DLL1, Notch1 및 Lfng I의 시공간적 분포의 시공간적 분포에 따라 시간적 순서에 따라 배열된다 (H) , DLL1과 Notch1 식 역학의 진동 특성을 강조. 꼬리 PSM의 (J) 박동 Notch1 단백질 식 (I)에 도시 된 가상 kymograph 내에서 구분 영역의 확대에 의해 강조된다. 참조 (12)에서 수정 된 것은 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2 : DLL1과 Notch1 단백질 발현의 공간 - 시간적 역학의 정량화. (A)는 예를 들어 강도 플롯 PSM 걸쳐 신호 강도의 축 방향의 변화를 도시한다. 데이터는에서 반 이식편의 반대편 이식편의 절반 Notch1 단백질 (빨간색)뿐만 아니라 Lfng 사전의 mRNA (검은 색 실선)에 비해 한 이식편에 Lfng 사전의 mRNA (블랙 해시 라인)를 나타내는 두 개의 이식편 쌍에서 플롯 두 번째 꼬리의 반대측 이식편의 절반 DLL1 단백질 (녹색)에 비해 두 번째 꼬리. 측정 된 강도 (y 축)의 축 방향 위치에 대해 도시되어있다 (X 축, 입쪽 [A] 왼쪽 오른쪽 꼬리 [P])이다. (BH) Kymographs 수많은 된 PSM 걸쳐 Notch1, DLL1, NICD 및 Lfng (I)의 공간적 분포를 나타낸다. (B 및 C) NICD (B) 및 DLL1 (C) PSM 부에서의 발현; (D 및 E) Lfng (ⅰ) (D) 및 DLL1 (<반대측 이식편 반쪽 강한> E); (F 및 G) 반대측 이식편의 절반에 Lfng (ⅰ) (F) 및 Notch1 (G). 참조 (12)에서 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
프로토콜 내에서 중요한 단계
본 프로토콜은 E10.5 마우스 PSM 외식에 낮은 수준의 단백질 발현 및 진동 역학의 정량 분석을 수행 할 수있는 민감한 방법을 설명합니다. 계내 혼성화 (FISH) 모두 면역 형광위한 강력한 프로토콜은 공 촛점 이미지를 전체 마운트 고해상도 뒤에 다음 이미지 분석 kymographs 시간적 분할하여 PSM 걸쳐 단백질 발현의 시공간 맵을 생성하기. 단백질 및 mRNA의 검출에 높은 신호 대 잡음비는이 기술의 성공을 위해 필수적이다. 케어 3 단계의 중요한 단계에서 65 ° C 세척의 온도를 충분히 세척 단계 동안 효과적으로 모든 솔루션을 교환하고 유지하기 위해주의해야 대하여 효과적인 항체 및 RNA 프로브를 소스에 시간이 걸리는 것이 가장 바람직하다 관심의 대상이 시약을 테스트철저하게이 프로토콜을 시작하기 전에 전체 마운트 샘플에.
수정 및 문제 해결
이 프로토콜을 수행 할 때 발생할 수있는 가장 큰 문제점이 불량 검출 신호 강도 및 품질에서 발생한다. 이것은 각각의 프로토콜의 면역 또는 FISH 단계에 사용되는 항체 또는 RNA 프로브의 효능에 따라 크게 좌우된다. 충분한 신호 검출이 달성되기 전에 상이한 다수의 단계의 최적화를 필요로 할 수있다. 가난한 신호 검출에 대한 하나의 일반적인 원인은 부적절한 고정입니다; 신선한 PFA 또는 PFA 중 하나가 샘플을 고정하는 데 사용됩니다 더 이상보다 주 동안 4 ° C에서 보관하는 것이 필수적이다. 고정 길이도 사용될 항체 또는 RNA 프로브에 따라 최적화를 필요로 할 수있다. 항체를 들어, 가능한 RNA 프로브, 우리는 출판 문학의 상담을 조언하면서, 제조업체의 지침을 따르도록 권장합니다.
, 우리는 특히 클록 Lfng 유전자의 mRNA의 전을 검출하는 RNA 프로브를 사용했다. 인해 다량의 상대적 부족으로 Lfng 프리 mRNA를 검출 양호한 신호 검출을위한 배 식염수 구연산 나트륨 (SSC)를 포함하는 혼성화 믹스 프로브와 인큐베이션 장시간을 필요로한다. 동일한 조건 약하게 발현 된 mRNA를 검출 다른 프로브에 적용 할 수 있으나, 우리의 경험에서보다 안정적인 mRNA의 타겟의 검지는 혼성화 믹스 짧은 프로브 혼성화 공정 및 하부 SSC 농도 (예를 들면, 1.3 배의 SSC)를 요구할 수있다. 면역 및 FISH 모두에 대해, 프로토콜은 제 전체 배아 최적화되어야하고, 항체 또는 프로브의 최적 농도는 실험적으로 결정되어야한다.
기술의 한계
전술 한 바와 같이,이 기술의 성공은 단백질 및 mRNA의 검출의 품질에 크게 의존한다. WE 단백질 및 mRNA의 검출을 향상시킬 수있는 방법에 대한 몇 가지 제안을 설명하지만, 고품질의 형광 신호 검출의 부재 하에서 상기 실험을 진행할 수있는 방법이 없다. 각각의 조직 샘플에서 분석 될 수있는 단백질 표적의 수는 공 초점 레이저 주사 현미경의 분광 분해능에 의해 사용 된 항체의 에피토프에 의해 제한된다. 본 연구에서는 각 샘플 (12)에 DNA 얼룩과 함께 단백질 검출을위한 세 가지 항원 결정기까지 사용할 수 있었다. 현재의 다른 방법은 최대 세 대상 (14)이 증가하기 위해 이용 될 수 있지만,이 프로토콜은 하나의 mRNA 표적의 검출을 허용한다.
기존 / 대체 방법에 대하여 기술의 중요성
여기에 설명 된 방법은 전체 산 PSM 이식편에서 낮은 수준의 단백질 변화를 감지하는 감지 기술을 제공한다. 이러한 역학의 정량화입니다반대측 외식 대응에 알려진 시계 유전자에 대한 FISH를 수행하여 가능합니다. kymographs의 라이브러리는 현재 프레임 내에서 관심 대상의 시공간 발현 동성을 강조 번 분할 클록 사이클에 걸쳐 구성 될 수 생성된다. 다른 것보다이 기술의 주요 차이점은 시간적으로 공평하게 분석 될 신규 클럭 성분의 시공간 식 역학 허용 대용량 데이터 세트를 주문할 계산 자동화의 사용이다. 예를 들어,이 기술은 DLL1 및 Notch1 단백질 및 진동 공동 규제 전체 PSM 걸쳐 얼마나에 대한 통찰력을 제공. 이 상황에서 다른 방법도 면역 염색에 의존하지만,이 방법을 사용하여 알 수 있었다 꼬리의 PSM에 DLL1과 Notch1 단백질 수준에서의 작은 변동을 감지하지 않았다. 대신, 그들은 주동이의 영역 (9)에 가장 강한 표현의 꾸준한 그라데이션을보고 , 10, 11. 단백질의 낮은 수준을 검출하기 위해 요구 될 수있다 - (밤새 달리 오일, 3)이이 프로토콜은 더 긴 차 항체 잠복기를 가지고 있다는 사실에 기인 할 수있다. DLL1 및 Notch1 발현 수준이 입쪽 PSM 비교적 높고,이 화상의 작성자보다 낮게 설정 노광의 샘플 꼬리 단백질 발현을 검출 할 필요가있을 것이다 영향을 미쳤을 수도있다. 하나 더 잠재적 인 불일치는 채프먼 등의 등의 연구에 고정되지 않은 조직의 사용에서 발생한다. ,있는 꼬리 PSM에 DLL1과 Notch1의 일시적 발현이 적은 9 잘 보존되어있다.
기술을 마스터 한 후 미래의 응용 프로그램 또는 방향
이 프로토콜은 마스터되면 높은 처리량 발현 분석 PSM의 관심있는 단백질을 행할 수있다.몇몇 마우스 새끼로부터 생성 PSM 이식편 분석에 필요한 높은 샘플 수를 생성하기 위해 한 번에 처리 될 수있다. 우리는 이들 연구에서 야생형 배아를 사용하고 있지만, 단백질 발현 역학에 하나 또는 그 이상의 요소의 중요성을 평가하기 위해 유전자 변형 된 배아를 사용하여이 분석을 수행 할 수있다. PSM 외에도이 프로토콜은 두 반대측 반부로 구성하고 민감하게 낮은 수준의 단백질 발현 및 진동 역학을 검출하는데 사용될 수있는 다른 시스템에 적용 할 수있다. 반대측 반부 생성 배양 및 노치 활성은 15 패터닝 본 중요한 두 것으로 밝혀졌다 할 수 있기 때문에,이 프로토콜이 적응 될 수있는 하나의 예는 마우스 신경관 동적 단백질 발현의 연구이다. 우리는 다른 시스템에이 프로토콜을 적용하고 향후 개선을위한 피드백을 제공하기 위해 다른 그룹을 권장합니다.
공개
The authors have nothing to disclose.
감사의 말
이 작품은 RAB에 MRC의 학생이기, CSLB에 MRC의 재학 및 JKD (WT089357MA)에 WT 프로젝트 기금에 의해 지원되었다. 이 연구는 또한 환영 신뢰 전략적인 수상 (097945 / Z / 11 / Z)에 의해 지원되었다. 우리는 Lfng RNA 프로브에 대한 DLL1 항체 박사 O. Pourquie의 종류 선물 박사 E. Kremmer 감사합니다.
자료
| Name | Company | Catalog Number | Comments |
| DMEM-F12 | Gibco (ThermoFisher Scientific) | 11320033 | |
| GlutaMAX™-1 (100x) | Gibco (ThermoFisher Scientific) | 35050 | |
| Fetal Bovine Serum, qualified, E.U.-approved, South America origin | Gibco (ThermoFisher Scientific) | 10270106 | |
| Recombinant Human FGF-basic (154 a.a.) | Peprotech | 100-18B | |
| Penicillin/Streptomycin | Gibco (ThermoFisher Scientific) | 15140122 | |
| anti-mouse monoclonal Notch1 antibody^ | BD Pharmingen | 552466 | |
| anti-rat polyclonal Dll1 antibody^* | N/A | N/A | |
| Lfng intronic anti-sense RNA probe^* | N/A | N/A | |
| 16% paraformaldehyde | Pierce (ThermoFischer Scientific) | PI28908 | |
| Proteinase K, recombinant, PCR grade | Roche (Sigma-Aldrich) | 31158 | |
| Phosphate buffered saline (PBS), pH 7.4 | Made in house | N/A | |
| Triton-X 100 | Sigma-Aldrich | T8787 | |
| Bovine serum albumin (BSA) | Sigma-Aldrich | 5470 | |
| Normal goat serum (NGS) (heat-treated) | Gibco (ThermoFisher Scientific) | 16210072 | |
| Hoechst 33342 | ThermoFischer Scientific | H3570 | |
| Tween-20 | Sigma-Aldrich | P9416 | |
| Ethanol | Sigma-Aldrich | 46139 | |
| Glutaraldehyde | Sigma-Aldrich | 340855 | |
| Formamide | Sigma-Aldrich | F9037 | |
| Saline-sodium citrate (SSC) | Sigma-Aldrich | 93017 | |
| EDTA | Sigma-Aldrich | 798681 | |
| tRNA | Roche (Sigma-Aldrich) | 101095 | |
| Heparin | Sigma-Aldrich | H3149 | |
| Tris-buffered saline (TBS) | Made in house | N/A | |
| Blocking Buffer Reagent | Roche (Sigma-Aldrich) | 11096176001 | |
| anti-DIG horseradish peroxidase (HRP) conjugated antibody | Roche (Sigma-Aldrich) | 11207733910 | |
| Tyramide signal amplification (TSA) kit | Perkin Elmer | NEL744001KT | |
| *The Dll1 antibody and RNA probe used in this study are not commercially available. Please see acknowledgements for sources. | |||
| ^Antibodies/RNA probes should be sourced which are applicable to the research interests of the reader. | |||
참고문헌
- Oates, A. C., Morelli, L. G., Ares, S. Patterning embryos with oscillations: structure, function and dynamics of the vertebrate segmentation clock. Development. 139 (4), 625-639 (2012).
- Krol, A. J., Roellig, D., et al. Evolutionary plasticity of segmentation clock networks. Development. 138 (13), 2783-2792 (2011).
- Dequéant, M. -. L., Ahnert, S., et al. Comparison of Pattern Detection Methods in Microarray Time Series of the Segmentation Clock. PLoS ONE. 3 (8), 2856 (2008).
- Bailey, C., Dale, K. . Somitogenesis in Vertebrate Development. , 1-15 (2015).
- Maroto, M., Bone, R. A., Somitogenesis Dale, J. K. Somitogenesis. Development. 139 (14), 2453-2456 (2012).
- Kageyama, R., Masamizu, Y., Niwa, Y. Oscillator mechanism of notch pathway in the segmentation clock. Developmental Dynamics. 236 (6), 1403-1409 (2007).
- Ferjentsik, Z., Hayashi, S., et al. Notch Is a Critical Component of the Mouse Somitogenesis Oscillator and Is Essential for the Formation of the Somites. PLoS Genetics. 5 (9), 1000662 (2009).
- Wiedermann, G., Bone, R. A., Silva, J. C., Bjorklund, M., Murray, P. J., Dale, J. K. A balance of positive and negative regulators determines the pace of the segmentation clock. eLife. 4, 05842 (2015).
- Chapman, G., Sparrow, D. B., Kremmer, E., Dunwoodie, S. L. Notch inhibition by the ligand DELTA-LIKE 3 defines the mechanism of abnormal vertebral segmentation in spondylocostal dysostosis. Human Molecular Genetics. 20 (5), 905-916 (2011).
- Sparrow, D. B., Chapman, G., et al. A Mechanism for Gene-Environment Interaction in the Etiology of Congenital Scoliosis. Cell. 149 (2), 295-306 (2012).
- Okubo, Y., Sugawara, T., Abe-Koduka, N., Kanno, J., Kimura, A., Saga, Y. Lfng regulates the synchronized oscillation of the mouse segmentation clock via trans-repression of Notch signalling. Nature communications. 3, 1141 (2012).
- Bone, R. A., Bailey, C. S. L., et al. Spatiotemporal oscillations of Notch1, Dll1 and NICD are coordinated across the mouse PSM. Development. 141 (24), 4806-4816 (2014).
- Shea, K., Geijsen, N. Dissection of 6.5 dpc mouse embryos. Journal of Visualized Experiments. (2), 160 (2007).
- Denkers, N., García-Villalba, P., Rodesch, C. K., Nielson, K. R., Mauch, T. J. FISHing for chick genes: Triple-label whole-mount fluorescence in situ hybridization detects simultaneous and overlapping gene expression in avian embryos. Developmental Dynamics. 229 (3), 651-657 (2004).
- Stasiulewicz, M., Gray, S. D., et al. A conserved role for Notch signaling in priming the cellular response to Shh through ciliary localisation of the key Shh transducer Smo. Development. 142 (13), 2291-2303 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유