Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Inducible T7 RNA polimerasa mediada Multigene Expression System, pMGX
En este artículo
Resumen
Este estudio describe métodos para la co-expresión mediada por T7 de múltiples genes de un único plásmido en Escherichia coli usando el sistema de plásmido pMGX.
Resumen
La co-expresión de múltiples proteínas es cada vez más esencial para la biología sintética, estudiando los complejos proteína-proteína, y caracterizando y aprovechando las vías biosintéticas. En este manuscrito, se describe el uso de un sistema altamente eficaz para la construcción de operones sintéticos multigénicos bajo el control de una ARN polimerasa T7 inducible. Este sistema permite que muchos genes se expresen simultáneamente a partir de un plásmido. En este caso, un conjunto de cuatro vectores relacionados, pMGX-A, pMGX-hisA, pMGX-K y pMGX-hisK, con el marcador seleccionable de resistencia a la ampicilina o la kanamicina (A y K) y que poseen o carecen de una hexahistidina N-terminal Etiqueta (su) se describen. Se proporcionan protocolos detallados para la construcción de operones sintéticos usando este sistema vectorial junto con los datos correspondientes, mostrando que un sistema basado en pMGX que contiene cinco genes puede ser fácilmente construido y usado para producir las cinco proteínas codificadas en Escherichia coli . Este sistemaEm y el protocolo permite a los investigadores a rutinariamente expresar complejos módulos de múltiples componentes y vías en E. coli .
Introducción
La co-expresión de múltiples proteínas es cada vez más esencial, particularmente en las aplicaciones de biología sintética, donde se deben expresar múltiples módulos funcionales 1 ; En el estudio de complejos proteína-proteína, donde la expresión y la función a menudo requieren co-expresión 2 , 3 ; Y en la caracterización y el aprovechamiento de las vías biosintéticas, en las que cada gen en la vía debe expresarse 4 , 5 , 6 , 7 , 8 . Se han desarrollado varios sistemas para la co-expresión, particularmente en el organismo huésped Escherichia coli , el caballo de trabajo para la expresión de proteínas recombinantes en laboratorio 9 . Por ejemplo, se pueden usar múltiples plásmidos con diferentes marcadores seleccionables para expresar proteínas individuales utilizando una gran cantidad de diferentes ex Vectores de presión 10 , 11 . Los sistemas individuales de plásmido para la expresión de múltiples proteínas han utilizado múltiples promotores para controlar la expresión de cada gen 10 , 12 ; Operones sintéticos, donde múltiples genes se codifican en un solo transcrito 2 , 13 ; O, en algunos casos, un solo gen que codifica un polipéptido que se procesa en última instancia proteolíticamente, produciendo las proteínas deseadas de interés 14 .
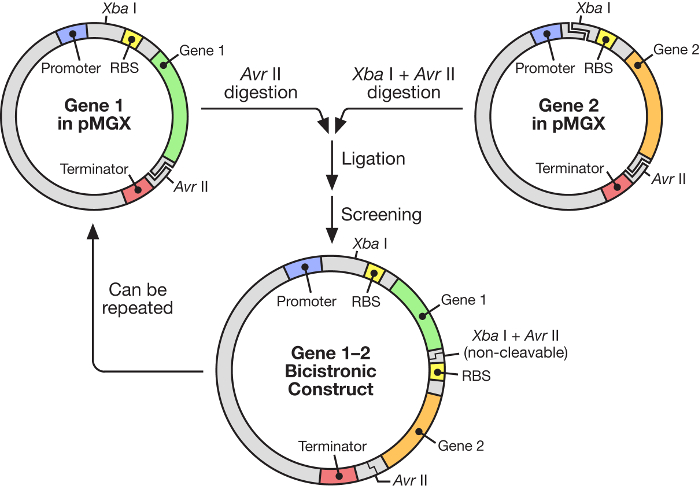
Figura 1: Flujo de trabajo pMGX mostrando la construcción de un vector policistrónico. El sistema pMGX proporciona una estrategia flexible y fácil de usar para la construcción de operones sintéticos bajo el control de un promotor T7 inducible.E.com/files/ftp_upload/55187/55187fig1large.jpg "target =" _ blank "> Haga clic aquí para ver una versión más grande de esta figura.
En este manuscrito, se describe el uso de un sistema altamente eficaz para la construcción de operones sintéticos multigénicos bajo el control de una ARN polimerasa T7 inducible ( Figura 1 ). Este sistema permite que muchos genes se expresen simultáneamente a partir de un plásmido. Se basa en un sistema de plásmidos, originalmente llamado pKH22, que se ha utilizado con éxito para un número de diferentes aplicaciones 6 , 7 , 8 . En este caso, este conjunto de plásmidos se amplía para incluir cuatro vectores relacionados: pMGX-A, un vector de expresión que carece de etiquetas C o N-terminales y con el marcador de resistencia a la ampicilina; PMGX-hisA, un vector de expresión que codifica una etiqueta de hexahistidina N-terminal y con el marcador de resistencia a la ampicilina; PMGX-K, un vector de expresión lMarcar cualquier marcador C o N-terminal y con el marcador de resistencia a la kanamicina; Y pMGX-hisK, un vector de expresión que codifica una etiqueta de hexahistidina N-terminal y con el marcador de resistencia a la kanamicina. En este estudio, se demuestra el método para generar un vector policistrónico que contiene cinco genes usando el sistema pMGX, específicamente pMGX-A, junto con la producción exitosa de cada proteína individual en Escherichia coli .
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Obtención de genes de interés
- Diseño de genes sintéticos.
- Optimizar una secuencia génica para la expresión de E. coli .
- Elimine cualquier sitio de restricción problemático de la secuencia (AvrII, NdeI, EcoRI y XbaI).
- Incorporar sitios de restricción para la clonación; Se recomienda un sitio 5'-NdeI y un sitio 3'-EcoRI. Otros sitios pueden ser usados, si es necesario; Se refieren a la región de clonación múltiple del plásmido seleccionado ( Figura S1 - S4 ). Si se desea, incluir una etiqueta de codificación 5 'o 3' para la detección de transferencia Western.
- Ordena comercialmente el gen diseñado.
- Tanto el clon contundente del gen en un vector romo usando un kit de clonación contundente, según las instrucciones del fabricante; Diseñar cebadores para amplificar el gen (luego proceda a la Etapa 1.2.2); O añadir los extremos 5 'y 3' adicionales para la clonación directa en un plásmido pMGX y proceder al Paso 2. Aquí, thLos resultados representativos son de clonación contundente de los genes de interés.
- Amplificar el gen deseado (a partir de un gen sintético diseñado y optimizado o de ADN molde que contenga el gen deseado) 15 .
- Diseño de cebadores con sitios de restricción para la clonación; Se recomienda un sitio 5'-NdeI y un sitio 3'-EcoRI. Otros sitios pueden ser usados, si es necesario; Se refieren a la región de clonación múltiple del plásmido seleccionado ( Figura S1 - S4 ). Si se desea, incluya una etiqueta de codificación 5 'o 3' para la detección de transferencia Western.
- PCR amplificar el gen deseado 15 .
- Analizar la PCR por electroforesis en gel de agarosa [ 16] .
- Si hay amplificación inespecífica, limpiar el gen amplificado por extracción de gel. Si no, utilice un kit de limpieza enzimática.
- Cuantificar el ADN utilizando un espectrofotómetro comprobando el absorbancE a 260 nm; En blanco con tampón de elución 17 .
2. Clonación de genes de interés en un sistema de expresión multigénico Vector, pMGX 18
- La restricción digiere el gen obtenido de interés y el vector deseado, pMGX, con NdeI y EcoRI.
NOTA: Si se obtiene una gran cantidad de plásmidos recircularizados, permita que la reacción NdeI continúe durante 1 h antes de añadir EcoRI.- Utilizar una reacción digestiva de 40 μl que contenga 0,5-1,5 μg de ADN y 1 μl de cada enzima con 4 μl del tampón 10x apropiado.
- Para un digesto de NdeI y EcoRI, utilice tampón EcoRI y primero añada endonucleasa NdeI. Recuento durante 1 h a 37 ° C. Luego, añada la endonucleasa EcoRI, y permita que la digestión continúe durante una hora adicional. NdeI es sensible a las divisiones cerca del final del ADN, por lo que la digestión inicial por EcoRI puede prevenir la digestión eficaz por NdeI.
- ElectrOphorese la digestión de restricción en gel de agarosa (para un gen de 1 kb, use un gel de agarosa al 0,7% a 110 V durante 55 min, para seleccionar el porcentaje de gel de agarosa para diferentes tamaños de genes, consulte la referencia 16 .
- Inserte las bandas del inserto y del vector usando un bisturí limpio o una cuchilla de afeitar y coloque el segmento de gel extirpado en un tubo de 1,5 ml.
- Extraer el ADN del gel de agarosa usando un kit de extracción de gel de acuerdo con el protocolo del fabricante.
- Ligar el gen de interés en el vector pMGX usando una relación de inserto a vector de 3: 1; Establecer una ligación negativa de pMGX digerido sin el inserto.
- Se prepara una reacción de ligadura de 20 μL que contiene 1 μl de ADN ligasa T4, 0,15-0,5 μg de ADN vector (~ 5 μl) y una cantidad apropiada de inserto basada en una relación inserto-vector de 3: 1 y el tamaño de El gen que se inserta; Las espinas dorsales pMGX tienen entre 5,312 y 5,504 pb de tamaño. Incluir una reacción de control negativo que contiene todo buT el gen que se está insertando. Asegúrese de que la cantidad de ADN vector es equivalente en la ligación de control negativo y en las reacciones de ligamiento de inserto más vector.
- Transformar reacciones de ligación en células de E. coli químicamente competentes XL1-Blue y plásmidos ligados pMGX- yfg1 (que contiene el gen de interés 1), pMGX- yfg2 (que contiene el gen de interés 2) ... pMGX- yfgn (que contiene el gen de interés n). Use técnica aséptica (dentro de un gabinete de bioseguridad o bajo una llama).
- Descongelar 100 μL de alícuotas de E. coli XL1-Blue químicamente competente en hielo durante 5 min y luego añadir 5 μl de las reacciones de ligación. Incubar durante 30 minutos en hielo.
- Calentar el calor de las células durante 45 s en un baño de agua se mantiene a 42 ° C y luego añadir 200 μ l de frío LB. Incubarlos en hielo durante 2 min.
- Agitar las células a 37 ° C, 220 rpm durante 1 h y luego extender la placa de 100 μL sobre una placa de LB-agar que contiene unMarcador seleccionable apropiado (ya sea ampicilina o kanamicina).
- Examinar los clones de los transformantes positivos.
- Comparar los recuentos de colonias en las placas de control negativo y ligadura. Se desea una proporción de recuento de colonias mayor que 1: 2. Si hay un gran número de colonias en la placa de control negativo, vuelva al Paso 2.1 y revise la nota.
- Se seleccionan 4-8 colonias individuales (dependiendo del control negativo: relación de ligación) de cada reacción de ligación de los diferentes genes insertados para el cribado (1 n) e inoculan un LB de 4 ml y el cultivo / colonia individual de antibiótico durante la noche. Cultivar los cultivos durante la noche con agitación a 37 ° C y 220 rpm.
- Aislar el ADN del plásmido usando un kit de aislamiento de ADN plasmídico de acuerdo con el protocolo del fabricante.
- Se prepara una reacción de digestión con NdeI + EcoRI de 20 μl que contiene 150-500 ng de ADN y 1 μl de cada enzima con 2 μl del tampón 10x apropiado. EnEs caso, añadir NdeI y EcoRI al mismo tiempo que el gen de interés estará entre los sitios de restricción. Se recomienda un control negativo con el vector pMGX vacío. Se realiza la digestión durante 2 ha 37 ° C en un baño de agua.
3. Inserción del gen 2 en el vector pMGX que contiene el gen 1, pMGX- yfg1
- Restriction digerir pMGX- yfg1 con AvrII y tratar con fosfatasa intestinal de ternera (CIP).
- Utilice una reacción digestiva de 40 μl que contenga 0,5-1,5 μg de ADN vectorial (~ 5-10 μL de ADN aislado) y 1 μl de AvrII con 4 μl del tampón 10x apropiado. Digerir durante 1,5 h a 37 ° C y luego añadir 1,5 μ l de CIP. Dejar a 37 ° C durante 30 minutos más.
- Restriction digerir pMGX- yfg2 con AvrII y XbaI para liberar el gen de interés.
- Utilizar una reacción digestiva de 40 μl que contenga 0,5-1,5 μg de ADN y 1 μ; L de cada enzima con 4 μl del apropiado tampón 10x. Digerir durante 2 h a 37 ° C.
- La restricción electroforosa se digiere sobre un gel de agarosa de porcentaje (0,7%) y elimina las bandas de inserto y vector usando un bisturí limpio / cuchilla de afeitar (consulte los pasos 2.2-2.3).
- Extraer el ADN utilizando un kit de extracción de gel de acuerdo con el protocolo del fabricante y cuantificar el ADN 17 .
- Ligate gen 2 en pMGX- yfg1 utilizando una proporción de inserto a vector de 3: 1. Establezca una ligación negativa de pMGX - yfg1 digerido sin el inserto adicional. Configure como se indica en el Paso 2.5.
- Transformar 5 μL de las reacciones de ligación en células de E. coli químicamente competentes de XL1-Blue, plasmidos ligados pMGX- yfg1,2 (que contienen el gen de interés 1 y 2) y control negativo de pMGX- yfg1 , como se observa en el Paso 2.6.
- Comparar el recuento de colonias en las placas de control negativo y ligadura. Una proporción de recuento de colonias gMás de 1: 2. Si hay un gran número de colonias en la placa de control negativo, vuelva al Paso 3.1 y revise el tratamiento con CIP.
- Se seleccionan 4-8 colonias individuales (dependiendo del control negativo: relación de ligación) de la reacción de ligación e inoculan 4 mL de LB más el antibiótico apropiado por colonia individual y crecen durante la noche a 37ºC y 220 rpm.
- Aislar el ADN plasmídico utilizando un kit de aislamiento de ADN plasmídico de acuerdo con el protocolo del fabricante y cuantificar el ADN 17 .
- Examinar la inserción efectiva del segundo gen mediante la realización de una digestión de restricción de pMGX- yfg1,2 con EcoRI.
- Utilizar una reacción digestiva de 20 μL que contenga 150-500 ng de ADN y 1 μl de enzima EcoRI con 2 μl de tampón EcoRI 10x. Digerir durante 2 h a 37 ° C.
- Electroforese el digesto de restricción en un gel de agarosa de porcentaje apropiado; Busca una banda que correspondaS al tamaño del gen 2 (véase el paso 2.2). Un gen puede insertarse en el vector en la orientación no deseada, invertida.
4. Adición de un tercer gen en el vector pMGX que contiene los genes 1 y 2, pMGX- yfg1,2
- Restriction digerir pMGX- yfg1,2 con AvrII y tratar con CIP, como se ve en el paso 3.1.
- Restriction digerir pMGX- yfg3 con AvrII y XbaI, como se ve en el paso 3.2.
- La electroforesis de la restricción se digiere en un gel de porcentaje de agarosa apropiado y en inserciones de inserto y bandas de vectores usando una cuchilla limpia de bisturí / navaja; Consulte los pasos 2.2-2.3.
- Extraer el ADN del gel de agarosa utilizando un kit de extracción de gel y cuantificar el ADN 17 .
- Ligar el gen 3 en pMGX- yfg1,2 usando una relación de inserto a vector de 3: 1 y establecer una ligación negativa de pMGX - yfg1,2 digerido sin un inserto adicional, como se observa en el Paso 3.5.
- Transformar 5 μL de las reacciones de ligación en XLCélulas de E. coli químicamente competentes de 1-Blue, plásmido ligado pMGX- yfg1,2,3 (que contiene genes de interés 1, 2 y 3), y control negativo de pMGX- yfg1,2 , como se observa en el Paso 2.6.
- Comparar el recuento de colonias en las placas de control negativo y ligadura. Si hay un gran número de colonias en la placa de control negativo, vuelva al paso 4.1 y revise el tratamiento con CIP.
- Seleccionar de 4 a 8 colonias individuales (dependiendo del control negativo: relación de ligación) de la reacción de ligación e inocular 4 ml de LB más el antibiótico apropiado por colonia individual; Crecen durante la noche a 37 ° C y 220 rpm.
- Aísle el ADN plasmídico usando un kit de aislamiento de ADN plasmídico de acuerdo con el protocolo del fabricante.
- Examinar la inserción efectiva del tercer gen mediante la realización de una digestión de restricción de pMGX- yfg1,2,3 con EcoRI, como se observa en el Paso 3.10.
- Electroforese el digesto de restricción en un gel de agarosa de porcentaje apropiado; MiraPara bandas que corresponden a los tamaños del gen 2 y del gen 3 (véase el paso 2.2). Nota: el gen 3 puede insertarse en el vector en la orientación invertida no deseada. Si los genes 2 y 3 son del mismo tamaño, se debe seleccionar otro sitio de digestión de restricción apropiado para el cribado.
NOTA: Repita según sea necesario para cada nuevo gen.
5. Producción de proteínas de interés utilizando un sistema de expresión multigénico y evaluación de la producción mediante Western Blot
- Transforma el clon positivo que contiene todos los genes de interés en E. coli de producción de proteína químicamente competente, tal como BL21- (λDE3).
- Descongelar 100 μL de alícuotas de E. coli BL21- (λDE3) químicamente competente sobre hielo durante 5 min y luego añadir 1 μl del ADN del plásmido clonado positivo; Incubar durante 30 minutos sobre hielo.
- Calentar térmicamente las células durante 45 s en un baño de agua mantenido a 42 ° C y luego añadir 200 μ l de frío LB. Incubar en hielo durante 2 min.
- Agitar las células a 37ºC y 220 rpm durante 1 h y luego extender la placa 100 mu l sobre una placa de LB - agar que contiene el marcador seleccionable apropiado (ya sea ampicilina o kanamicina).
- Expresar la proteína por inducción de isopropil-β-D-1-tiogalactopiranósido (IPTG).
- Seleccionar una colonia aislada de la placa de transformación B21- (λDE3) e inocular 4 mL de LB más el antibiótico apropiado; Crecen durante la noche, agitando a 37 ° C y 220 rpm.
- Inocular una LB de 100 mL más el cultivo antibiótico apropiado usando 1 mL de cultivo durante la noche.
- Crecer a 37 ° C con agitación a 220 rpm a un OD 600 de 0,6.
- Inducir el cultivo con 100 μ M IPTG y crecer durante 15 horas a 25 ° C y 220 rpm.
- Eliminar 1 ml del cultivo y centrifugarlo a 13.000 xg durante 1 min; Desechar el sobrenadante.
- Lyse las células utilizando solución de lisis según las instrucciones del fabricanteY preformar una transferencia Western del lisado celular soluble para determinar si todas las proteínas fueron producidas con éxito [ 19] .
Access restricted. Please log in or start a trial to view this content.
Resultados
En este estudio, el objetivo era co-expresar cinco proteínas de un solo plásmido. Los fragmentos de gen sintéticos optimizados con cinco codones que codifican etiquetas de hexahistidina N- o C-terminal se compraron comercialmente. Los genes sintéticos se amplificaron por PCR y se clonaron individualmente en un vector PCR-blunt y se secuenciaron. Para generar el plásmido policistrónico, los cinco genes de interés se clonaron primero en un plásmido pMGX adecuado, pMGX-A.
Access restricted. Please log in or start a trial to view this content.
Discusión
La co-expresión de múltiples genes es cada vez más esencial, en particular en la caracterización y reconstitución complejas, multigénicas vías metabólicas [ 3 , 4 , 5] . El sistema pMGX hace co-expresión multigén en la rutina de E. coli 6 , 7 , 8 y accesible a diversos investigadores. En este ...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por el Consejo de Investigación de Ciencias Naturales e Ingeniería de Canadá.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
Referencias
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www.jove.com/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados