A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
T7 RNA פולימראז בתיווך Multigene הביטוי מערכת, pMGX
In This Article
Summary
מחקר זה מתאר שיטות עבור T7 בתיווך ביטוי משותף של גנים מרובים מ פלסמיד בודד ב Escherichia coli באמצעות מערכת pMGX פלסמיד.
Abstract
ביטוי משותף של חלבונים מרובים הוא חיוני יותר ויותר עבור ביולוגיה סינתטית, חקר קומפלקסים חלבונים חלבון, אפיון ורתום מסלולים biosynthetic. בכתב יד זה, את השימוש של מערכת יעילה מאוד לבניית multonene סינתטי אופרנים תחת שליטה של פולימרז T7 RNA inducable מתואר. מערכת זו מאפשרת גנים רבים להתבטא בו זמנית פלסמיד אחד. הנה, קבוצה של ארבעה וקטורים הקשורים, pMGX-A, pMGX-hea, pMGX-K, ו pMGX-hek, עם או ampicillin או קנמיצין התנגדות סמן לבחירה (A ו K) או גם בעל או חסר N- מסוף hexahistidine התג (שלו) נחשפים. פרוטוקולים מפורטים לבניית אופטונים סינתטיים באמצעות מערכת וקטורית זו מסופקים יחד עם הנתונים המתאימים, מראים כי מערכת pMGX מבוסס המכיל חמישה גנים ניתן לבנות בקלות ומשמש לייצר את כל חמש חלבונים מקודדים ב Escherichia coli . זה systEm ופרוטוקול מאפשר לחוקרים באופן שגרתי להביע מורכבים מרובי רכיב רכיבים ומסלולים ב E. coli .
Introduction
ביטוי משותף של חלבונים מרובים הוא חיוני יותר ויותר, במיוחד ביישומים ביולוגיה סינתטית, שבו מודולים פונקציונליים מרובים חייב לבוא לידי ביטוי 1 ; בחקר חלבונים חלבונים, שבו הביטוי ואת הפונקציה דורשים לעתים קרובות שיתוף ביטוי 2 , 3 ; וכן אפיון ורתום מסלולים ביוסינתטיים, שבו כל גן במסלול חייב לבוא לידי ביטוי 4 , 5 , 6 , 7 , 8 . מספר מערכות פותחו עבור שיתוף ביטוי, במיוחד האורגניזם המארח Escherichia coli , סוס העבודה עבור ביטוי חלבון רקומביננטי מעבדה 9 . לדוגמה, פלסמידים מרובים עם סמנים לבחירה שונים ניתן להשתמש כדי לבטא חלבונים בודדים באמצעות שפע של שונים לשעבר 12 הצ 12 , 12 . מערכות פלסמיד יחיד עבור ביטוי חלבון מרובים השתמשו גם מספר האמרגן לשלוט על הביטוי של כל גן 10 , 12 ; אופטונים סינתטיים, שבהם מקודדים גנים מרובים על תמליל יחיד 2 , 13 ; או, במקרים מסוימים, גן יחיד קידוד פוליפפטיד כי הוא מעובד proteolytically בסופו של דבר, מניב את החלבונים הרצוי של עניין 14 .
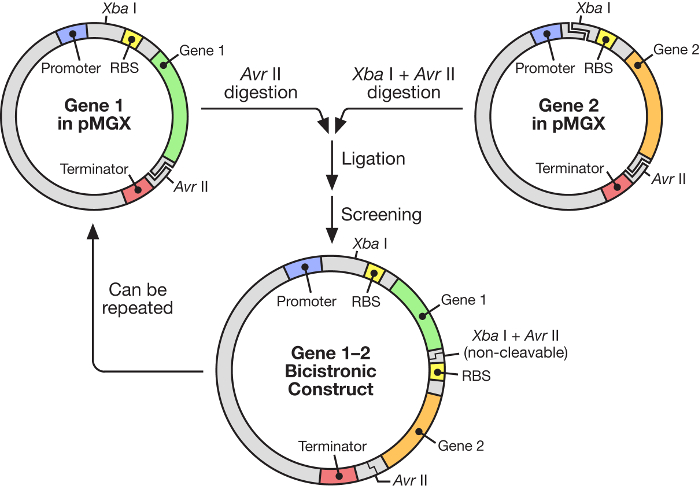
איור 1: זרימת עבודה pMGX מראה את הבנייה של וקטור polycistronic. מערכת pMGX מספקת אסטרטגיה גמישה וקלה לשימוש לבניית אופרונים סינתטיים בשליטתו של מקדם T7."לחץ כאן כדי להציג גרסה גדולה יותר של דמות זו." Target = "_ blank"
בכתב יד זה, את השימוש של מערכת יעילה מאוד לבניית multonene אופטונים סינתטיים תחת שליטה של פולימרז T7 RNA inducible ( איור 1 ) מתואר. מערכת זו מאפשרת גנים רבים להתבטא בו זמנית פלסמיד אחד. הוא מבוסס על מערכת פלסמיד, שנקרא במקור pKH22, כי כבר בשימוש בהצלחה עבור מספר יישומים שונים 6 , 7 , 8 . הנה, זה להגדיר פלסמיד מורחבת לכלול ארבעה וקטורים הקשורים: pMGX-A, וקטור ביטוי חסר כל C- או N- מסוף תגים עם סמן עמיד ampicillin; PMGX-heA, ביטוי וקטור קידוד תג N- מסוף hexahistidine ועם סמן עמיד ampicillin; PMGX-K, וקטור ביטוי lAcking כל C או N- מסוף תגים עם סמן התנגדות kanamycin; ו pMGX-hek, ביטוי וקטור קידוד N- מסוף hexahistidine תג עם סמן התנגדות kanamycin. במחקר זה, השיטה להפקת וקטור polycistronic המכיל חמישה גנים באמצעות מערכת pMGX, במיוחד pMGX-A, הוא הפגינו יחד עם הייצור המוצלח של כל חלבון הפרט Escherichia coli .
Access restricted. Please log in or start a trial to view this content.
Protocol
1. קבלת גנים של עניין
- עיצוב גנים סינתטיים.
- ייעל רצף גנים עבור ביטוי E. coli .
- הסר את כל אתרי ההגבלות הבעייתיים מהרצף (AvrII, NdeI, EcoRI ו- XbaI).
- שילוב אתרי הגבלה לשיבוט; מומלץ לבנות אתר של 5'-NdeI ואתר 3'-EcoRI. ניתן להשתמש באתרים אחרים, במידת הצורך; מתייחסים לאזור multicloning של הפלסמיד הנבחר ( איור S1- S4 ). אם תרצה, הוסף תג קידוד בגודל 5 או 3 עבור זיהוי כתמים מערביים.
- באופן מסחרי להזמין את הגן המעוצב.
- או בוטה לשכפל את הגן לתוך וקטור קהה באמצעות ערכת שיבוט קהה, בהתאם להוראות היצרן; פריימרים בעיצוב כדי להגביר את הגן (ולאחר מכן להמשיך לשלב 1.2.2); או להוסיף 5 'ו 3' מסתיים נוספים שיבוט ישיר לתוך pMGX פלסמיד ולהמשיך לשלב 2. כאן,התוצאות הייצוגיות הן מתוך שיבוט קהה של הגנים המעניינים.
- להגביר את הגן הרצוי (מגן מתוכנן וממוטב של סינתטי או מדגם DNA המכיל את הגן הרצוי).
- עיצוב primers עם אתרי הגבלה לשיבוט; מומלץ לבנות אתר של 5'-NdeI ואתר 3'-EcoRI. ניתן להשתמש באתרים אחרים, במידת הצורך; מתייחסים לאזור multicloning של הפלסמיד הנבחר ( איור S1- S4 ). אם תרצה, הוסף תג קידוד של 5 או 3 לזיהוי כתמים מערביים.
- PCR להגביר את הגן הרצוי 15 .
- ניתוח PCR על ידי agarose ג'ל אלקטרופורזה 16 .
- אם יש הגברה unspecification, לנקות את הגן מוגבר על ידי מיצוי ג 'ל. אם לא, השתמש ערכת אנזים ניקוי.
- לכמת את ה- DNA באמצעות ספקטרופוטומטר ידי בדיקת absorbancE ב 260 ננומטר; ריק עם חיץ elution 17 .
2. שיבוט גנים של עניין לתוך מערכת ביטוי multigene וקטור, pMGX 18
- הגבלת digest הגן שהושג של עניין ואת וקטור הרצוי, pMGX, עם NdeI ו EcoRI.
הערה: אם כמות גדולה של פלסמידים recircularized מתקבלים, לאפשר תגובה NdeI להמשיך במשך שעה 1 לפני הוספת EcoRI.- השתמש בתגובת 40 לעכל μL המכיל 0.5-1.5 מיקרוגרם של ה- DNA ו 1 μL של אנזים עם 4 μL של חיץ 10x המתאים.
- עבור Ndei ו- EcoRI digest, השתמש במאגר EcoRI והוסף תחילה endonuclease NDEI. תקציר עבור 1 שעה ב 37 ° C. לאחר מכן להוסיף endrouclease EcoRI, ולאפשר digest להמשיך במשך שעה נוספת. NdeI הוא רגיש מחשופים קרוב לסוף הדנ"א, ולכן העיכול הראשוני על ידי EcoRI יכול למנוע עיכול יעיל על ידי NdeI.
- חשמלOphorese הגבלת digest על agarose ג'ל (עבור גן 1 kb, השתמש ג'ל agarose 0.7% ב 110 V עבור 55 דקות, כדי לבחור את אחוז ג'ל agarose עבור גנים שונים בגן, עיין התייחסות 16 .
- הבלו להוסיף להקות וקטור באמצעות אזמל נקי או סכין גילוח ומקום קטע ג'ל שנעלמו לתוך צינור 1.5 מ"ל.
- תמצית דנ"א מג'ל agarose באמצעות ערכת מיצוי ג'ל בהתאם לפרוטוקול היצרן.
- לקשר את הגן של עניין לתוך וקטור pMGX באמצעות יחס 3: 1 להוסיף וקטור; להקים קשירת שלילי של pMGX מתעכל ללא הכנס.
- הגדרת תגובה 20 קשירת μL המכיל 1 μL של ליגז DNA T4, 0.15-0.5 מיקרוגרם של DNA וקטורית (~ 5 μL), ואת הכמות המתאימה של הוספה על בסיס יחס של 3: 1 להוסיף וקטור ואת גודל הגן מוחדר; את pMGX backbones הם בין 5,312 ו 5,504 bp בגודל. כלול תגובה שלילית שליטה המכיל הכל buלא את הגן מוחדר. ודא כי כמות ה- DNA הווקטורית היא המקבילה של קשירת שליטה שלילית ואת וקטור פלוס להוסיף קשירת התגובות.
- שינוי תגובות קשירת לתוך XL1 כחול כחול מוכשר כימית תאים E. coli ו ligated פלסמידים pMGX- yfg1 (המכיל גן של עניין 1), pMGX- yfg2 (המכיל גן של עניין 2) ... pMGX- yfgn (המכיל גן של עניין n). השתמש בטכניקה aseptic (בתוך ארון biosafety או תחת להבה).
- הפשרה 100 aliquots μL של המוסמכת כימית XL1-Blue E. coli על הקרח במשך 5 דקות ולאחר מכן להוסיף 5 μL של התגובות קשירת. דגירה במשך 30 דקות על הקרח.
- מחממים הלם התאים במשך 45 ים באמבט מים שנערך ב 42 מעלות צלזיוס ולאחר מכן להוסיף 200 μL של LB קר. דגירה אותם על הקרח במשך 2 דקות.
- לנער את התאים ב 37 מעלות צלזיוס, 220 סל"ד עבור 1 שעות ולאחר מכן להפיץ צלחת 100 μL על צלחת LB אגר המכיל אישורסמן לבחירה (או אמפיצילין או קנמיצין).
- מסך שיבוטים עבור transformants חיובי.
- השווה את המושבה סופרת על שליטה שלילית צלחות קשירת. יחס ספירת מושבה גדול מ 1: 2 הוא הרצוי. אם יש מספר גדול של מושבות על לוח הבקרה השלילי, לחזור לשלב 2.1 ולסקור את ההערה.
- בחר 4-8 מושבות בודדות (בהתאם שליטה שלילי: קשירת יחס) מכל תגובה קשירת כל הגנים השונים שהוכנסו להקרנה (1 n) ו לחסן 4 מ"ל LB ואת התרבות המתאימה אנטיביוטיקה לילה / מושבה אישית. לגדול תרבויות לילה עם רועד על 37 מעלות צלזיוס 220 סל"ד.
- בידוד DNA פלסמיד באמצעות ערכת בידוד DNA פלסמיד על פי פרוטוקול של היצרן.
- הגדרת 20 NDEI + תגובה EcoRI digest μL המכיל 150-500 ng של DNA ו 1 μL של כל אנזים עם μL 2 של חיץ 10x המתאים. בהוא מקרה, להוסיף NdeI ו EcoRI באותו זמן כי הגן של עניין יהיה בין אתרי הגבלה. שליטה שלילית עם וקטור pMGX ריק מומלץ. תקציר עבור 2 שעות ב 37 מעלות צלזיוס באמבט מים.
3. הוספת ג'ין 2 לתוך pMGX וקטור המכיל את ג 'ין 1, pMGX- yfg1
- הגבלת digest pMGX- yfg1 עם AvrII ולטפל עם phosphatase המעי העגל (CIP).
- השתמש בתגובת 40-μL digest המכיל 0.5-1.5 מיקרוגרם של DNA וקטורית (~ 5-10 μL של DNA בודד) ו 1 μL של AvrII עם 4 μL של חיץ 10x המתאים. תקציר עבור 1.5 שעות ב 37 ° C ולאחר מכן להוסיף 1.5 μL של CIP. להשאיר אותו על 37 מעלות צלזיוס למשך 30 דקות נוספות.
- הגבלה digest pMGX- yfg2 עם AvrII ו XbaI לשחרר את הגן של עניין.
- השתמש בתגובת 40 לעכל μL המכיל 0.5-1.5 מיקרוגרם של DNA ו 1 μ, L של כל אנזים עם 4 μL של חיץ המתאים 10x. תקציר עבור 2 שעות ב 37 ° C.
- הגבלה אלקטרופורזה מתעכל על אחוז המתאים (0.7%) agarose ג'ל ובלו את להקות להוסיף וקטור באמצעות סכין מנתח נקי / סכין גילוח (עיין צעדים 2.2-2.3).
- חלץ את ה- DNA באמצעות ערכת מיצוי ג'ל בהתאם לפרוטוקול של היצרן לכמת את ה- DNA 17 .
- לקשור גן 2 לתוך pMGX- yfg1 באמצעות יחס 3: 1 להוסיף וקטור. הגדרת קשירת שלילי של מתעכל pMGX- yfg1 ללא הוספה נוספת. הגדר כנ"ל בשלב 2.5.
- המרה 5 μL של התגובות קשירת לתוך XL1 כחול כחול מוכשר כימית תאים E. coli , פלסמידים ligated pMGX- yfg1,2 (המכיל גן של עניין 1 ו -2), ושליטה שלילית pMGX- yfg1 , כפי שניתן לראות בשלב 2.6.
- השווה את המושבה לספור על שליטה שלילית צלחות קשירת. יחס ספירת מושבות זReater יותר מ 1: 2 הרצוי. אם יש מספר רב של מושבות על לוח הבקרה השלילי, לחזור שלב 3.1 ולסקור את הטיפול CIP.
- בחר 4-8 מושבות בודדות (בהתאם שליטה שלילית: יחס קשירת) מן התגובה קשירת לחסן 4 מ"ל של LB בתוספת אנטיביוטיקה המתאימה לכל מושבה הפרט לגדול בן לילה ב 37 מעלות צלזיוס 220 סל"ד.
- בידוד DNA פלסמיד באמצעות ערכת בידוד DNA פלסמיד על פי פרוטוקול של היצרן לכמת את ה- DNA 17 .
- מסך ההכנסה יעילה של הגן השני על ידי ביצוע digest הגבלה של pMGX- yfg1,2 עם EcoRI.
- השתמש בתגובת 20 לעכל μL המכיל 150-500 ng של DNA ו 1 μL של אנזים EcoRI עם μL 2 של חיץ EcoRI 10x. תקציר עבור 2 שעות ב 37 ° C.
- Electrophorese הגבלה digest על אחוז המתאים agarose ג'ל; לחפש להקה שמתאימהS לגודל הגן 2) ראה שלב 2.2 (. גן יכול להכניס לתוך הווקטור בכיוון הלא רצוי, הפוכה.
4. הוספת ג 'ין השלישי לתוך וקטור pMGX המכילים גנים 1 ו -2, pMGX- yfg1,2
- הגבלה digest pMGX- yfg1,2 עם AvrII ולטפל עם CIP, כפי שניתן לראות בשלב 3.1.
- הגבלה digest pMGX- yfg3 עם AvrII ו XbaI, כפי שניתן לראות בשלב 3.2.
- Electrophorese הגבלה digests על אחוז המתאים agarose ג'ל ו הבלו להוסיף ווקטור להקות באמצעות סכין מנתח נקי / סכין גילוח; עיין בשלבים 2.2-2.3.
- חלץ את ה- DNA מן ג'ל agarose באמצעות ערכת מיצוי ג'ל לכמת את ה- DNA 17 .
- לקשור גן 3 לתוך pMGX- yfg1,2 באמצעות יחס 3: 1 להוסיף וקטור ולהגדיר קשירת שלילי של מתעכל pMGX- yfg1,2 ללא הוספה נוספת, כפי שניתן לראות בשלב 3.5.
- המרה 5 μL של תגובות קשירת לתוך XL1-כחול מוכשר כימית תאים E. coli , ligated פלסמיד pMGX- yfg1,2,3 (המכיל גנים של עניין 1, 2, ו -3), וכן שלילי pMGX- yfg1,2 שליטה, כפי שניתן לראות בשלב 2.6.
- השווה את המושבה לספור על שליטה שלילית צלחות קשירת. אם יש מספר רב של מושבות על לוח הבקרה השלילי, לחזור שלב 4.1 ולסקור את הטיפול CIP.
- בחר 4-8 מושבות בודדות (בהתאם שליטה שלילית: יחס קשירת) מן התגובה קשירת לחסן 4 מ"ל של LB בתוספת אנטיביוטיקה המתאימה לכל מושבה הפרט; לגדול בן לילה ב 37 מעלות צלזיוס 220 סל"ד.
- לבודד את ה- DNA פלסמיד באמצעות ערכת בידוד DNA פלסמיד על פי פרוטוקול של היצרן.
- מסך ההכנסה יעילה של הגן השלישי על ידי ביצוע תקציר digest של pMGX- yfg1,2,3 עם EcoRI, כפי שניתן לראות בשלב 3.10.
- Electrophorese הגבלה digest על אחוז המתאים agarose ג'ל; תראהעבור להקות המתאימות לגדלים של גן 2 ו גן 3 (ראה שלב 2.2). הערה: הגן 3 יכול להכניס לתוך הווקטור בכיוון הלא רצוי, הפוכה. אם הגנים 2 ו- 3 הם באותו גודל, יש לבחור אתר אחר של עיכוב הגבלה מתאים להקרנה.
הערה: חזור על פי הצורך בכל גן חדש.
5. הפקת חלבונים של עניין באמצעות מערכת ביטוי multigene ו הערכת הייצור על ידי סופג המערבי
- לשנות את שיבוט חיובי המכיל את כל הגנים של עניין לתוך מוכשר מבחינה כימית, חלבון-ייצור E. coli , כגון BL21- (λDE3).
- הפשרה 100 aliquots μL של BL21- מוכשר כימית (λDE3) E. coli על קרח במשך 5 דקות ולאחר מכן להוסיף 1 μL של DNA פלסמיד משובץ חיובי; דגירה במשך 30 דקות על הקרח.
- הלם חום התאים במשך 45 ים באמבט מים שנערך ב 42 מעלות צלזיוס ולאחר מכן להוסיף 200 μL של LB קר. דגירה על קרח במשך 2 דקות.
- לנער את התאים ב 37 מעלות צלזיוס 220 סל"ד במשך 1 שעות ולאחר מכן להפיץ צלחת 100 μL על צלחת LB אגר המכיל את סמן לבחירה המתאים (או ampicillin או kanamycin).
- להביע את החלבון על ידי איזופרופיל-β-D-1-thiogalactopyranoside (IPTG) אינדוקציה.
- בחר מושבה מבודדת מן B21- (λDE3) שינוי צלחת לחסן 4 מ"ל של LB בתוספת אנטיביוטי מתאים; לגדול בן לילה, רועד על 37 מעלות צלזיוס 220 סל"ד.
- לחסן 100 מ"ל LB בתוספת תרבות אנטיביוטי מתאים באמצעות 1 מ"ל של תרבות לילה.
- לגדול ב 37 מעלות צלזיוס עם רועד ב 220 סל"ד ל 600 OD של 0.6.
- לעורר את התרבות עם 100 מיקרומטר IPTG ולגדול במשך 15 שעות ב 25 ° C ו 220 סל"ד.
- הסר 1 מ"ל של התרבות צנטריפוגה זה ב XG 13,000 במשך דקה 1; להשליך את supernatant.
- Lyse התאים באמצעות פתרון תמוגה לפי הוראות היצרןו preform כתם המערבי של lysate תא מסיס כדי לקבוע אם כל החלבונים הופקו בהצלחה 19 .
Access restricted. Please log in or start a trial to view this content.
תוצאות
במחקר זה, המטרה היתה לשיתוף חמישה חלבונים מתוך פלסמיד אחד. חמש קודון אופטימיזציה שברי גנים סינתטיים קידוד או N- או מסוף hexahistidine תגים נרכשו מסחרית. הגנים הסינתטיים היו מוגבר על ידי PCR ו משובטים בנפרד לתוך וקטור PCR-blunt ו sequenced. כדי ליצור את polycistronic פלסמיד, ?...
Access restricted. Please log in or start a trial to view this content.
Discussion
ביטוי משותף של גנים מרובים הוא חיוני יותר ויותר, במיוחד באפיון מחדש מסלולים מורכבים, multigene מטבולי 3 , 4 , 5 . מערכת pMGX עושה ביטוי multigene שיתוף ב E. קולי שגרת 6 , 7 , 8 ונגי...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין מה לגלות.
Acknowledgements
עבודה זו נתמכה על ידי מדעי הטבע והנדסה מועצת המחקר של קנדה.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| Enzymes | |||
| Alkaline Phosphatase, Calf Intestinal (CIP) | New England Biolabs | M0290S | |
| AvrII | New England Biolabs | R0174S | |
| EcoRI | New England Biolabs | R0101S | |
| NdeI | New England Biolabs | R0111S | |
| XbaI | New England Biolabs | R0145S | |
| Herculase II Fusion DNA Polymerase | Agilent Technologies | 600677 | |
| T4 DNA Ligase | New England Biolabs | M0202S | |
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 1 kb DNA ladder | New England Biolabs | N3232L | |
| 4-20% Mini-PROTEAN TGX Stain-Free Protein Gels | Bio-Rad | 456-8095 | |
| 50x TAE | Fisher Thermo Scientific | BP1332-4 | |
| Agar | Fisher Thermo Scientific | BP1423-500 | |
| Agarose | Fisher Thermo Scientific | BP160-500 | |
| Ampicilin | Sigma-Alrich | A9518-5G | |
| BL21 (DE3) chemically comeptent cells | Comeptent cell prepared in house | ||
| B-PER Bacterial Protein Extraction Reagent | Fisher Thermo Scientific | PI78243 | |
| dNTP mix | Agilent Technologies | Supplied with polymerase | |
| Gel Extraction Kit | Omega | D2500-02 | E.Z.N.A Gel Extraction, supplied by VWR Cat 3: CA101318-972 |
| Glycine | Fisher Thermo Scientific | BP381-1 | |
| His Tag Antibody [HRP], mAb, Mouse | GenScript | A00612 | |
| Immobilon Western Chemiluminescent HRP Substrate | EMD Millipore | WBKLS0100 | |
| IPTG | Sigma-Alrich | 15502-10G | |
| LB | Fisher Thermo Scientific | BP1426-500 | |
| Methanol | Fisher Thermo Scientific | A411-20 | |
| Pasteurized instant skim milk powder | Local grocery store | No-name grocery store milk is adequate | |
| Nitrocellulose membrane | Amersham Protran (GE Healthcare Life Sciences) | 10600007 | Membrane PT 0.45 µm 200 mm x 4 m, supplied by VWR Cat #: CA10061-086 |
| Plasmid DNA Isolation Kit | Omega | D6943-02 | E.Z.N.A Plasmid DNA MiniKit I, supplied by VWR Cat #: CA101318-898 |
| pMGX | Boddy Lab | Request from the Boddy Lab Contact cboddy@uottawa.ca | |
| Primers | Intergrated DNA Technologies | Design primers as needed for desired gene | |
| Synthetic Gene | Life Technologies | Design and optimize as needed | |
| Thick Blot Filter Paper | Bio-Rad | 1703932 | |
| Tris base | BioShop | TRS001.1 | |
| Tween-20 | Sigma-Alrich | P9416-50ML | |
| XL1-Blue chemically competent cells | Comeptent cell prepared in house | ||
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| BioSpectrometer | Eppendorf | RK-83600-07 | |
| Gel box - PAGE | Bio-Rad | 1658005 | Mini-PROTEIN Tetra Vertical Electrophoresis Cell |
| Gel Imager | Alpha Innotech | AlphaImager EC | |
| Incubator-oven | Fisher Thermo Scientific | 11-690-650D | Isotemp |
| Incubator-shaker | Fisher Thermo Scientific | SHKE6000-7 | MaxQ 6000 |
| Personna Razors | Fisher Thermo Scientific | S04615 | |
| Power Pack | Bio-Rad | S65533Q | FB300 |
| Transilluminator | VWR International | M-10E,6W | |
| Thermocylcer | Eppendorf | Z316091 | Mastercycler Personal, supplied by Sigma |
| UV Face-Shield | 18-999-4542 | ||
| Waterbath | Fisher Thermo Scientific | 15-460-2SQ | |
| Western Transfer Apparatus | Bio-Rad | 1703935 | Mini-Trans Blot Cell |
References
- Purnick, P. E. M., Weiss, R. The second wave of synthetic biology: from modules to systems. Nat. Rev. Mol. Cell Biol. 10 (6), 410-422 (2009).
- Bieniossek, C., et al. Automated unrestricted multigene recombineering for multiprotein complex production. Nat. Methods. 6 (6), 447-450 (2009).
- Jochimsen, B., et al. Five phosphonate operon gene products as components of a multi-subunit complex of the carbon-phosphorus lyase pathway. Proc. Natl. Acad. Sci. USA. 108 (28), 11393-11398 (2011).
- Martin, V. J. J., Pitera, D. J., Withers, S. T., Newman, J. D., Keasling, J. D. Engineering a mevalonate pathway in Escherichia coli for production of terpenoids. Nat. Biotechnol. 21 (7), 796-802 (2003).
- Ajikumar, P. K., et al. Isoprenoid pathway optimization for Taxol precursor overproduction in Escherichia coli. Science. 330 (6000), 70-74 (2010).
- Boddy, C. N., Hotta, K., Tse, M. L., Watts, R. E., Khosla, C. Precursor-directed biosynthesis of epothilone in Escherichia coli. J. Am. Chem. Soc. 126 (24), 7436-7437 (2004).
- Lundgren, B. R., Boddy, C. N. Sialic acid and N-acyl sialic acid analog production by fermentation of metabolically and genetically engineered Escherichia coli. Org. Biomol. Chem. 5 (12), 1903-1909 (2007).
- Horsman, M. E., Lundgren, B. R., Boddy, C. N. N-Acetylneuraminic Acid Production in Escherichia coli Lacking N-Acetylglucosamine Catabolic Machinery. Chem. Eng. Commun. 203 (10), 1326-1335 (2016).
- Romier, C., et al. Co-expression of protein complexes in prokaryotic and eukaryotic hosts: experimental procedures, database tracking and case studies. Acta Crystallogr. D, Biol. Crystallogr. 62 (Pt 10), 1232-1242 (2006).
- Tolia, N. H., Joshua-Tor, L. Strategies for protein coexpression in Escherichia coli. Nat. Methods. 3 (1), 55-64 (2006).
- Chanda, P. K., Edris, W. A., Kennedy, J. D. A set of ligation-independent expression vectors for co-expression of proteins in Escherichia coli. Protein Expr. Purif. 47 (1), 217-224 (2006).
- Kim, K. -J., et al. Two-promoter vector is highly efficient for overproduction of protein complexes. Protein Sci. 13 (6), 1698-1703 (2004).
- Tan, S., Kern, R. C., Selleck, W. The pST44 polycistronic expression system for producing protein complexes in Escherichia coli. Protein Expr. Purif. 40 (2), 385-395 (2005).
- Chen, X., Pham, E., Truong, K. TEV protease-facilitated stoichiometric delivery of multiple genes using a single expression vector. Protein Sci. 19 (12), 2379-2388 (2010).
- Lorenz, T. C. Polymerase chain reaction: basic protocol plus troubleshooting and optimization strategies. J. Vis. Exp. (63), e3998(2012).
- Lee, P. Y., Costumbrado, J., Hsu, C. -Y., Kim, Y. H. Agarose gel electrophoresis for the separation of DNA fragments. J. Vis. Exp. (62), (2012).
- Sukumaran, S. Concentration determination of nucleic acids and proteins using the micro-volume BioSpec-nano-spectrophotometer. J. Vis. Exp. (48), (2011).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. Molecular Cloning. J. Vis. Exp. , Available from: https://www.jove.com/science-education/5074/molecular-cloning (2016).
- JoVE Science Education Database. Basic Methods in Cellular and Molecular Biology. The Western Blot. J. Vis. Exp. , Available from: https://www.jove.com/science-education/5065/the-western-blot (2016).
- Laible, M., Boonrod, K. Homemade site directed mutagenesis of whole plasmids. J. Vis. Exp. (27), (2009).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved