Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Método de muestreo hisopo para la Detección de norovirus humano en superficies
En este artículo
Resumen
A macrofoam based sampling methodology was developed and evaluated for the detection and quantification of norovirus on environmental hard surfaces.
Resumen
norovirus humanos son una causa principal de la epidemia y la gastroenteritis esporádica en todo el mundo. Debido a que la mayoría de las infecciones se propagan ya sea directamente a través de la ruta de persona a persona o indirectamente a través de las superficies ambientales o alimentos, fómites contaminados y superficies inanimadas son vehículos importantes para la propagación del virus durante los brotes de norovirus.
Desarrollamos y evaluamos un protocolo utilizando hisopos macroespuma para la detección y tipificación de los norovirus humanos de las superficies duras. En comparación con hisopos con punta de fibra o toallitas antiestáticas, hisopos macroespuma permiten la recuperación de virus (rango de 1,2 a 33,6%) de las superficies de asiento de inodoro de hasta 700 cm 2. El protocolo incluye los pasos para la extracción del virus de los hisopos y una mayor concentración del ARN viral usando columnas de centrifugación. En total, 127 (58,5%) de 217 muestras de frotis que habían sido recogidos de las superficies en los cruceros y centros de atención a largo plazo donde había estado la gastroenteritis por norovirusinformaron dado positivo por norovirus GII por RT-qPCR. De estos 29 (22,8%) podría ser genotipo con éxito. En conclusión, la detección de norovirus en superficies ambientales utilizando el protocolo que hemos desarrollado puede ayudar a determinar el nivel de contaminación del medio ambiente durante los brotes, así como la detección de virus cuando las muestras clínicas no están disponibles; también puede facilitar el seguimiento de la eficacia de las estrategias de remediación.
Introducción
Norovirus humanos son una causa principal de la epidemia y la gastroenteritis aguda esporádica en todo el mundo 1, 2, 3. El virus es muy contagioso y la transmisión ocurre a través de la interacción persona directa a persona o indirectamente por contacto con alimentos contaminados, agua o superficies ambientales. Los norovirus se pueden desprender durante períodos prolongados y prolongan la supervivencia del virus en las superficies ambientales se ha documentado 1, 2, 3. Durante derramamiento pico, miles de millones de partículas de virus son liberadas por gramo de heces, y vómito también contiene un número suficiente de partículas virales para causar la infección 4, 5, 6, 7, 8,ef "> 9, 10. Además, la transferencia del virus entre las superficies inanimadas y la piel humana puede ocurrir fácilmente 2, 11, 12. Por lo tanto, el control de la contaminación ambiental puede ayudar en la investigación de brotes y en la evaluación de la eficacia de la limpieza y procedimientos de desinfección.
Varios protocolos de muestreo ambientales se han descrito para la detección de rotavirus, colifago MS2, calicivirus felino (FCV), y bacteriófago P22 13, 14, 15, 16. Sin embargo, las condiciones de validación descritas en estos estudios, incluyendo la desecación rápida (<1 hora) y la escasez de superficie (25 x 100 cm 2), pueden no representar adecuadamente la configuración del campo. Además, el bajos niveles de contaminación de Enviro esperadasuperficies nmental requieren protocolos que son capaces de detectar muy pocas partículas de virus.
Hemos desarrollado un método de muestreo de superficie a base de macroespuma para la detección y tipificación de norovirus. Este método ha sido validado durante varios brotes de norovirus. El protocolo incluye 1) cómo recolectar muestras de frotis de superficies ambientales (2) mejor manera de mantener la integridad de las muestras durante la recogida y el envío al laboratorio, y 3) las pruebas de laboratorio y tipificación de norovirus.
Protocolo
1. torunda de muestreo en el campo
- Use un par de guantes limpios.
- Medir el tamaño de la zona de muestreo sin tocar la superficie usando una cinta de medición o una regla. Tratar de estimar el área con la mayor precisión posible y llenar un formulario de informe (Cuadro 1).
- Compruebe el kit de raspado de posibles fugas y bolsas de transporte de muestras de etiquetas y kits de hisopos.
- Mueva el hisopo por el área de muestreo como sigue: un golpe en la dirección horizontal, de un solo golpe en la dirección vertical, y un golpe en una dirección diagonal. No limpie un área superficial mayor que 700 cm 2.
- Coloque cada hisopo en un tubo y apretar bien la tapa.
2. Almacenamiento y Transporte de hisopos al laboratorio
- Mantenga hisopos a 4 ° C durante un máximo de 48 h. Si se requiere el almacenamiento durante periodos más largos, almacenar los hisopos a -20 ° C (-70 ° C o).
- Mantener los tubos a 0-4 ° C (es decir,. , Utilizar bolsas de hielo) en un recipiente aislado durante el transporte al laboratorio y se envían dentro de las 24-48 horas de la recolección.
3. La concentración de virus, el ARN viral Extracción y Purificación
NOTA: Todos los pasos de centrifugación usar una centrífuga de mesa a 5.000 xg durante 5 minutos a temperatura ambiente, a menos que se indique lo contrario. Tenga mucho cuidado cuando se trabaja con el tampón universal de extracción de ácidos nucleicos (UNEX). Use gafas o careta.
- Etiquetar un tubo de 15 ml y una columna de ARN Midi para cada muestra. Incluir un control negativo de extracción en cada experimento.
- Para preparar la solución de lisis durante 10 hisopos, mezclar 25 ml de tampón de UNEX con 25 ml de PBST (PBS pH 7,2 que contiene 0,02% de Tween-80).
- Añadir 50 l de suspensión de colifago MS2 (10 6 PFU / l) a 50 ml de solución de lisis.
- Coloque una torunda en un tubo de 15 ml y añadir 5 ml de solución de lisis. Mezclar e incubar durante 10 min a temperatura ambiente.
- Añadir 5 ml de etanol 100% a cada tubo y agitar durante 10 s.
- Retire cuidadosamente el hisopo del tubo de 15 ml (que contiene tampón UNEX) presionando suavemente contra el lado del tubo para eliminar el exceso de líquido y luego desechar el hisopo. Los volúmenes restantes deben ser de entre 9-10 ml.
4. Columna Midi Viral Nucleic Acid Extracción
- Transferir cuidadosamente 4.5 / mezcla de etanol ml UNEX desde el paso 3.6 en una columna de midi, centrifugar y desechar el filtrado.
- Cargar otros 4,5 ml de la misma mezcla en la misma columna, se centrifuga, y desechar el filtrado.
- Para el primer lavado, añadir 3,5 ml de etanol al 70% en la región de Midi columna, centrifugar y desechar el filtrado.
- Para el segundo lavado, añadir otros 3,5 ml de etanol al 70% sobre la columna de Midi. Centrífuga y desechar el filtrado.
- Para el seco-spin, centrifugar las columnas Midi para eliminar todos los restos de etanol (que puede afectar negativamente a su recuperación de ARN viral).
- Colocar la columna de Midi en un nuevo tubo de centrífuga de 15 ml. Añadir 250 l de tampón de elución en la columna de Midi; esperar a 1 min antes de la centrifugación para obtener la mayor recuperación de ARN viral.
- Almacenar el ácido nucleico extraído a -70 ° C o proceder inmediatamente al paso 5.
5. La concentración de ácido nucleico viral utilizando ARN Clean y Concentrador Kits
- Añadir 500 l de tampón de unión de ARN a 250 l de ARN eluido en el paso 4.7 anterior y agitar durante 10 s.
- Añadir 750 l de etanol 100% y agitar durante 10 s.
- Etiquetar una columna de giro para cada muestra. Cargar la muestra de 750 l en una columna de centrifugado.
- Centrifugar las columnas de centrifugación a 12.000 xg durante 1 min. Descartar el flujo continuo.
- Cargar los 750 l restantes sobre una columna de centrifugación y se centrifuga a 12.000 xg durante 1 min.
- Para el pre-lavado, añadir 400 l de tampón RNA Prep a cada columna de centrifugación y centrifugar a 12.000 xg durante 1 min. Descartar el flujo continuo.
- Para el primer lavado, añadir 800 l de ARN tampón de lavado a cada columna de centrifugación y se centrifuga a 12.000 xg durante 1 min. Descartar el flujo continuo.
- Para el segundo lavado, añadir 400 l de tampón de lavado de ARN para hacer girar la columna y se centrifuga a 12.000 xg durante 1 min. Descartar el flujo continuo.
- Para el seco-spin, centrifugar la columna de centrifugación para eliminar todos los restos de tampón de lavado a 12.000 xg durante 2 min.
- Transferir cuidadosamente la columna de centrifugación a un tubo de microcentrífuga de 1,5 ml limpio.
- Añadir tampón de elución de 25 l en la columna de centrifugación y se incuba durante 1 min a temperatura ambiente. Esto aumentará la recuperación del ARN viral de la columna.
- Recoger ARN por centrifugación la columna de centrifugación a 10.000 xg durante 1 min.
- O bien proceder directamente a RT-qPCR (paso 6) o tienda de ARN a -70 ° C.
6. Mutiplex RT-qPCR Detección de Genogrupo I y II Los norovirus, y colifago MS 2 (Cuadro 2)
- superficies de trabajo limpias, pipeteeES, y centrífugas con RNasa solución de descontaminación para reducir la posible contaminación.
- Descongelar los reactivos RT-qPCR en hielo. ARN deshielo en hielo (en un área / habitación separada).
- Determinar el número de reacciones y añadir al menos un 10% más de ellos (por ejemplo, si se necesitan 10 reacciones, que mezcla maestra de 11).
- Vortex componentes de la mezcla maestra individuales, a excepción de 25x enzima RT-qPCR, durante 5 s. Mezclar con cuidado la enzima con un movimiento rápido del tubo con un dedo.
- Brevemente componentes de la mezcla centrífuga maestros para 5 s.
- Preparar la mezcla maestra para la detección en tiempo real de RT-PCR de GI norovirus y GII de acuerdo con las instrucciones del kit y añadir cebadores y sondas de oligonucleótidos de norovirus específico en un 1,5 ml tubo de microcentrífuga (Tabla 3).
- Mezclar la mezcla maestra pipeteando 5-10 veces arriba y abajo (no se recomienda agitación). Alícuota de 22 l de la mezcla maestra en cada pocillo de PCR en tiempo real placa de 96 pocillos.
- Vortex ARN de la muestradurante 5 segundos, y cobrar por breve (5 s) centrifugación.
- Añadir 3 l de muestra de RNA y GI y GII controles positivos a la placa de RT-qPCR (siga la plantilla de la Tabla 2). Añadir 3 l de nucleasa freewater en los pozos sin molde y controles (NTC).
- Sellar la placa en tiempo real con película adhesiva óptica.
- centrifugar con cuidado la placa en tiempo real en 1300 xg durante 1 min para eliminar las burbujas de aire o las gotas de líquido que pueden estar presentes en los pocillos.
- Establecer un instrumento en tiempo real PCR y de establecimiento de las siguientes condiciones de termociclado: 1) etapa de RT durante 10 min. a 45 ° C (2) la activación de Taq polimerasa, 10 min. a 95 ° C y (3) 45 ciclos de 15 s a 95 ° C, y 60 s a 60 ° C.
7. La cuantificación de norovirus en muestras de frotis
- Comprobar los resultados de los controles positivos y negativos para validar los resultados de RT-qPCR (Tabla 3).
- Determina si cada curva estándar se encuentra con elLos valores aceptables dan en la tabla complementaria 2 y calcular la desviación estándar global para la curva estándar utilizando la Ecuación 1.
(Ecuación 1)% de eficiencia = 100 x 10 (promedio de valores de Ct-Intercepción) / pendiente. - Si el software termociclador de PCR no calcula la pendiente de cada curva patrón, determinar la pendiente y los valores R 2 de la regresión utilizando el software estadístico (por ejemplo, Excel, SPSS o SAS). Además, se calcula el% de eficiencia para las curvas de calibración siguiente ecuación I
- Registre el número de copias de ARN calculado por el software termociclador para todas las muestras de prueba en base a las curvas de calibración que cumplen los criterios especificados en la Tabla 4 complementario. Vuelva a ejecutar ninguna de las muestras con las curvas de calibración t sombrero no cumplen con los criterios o tienen controles positivos falsos. Comprobar los valores de Ct de colifago MS2, que se añadió como un control interno para controlar la inhibición de la PCR.
- Determinar el número total de copias de ARN pmuestra er por la multiplicación del número de copias de ARN calculada en la etapa 7.2 por la relación del volumen (25 a 50 l) de eluyente de ARN total a la del ARN (3-5 l) utilizado para la reacción RT-qPCR. Alternativamente, el cálculo de la densidad de RNA dividiendo el número total de copias de ARN por el área de superficie del objeto (cm 2) analizada.
8. El genotipado de muestras en tiempo real RT-PCR positivos por Hemi anidada amplificación por PCR convencional
- Preparar la primera ronda de mezcla maestra para cada conjunto de cebadores de la RT-PCR ensayo (que complementa los cuadros 2 y 5).
- Añadir 5 l de ARN norovirus positiva a 20 l de mezcla maestra.
- Ejecutar la RT-PCR en las siguientes condiciones: (1) RT paso durante 30 min a 42 ° C (2) la activación de Taq polimerasa, 15 min a 95 ° C, y (3) 40 ciclos de 30 s a 95 ° C , 30 s a 50 ° C y 60 seg a 72 ° C. Después de 40 ciclos, se incuba durante 10 min adicionales a 72 ° C.
- Preparar la segunda ronda de mezcla mater para cada conjunto de cebadores de la RT-PCR ensayo (que complementa los cuadros 2 y 5).
- Añadir 23 l maestro de la mezcla y añadir 2 l de productos de primera ronda de RT-PCR (de primera ronda) a cada tubo (lo ideal es el producto de primera ronda debe ser diluido 1/10 en agua libre de RNasa).
- Repita el paso 8.3.
- Preparar un gel de agarosa al 2% en EDTA 100 ml 1x Tris-acetato (Tris 40 mM, ácido acético 20 mM, y EDTA 1 mM, a pH 8,3). Después de disolver de agarosa por calentamiento de la mezcla en un horno de microondas, añadir 10 l de colorante de ácidos nucleicos por 100 ml de gel preparado después de enfriar la mezcla a 60-70 ° C, se vierte el gel e insertar los peines. Deje que el gel se conforme con al menos 30 minutos.
- Mezclar 15 l de cada producto de RT-PCR con 3 l de colorante de carga 6x. Cargar 15 l de cada muestra y a electroforesis el gel de agarosa a 100 V durante 1 h.
- fragmentos objetivo Impuestos Especiales RT-PCR de tamaño apropiado (330 pb para GI y 341 pb para GII) a partir de un gel de lad purificar ARN usando un kit de extracción de gel comercial. El producto de PCR purificado se puede utilizar ahora para la secuenciación de Sanger.
Resultados
La Figura 1 presenta un diagrama de flujo del protocolo de muestreo hisopo. Este protocolo consiste en cuatro pasos principales; 1) la recogida de muestras, 2) de almacenamiento de muestras y el transporte, 3) la purificación de ARN viral y la concentración y 4) ensayo de RT-qPCR y genotipado.
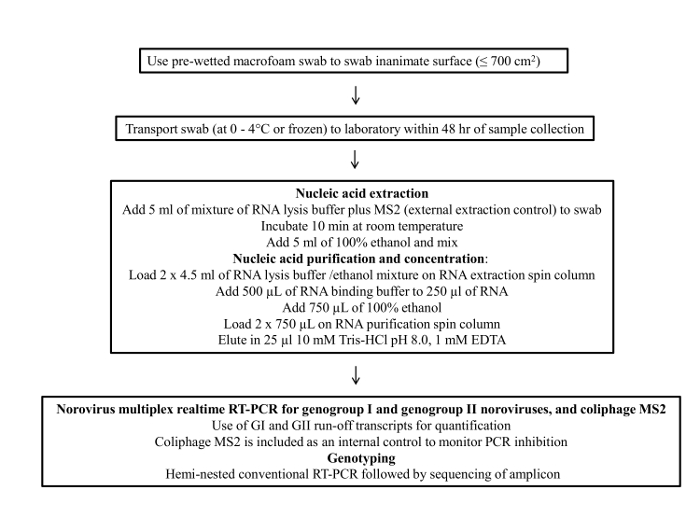
Figura 1: Diagrama de flujo del prot...
Discusión
Los norovirus tienen una dosis infecciosa humana 50% entre 18 y 10 3 20 partículas de virus. Por lo tanto, incluso la contaminación de bajo nivel de las superficies puede suponer un riesgo para la salud pública. Se evaluaron varios aspectos del protocolo de muestreo hisopo incluyendo: 1) diferentes materiales de hisopo, 2) hisopos de condiciones de almacenamiento durante el transporte, 3) la concentración de ARN viral, y 4) colífagos MS2 como el control de la extracción interna. ...
Divulgaciones
Authors have no conflicting interest. The findings and conclusions in this report are those of the authors and do not necessarily represent the official position of the Centers for Disease Control and Prevention.
Agradecimientos
The authors have no acknowledgements.
Materiales
| Name | Company | Catalog Number | Comments |
| Generic name for kits | |||
| Macrofoam swab | Premoistened EnviroMax Swab kit | Puritan | 2588060PFUW |
| RNA Lysis buffer | CDC UNEX buffer | Microbiologics | Cat No MR0501 |
| RNA extraction spin column | Midi column | Omega Biotek | Cat No R6664-02 |
| RNA purification spin column | Zymol RNA Clean and Concentrator kit | Zymo Research | Cat No R1016 |
| Real time RT-PCR kit | AgPath kit One-Step RT-PCR Kit | Life Technologies | Cat No 4387391 |
| Conventional RT-PCR kit | Qiagen one step RT-PCR kit | Qiagen kit | Cat No 210212 |
| Gel extraction kit | Qiagen QIAquick gel extraction kit | Qiagen kit | Cat No 28704 or 28706 |
| Coliphage MS2 | ATCC | Cat No 15597-B1 | |
| RNA run-off transcripts | |||
| Realtime PCR platform | Applied Biosystems | Model ABI 7500 | |
| Optical 96-well reaction plate | Thermo Scientific | Cat No 4316813 | |
| MicroAmp Clear Adhesive Film | Thermo Scientific | Cat No 4306311 |
Referencias
- Isakbaeva, E. T., et al. Norovirus transmission on cruise ship. Emerg. Infect. Dis. 11, 154-158 (2005).
- Lopman, B. A., Gastañaduy, P., Park, G. W., Hall, A. J., Parashar, U. D., Vinjé, P. Environmental transmission of norovirus gastroenteritis. Curr. Opin. Virol. 2 (1), 1-7 (2011).
- Malek, M., et al. Outbreak of norovirus infection among river rafters associated with packaged delicatessen meat, Grand Canyon, 2005. Clin Infect Dis. 48 (1), 31-37 (2009).
- Atmar, R. L., et al. Norwalk virus shedding after experimental human infection. Emerg. Infect. Dis. 14 (10), 1553-1557 (2008).
- Glass, R. I., Parashar, U. D., Estes, M. K. Norovirus gastroenteritis. N. Engl. J. Med. 361 (18), 1776-1785 (2009).
- Park, G. W., et al. Evaluation of a New Environmental Sampling Protocol for Detection of Human Norovirus on Inanimate Surfaces. Appl. Environ. Microbiol. 81 (17), 5987-5992 (2015).
- Barker, J., Jones, M. V. The potential spread of infection caused by aerosol contamination of surfaces after flushing a domestic toilet. J. Appl. Microbiol. 99, 339-347 (2005).
- Tung-Thompson, G., Libera, D. A., Koch, K. L., de Los Reyes, F. L., Jaykus, L. A. Aerosolization of a Human Norovirus Surrogate, Bacteriophage MS2, during Simulated Vomiting. PloS one. 10, 0134277 (2015).
- Atmar, R. L., et al. Determination of the 50% human infectious dose for Norwalk virus. J. Infect. Dis. 209 (7), 1016-1022 (2014).
- Petrignani, M., van Beek, J., Borsboom, G., Richardus, J. H., Koopmans, M. Norovirus introduction routes into nursing homes and risk factors for spread: a systematic review and meta-analysis of observational studies. J. Hosp. Infect. 89 (3), 163-178 (2015).
- . Centers for Disease Control Prevention. Norovirus outbreak in an elementary school--District of Columbia, February 2007. MMWR. Morb. Mortal. Wkly. Rep. 56 (51-52), 1340-1343 (2008).
- Cheesbrough, J. S., Barkess-Jones, L., Brown, D. W. Possible prolonged environmental survival of small round structured viruses. J. Hosp. Infect. 35, 325-326 (1997).
- Julian, T. R., Tamayo, F. J., Leckie, J. O., Boehm, A. B. Comparison of surface sampling methods for virus recovery from fomites. Appl. Environ. Microbiol. 77, 6918-6925 (2011).
- Taku, A., et al. Concentration and detection of caliciviruses from food contact surfaces. J. Food. Prot. 65, 999-1004 (2002).
- Scherer, K., Ellerbroek, L., Schulenburg, J., Johne, R., Klein, G. Application of a swab sampling method for the detection of norovirus and rotavirus on artifically contaminated food and environmental surfaces. Food. Environ. Virol. 1 (42), 42-49 (2009).
- Herzog, A. B., et al. Evaluation of sample recovery efficiency for bacteriophage P22 on fomites. Appl. Environ. Microbiol. 78, 7915-7922 (2012).
- Vega, E., et al. CaliciNet: A Novel Surveillance Network for Norovirus Gastroenteritis Outbreaks in the United States. Emerging Infectious Diseases. 17 (8), 1389-1395 (2011).
- Rolfe, K. J., et al. An internally controlled, one-step, real-time RT-PCR assay for norovirus detection and genogrouping. J Clin Virol. 39 (4), 318-321 (2007).
- Kittigul, L., et al. Norovirus GII-4 2006b variant circulating in patients with acute Thailand during a 2006-2007 study. J. Med. Virol. 82 (5), 854-860 (2010).
- Teunis, P. F., et al. Norwalk virus: how infectious is it. J. Med. Virol. 80 (8), 1468-1476 (2008).
- Wollants, E., et al. Evaluation of a norovirus sampling method using sodium dodecyl sulfate/EDTA-pretreated chromatography paper strips. J. Virol. Methods. 122, 45-48 (2004).
- Weir, M. H., Shibata, T., Masago, Y., Cologgi, D., Rose, J. B. The Effect of Surface Sampling and Recovery of Viruses and Non-Spore Forming Bacteria on a QMRA Model for Fomites. Environ. Sci. Technol. 50 (11), 5945-5952 (2016).
- . Microbiology of food and animal feed-Horizontal method for determination of hepatitis A virus and norovirus in food using real-time RT-PCR. International Organization for Standardization (ISO). , (2013).
- Huslage, K., Rutala, W. A., Sickbert-Bennett, E., Weber, D. J. A quantitative approach to defining "high-touch" surfaces in hospitals. Infect. Control. Hosp. Epidemiol. 31 (8), 850-853 (2010).
- Wu, H. M., et al. A norovirus outbreak at a long-term-care facility: the role of environmental surface contamination. Infect. Control. Hosp. Epidemiol. 26 (10), 802-810 (2005).
- Ikner, L. A., Gerba, C. P., Bright, K. R. Concentration and recovery of viruses from water: a comprehensive review. Food Environ. Virol. 4 (2), 41-67 (2012).
- Gallimore, C. I., et al. Environmental monitoring for gastroenteric viruses in a pediatric primary immunodeficiency unit. J. Clin. Microbiol. 44 (2), 395-399 (2006).
- Ganime, A. C., et al. Dissemination of human adenoviruses and rotavirus species A on fomites of hospital pediatric units. Am J Infect Control. , (2016).
- Verani, M., Bigazzi, R., Carducci, A. Viral contamination of aerosol and surfaces through toilet use in health care and other settings. Am J Infect Control. 42 (7), 758-762 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados