Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Swab Sampling Verfahren zum Nachweis von humanem Norovirus auf Oberflächen
In diesem Artikel
Zusammenfassung
A macrofoam based sampling methodology was developed and evaluated for the detection and quantification of norovirus on environmental hard surfaces.
Zusammenfassung
Menschliche Noroviren sind eine führende Ursache für Epidemie und sporadische Gastroenteritis weltweit. Da die meisten Infektionen entweder direkt über die Person-to-Person Route oder indirekt durch Umwelt Oberflächen oder Lebensmittel übertragen werden, kontaminierte fomites und leblose Oberflächen sind wichtige Vehikel für die Verbreitung des Virus während Norovirusausbrüchen.
Wir entwickelt und evaluiert ein Protokoll Makroschaums Tupfer zum Nachweis und zur Typisierung von humanen Noroviren von harten Oberflächen verwendet wird. Im Vergleich zu faser bestückte Tupfer oder antistatische Wischtücher ermöglichen Makroschaum Tupfer Virus Recovery (Bereich 1,2 bis 33,6%) von Toilettensitzflächen von bis zu 700 cm 2. Das Protokoll enthält die Schritte zur Extraktion des Virus aus den Tupfer und eine weitere Konzentration der viralen RNA unter Verwendung von Spin-Säulen. Insgesamt 127 (58,5%) von 217 Tupferproben, die von den Oberflächen auf Kreuzfahrtschiffen und Langzeitpflegeeinrichtungen, wo Norovirus-Gastroenteritis war gesammelt worden warenpositiv getestet GII Norovirus durch RT-qPCR berichtet. Von diesen 29 (22,8%) erfolgreich genotypisiert werden konnte. Abschließend Nachweis von Noroviren auf Umgebungsoberflächen unter Verwendung des Protokolls wir bei der Bestimmung des Grades der Umweltverschmutzung während der Ausbrüche sowie Nachweis von Virus kann helfen entwickelt, wenn klinische Proben nicht zur Verfügung stehen; es kann auch die Überwachung der Wirksamkeit von Sanierungsstrategien erleichtern.
Einleitung
Menschliche Noroviren sind eine führende Ursache für Epidemie und sporadischen akuten Gastroenteritis weltweit 1, 2, 3. Das Virus ist sehr ansteckend und Übertragung erfolgt durch direkten Mensch zu Mensch Interaktion oder indirekt durch Kontakt mit kontaminierten Lebensmitteln, Wasser oder Umwelt-Oberflächen. Noroviren können für längere Zeit vergossen werden und verlängert das Überleben des Virus auf Umwelt Oberflächen hat 1 dokumentiert, 2, 3. Während der Spitzenabwurf, Milliarden von Viruspartikeln pro Gramm Faeces freigesetzt werden, und Erbrochenem enthält auch eine ausreichende Anzahl von Viruspartikeln 4 Infektion zu verursachen, 5, 6, 7, 8,ef "> 9, 10. Darüber hinaus wird die Übertragung des Virus zwischen leblosen Oberflächen und die menschliche Haut kann leicht 2, 11, 12. Daher Überwachung der Umweltverschmutzung in Ausbruchsuntersuchungen auftreten unterstützen können und in der Wirksamkeit der Clean-up - Bewertung und Desinfektionsverfahren.
Mehrere Umweltprobenahme Protokolle zum Nachweis von Rotavirus, Coliphage MS2, feline Calicivirus (FCV) und Bakteriophagen P22 13, 14, 15, 16 beschrieben worden. Jedoch sind die Validierungsbedingungen in diesen Studien beschrieben, einschließlich der schnellen Austrocknung (<1 h) und kleine Oberflächenbereiche (25 x 100 cm 2), möglicherweise nicht ausreichend Feldeinstellungen repräsentieren. Darüber hinaus ist die geringe Belastung von Enviro erwartetnmental Oberflächen erfordern Protokolle, die der Lage sind, sehr wenige Viruspartikel zu detektieren.
Wir entwickelten eine Makroschaum-basierte Oberfläche Probenahmeverfahren zum Nachweis und zur Typisierung von Norovirus. Dieses Verfahren wurde bei mehreren Norovirusausbrüchen validiert. Das Protokoll enthält 1), wie Tupferproben von Umwelt Oberflächen (2), wie die Integrität der Proben während der Entnahme und Versand an das Labor, und 3) Labortests und Typisierung von Norovirus am besten halten zu sammeln.
Protokoll
1. Swab Probenahme im Feld
- Tragen Sie ein sauberes Paar Handschuhe.
- Messen Sie die Größe des Beprobungsfläche ohne Berührung der Oberfläche mit einem Maßband oder Lineal. Versuchen Sie, den Bereich so genau wie möglich zu ermitteln und füllen Sie ein Berichtsformular (Supplementary Tabelle 1).
- Überprüfen Sie den Tupfer-Kit für mögliche Leckagen und Etikettenprobentransporttaschen und Tupfer-Kits.
- Bewegen des Tupfers über die Flächenabtastung folgendermaßen: einen Hub in horizontaler Richtung, einen Hub in vertikaler Richtung und einem Hub in einer diagonalen Richtung. Seien Sie nicht eine Fläche größer als 700 cm 2 Tupfer.
- Legen Sie jedes Tupfer in ein Röhrchen und ziehen Sie die Kappe.
2. Lagerung und Transport von Putzlappen zum Labor
- Halten Tupfer bei 4 ° C für bis zu 48 h. Falls eine Lagerung für einen längeren Zeitraum erforderlich ist, lagern die Abstriche bei -20 ° C (oder -70 ° C).
- Halten Sie die Röhrchen bei 0-4 ° C (dh. verwenden, Kältepackungen) in einem isolierten Behälter während des Transports zum Labor und werden innerhalb von 24 bis 48 h der Sammlung.
3. Viruskonzentration, Viral RNA Extraktion und Reinigung
HINWEIS: Alle Zentrifugationsschritte verwenden, um eine Tischzentrifuge bei 5.000 × g für 5 Minuten bei Raumtemperatur, sofern nicht anders angegeben. Seien Sie besonders vorsichtig, wenn Sie mit der universellen Nukleinsäure-Extraktion (UNEX) Puffer arbeiten. Schutzbrille oder Gesichtsschutz.
- Beschriften Sie ein 15 ml-Röhrchen und einer RNA-Midi-Säule für jede Probe. Fügen Sie eine negative Extraktionskontrolle in jedem Experiment.
- Die Lyse-Lösung für 10 Tupfer vorzubereiten, mit 25 ml PBST (PBS, pH 7,2, enthaltend 0,02% Tween-80) 25 ml UNEX Puffer mischen.
- Je 50 ul der Coliphage MS2 - Suspension (10 & sup6 ; PFU / & mgr; l) zu 50 ml Lysis - Lösung.
- Legen Sie einen Tupfer in einem 15 ml-Röhrchen und mit 5 ml Lyselösung. Mischen und Inkubation für 10 min bei Raumtemperatur.
- 5 ml 100% Ethanol zu jedem Röhrchen und Vortex für 10 s.
- Vorsichtig den Tupfer aus dem 15 ml-Röhrchen zu entfernen (es enthält UNEX Puffer) es leicht gegen die Seite des Schlauchpress überschüssige Flüssigkeit zu entfernen und dann den Tupfer verwerfen. Die restlichen Mengen sollten zwischen 9-10 ml sein.
4. Midi Säule viralen Nukleinsäureextraktion
- Transfer vorsichtig 4,5 ml UNEX / Ethanol-Mischung aus Schritt 3.6 auf einem Midi-Säule, Zentrifuge und Filtrat verwerfen.
- Legen Sie eine andere 4,5 ml aus der gleichen Mischung auf die gleiche Säule, Zentrifuge und verwerfen Sie das Filtrat.
- Für die erste Wäsche, fügen Sie 3,5 ml 70% Ethanol auf die Midi-Säule, Zentrifuge und das Filtrat verwerfen.
- Für die zweite Wäsche, weitere 3,5 ml 70% Ethanol auf die Midi-Spalte hinzufügen. Zentrifuge und Filtrat verwerfen.
- Für die Trockenschleuder Zentrifugation der Midi Spalten alle Spuren von Ethanol zu entfernen (was sich negativ auf Ihre virale RNA-Gewinnung beeinflussen können).
- Legen Sie die Midi-Spalte in einer neuen 15 ml Zentrifugenröhrchen. In 250 ul Elutionspuffer auf die Midi-Säule; 1 min warten, bevor das Zentrifugieren der höchsten viralen RNA-Gewinnung zu erhalten.
- Die extrahierten Nukleinsäure bei -70 ° C oder sofort zu Schritt 5.
5. Die Konzentration der viralen Nukleinsäure Verwendung von RNA-Clean and Concentrator Kits
- In 500 & mgr; l RNA Binding Buffer zu 250 & mgr; l in Schritt eluierte RNA 4.7 oben und Vortex für 10 s.
- In 750 & mgr; l 100% Ethanol und Vortex für 10 s.
- Beschriften Sie eine Spin-Säule für jede Probe. Laden Sie die 750 & mgr; l Probe auf eine Spin-Säule.
- Zentrifugieren Sie die Spin-Säulen bei 12.000 × g für 1 min. Verwerfen Sie den Durchfluss durch.
- Legen Sie die restlichen 750 & mgr; l auf eine Spin-Säule und Zentrifuge bei 12.000 g für 1 min.
- Für die Vorwäsche, werden 400 & mgr; l RNA Prep Puffer zu jeder Spin-Säule und Zentrifuge bei 12.000 g für 1 min. Verwerfen Sie den Durchfluss durch.
- Für die erste Wäsche, fügen 800 & mgr; l RNA Waschpuffer in jede Spin-Säule und Zentrifuge bei 12.000 g für 1 min. Verwerfen Sie den Durchfluss durch.
- Für das zweite Waschen werden 400 & mgr; l RNA Waschpuffer Säule und Zentrifuge bei 12.000 g für 1 min zu spinnen. Verwerfen Sie den Durchfluss durch.
- Für die Trockenschleuder Zentrifugation der Spin-Säule alle Spuren von Waschpuffer für 2 min bei 12.000 xg zu entfernen.
- Transfer vorsichtig Spin-Säule in ein sauberes 1,5 ml Mikrozentrifugenröhrchen.
- Zugeben von 25 & mgr; l Elutionspuffer auf die spin-Säule und Inkubation für 1 min bei Raumtemperatur. Dadurch wird die Rückgewinnung von viraler RNA aus der Säule zu erhöhen.
- Sammeln RNA durch die Spin-Säule bei 10.000 × g für 1 min zentrifugiert.
- Entweder gehen Sie direkt auf RT-qPCR (Schritt 6) oder Speicher-RNA bei -70 ° C.
6. Mutiplex RT-qPCR Nachweis von Genogruppe I und II Noroviren und Coliphage MS 2 (Supplementary Tabelle 2)
- Saubere Arbeitsflächen, pipettes und Zentrifugen mit RNase Dekontaminationslösung auf eine mögliche Kontamination zu reduzieren.
- Auftauen RT-qPCR-Reagenzien auf Eis. Thaw RNA auf Eis (in einem separaten Bereich / Zimmer).
- Bestimmen Sie die Anzahl der Reaktionen und fügen Sie mindestens 10% mehr von ihnen (zB wenn 10 Reaktionen benötigt werden, machen Master - Mix für 11).
- Vortex einzelnen Master-Mix-Komponenten, mit Ausnahme von 25x RT-qPCR Enzym, für 5 s. durch Ausklopfen der Röhre mit einem Finger vorsichtig das Enzym mischen.
- Kurz Zentrifuge Master-Mix-Komponenten für 5 s.
- Bereiten Sie den Master - Mix für die Echtzeit - RT-PCR - Nachweis von Noroviren GI und GII gemäß dem Kit - Anweisungen und Norovirus-spezifischen Oligonukleotid - Primer und Sonden in einem 1,5 ml Mikrozentrifugenröhrchen (Supplementary Tabelle 3) hinzuzufügen.
- Mischen Sie den Master-Mix durch Pipettieren von 5-10 mal nach oben und unten (Verwirbelung wird nicht empfohlen). Aliquot 22 ul des Master-Mix in jeder Vertiefung der Echtzeit-PCR-96-Well-Platte.
- Vortex Probe RNA5 Sekunden, und sammeln Sie durch kurzes (5 s) Zentrifugation.
- In 3 ul der Probe RNA und GI und GII positive Kontrollen an den RT-qPCR Platte (folgen Vorlage aus Tabelle 2). In 3 ul Nuklease-Freewater in den No-Template-Steuerung (NTC) Brunnen.
- Verschließen Sie die Echtzeit-Platte mit optischen Klebefilm.
- Zentrifuge vorsichtig die Echtzeit-Platte bei 1300 × g für 1 min alle Luftblasen oder Flüssigkeitstropfen zu entfernen, die in den Vertiefungen vorhanden sein können.
- Stellen Sie eine Echtzeit-PCR-Instrument und Set-up folgende Thermozyklisierung Bedingungen: 1) RT-Schritt 10 min. bei 45 ° C (2) Aktivierung der Taq-Polymerase, 10 min. bei 95 ° C und (3) 45 Zyklen von 15 s bei 95 ° C und 60 s bei 60 ° C.
7. Quantifizierung von Norovirus in Tupferproben
- Überprüfen Sie die Ergebnisse der positiven und negativen Kontrollen RT-qPCR Ergebnisse (Ergänzende Tabelle 3) zu validieren.
- Bestimmen Sie, ob jede Standardkurve erfüllt dieakzeptable Werte in Ergänzungs Tabelle 2 und berechnen die Gesamtstandardabweichung für die Standardkurve unter Verwendung von Gleichung 1 gegeben.
(Gleichung 1)% Wirkungsgrad = 100 x 10 (Durchschnitt CT - Werte-Intercept) / Slope. - Wenn die PCR - Thermocycler - Software wird die Steigung nicht für jede Standardkurve zu berechnen, bestimmen Steigung und R 2 Werte , die durch Regression Statistik - Software (zB Excel, SPSS oder SAS). Darüber hinaus berechnen für Standardkurven folgende Gleichung I die% Wirkungsgrad
- Notieren Sie sich die RNA - Kopienzahl von Thermocycler - Software berechnet für alle Testproben auf der Basis von Standardkurven, die den Kriterien in Ergänzende Tabelle 4 erfüllen. Rerun alle Proben mit Standard - t Hut Kurven erfüllen nicht die Kriterien oder haben falsch-positive Kontrollen. Überprüfen Ct-Werte von Coliphage MS2, die als interne Kontrolle zur Überwachung PCR Hemmung aufgenommen.
- Bestimmen Sie die Gesamtzahl der RNA-Kopien per Probe durch die in Schritt 7.2 berechnet, um die RNA-Kopienzahl multipliziert mit dem Verhältnis des Volumens (25 bis 50 & mgr; l) der gesamten RNA Eluent derjenigen der RNA (3 bis 5 & mgr; l), die für die RT-qPCR-Reaktion. Alternativ berechnen die RNA - Dichte durch die Gesamt - RNA - Kopienzahl durch die Objektfläche (cm 2) analysiert geteilt wird .
8. Genotypisierung von Echtzeit-RT-PCR-positiven Proben von Hemi Nested Herkömmliche PCR-Amplifikation
- Bereiten Sie die erste Runde des Master - Mix für jeden Primersatz der RT-PCR - Assays (Supplementary Tabellen 2 und 5).
- In 5 ul Norovirus positive RNA zu 20 & mgr; l Master-Mix.
- Führen Sie die RT-PCR unter den folgenden Bedingungen: (1) RT-Schritt 30 min bei 42 ° C (2) Aktivierung der Taq-Polymerase, 15 min bei 95 ° C, und (3) 40 Zyklen von 30 s bei 95 ° C , 30 s bei 50 ° C und 60 sec bei 72 ° C. Nach 40 Zyklen bei 72 ° C für weitere 10 min inkubiert.
- Bereiten Sie die zweite Runde der mater - Mix für jeden Primersatz der RT-PCR - Assays (Supplementary Tabellen 2 und 5).
- In 23 & mgr; l Master-Mix und mit 2 & mgr; l der ersten Runde RT-PCR-Produkte (von der ersten Runde) zu jedem Röhrchen (idealerweise der ersten Runde Produkt 1/10 in RNase-freiem Wasser verdünnt werden sollte).
- Wiederholen Sie Schritt 8.3.
- Vorbereiten eines 2% igen Agarose-Gel in 100 ml 1x Tris-Acetat-EDTA (40 mM Tris, 20 mM Essigsäure und 1 mM EDTA bei pH 8,3). Nach dem Auflösen von Agarose, indem die Mischung in einem Mikrowellenofen erhitzt wird, 10 ul Nukleinsäurefarbstoff pro 100 ml der vorbereiteten Gel hinzu, nachdem die Mischung auf 60-70 ° C abgekühlt, um das Gel gießen und die Kämme einzufügen. Lassen Sie das Gel für mindestens 30 min absetzen.
- Mischen Sie 15 ul jeder RT-PCR-Produkt mit 3 & mgr; l 6-fachem Beladungs Farbstoff. Last 15 ul jeder Probe und dem Agarosegel elektrophoretisch bei 100 V für 1 h.
- Excise Ziel RT-PCR-Fragmente von geeigneter Größe (330 bp für GI und 341 bp für GII) aus dem Gel eind RNA unter Verwendung eines kommerziellen Gel-Extraktions-Kits reinigen. Das gereinigte PCR-Produkt kann jetzt Sanger-Sequenzierung verwendet werden.
Ergebnisse
Abbildung 1 zeigt ein Flussdiagramm des Tupferprobenahmeprotokoll. Dieses Protokoll besteht aus vier Hauptschritten; 1) Probenentnahme, 2) Probenlagerung und Transport, 3) die virale RNA-Reinigung und Konzentration und 4) RT-qPCR-Assay und die Genotypisierung.
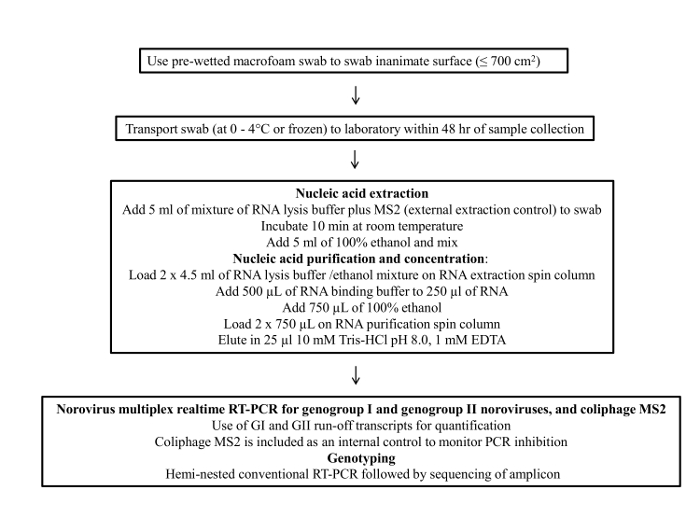
Abbildung 1: Ablaufdiagramm des endgültigen Protokolls für Umweltob...
Diskussion
Noroviren haben eine 50% human infektiöse Dosis zwischen 18 und 10 drei Viruspartikel 20. Deshalb, auch Low-Level-Kontamination von Oberflächen kann eine Gefahr für die Gesundheit darstellen. Verschiedene Aspekte der Tupfer Probenahmeprotokoll wurden einschließlich ausgewertet: 1) verschiedene Tupfer Materialien, 2) Lagerbedingungen Tupfer während des Transports, 3) Virus-RNA-Konzentration und 4) Coliphage MS2 als interne Extraktionskontrolle.
Bis vor ...
Offenlegungen
Authors have no conflicting interest. The findings and conclusions in this report are those of the authors and do not necessarily represent the official position of the Centers for Disease Control and Prevention.
Danksagungen
The authors have no acknowledgements.
Materialien
| Name | Company | Catalog Number | Comments |
| Generic name for kits | |||
| Macrofoam swab | Premoistened EnviroMax Swab kit | Puritan | 2588060PFUW |
| RNA Lysis buffer | CDC UNEX buffer | Microbiologics | Cat No MR0501 |
| RNA extraction spin column | Midi column | Omega Biotek | Cat No R6664-02 |
| RNA purification spin column | Zymol RNA Clean and Concentrator kit | Zymo Research | Cat No R1016 |
| Real time RT-PCR kit | AgPath kit One-Step RT-PCR Kit | Life Technologies | Cat No 4387391 |
| Conventional RT-PCR kit | Qiagen one step RT-PCR kit | Qiagen kit | Cat No 210212 |
| Gel extraction kit | Qiagen QIAquick gel extraction kit | Qiagen kit | Cat No 28704 or 28706 |
| Coliphage MS2 | ATCC | Cat No 15597-B1 | |
| RNA run-off transcripts | |||
| Realtime PCR platform | Applied Biosystems | Model ABI 7500 | |
| Optical 96-well reaction plate | Thermo Scientific | Cat No 4316813 | |
| MicroAmp Clear Adhesive Film | Thermo Scientific | Cat No 4306311 |
Referenzen
- Isakbaeva, E. T., et al. Norovirus transmission on cruise ship. Emerg. Infect. Dis. 11, 154-158 (2005).
- Lopman, B. A., Gastañaduy, P., Park, G. W., Hall, A. J., Parashar, U. D., Vinjé, P. Environmental transmission of norovirus gastroenteritis. Curr. Opin. Virol. 2 (1), 1-7 (2011).
- Malek, M., et al. Outbreak of norovirus infection among river rafters associated with packaged delicatessen meat, Grand Canyon, 2005. Clin Infect Dis. 48 (1), 31-37 (2009).
- Atmar, R. L., et al. Norwalk virus shedding after experimental human infection. Emerg. Infect. Dis. 14 (10), 1553-1557 (2008).
- Glass, R. I., Parashar, U. D., Estes, M. K. Norovirus gastroenteritis. N. Engl. J. Med. 361 (18), 1776-1785 (2009).
- Park, G. W., et al. Evaluation of a New Environmental Sampling Protocol for Detection of Human Norovirus on Inanimate Surfaces. Appl. Environ. Microbiol. 81 (17), 5987-5992 (2015).
- Barker, J., Jones, M. V. The potential spread of infection caused by aerosol contamination of surfaces after flushing a domestic toilet. J. Appl. Microbiol. 99, 339-347 (2005).
- Tung-Thompson, G., Libera, D. A., Koch, K. L., de Los Reyes, F. L., Jaykus, L. A. Aerosolization of a Human Norovirus Surrogate, Bacteriophage MS2, during Simulated Vomiting. PloS one. 10, 0134277 (2015).
- Atmar, R. L., et al. Determination of the 50% human infectious dose for Norwalk virus. J. Infect. Dis. 209 (7), 1016-1022 (2014).
- Petrignani, M., van Beek, J., Borsboom, G., Richardus, J. H., Koopmans, M. Norovirus introduction routes into nursing homes and risk factors for spread: a systematic review and meta-analysis of observational studies. J. Hosp. Infect. 89 (3), 163-178 (2015).
- . Centers for Disease Control Prevention. Norovirus outbreak in an elementary school--District of Columbia, February 2007. MMWR. Morb. Mortal. Wkly. Rep. 56 (51-52), 1340-1343 (2008).
- Cheesbrough, J. S., Barkess-Jones, L., Brown, D. W. Possible prolonged environmental survival of small round structured viruses. J. Hosp. Infect. 35, 325-326 (1997).
- Julian, T. R., Tamayo, F. J., Leckie, J. O., Boehm, A. B. Comparison of surface sampling methods for virus recovery from fomites. Appl. Environ. Microbiol. 77, 6918-6925 (2011).
- Taku, A., et al. Concentration and detection of caliciviruses from food contact surfaces. J. Food. Prot. 65, 999-1004 (2002).
- Scherer, K., Ellerbroek, L., Schulenburg, J., Johne, R., Klein, G. Application of a swab sampling method for the detection of norovirus and rotavirus on artifically contaminated food and environmental surfaces. Food. Environ. Virol. 1 (42), 42-49 (2009).
- Herzog, A. B., et al. Evaluation of sample recovery efficiency for bacteriophage P22 on fomites. Appl. Environ. Microbiol. 78, 7915-7922 (2012).
- Vega, E., et al. CaliciNet: A Novel Surveillance Network for Norovirus Gastroenteritis Outbreaks in the United States. Emerging Infectious Diseases. 17 (8), 1389-1395 (2011).
- Rolfe, K. J., et al. An internally controlled, one-step, real-time RT-PCR assay for norovirus detection and genogrouping. J Clin Virol. 39 (4), 318-321 (2007).
- Kittigul, L., et al. Norovirus GII-4 2006b variant circulating in patients with acute Thailand during a 2006-2007 study. J. Med. Virol. 82 (5), 854-860 (2010).
- Teunis, P. F., et al. Norwalk virus: how infectious is it. J. Med. Virol. 80 (8), 1468-1476 (2008).
- Wollants, E., et al. Evaluation of a norovirus sampling method using sodium dodecyl sulfate/EDTA-pretreated chromatography paper strips. J. Virol. Methods. 122, 45-48 (2004).
- Weir, M. H., Shibata, T., Masago, Y., Cologgi, D., Rose, J. B. The Effect of Surface Sampling and Recovery of Viruses and Non-Spore Forming Bacteria on a QMRA Model for Fomites. Environ. Sci. Technol. 50 (11), 5945-5952 (2016).
- . Microbiology of food and animal feed-Horizontal method for determination of hepatitis A virus and norovirus in food using real-time RT-PCR. International Organization for Standardization (ISO). , (2013).
- Huslage, K., Rutala, W. A., Sickbert-Bennett, E., Weber, D. J. A quantitative approach to defining "high-touch" surfaces in hospitals. Infect. Control. Hosp. Epidemiol. 31 (8), 850-853 (2010).
- Wu, H. M., et al. A norovirus outbreak at a long-term-care facility: the role of environmental surface contamination. Infect. Control. Hosp. Epidemiol. 26 (10), 802-810 (2005).
- Ikner, L. A., Gerba, C. P., Bright, K. R. Concentration and recovery of viruses from water: a comprehensive review. Food Environ. Virol. 4 (2), 41-67 (2012).
- Gallimore, C. I., et al. Environmental monitoring for gastroenteric viruses in a pediatric primary immunodeficiency unit. J. Clin. Microbiol. 44 (2), 395-399 (2006).
- Ganime, A. C., et al. Dissemination of human adenoviruses and rotavirus species A on fomites of hospital pediatric units. Am J Infect Control. , (2016).
- Verani, M., Bigazzi, R., Carducci, A. Viral contamination of aerosol and surfaces through toilet use in health care and other settings. Am J Infect Control. 42 (7), 758-762 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten