É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Swab método de amostragem para a detecção do norovírus Humano em Surfaces
Neste Artigo
Resumo
A macrofoam based sampling methodology was developed and evaluated for the detection and quantification of norovirus on environmental hard surfaces.
Resumo
noroviruses humanos são uma das principais causas da epidemia e gastroenterite esporádica em todo o mundo. Porque a maioria das infecções são ou transmitida diretamente por via da pessoa-a-pessoa ou indirectamente através de superfícies ambientais ou alimentos, fômites contaminados e superfícies inanimadas são veículos importantes para a propagação do vírus durante surtos de norovírus.
Nós desenvolvido e avaliado um protocolo com cotonetes macro-espuma para a detecção e tipagem de norovírus humanos de superfícies duras. Comparado com cotonetes com ponta de fibra ou toalhetes anti-estáticos, compressas macro-espuma permitir a recuperação de vírus (intervalo 1,2-33,6%) das superfícies de assento de sanita de até 700 cm2. O protocolo inclui passos para a extracção do vírus a partir dos cotonetes e ao aumento da concentração do RNA viral utilizando colunas de centrifugação. No total, 127 (58,5%) das 217 amostras de esfregaço que tinham sido recolhidas a partir de superfícies em navios de cruzeiro e instalações de cuidados de longo prazo, onde norovirus gastroenterite tinham sidorelatado testou positivo para GII norovirus por RT-qPCR. Destes 29 (22,8%) poderia ser genotipados com sucesso. Em conclusão, a detecção de norovírus em superfícies ambientais utilizando o protocolo desenvolvido que pode ajudar a determinar o nível de contaminação do meio ambiente durante surtos, bem como a detecção de vírus quando as amostras não estão disponíveis clínicos; pode também facilitar o acompanhamento da eficácia de estratégias de remediação.
Introdução
Noroviruses humanos são uma das principais causas da epidemia e gastroenterite aguda esporádica em todo o mundo 1, 2, 3. O vírus é extremamente contagiosa ea transmissão ocorre por meio de pessoa direta a interacção pessoa ou indirectamente, através de contacto com os alimentos contaminados, água ou superfícies ambientais. Norovírus pode ser derramado por longos períodos e prolongada sobrevivência do vírus em superfícies ambientais tem sido documentada 1, 2, 3. Durante derramamento de pico, bilhões de partículas de vírus são liberados por grama de fezes e vômito também contém um número suficiente de partículas virais para causar a infecção 4, 5, 6, 7, 8,ef "> 9, 10. Além disso, a transferência do vírus entre as superfícies inanimadas e pele humana pode ocorrer facilmente 2, 11, 12. Por isso, o monitoramento de contaminação ambiental pode ajudar na investigação de surtos e na avaliação da eficácia da limpeza e procedimentos de desinfecção.
Vários protocolos de amostragem ambientais foram descritos para a detecção de rotavírus, colifago MS2, calicivírus felino (FCV), e bacteriófago P22 13, 14, 15, 16. No entanto, as condições de validação descritos nestes estudos, incluindo a dessecação rápida (<1 hora) e da superfície reduzida (25 x 100 cm 2), pode não representar adequadamente as configurações de campo. Além disso, a baixos níveis de contaminação de Enviro esperadosuperfícies nmental requerem protocolos que são capazes de detectar muito poucas partículas de vírus.
Foi desenvolvido um método de amostragem de superfície à base de macro-espuma para a detecção e tipagem de norovírus. Este método foi validado durante vários surtos de norovírus. O protocolo inclui 1) como coletar amostras de esfregaço de superfícies ambientais (2) a melhor forma de manter a integridade das amostras durante a recolha e envio para o laboratório, e 3) testes de laboratório e tipagem de norovirus.
Protocolo
1. Swab Amostragem no campo
- Usar um par limpo de luvas.
- Medir o tamanho da área de amostragem, sem tocar na superfície utilizando uma fita de medição ou régua. Tente estimar a área com a maior precisão possível e preencher um formulário de relatório (Tabela Suplementar 1).
- Verifique o kit cotonete para possíveis fugas e amostras rótulo sacos de transporte e kits de esfregaço.
- Mover a haste de toda a área de amostragem, como segue: um acidente vascular cerebral em direcção horizontal, um acidente vascular cerebral em direcção vertical, e um acidente vascular cerebral em um sentido diagonal. Não pincelar uma maior área de superfície de 700 cm2.
- Coloque cada mecha em um tubo e aperte a tampa de segurança.
2. Armazenamento e Transporte de cotonetes para o Laboratório
- Manter cotonetes a 4 ° C durante até 48 h. Se for necessário o armazenamento por períodos mais longos, armazenar os cotonetes a -20 ° C (ou -70 ° C).
- Mantenha os tubos a 0-4 ° C (ou seja,. , Use compressas frias) em um recipiente isolado durante o transporte para o laboratório e navio dentro de 24-48 h da coleta.
3. concentração de vírus, Viral RNA extração e purificação
NOTA: Todos os passos de centrifugação usar uma centrífuga de bancada a 5000 xg durante 5 min à temperatura ambiente, a menos que indicado de outra forma. Tenha cuidado extra quando se trabalha com o tampão universal extração de ácidos nucléicos (UNEX). Usar óculos ou viseira.
- Rotular um tubo de 15 mL e uma coluna RNA Midi para cada amostra. Incluir um controlo de extração negativo em cada experimento.
- Para preparar a solução de lise durante 10 esfregaços, misturar 25 mL de tampão de UNEX com 25 mL de PBST (PBS, pH 7,2, contendo 0,02% de Tween-80).
- Adicionar 50 ul de suspensão colifago MS2 (10 6 PFU / mL) a 50 ml de solução de lise.
- Coloque um algodão embebido em um tubo de 15 ml e 5 ml da solução de Lise. Misturar e incubar durante 10 min à temperatura ambiente.
- Adicionar 5 ml de etanol a 100% a cada tubo e agitar com vortex durante 10 s.
- Cuidadosamente retire o cotonete do tubo de 15 mL (contém tampão UNEX) pressionando-o suavemente contra o lado do tubo para remover o excesso de líquido e, em seguida, descartar o cotonete. Os volumes restantes deve ser entre 9-10 mL.
4. Coluna Midi viral Nucleic Acid Extraction
- Transferir cuidadosamente 4,5 mL UNEX mistura / etanol a partir do passo 3.6 para uma coluna de midi, centrífuga e descartar o filtrado.
- Carregar mais 4,5 ml a partir da mesma mistura na mesma coluna, centrifugação, e descartar o filtrado.
- Para a primeira lavagem, adicionar 3,5 mL de etanol a 70% para a coluna Midi, centrifugar e rejeitar-se o filtrado.
- Para a segunda lavagem, adicionar outros 3,5 mL de etanol a 70% para a coluna Midi. Centrífuga e descartar o filtrado.
- Para o spin seca, centrifugar as colunas Midi para remover todos os vestígios de etanol (que pode afetar negativamente a sua recuperação RNA viral).
- Colocar a coluna Midi em um novo tubo de centrífuga de 15 ml. Adicionar 250 ul de tampão de eluição na coluna Midi; aguarde 1 min antes da centrifugação para obter o maior recuperação de RNA viral.
- Armazenar o ácido nucleico extraído a -70 ° C ou prosseguir imediatamente para a etapa 5.
5. Concentração de Ácido Nucleico Utilizando Viral RNA limpo e Concentrador Kits
- Adicionar 500 uL de ARN de Tampão de Ligação para 250 ul de ARN eluída no passo 4.7 acima e vortex durante 10 s.
- Adicionar 750 uL de 100% de etanol e agitar com vortex durante 10 s.
- Rotular uma coluna de centrifugação para cada amostra. Coloque a amostra de 750 mL para uma coluna de spin.
- Centrifugar as colunas de spin a 12.000 xg por 1 min. Descartar o fluxo de passagem.
- Carregar os restantes 750 uL numa coluna de centrifugação e centrifugar a 12000 xg durante 1 min.
- Para a pré-lavagem, adicionar 400 ul de tampão de ARN Prep para cada coluna de centrifugação e centrifugar a 12000 xg durante 1 min. Descartar o fluxo de passagem.
- Para a primeira lavagem, adicionar 800 uL de ARN tampão de lavagem para cada coluna de centrifugação e centrifugar a 12000 xg durante 1 min. Descartar o fluxo de passagem.
- Para a segunda lavagem, adicionar 400 uL de ARN de Tampão de Lavagem a girar coluna e centrifugar a 12000 xg durante 1 min. Descartar o fluxo de passagem.
- Para o spin seco, centrifugar a coluna de centrifugação para remover todos os vestígios de tampão de lavagem a 12.000 xg durante 2 min.
- Transferir cuidadosamente coluna de rotação para um tubo de microcentrífuga limpo 1,5 mL.
- Adicionar 25 ul de tampão de eluição na coluna de rotação e incuba-se durante 1 minuto à temperatura ambiente. Isto irá aumentar a recuperação de RNA viral a partir da coluna.
- Recolha de ARN por centrifugação a coluna de centrifugação a 10.000 xg durante 1 min.
- Ou prosseguir directamente para RT-qPCR (passo 6) ou ARN armazenar a -70 ° C.
6. MULTIPLEX Detecção RT-qPCR de genogrupo I e II norovírus, e colifagos MS 2 (Tabela Complementar 2)
- superfícies de trabalho limpas, pipetaes, e centrífugas com solução de descontaminação RNase para reduzir a possibilidade de contaminação.
- Descongelar reagentes RT-qPCR em gelo. RNA Thaw no gelo (em uma área / sala separada).
- Determinar o número de reacções e adicionar pelo menos 10% mais deles (por exemplo, se são necessários 10 reacções, certifique-master mix para 11).
- componentes master mix Vortex individuais, com exceção de 25x enzima RT-qPCR, por 5 s. Misturar cuidadosamente a enzima sacudindo o tubo com um dedo.
- Resumidamente componentes Mix Master centrífuga por 5 s.
- Preparar a mistura de mestre para a detecção em tempo real de RT-PCR do GI e GII norovírus de acordo com as instruções do kit e adicionar iniciadores oligonucleotídicos específicos do norovírus e sondas num tubo de microcentrífuga de 1,5 ml (Tabela 3 Complementar).
- Misture a mistura principal pipetando 5-10 vezes para cima e para baixo (vórtice não é recomendado). Aliquota de 22 uL da mistura principal em cada poço de PCR em tempo real placa de 96 poços.
- RNA amostra Vortexdurante 5 seg, e recolher por breve (5 s) de centrifugação.
- Adicionar 3 mL da amostra de RNA e GI e GII controlos positivos para a placa de RT-qPCR (siga molde a partir da Tabela 2). Adicionar 3 mL de nuclease-freewater nos poços não-molde-controlo (NTC).
- Selar a placa em tempo real com película adesiva óptica.
- Cuidadosamente centrifugar a placa em tempo real a 1.300 xg durante 1 min para remover quaisquer bolhas de ar ou gotas de líquido que podem estar presentes nos poços.
- Configurar um tempo real instrumento de PCR e set-up as seguintes condições ciclos térmicos: 1) etapa RT por 10 min. a 45 ° C (2) activação da polimerase Taq, 10 min. a 95 ° C e (3) 45 ciclos de 15 s a 95 ° C, e 60 s a 60 ° C.
7. Quantificação de norovírus em amostras de swab
- Confira os resultados dos controlos positivos e negativos para validar os resultados RT-qPCR (Tabela Complementar 3).
- Determinar se cada curva padrão encontra ovalores aceitáveis dada no quadro suplementar 2 e calcular o desvio padrão geral para a curva padrão, usando a Equação 1.
(Equação 1)% de eficiência = 100 x 10 (média Ct Valores-Intercept) / Slope. - Se o software termociclador PCR não calcula a inclinação para cada curva padrão, determinar encosta e R 2 valores por regressão usando software estatístico (por exemplo, Excel, SPSS ou SAS). Além disso, o cálculo da% de eficiência para as curvas de padrão seguinte equação I
- Grave o número de cópias de RNA calculado pelo software termociclador para todas as amostras de teste com base em curvas padrão que atendem aos critérios especificados na Tabela Complementar 4. Execute novamente todas as amostras com curvas padrão chapéu de t, não satisfazem os critérios ou ter controles de falso-positivos. Verifique os valores Ct de colifago MS2, que foi adicionado como um controlo interno para monitorar a inibição PCR.
- Determinar o número total de cópias de RNA per amostra pela multiplicação do número de cópias de ARN calculada no passo 7.2 pela razão entre o volume (25 a 50 ul) de eluente de ARN total para o ARN que de (3-5 uL) utilizado para a reacção de RT-qPCR. Em alternativa, o cálculo da densidade ARN dividindo o número total de cópias de ARN por área de superfície do objecto (2 cm) analisados.
8. A genotipagem de tempo-real amostras RT-PCR positivos por Hemi Nested convencional PCR Amplification
- Preparar a primeira rodada de master mix para cada conjunto de primers do ensaio de RT-PCR (Tabelas complementares 2 e 5).
- Adicionar 5 mL de RNA positiva norovirus para 20 l de mistura principal.
- Executar o RT-PCR sob as seguintes condições: (1) passo de RT durante 30 min a 42 ° C (2) activação da polimerase Taq, em 15 min a 95 ° C, e (3) 40 ciclos de 30 s a 95 ° C , 30 s a 50 ° C, e 60 seg a 72 ° C. Após 40 ciclos, incubar durante um adicional de 10 min a 72 ° C.
- Prepare a segunda rodada de mix mater para cada conjunto de primers do ensaio de RT-PCR (Tabelas complementares 2 e 5).
- Adicionar 23 mL master mix e adicionar 2 mL de primeira rodada produtos de RT-PCR (da primeira rodada) a cada tubo (o ideal é o produto da primeira rodada deve ser diluído 1/10 em água RNase-free).
- Repita o passo 8.3.
- Preparar um gel de agarose a 2% em 100 ml de EDTA 1x Tris-acetato (Tris 40 mM, ácido acético 20 mM, e EDTA 1 mM, a pH 8,3). Após a dissolução de agarose por aquecimento da mistura num forno de micro-ondas, adicionam-se 10 ul de mancha de ácido nucleico por 100 ml de gel preparada depois de se arrefecer a mistura para 60-70 ° C, verter o gel e inserir os pentes. Deixe o gel se contentar com, pelo menos, 30 min.
- Misturar 15 uL de cada produto de RT-PCR com 3 ul de corante de carga de 6x. Carga de 15 uL de cada amostra e electroforese do gel de agarose, a 100 V durante 1 h.
- fragmentos alvo especial sobre o consumo de RT-PCR de tamanho adequado (330 pb para GI e 341 pb para GII) a partir do geld purificar RNA usando um kit de extracção de gel comercial. O produto de PCR purificado pode agora ser utilizada para a sequenciação de Sanger.
Resultados
A Figura 1 apresenta um fluxograma do protocolo de amostragem cotonete. Este protocolo consiste em quatro etapas principais; 1) coleta de amostra, 2) o armazenamento de amostras e transporte, 3) purificação viral RNA e concentração e 4) ensaio de RT-qPCR e genotipagem.
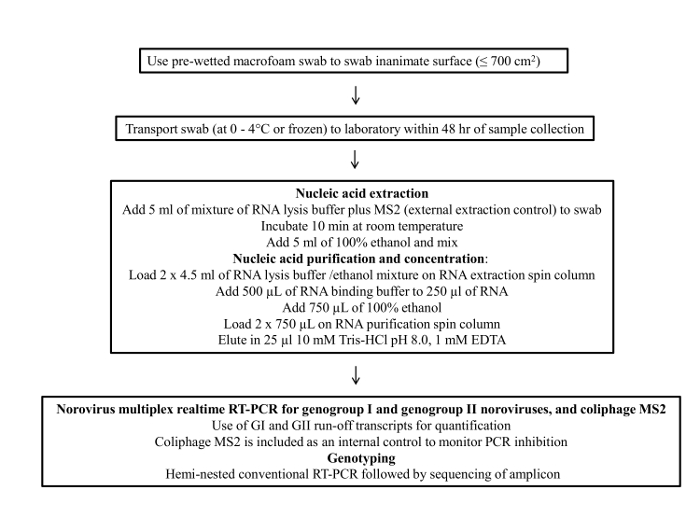
Figura 1: Fluxograma do protocolo final para amostragem de ...
Discussão
Os norovírus têm uma dose infecciosa humana 50% entre 18 e 10 3 partículas de vírus 20. Portanto, mesmo a contaminação de baixo nível de superfícies podem representar um risco para a saúde pública. Vários aspectos do protocolo de amostragem swab foram avaliados, incluindo: 1) diferentes materiais de esfregaço, 2) cotonetes condição de armazenamento durante o transporte, 3) concentração de RNA viral, e 4) colifago MS2 como controle de extração interno.
Divulgações
Authors have no conflicting interest. The findings and conclusions in this report are those of the authors and do not necessarily represent the official position of the Centers for Disease Control and Prevention.
Agradecimentos
The authors have no acknowledgements.
Materiais
| Name | Company | Catalog Number | Comments |
| Generic name for kits | |||
| Macrofoam swab | Premoistened EnviroMax Swab kit | Puritan | 2588060PFUW |
| RNA Lysis buffer | CDC UNEX buffer | Microbiologics | Cat No MR0501 |
| RNA extraction spin column | Midi column | Omega Biotek | Cat No R6664-02 |
| RNA purification spin column | Zymol RNA Clean and Concentrator kit | Zymo Research | Cat No R1016 |
| Real time RT-PCR kit | AgPath kit One-Step RT-PCR Kit | Life Technologies | Cat No 4387391 |
| Conventional RT-PCR kit | Qiagen one step RT-PCR kit | Qiagen kit | Cat No 210212 |
| Gel extraction kit | Qiagen QIAquick gel extraction kit | Qiagen kit | Cat No 28704 or 28706 |
| Coliphage MS2 | ATCC | Cat No 15597-B1 | |
| RNA run-off transcripts | |||
| Realtime PCR platform | Applied Biosystems | Model ABI 7500 | |
| Optical 96-well reaction plate | Thermo Scientific | Cat No 4316813 | |
| MicroAmp Clear Adhesive Film | Thermo Scientific | Cat No 4306311 |
Referências
- Isakbaeva, E. T., et al. Norovirus transmission on cruise ship. Emerg. Infect. Dis. 11, 154-158 (2005).
- Lopman, B. A., Gastañaduy, P., Park, G. W., Hall, A. J., Parashar, U. D., Vinjé, P. Environmental transmission of norovirus gastroenteritis. Curr. Opin. Virol. 2 (1), 1-7 (2011).
- Malek, M., et al. Outbreak of norovirus infection among river rafters associated with packaged delicatessen meat, Grand Canyon, 2005. Clin Infect Dis. 48 (1), 31-37 (2009).
- Atmar, R. L., et al. Norwalk virus shedding after experimental human infection. Emerg. Infect. Dis. 14 (10), 1553-1557 (2008).
- Glass, R. I., Parashar, U. D., Estes, M. K. Norovirus gastroenteritis. N. Engl. J. Med. 361 (18), 1776-1785 (2009).
- Park, G. W., et al. Evaluation of a New Environmental Sampling Protocol for Detection of Human Norovirus on Inanimate Surfaces. Appl. Environ. Microbiol. 81 (17), 5987-5992 (2015).
- Barker, J., Jones, M. V. The potential spread of infection caused by aerosol contamination of surfaces after flushing a domestic toilet. J. Appl. Microbiol. 99, 339-347 (2005).
- Tung-Thompson, G., Libera, D. A., Koch, K. L., de Los Reyes, F. L., Jaykus, L. A. Aerosolization of a Human Norovirus Surrogate, Bacteriophage MS2, during Simulated Vomiting. PloS one. 10, 0134277 (2015).
- Atmar, R. L., et al. Determination of the 50% human infectious dose for Norwalk virus. J. Infect. Dis. 209 (7), 1016-1022 (2014).
- Petrignani, M., van Beek, J., Borsboom, G., Richardus, J. H., Koopmans, M. Norovirus introduction routes into nursing homes and risk factors for spread: a systematic review and meta-analysis of observational studies. J. Hosp. Infect. 89 (3), 163-178 (2015).
- . Centers for Disease Control Prevention. Norovirus outbreak in an elementary school--District of Columbia, February 2007. MMWR. Morb. Mortal. Wkly. Rep. 56 (51-52), 1340-1343 (2008).
- Cheesbrough, J. S., Barkess-Jones, L., Brown, D. W. Possible prolonged environmental survival of small round structured viruses. J. Hosp. Infect. 35, 325-326 (1997).
- Julian, T. R., Tamayo, F. J., Leckie, J. O., Boehm, A. B. Comparison of surface sampling methods for virus recovery from fomites. Appl. Environ. Microbiol. 77, 6918-6925 (2011).
- Taku, A., et al. Concentration and detection of caliciviruses from food contact surfaces. J. Food. Prot. 65, 999-1004 (2002).
- Scherer, K., Ellerbroek, L., Schulenburg, J., Johne, R., Klein, G. Application of a swab sampling method for the detection of norovirus and rotavirus on artifically contaminated food and environmental surfaces. Food. Environ. Virol. 1 (42), 42-49 (2009).
- Herzog, A. B., et al. Evaluation of sample recovery efficiency for bacteriophage P22 on fomites. Appl. Environ. Microbiol. 78, 7915-7922 (2012).
- Vega, E., et al. CaliciNet: A Novel Surveillance Network for Norovirus Gastroenteritis Outbreaks in the United States. Emerging Infectious Diseases. 17 (8), 1389-1395 (2011).
- Rolfe, K. J., et al. An internally controlled, one-step, real-time RT-PCR assay for norovirus detection and genogrouping. J Clin Virol. 39 (4), 318-321 (2007).
- Kittigul, L., et al. Norovirus GII-4 2006b variant circulating in patients with acute Thailand during a 2006-2007 study. J. Med. Virol. 82 (5), 854-860 (2010).
- Teunis, P. F., et al. Norwalk virus: how infectious is it. J. Med. Virol. 80 (8), 1468-1476 (2008).
- Wollants, E., et al. Evaluation of a norovirus sampling method using sodium dodecyl sulfate/EDTA-pretreated chromatography paper strips. J. Virol. Methods. 122, 45-48 (2004).
- Weir, M. H., Shibata, T., Masago, Y., Cologgi, D., Rose, J. B. The Effect of Surface Sampling and Recovery of Viruses and Non-Spore Forming Bacteria on a QMRA Model for Fomites. Environ. Sci. Technol. 50 (11), 5945-5952 (2016).
- . Microbiology of food and animal feed-Horizontal method for determination of hepatitis A virus and norovirus in food using real-time RT-PCR. International Organization for Standardization (ISO). , (2013).
- Huslage, K., Rutala, W. A., Sickbert-Bennett, E., Weber, D. J. A quantitative approach to defining "high-touch" surfaces in hospitals. Infect. Control. Hosp. Epidemiol. 31 (8), 850-853 (2010).
- Wu, H. M., et al. A norovirus outbreak at a long-term-care facility: the role of environmental surface contamination. Infect. Control. Hosp. Epidemiol. 26 (10), 802-810 (2005).
- Ikner, L. A., Gerba, C. P., Bright, K. R. Concentration and recovery of viruses from water: a comprehensive review. Food Environ. Virol. 4 (2), 41-67 (2012).
- Gallimore, C. I., et al. Environmental monitoring for gastroenteric viruses in a pediatric primary immunodeficiency unit. J. Clin. Microbiol. 44 (2), 395-399 (2006).
- Ganime, A. C., et al. Dissemination of human adenoviruses and rotavirus species A on fomites of hospital pediatric units. Am J Infect Control. , (2016).
- Verani, M., Bigazzi, R., Carducci, A. Viral contamination of aerosol and surfaces through toilet use in health care and other settings. Am J Infect Control. 42 (7), 758-762 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados