JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
표면에 인간의 노로 바이러스의 검출을위한 면봉 표본 추출 방법
요약
A macrofoam based sampling methodology was developed and evaluated for the detection and quantification of norovirus on environmental hard surfaces.
초록
인간의 noroviruses는 전염병이 전 세계적으로 산발적 위장염의 주요 원인입니다. 대부분의 감염이 어느 환경 표면 또는 음식을 통해 간접적으로 개인 대 개인 경로를 통해 직접 전파 또는 때문에, 오염 fomites과 무생물 표면은 노로 바이러스의 발생시 바이러스의 확산을위한 중요한 차량이다.
우리는 개발 검출 및 하드 표면에서 인간의 noroviruses의 입력에 대한 macrofoam 면봉을 사용하여 프로토콜을 평가 하였다. 섬유로 만들어진 면봉 또는 정전기 방지 물티슈에 비해 macrofoam 면봉는 최대 700cm 2의 변기 표면에서 바이러스 복구 (범위 1.2-33.6 %)을 할 수 있습니다. 이 프로토콜은 스핀 열을 사용하여 바이러스 성 RNA의 면봉에서 바이러스의 추출과 더 집중하는 단계를 포함한다. 노로 바이러스 위장염이 있었다 유람선 및 장기 요양 시설의 표면에서 수집 한 217 면봉 샘플의 총, 127 (58.5 %)RT-qPCR에 의한 GII의 노로 바이러스 양성 반응보고했다. 이 29 (22.8 %)의 성공적 유전자형을 할 수있다. 결론적으로, 우리는 임상 샘플을 사용할 수없는 경우 바이러스의 검출뿐만 아니라 발생시 환경 오염의 수준을 결정하는데 도움이 될 수 개발 된 프로토콜을 사용하여 환경 표면 노로 바이러스의 검출; 또한 치료 전략의 효과의 모니터링을 용이하게 할 수있다.
서문
인간의 noroviruses는 전염병과 산발적 급성 위장염 전 세계적으로 1, 2, 3의 주요 원인이다. 이 바이러스는 매우 전염성이 전송은 사람의 상호 작용으로 또는 간접적으로 오염 된 음식, 물 또는 환경 표면과 접촉을 통해 직접 사람을 통해 발생합니다. Noroviruses는 장시간 흘리고 연장 환경면에서의 바이러스 생존 1, 2, 3에 설명 된 수있다. 피크 흘리는 동안 바이러스 입자 수십억 대변 그램 당 방출하고, 구토 같은 감염 4, 5, 6, 7, 8 일으키는 바이러스 입자의 충분한 개수를 포함EF "> 9,10. 또한 무생물 표면과 사람의 피부의 바이러스의 이동이 발생하기 쉬운 2, 11, 12. 따라서, 환경 오염의 모니터링이 발생 조사 돕기 및 정화의 효과를 평가할 수 있으며 소독 절차.
몇몇 환경 샘플링 프로토콜 로타 바이러스의 검출에 대해 설명되었지만, coliphage MS2, 고양이 calicivirus (FCV) 및 박테리오파지 P22 13, 14, 15, 16. 그러나, 고속 탈수 (<1 시간) 작은 표면적을 포함하여이 연구에 기술 검증 조건 (25 X 100cm 2), 적절 필드 설정을 표현하지 않을 수 있습니다. 또한 ENVIRO 낮은 오염 수준을 예상nmental 표면은 거의 바이러스 입자를 감지 할 수있는 프로토콜을 필요로한다.
우리는 노로 바이러스의 검출 및 입력을위한 macrofoam 기반 표면 샘플링 방법을 개발했다. 이 방법은 여러 노로 바이러스 발생시 검증되었습니다. 프로토콜이 포함 1) (2) 최선의 방법 실험실에 수집 및 운송 중에 샘플의 무결성을 유지하고, 노로 바이러스 3) 실험실 테스트 및 타이핑 환경면에서 면봉 샘플을 수집하는 방법에 대해 설명합니다.
프로토콜
필드 1. 면봉 샘플링
- 장갑의 깨끗한 쌍을 착용 할 것.
- 측정 테이프자를 사용하여 표면을 만지지 않고 샘플링 에리어의 크기를 측정한다. 가능한 한 정확하게 영역을 추정하고 보고서 양식 (보충 표 1)를 작성하려고합니다.
- 가능한 누출 및 라벨 샘플 수송 가방과 면봉 키트에 대한 면봉 키트를 확인합니다.
- 다음 샘플링 에리어 걸쳐 면봉 이동 : 수평 방향의 하나의 선, 세로 방향으로 하나의 스트로크와, 대각 방향에 하나의 획. 700cm 2보다 표면적이 큰 면봉하지 마십시오.
- 튜브에 각각 면봉을 놓고 안전하게 뚜껑을 조입니다.
실험실에 면봉 2. 보관 및 운송
- 최대 48 시간 동안 4 ° C에서 면봉을 유지합니다. 오랜 기간 동안 보관이 필요한 경우, -20 ° C에서 면봉을 저장 (또는 -70 ° C).
- 0-4 ℃에서 튜브를 유지 (즉,. 컬렉션의 24 ~ 48 시간 이내에 실험실 및 선박에 운송 중 절연 된 용기에) 콜드 팩을 사용합니다.
3. 바이러스 농도 바이러스 RNA 추출 및 정제
주 : 달리 명시되지 않는 한 모든 원심 분리 단계는 실온에서 5 분 동안 5,000 XG에서 테이블 탑 원심 분리기를 사용한다. 보편적 인 핵산 추출 (UNEX) 버퍼로 작업 할 때 추가로주의해야합니다. 고글 또는 안면 보호대를 착용하십시오.
- 한 15 ML의 튜브와 각 샘플에 대해 하나의 RNA 미디 열 레이블. 각 실험에 부정적인 추출 컨트롤을 포함합니다.
- 10 면봉 대한 분해 용액을 제조 PBST 25 mL로 UNEX 완충액 25 ㎖를 혼합하여 (0.02 %를 함유하는 PBS pH를 7.2 트윈 80).
- 용해 액 50 ㎖에 coliphage의 MS2 서스펜션 (10 6 PFU / μL) 50 μL를 추가합니다.
- 15 ㎖의 튜브에 면봉을 넣고 5 ml의 용해 액을 추가합니다. 혼합하고 실온에서 10 분 동안 배양한다.
- 10 초 동안 각 튜브와 소용돌이에 5 ml의 100 % 에탄올을 추가합니다.
- 조심스럽게 15 ML의 튜브에서 면봉을 제거 초과 액체를 제거하고 면봉을 폐기 튜브의 측면에 살짝 누르면 (이 UNEX 버퍼를 포함). 나머지 볼륨은 9 ~ 10 mL의 사이에 있어야한다.
4. 미디 열 바이러스 핵산 추출
- 조심스럽게 미디 컬럼, 원심 분리기에 단계 3.6에서 4.5 mL의 UNEX / 에탄올 혼합물을 전송하고, 여과 액을 버린다.
- 동일한 열, 원심 분리기에 같은 혼합물로부터 또 다른 4.5 mL로로드하고, 여과 액을 버린다.
- 제 씻지 미디 칼럼, 원심 분리기에 70 % 에탄올 3.5 mL를 추가하고, 여과 액을 버린다.
- 제 씻지 미디 컬럼에 70 % 에탄올의 또 다른 3.5 mL의 추가. 원심 분리기 여과 액을 버리고.
- 건조 스핀를 들어, 에탄올의 모든 흔적을 (부정적인 바이러스 성 RNA 복구에 영향을 미칠 수있는)을 제거하기 위해 미디 열을 원심 분리기.
- 새로운 15 ML의 원심 분리기 튜브의 미디 열을 놓습니다. 미디 칼럼에 250 μL 용출 버퍼를 추가; 가장 높은 바이러스 성 RNA 복구를 얻기 위해 원심 분리하기 전에 1 분 동안 기다립니다.
- -70 ° C에서 추출 된 핵산을 저장하거나 5 단계로 바로 진행합니다.
RNA 깨끗하고 집중 키트를 사용하여 바이러스 핵산의 5 농도
- RNA는 10 초 동안 위의 단계 4.7과 와동에 용출 된 RNA의 250 μL에 버퍼를 바인딩의 500 μL를 추가합니다.
- 10 초 동안 750 μL 100 %를 에탄올과 소용돌이를 추가합니다.
- 각 샘플에 대한 스핀 열 레이블. 스핀 칼럼에 750 μL 샘플을로드합니다.
- 1 분 동안 12,000 XG에 스핀 컬럼을 원심 분리기. 흐름 -을 통해 폐기하십시오.
- 1 분 동안 12,000 XG에 스핀 컬럼 및 원심 분리기 상에 남아있는 750 μL를로드합니다.
- 프리 워시를 들어, 1 분 동안 12,000 XG에서 각 스핀 컬럼 및 원심 분리기 400 μL RNA 준비 버퍼를 추가합니다. 흐름 -을 통해 폐기하십시오.
- 첫 번째 세척를 들어, 1 분 동안 12,000 XG에서 각 스핀 컬럼 및 원심 분리기 800 μL RNA 세척 버퍼를 추가합니다. 흐름 -을 통해 폐기하십시오.
- 두 번째 세척를 들어, 1 분 동안 12,000 XG에서 열 원심 분리기를 회전 400 μL RNA 세척 버퍼를 추가합니다. 흐름 -을 통해 폐기하십시오.
- 건조 스핀를 들어, 2 분 동안 12,000 XG에 세척 버퍼의 모든 흔적을 제거하는 스핀 컬럼을 원심 분리기.
- 조심스럽게 깨끗한 1.5 ML의 microcentrifuge 관에 스핀 컬럼을 전송합니다.
- 스핀 컬럼 상에 25 μL의 용출 완충액을 첨가하고 실온에서 1 분 동안 배양한다. 이 컬럼으로부터 RNA 바이러스의 회수를 증가시킬 것이다.
- 1 분 동안 10,000 XG에 스핀 컬럼을 원심 분리하여 RNA를 수집합니다.
- 어느 -70 ° C에서 RT-qPCR에 (6 단계) 또는 저장 RNA에 직접 진행합니다.
6. 유전자형 I의 Mutiplex RT-qPCR에 탐지 및 II Noroviruses 및 Coliphage MS 2 (보충 표 2)
- 청소 작업 표면 pipettES,의 RNase의 오염 제거 용액와 원심 분리기는 오염 가능성을 줄일 수 있습니다.
- 얼음에 RT-qPCR에 시약을 녹여. (별도의 영역 / 방에) 얼음에 해동 RNA.
- 반응의 수를 결정하고 적어도 10 % 이상을 추가 (열 반응이 필요한 경우, 예를 들면, 11의 마스터 믹스를 확인).
- 5 초 동안 25 배 RT-qPCR에 효소를 제외 소용돌이 개별 마스터 믹스 구성 요소. 조심스럽게 손가락으로 튜브를 쓸어 넘겨 효소를 혼합한다.
- 5 초 동안 간단히 원심 분리기 마스터 믹스 구성 요소.
- 키트 지침에 따라 노로 바이러스 GI과 GII의 실시간 RT-PCR 검출을위한 마스터 믹스를 준비하고 1.5 ml의 microcentrifuge 관 (보충 표 3)에서 노로 바이러스 특이 적 올리고 뉴클레오티드 프라이머 및 프로브를 추가합니다.
- 위아래로 5 ~ 10 회 피펫 팅하여 마스터 믹스 믹스 (텍싱하지 않는 것이 좋습니다). 실시간 PCR을 96 웰 플레이트의 각 웰에서의 마스터 믹스를 22 μL 분취.
- 소용돌이 샘플 RNA5 초 동안, 그리고 짧은 (5 초) 원심 분리하여 수집합니다.
- RT-qPCR에 판 (표 2에서 템플릿을 따라)에 샘플 RNA와 GI과 GII 양성 대조군의 3 μL를 추가합니다. 노 템플릿 제어 (NTC) 우물에 클레아 제-freewater의 3 μL를 추가합니다.
- 광학 접착제 필름 실시간 플레이트를 밀봉.
- 1 분 웰에 존재할 수있는 임의의 기포 또는 액체 방울을 제거하기 위해주의 깊게 1300 XG에서 실시간 플레이트를 원심 분리.
- 10 분 동안 1) RT 단계 : 실시간 PCR 장비 및 설정 다음 열 순환 조건을 설정합니다. Taq 중합 효소의 45 ° C (2) 활성화, 10 분에서. 95 ° C에서 및 (3) 95 ℃에서 15 초간 45 사이클, 그리고 60 ℃에서 60 초.
면봉 샘플에서 노로 바이러스의 7 정량화
- RT-qPCR의 결과 (보충 표 3)의 유효성을 검사 양성 및 음성 대조군의 결과를 확인합니다.
- 각각의 표준 곡선이 사항을 만족하는지 확인사용할 수있는 값은 보조 표 2에 주어진 수학 식 (1)를 사용하여 표준 곡선의 전체 표준 편차를 계산합니다.
(식 1) %의 효율 = 100 × 10 (평균 코네티컷 값 절편) / 경사. - PCR을 열 순환기 소프트웨어는 각각의 표준 곡선에 대한 기울기를 계산하지 않으면 기울기 R 통계 소프트웨어를 이용하여 회귀 2 값 (예를 들어 엑셀, SPSS 또는 SAS)를 결정한다. 또한, 방정식 나는 다음과 같은 표준 곡선에 대한 %의 효율을 계산
- 보충 표 4에 명시된 기준에 맞는 표준 곡선에 기초하여 모든 테스트 샘플에 대한 열 순환기 소프트웨어에 의해 산출 된 RNA 카피 수를 기록한다. 기준을 충족하거나 거짓 양성 대조군이없는 표준 곡선 t 모자와 함께 어떤 샘플을 다시 실행하십시오. PCR 억제를 모니터링 할 수있는 내부 통제로 추가 된 coliphage MS2의 코네티컷 값을 확인합니다.
- RNA 카피 (P)의 총 개수를 결정는 RT-qPCR의 반응에 사용 된 RNA (3 μL 5)의 것과 총 RNA의 용출 부피 (25 내지 50 μL)의 비율로 단계 7.2에서 계산 된 RNA 복사 개수를 곱하여 ER 샘플. 대안 적으로, 분석 대상물의 표면적 (cm 2)에 의해 총 RNA 복사 개수를 분할함으로써 RNA 농도를 계산한다.
헤미 중첩 된 기존의 PCR 증폭에 의해 실시간 RT-PCR 긍정적 인 샘플의 8 유전자형
- RT-PCR 분석의 각 프라이머 세트에 대한 마스터 믹스의 첫 번째 라운드를 준비합니다 (보충 표 2, 5).
- 마스터 믹스 20 μL에 노로 바이러스 양성 RNA의 5 μL를 추가합니다.
- 다음과 같은 조건에서 RT-PCR을 실행하여, (1) 실온에서 95 ° C에서 Taq 중합 효소의 42 ° C (2) 활성화, 15 분에서 30 분 동안 단계, 및 (3) 95 ℃에서 30 초, 40주기 50 ℃에서 30 초, 72 ℃에서 60 초. 40 사이클 후, 72 ℃에서 추가로 10 분 동안 배양한다.
- RT-PCR 분석 각각의 프라이머 세트 (보충 표 2와 5)에 대한 이잖아요 믹스의 두 번째 라운드를 준비합니다.
- 23 μL 마스터 믹스를 추가하고 각 튜브 (이상적으로 1 차 제품의 RNase없는 물에 1/10 희석한다)에 (첫 라운드에서) 1 라운드 RT-PCR 제품의 2 μL를 추가합니다.
- 단계를 반복 8.3.
- (PH 8.3에서 40 mM 트리스, 20 mM의 아세트산 및 1 mM의 EDTA) 100 ㎖ 1X 트리스 - 아세테이트 EDTA 2 % 아가 로스 겔을 제조. 전자 레인지의 혼합물을 가열하여 아가로 용해한 후, 60 ~ 70 ℃로 냉각 한 후, 혼합물을 제조 된 겔을 100㎖ 당 핵산 얼룩 10 μL를 추가 겔 붓고 콤 삽입. 겔은 적어도 30 분 동안 정착하자.
- 6X 로딩 염료 3 μL 각 RT-PCR 생성물의 15 μl를 섞는다. 로드 각 시료 15 μL 및 1 시간 동안 100 V의 아가로 오스 겔 electrophorese.
- 겔에서 적절한 크기 (GI 330 BP와 GII 341 BP)의 소비세 대상 RT-PCR 단편상업적 겔 추출 키트를 사용하여 RNA를 정제 거라고. 정제 된 PCR 생성물은 이제 생거 시퀀싱에 사용될 수있다.
결과
도 1은 면봉 샘플링 프로토콜의 흐름도를 나타낸다. 이 프로토콜은 네 가지 주요 단계로 구성; 1) 샘플 수집, 2) 시료의 저장 및 수송, 3) 바이러스 성 RNA의 정제 및 농도 4) RT-qPCR의 분석 및 유전자형.
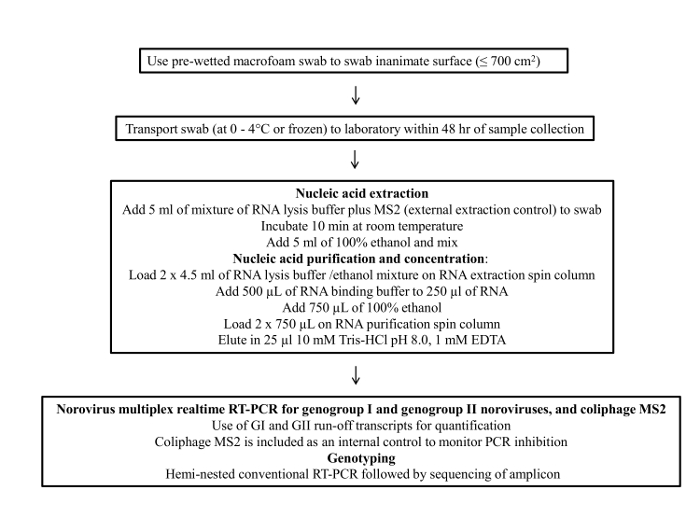
그림 1 : 노로 바이러스의 환경 표면 샘플링에 대한 최종...
토론
Noroviruses 18 10 3 바이러스 입자 20 ~ 50 %의 인간 감염 복용량을 가지고있다. 따라서, 표면도 낮은 수준의 오염은 공중 보건 위험을 초래할 수 있습니다. 내부 대조군으로서 추출 운송 중 1) 면봉 다른 재료, 2) 저장 조건 면봉 3) 바이러스 성 RNA 농도, 4) coliphage MS2 : 면봉 샘플링 프로토콜의 여러 측면을 포함하여 평가 하였다.
최근까지,면, 폴리 에스테르, ...
공개
Authors have no conflicting interest. The findings and conclusions in this report are those of the authors and do not necessarily represent the official position of the Centers for Disease Control and Prevention.
감사의 말
The authors have no acknowledgements.
자료
| Name | Company | Catalog Number | Comments |
| Generic name for kits | |||
| Macrofoam swab | Premoistened EnviroMax Swab kit | Puritan | 2588060PFUW |
| RNA Lysis buffer | CDC UNEX buffer | Microbiologics | Cat No MR0501 |
| RNA extraction spin column | Midi column | Omega Biotek | Cat No R6664-02 |
| RNA purification spin column | Zymol RNA Clean and Concentrator kit | Zymo Research | Cat No R1016 |
| Real time RT-PCR kit | AgPath kit One-Step RT-PCR Kit | Life Technologies | Cat No 4387391 |
| Conventional RT-PCR kit | Qiagen one step RT-PCR kit | Qiagen kit | Cat No 210212 |
| Gel extraction kit | Qiagen QIAquick gel extraction kit | Qiagen kit | Cat No 28704 or 28706 |
| Coliphage MS2 | ATCC | Cat No 15597-B1 | |
| RNA run-off transcripts | |||
| Realtime PCR platform | Applied Biosystems | Model ABI 7500 | |
| Optical 96-well reaction plate | Thermo Scientific | Cat No 4316813 | |
| MicroAmp Clear Adhesive Film | Thermo Scientific | Cat No 4306311 |
참고문헌
- Isakbaeva, E. T., et al. Norovirus transmission on cruise ship. Emerg. Infect. Dis. 11, 154-158 (2005).
- Lopman, B. A., Gastañaduy, P., Park, G. W., Hall, A. J., Parashar, U. D., Vinjé, P. Environmental transmission of norovirus gastroenteritis. Curr. Opin. Virol. 2 (1), 1-7 (2011).
- Malek, M., et al. Outbreak of norovirus infection among river rafters associated with packaged delicatessen meat, Grand Canyon, 2005. Clin Infect Dis. 48 (1), 31-37 (2009).
- Atmar, R. L., et al. Norwalk virus shedding after experimental human infection. Emerg. Infect. Dis. 14 (10), 1553-1557 (2008).
- Glass, R. I., Parashar, U. D., Estes, M. K. Norovirus gastroenteritis. N. Engl. J. Med. 361 (18), 1776-1785 (2009).
- Park, G. W., et al. Evaluation of a New Environmental Sampling Protocol for Detection of Human Norovirus on Inanimate Surfaces. Appl. Environ. Microbiol. 81 (17), 5987-5992 (2015).
- Barker, J., Jones, M. V. The potential spread of infection caused by aerosol contamination of surfaces after flushing a domestic toilet. J. Appl. Microbiol. 99, 339-347 (2005).
- Tung-Thompson, G., Libera, D. A., Koch, K. L., de Los Reyes, F. L., Jaykus, L. A. Aerosolization of a Human Norovirus Surrogate, Bacteriophage MS2, during Simulated Vomiting. PloS one. 10, 0134277 (2015).
- Atmar, R. L., et al. Determination of the 50% human infectious dose for Norwalk virus. J. Infect. Dis. 209 (7), 1016-1022 (2014).
- Petrignani, M., van Beek, J., Borsboom, G., Richardus, J. H., Koopmans, M. Norovirus introduction routes into nursing homes and risk factors for spread: a systematic review and meta-analysis of observational studies. J. Hosp. Infect. 89 (3), 163-178 (2015).
- . Centers for Disease Control Prevention. Norovirus outbreak in an elementary school--District of Columbia, February 2007. MMWR. Morb. Mortal. Wkly. Rep. 56 (51-52), 1340-1343 (2008).
- Cheesbrough, J. S., Barkess-Jones, L., Brown, D. W. Possible prolonged environmental survival of small round structured viruses. J. Hosp. Infect. 35, 325-326 (1997).
- Julian, T. R., Tamayo, F. J., Leckie, J. O., Boehm, A. B. Comparison of surface sampling methods for virus recovery from fomites. Appl. Environ. Microbiol. 77, 6918-6925 (2011).
- Taku, A., et al. Concentration and detection of caliciviruses from food contact surfaces. J. Food. Prot. 65, 999-1004 (2002).
- Scherer, K., Ellerbroek, L., Schulenburg, J., Johne, R., Klein, G. Application of a swab sampling method for the detection of norovirus and rotavirus on artifically contaminated food and environmental surfaces. Food. Environ. Virol. 1 (42), 42-49 (2009).
- Herzog, A. B., et al. Evaluation of sample recovery efficiency for bacteriophage P22 on fomites. Appl. Environ. Microbiol. 78, 7915-7922 (2012).
- Vega, E., et al. CaliciNet: A Novel Surveillance Network for Norovirus Gastroenteritis Outbreaks in the United States. Emerging Infectious Diseases. 17 (8), 1389-1395 (2011).
- Rolfe, K. J., et al. An internally controlled, one-step, real-time RT-PCR assay for norovirus detection and genogrouping. J Clin Virol. 39 (4), 318-321 (2007).
- Kittigul, L., et al. Norovirus GII-4 2006b variant circulating in patients with acute Thailand during a 2006-2007 study. J. Med. Virol. 82 (5), 854-860 (2010).
- Teunis, P. F., et al. Norwalk virus: how infectious is it. J. Med. Virol. 80 (8), 1468-1476 (2008).
- Wollants, E., et al. Evaluation of a norovirus sampling method using sodium dodecyl sulfate/EDTA-pretreated chromatography paper strips. J. Virol. Methods. 122, 45-48 (2004).
- Weir, M. H., Shibata, T., Masago, Y., Cologgi, D., Rose, J. B. The Effect of Surface Sampling and Recovery of Viruses and Non-Spore Forming Bacteria on a QMRA Model for Fomites. Environ. Sci. Technol. 50 (11), 5945-5952 (2016).
- . Microbiology of food and animal feed-Horizontal method for determination of hepatitis A virus and norovirus in food using real-time RT-PCR. International Organization for Standardization (ISO). , (2013).
- Huslage, K., Rutala, W. A., Sickbert-Bennett, E., Weber, D. J. A quantitative approach to defining "high-touch" surfaces in hospitals. Infect. Control. Hosp. Epidemiol. 31 (8), 850-853 (2010).
- Wu, H. M., et al. A norovirus outbreak at a long-term-care facility: the role of environmental surface contamination. Infect. Control. Hosp. Epidemiol. 26 (10), 802-810 (2005).
- Ikner, L. A., Gerba, C. P., Bright, K. R. Concentration and recovery of viruses from water: a comprehensive review. Food Environ. Virol. 4 (2), 41-67 (2012).
- Gallimore, C. I., et al. Environmental monitoring for gastroenteric viruses in a pediatric primary immunodeficiency unit. J. Clin. Microbiol. 44 (2), 395-399 (2006).
- Ganime, A. C., et al. Dissemination of human adenoviruses and rotavirus species A on fomites of hospital pediatric units. Am J Infect Control. , (2016).
- Verani, M., Bigazzi, R., Carducci, A. Viral contamination of aerosol and surfaces through toilet use in health care and other settings. Am J Infect Control. 42 (7), 758-762 (2014).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유