Method Article
Un método de extracción y análisis de lípidos para caracterizar los microbios del suelo en experimentos con muchas muestras
En este artículo
Resumen
El artículo describe un método que incrementa el rendimiento mientras se equilibra el esfuerzo y precisión para la extracción de lípidos de las membranas celulares de los microorganismos para su uso en la caracterización tanto de los lípidos totales como de la abundancia relativa de los lípidos indicadores para determinar la estructura microbiana comunitaria en estudios con muchas muestras.
Resumen
Las comunidades microbianas son importantes impulsores y reguladores de los procesos de los ecosistemas. Para entender cómo el manejo de los ecosistemas puede afectar a las comunidades microbianas, una técnica relativamente precisa pero que requiere esfuerzo intensivo para analizar la composición de la comunidad microbiana es el análisis de ácidos grasos fosfolípidos (PLFA). El PLFA fue desarrollado para analizar biomarcadores de fosfolípidos, que pueden ser utilizados como indicadores de la biomasa microbiana y la composición de amplios grupos funcionales de hongos y bacterias. Se ha utilizado comúnmente para comparar suelos bajo comunidades vegetales alternativas, ecología y regímenes de manejo. Se ha demostrado que el método PLFA es sensible a la detección de cambios en la composición de la comunidad microbiana.
Se desarrolló un método alternativo de extracción y análisis de ésteres metílicos de ácidos grasos (MIDI-FA) para la extracción rápida de lípidos totales, sin separación de la fracción de fosfolípidos, de cultivos puros como técnica de identificación microbiana. Este método esRápido pero es menos adecuado para muestras de suelo porque carece de un paso inicial que separa las partículas del suelo y comienza en su lugar con una reacción de saponificación que probablemente produce artefactos de la materia orgánica de fondo en el suelo.
Este artículo describe un método que incrementa el rendimiento mientras se equilibra el esfuerzo y la precisión para la extracción de lípidos de las membranas celulares de los microorganismos para su uso en la caracterización de los lípidos totales y la abundancia relativa de los lípidos indicadores para determinar la estructura microbiana comunitaria en estudios con muchas muestras. El método combina la precisión obtenida mediante el perfil de PLFA mediante la extracción y concentración de los lípidos del suelo como primer paso, y una reducción del esfuerzo saponificando el material orgánico extraído y procesando con el método MIDI-FA como un segundo paso.
Introducción
Dado el papel clave de los microorganismos en el ciclo de los nutrientes 1 , la modificación de la composición de la comunidad vegetal 2 , la regulación de la productividad de las plantas 3 y la descomposición de la materia orgánica 4 , la comprensión de las comunidades microbianas del suelo es vital para comprender los ecosistemas terrestres.
Debido a su abundancia relativamente alta en el suelo, y su firma química, los biomarcadores de lípidos se pueden utilizar para el perfil de los grupos ecológicos dominantes que comprenden las comunidades microbianas del suelo 5 . Mediante la cuantificación de biomarcadores lipídicos que son característicos de diferentes grupos microbianos, podemos estimar los lípidos totales y luego separar estos lípidos en grupos ecológicamente relevantes tales como bacterias Gram positivas (Gm +) y Gram negativas (Gm-), micorrizas arbusculares (AM) y saprotrópicas Hongos y actinomicetos 5 , Ss = "xref"> 6 , 7 , 8 .
Existen muchos métodos para caracterizar aspectos de las comunidades microbianas. El método PLFA es comúnmente utilizado para comprender la estructura básica de la comunidad microbiana. Es una manera eficaz de evaluar la abundancia relativa de grupos microbianos, así como la biomasa microbiana total. Debido a la rápida rotación de lípidos, el perfil de PLFA también permite una detección relativamente rápida de cambios en la comunidad microbiana del suelo y proporciona información que permite comparar la función del ecosistema, por ejemplo, las relaciones fúngicas y bacterianas para evaluar las tasas de ciclo de nutrientes 1 , 9 , 10 . Sin embargo, si bien el método de extracción de PLFA es honrado y respetado, también consume mucho tiempo y no se presta bien a estudios a escala de ecosistemas que requieren un gran número de muestras de repeticiones a escala de campoF "> 11 , 12 .
Por el contrario, el método de extracción de éster metílico de ácidos grasos (MIDI-FA) tiene el potencial de permitir un rendimiento rápido. En este método, las muestras se saponifican, se convierten a FAMEs, se extraen y luego se analizan. El método MIDI-FA es rápido pero menos discriminante que el PLFA, que combina la extracción de lípidos con la separación de diferentes clases de lípidos 13 (fosfolípidos, lípidos neutros y glicolípidos).
En este protocolo, se describe un método que combina elementos de ambos PLFA y MIDI-FA perfilación de lípidos. Emplea la extracción de lípidos usando las etapas de extracción de cloroformo iniciales del método de Bligh y Dyer modificado, y después saponificación y conversión a FAMEs. Esto proporciona una manera robusta para detectar la estructura de la comunidad microbiana, mientras que la exclusión de gran parte del ruido de fondo de material no microbiano 5 , 14 15] . Al realizar la extracción inicial y aislar los componentes orgánicos solubles ( por ejemplo, lípidos) antes de realizar MIDI-FA, y completar esto con un paso de purificación, el protocolo ofrece un equilibrio entre velocidad y precisión.
Protocolo
NOTA: Siempre use equipo de protección personal (EPP) apropiado durante todo el procedimiento. Para evitar la posible contaminación de la muestra, no toque las piezas de vidrio con las manos desnudas. Use guantes adecuados cuando se estén ejecutando los pasos del protocolo que requieren el manejo de cloroformo.
1. Preparaciones (2 días para ~ 40 muestras)
- Recoja el suelo en bolsas estériles y transporte desde el campo en un refrigerador que contenga hielo. Si no es posible tamizar el suelo fresco y congelarlo inmediatamente, almacenar las muestras en un congelador de -80 ° C hasta que esté listo para comenzar el análisis.
- Preparar el suelo homogeneizando. Eliminar las raíces y las piedras y romper los terrones mediante tamizado grueso ( por ejemplo, 2 mm).
- Prepárese para la liofilización poniendo las submuestras en un recipiente apropiado según las instrucciones del manual de liofilizador y lávelas con hielo lo antes posible. Una vez que los suelos son liofilizados, almacenar en un recipiente sellado con desecante hasta la extracción. Es mejor sRasgó el suelo liofilizado a un mínimo de -20ºC, pero preferiblemente a -80ºC.
- En preparación para la extracción, eliminar los suelos liofilizados de almacenamiento y moler a una consistencia similar a la harina. Los métodos para moler incluyen molino de bolas, batidor de bola, y mortero y mano de mortero. Después de moler el suelo, guárdelo en un congelador (ver 1.3).
NOTA: La cantidad de muestra utilizada para la extracción depende del contenido de materia orgánica. Una pauta general es usar 0,5 a 1 g de un suelo que es de 12 a 18% en peso de carbono y de 3 a 5 g para un suelo de 1 a 3% en peso de carbono. - Pre-enjuague los tubos de centrífuga de 30 ml con hexano. Añadir aproximadamente 2 a 3 mL de hexano a los tubos y agitar durante 5 s. Decantar hexano a otro tubo, y vórtice. Se puede utilizar hexano (2 a 3 ml) para enjuagar en serie seis tubos. Guarde los tubos enjuagados con hexano invertidos en la campana extractora de humos y deseche el hexano usado en un contenedor de residuos apropiado.
- Envuelva la cristalería en 2 a 3 capas de papel de aluminio y colóquela en el horno de mufla. Hornear(Mufla) a 450 ° C durante 4,5 h.
- Prepare los reactivos.
- Para la cantidad de suelo utilizada, añadir los productos químicos en una proporción de volumen de 0,8: 1: 2 para tampón de fosfato (tampón P): CHCl $ ₃ $ : MeOH.
- Preparar solución tampón P: tampón fosfato: 0,1 M, pH 7,0.
- Agregue 61 mL de 1M de K 2 HPO 4 stock, estéril (el producto químico debe estar certificado por ACS o mejor). Añadir 39 mL de 1 M de KH 2 PO 4 stock, estéril (el producto químico debe tener un grado certificado ACS o mejor). Rellenar hasta 1000 mL con agua Tipo 1. Ajustar el pH a 7,0 con NaOH o HCl. Almacene la solución no utilizada hasta 7 días a temperatura ambiente o 30 días en el refrigerador.
- Alternativamente, pésese 10,62 g de K 2 HPO 4 y 5,31 g de KH 2 PO 4; Diluir a 1 L con agua Tipo 1. Compruebe el pH, ajuste si es necesario.
- Prepare el Reactivo 1, Reactivo de Saponificación.
- Dispense 300 mL de agua de Tipo 1.Añadir 300 ml de CH $ ₃ $ OH (grado HPLC o superior). Añadir 90,00 g de NaOH (ACS certificado o mejor). Añadir los gránulos de NaOH a la solución mientras se agita. Revuelva hasta que los gránulos se disuelvan. Se recomienda almacenar esta solución no más de 30 d.
- Prepare el Reactivo 2, Reactivo de Metilación.
- Dispense 275 ml de CH $ ₃ $ OH (grado de HPLC o superior). Agregue 325 mL de HCl certificada de 6,00 N mientras se agita. Se recomienda almacenar esta solución no más de 30 d.
- Prepare el reactivo 3, reactivo de extracción: 50% C 6 H 14 (Hexano), 50% C 5 H 12 O (MTBE).
- En la campana, combine 200 ml de C 6 H 14 (grado HPLC o superior) y 200 ml de C 5 H 12 O (grado HPLC o superior). Mezclar bien; Tapa firmemente. Almacene en el gabinete de los productos inflamables o en la campana extractora de humos durante no más de 1 año.
- Prepare el Reactivo 4, Reactivo de lavado base.
- Dispense 900Ml de agua de Tipo 1 a un vaso de precipitados. Añadir 10,80 g de NaOH (ACS certificado o mejor) mientras se agita. Revuelva hasta que los gránulos se disuelvan. Se recomienda almacenar esta solución no más de 30 d.
- Preparar la solución madre de la norma interna.
- Pesar 100 mg de la norma EE de 19: 0. Añadir a 50 ml de frasco volumétrico enjuagado con hexano. A~nadir ~ 15 ml de solución de hexano-MTBE (reactivo 3) al matraz, agitar para disolver. Retirar a 50 ml con solución de hexano-MTBE (reactivo 3). Cap y remolino para mezclar. Transferir a tubos de vidrio enjuagados con hexano con tapas revestidas con PTFE y almacenar a -20 ° C.
2. DIA 1 - Extracción de ácidos grasos del suelo (4 a 5 h para ~ 40 muestras)
- Prepare tres pipetas dispensadoras de 5 a 10 ml para el tampón P, cloroformo y metanol.
NOTA: Se han propuesto alternativas menos peligrosas al cloroformo como extractantes. Estos pueden dar como resultado tasas de recuperación de lípidos equivalentes 16 , 17 , pero no se evalúan en este protocolo. - Pesar los suelos liofilizados en tubos de centrífuga de 30 ml y registrar la masa del suelo.
- Hacer dos espacios en blanco e incluir un cheque estándar (un suelo que se ha extraído anteriormente).
- En la campana extractora, añadir los reactivos al suelo en el tubo de centrífuga en el siguiente orden: P-buffer, CHCl 3 y MeOH (Tabla 1). Permitir que el tiempo de suelos para mojar después de la adición-buffer P antes de añadir el CHCl3. Cubra los tubos de centrífuga firmemente y cubra para protegerlos de la luz.
- Colóquelos en el agitador horizontalmente asegurándose de que estén bien asegurados. Con la velocidad ajustada en 280 rpm, agitar durante 1 h 18 .
- Preparar dos 16 mm x 150 mm tubos de vidrio para cada muestra como sigue: Etiqueta del tubo y añadir el mismo volumen de CHCl3 y un volumen igual de P-buffer.
- Retire los tubos de centrifugación del agitador y centrifugue durante 10 min a 1.430Xg y 25ºC. La separación de fases debe ser visible en el tubo de vidrio.
- En la campana extractora, decantar el sobrenadante del tubo de centrifugación en uno de los tubos preparados en el paso 2.6. Repita los pasos 2.4 a 2.5 y decantar el sobrenadante en el segundo tubo.
- Cubra firmemente todos los tubos de vidrio de 16 mm × 150 mm con tapas con revestimiento de PTFE e invierta 10 x para mezclar.
- Deje que las muestras permanezcan inalteradas durante la noche para completar la separación de las dos fases. Para ello, mantenga las muestras en un armario / espacio oscuro o cubiertas con papel de aluminio a temperatura ambiente. Es aceptable permitir que los extractos se separen durante el fin de semana.
- Alternativamente, si se desea pasar directamente a la siguiente etapa, o si las muestras se alteran al día siguiente, se centrifugarán las muestras durante 10 min a 1.000 xg y 25ºC.
NOTA: Somete todas las muestras dentro de un grupo de comparación al mismo tiempo de espera de separación de fase.
- Alternativamente, si se desea pasar directamente a la siguiente etapa, o si las muestras se alteran al día siguiente, se centrifugarán las muestras durante 10 min a 1.000 xg y 25ºC.
3 DÍAS2 - Aislamiento de lípidos (3 a 4 h para ~ 40 muestras)
- Encienda el aparato de evaporación y el purgador de disolventes. Ajustar el aparato de evaporación a 33 ° C y precalentar.
- Instalar un aspirador de vacío en la campana: un matraz de brazo lateral conectado a una bomba de vacío, una longitud de tubo de alta pureza y una pipeta de vidrio.
- Aspirar la capa superior y la interfaz (aproximadamente 2/3 del camino hacia abajo) de los dos tubos de vidrio, reteniendo la capa acuosa. Repita el proceso con una pipeta limpia para cada muestra.
- Combinar las capas acuosas de extracto del segundo tubo con el del primer tubo por decantación cuidadosa. Asegúrese de que el tubo que está decantando está libre de arañazos o grietas.
- Una vez que los extractos de CHCl 3 se combinan, inspeccionar el líquido - debe ser claro. Si no agrega MeOH hasta que esté despejado.
- Seque todas las muestras utilizando el aparato de evaporación al vacío. Comienza con ajustes de temperatura 33 ° C, vórtice 26% y presión 400 mbar.
- PAGAtar las muestras en el sistema de evaporación y después de 10 min bajar lentamente la presión a 350 mbar.
- Controle el progreso y cuando el nivel del líquido en el tubo haya alcanzado el nivel del bloque de calefacción, disminuya la presión a 300 mbar.
- Continúe monitoreando la altura del líquido restante y cuando sea aproximadamente 1 cm de profundidad, disminuya la presión a 200 mbar.
- Después de que las muestras se hayan secado, retire las muestras y haga funcionar la bomba durante 10 minutos adicionales para eliminar los vapores.
- Cubrir los tubos con fuerza y almacenar los lípidos en un congelador a -80 ° C.
- Deseche el líquido aspirado de acuerdo con las normas de seguridad química aplicables.
4. DÍA 3 - Saponificación y Metilación (6 a 7 h para ~ 40 Muestras)
- Encienda los baños de agua. Coloque el baño de 1 a 95 ° C y bañe de 2 a 80 ° C.
- Coloque los dispensadores de la pipeta y asegúrese de que están dispensando el volumen correcto.
Reactivo 1: 1 ml por muestra
Reactivo 2: 2 ml por muestra
Reactivo 3: 1,25 ml por muestra
Reactivo 4: 3 ml por muestra
NOTA: El proceso de calentamiento generará presión en el tubo. No utilice tubos rayados, agrietados o astillados. - Usando un dispensador de pipeta, añada 1,0 mL del Reactivo 1 a los lípidos secos. Tape firmemente, vórtice durante 5 s y colóquelo en un estante.
- Coloque la rejilla de tubos de muestra en el baño de agua a 95 ° C durante 5 min. Retire la rejilla de tubos del baño y verifique si hay fugas en los tubos, indicados por burbujas en el tubo. Vuelva a apretar o reemplace las tapas de los tubos con fugas y compruebe de nuevo si hay grietas o virutas. Continuar calentando los tubos en el baño de agua durante 10 minutos adicionales.
- Retirar las muestras del baño 1 y colocarlas en el baño 2 (80 ° C) y continuar la incubación durante otros 15 min.
- Retire los tubos y enfríe las muestras colocando el estante en una cacerola de agua del grifo para enfriar.
- Añadir 2 ml de Reactivo 2 a cada muestra. Tape firmemente y agite durante 5 a 10 seg.
- Colocar la rejilla en el baño de agua a 80 ° C e incubar durante 10 min.
- Retire la rejilla de los tubos del baño de agua y poner en una cacerola de agua del grifo para el enfriamiento. Agite el rack de tubos para acelerar el proceso de enfriamiento.
NOTA: No exceda el tiempo y la temperatura. Demasiada calefacción podría degradar FAMEs. - Utilizando el dispensador de pipeta, añadir 1,25 ml de Reactivo 3 a cada tubo de muestra para extraer los ésteres metílicos de ácidos grasos. Tape firmemente y coloque los tubos en el agitador durante 10 min.
- Después de agitar, deje que la rejilla de tubos se asiente durante 10 minutos para que las fases se separen. Transferir la fase orgánica (capa superior) a un tubo de ensayo de vidrio de 16 mm x 100 mm usando una pipeta de vidrio.
- Repita la extracción añadiendo de nuevo el reactivo 3, agitando, permitiendo separar las fases y transfiriendo la fase superior.
NOTA No transfiera ninguna de las fases inferiores. Está bien dejar una pequeña cantidad de fase superior en el tubo. - Usando el dispensador de pipeta, agregue 3 mL de Reactivo 4 alY tubos de 16 mm x 100 mm.
- Tape los tubos de ensayo firmemente y agite durante 20-30 s.
- Después de agitar en vórtex, centrifugar durante 3 min a 1.000 xg.
- Usando una pipeta de vidrio limpia, aspirar la fase orgánica superior y transferir a un vial ámbar de 4 ml.
- Encienda el aparato de evaporación y el purgador de disolventes. Ajustar el aparato de evaporación a 30 ° C, la velocidad del vórtice al 26%, el vacío a 200 mbar, y presionar el precalentamiento.
- Supervise el progreso. Después de que las muestras se hayan secado, retire las muestras y haga funcionar la bomba durante 10 minutos adicionales para eliminar los vapores.
- Retire la pipeta de las botellas de reactivo 1 y 4 y bombee a través de algún ácido diluido ( por ejemplo, HCl al 1%), seguido por agua DI. Enjuague la pipeta usada para el Reactivo 2 bombeando a través de agua DI. En la campana, escurrir el disolvente de la pipeta utilizada para el Reactivo 3 en un recipiente apropiado y mantenerlo en la campana hasta que los residuos del disolvente se hayan evaporado.
- Almacene las pipetas invertidas, con los émbolos quitados para evitar el pegamentoG de las válvulas de retención.
- Utilice las pipetas de vidrio una vez y luego deséchelas en un recipiente apropiado.
- En el capó, permita que los residuos de disolvente en la cristalería se evaporen.
- Enjuague la cristalería con agua limpia y solución detergente.
5. DÍA 4 - Preparación de la solución de trabajo y transferencia de FAMEs a los viales de GC (2-3 h para ~ 40 muestras)
- Reúna los materiales: viales ámbar de 4 ml con muestras secas; Frascos de 2 ml GC ambar; Insertos y tapas de vidrio de fondo plano de 400 μl; Jeringa de vidrio de 500 μL; Matraz volumétrico de 50 ml; Solución madre - Nonadecanoato de etilo (estándar interno de EE de 19: 0) en hexano / MTBE 50/50 (Reactivo 3).
- Utilizando la jeringa de vidrio, añadir 500 μl de solución madre de nonadecanoato de etilo (19: 0 EE) a un matraz aforado de 50 ml.
- Llene el matraz con la puntuación volumétrica con el Reactivo 3.
- Tape el matraz e invierta 5x para mezclar.
- Transferir 3 ml a un vial de 4 ml limpio para un depósito de solución de trabajo.
- Usando elY la jeringa de vidrio, añadir 300 μL de la solución de trabajo a cada uno de los viales de 4 ml que contienen los ésteres metílicos de ácidos grasos secos y la tapa.
- Vortex la muestra durante 15 s y dejar de lado durante 15 min.
- Utilizando una pipeta Pasteur de vidrio, transfiera cuidadosamente los ésteres metílicos de ácidos grasos suspendidos a un vial GC de 2 ml que contiene el inserto de 400 μl.
- Guarde los viales de GC sellados en un congelador de -20 ° C antes del análisis.
- Presentar muestras para el análisis GC.
NOTA: El análisis debe llevarse a cabo utilizando una columna de GC específica y las condiciones que se describen en el Archivo Suplementario S1 . Es mejor que el análisis de GC se complete dentro de 2 semanas de metilación.
Resultados
Las tablas de datos de los informes pueden agruparse en una hoja de cálculo o una base de datos. Después de ajustar el factor de respuesta (un factor de corrección que normaliza la respuesta para diferentes longitudes de cadena), las áreas de los picos se pueden comparar con el área del pico de las normas externas o internas para llegar a una concentración en el extracto. Al dividir por la masa de suelo extraída, los datos pueden expresarse como masa de FAME por gramo de suelo o, utilizando el peso molecular de cada FAME, el nmol más comúnmente comunicado por gramo de suelo. La suma de FAME microbianos es indicativa de la biomasa microbiana total, y puede ser comparada entre los tratamientos ( Figura 1 ). Ciertas FAME también pueden estar asociadas con grupos microbianos particulares tales como bacterias Gram-positivas o Gram-negativas, actinomicetos, hongos micorrízicos arbusculares y hongos saprotróficos 19 , 20 , 21 , 22 , 23 , 24 . Las proporciones de la masa de biomarcadores específicos pueden reflejar la abundancia relativa de estos grupos ( Figura 2 ]. En general los patrones de abundancia de FAME crean huellas dactilares a nivel comunitario, lo que permite comparar la disimilitud de la comunidad microbiana a través de técnicas multivariantes como la ordenación ( Figura 3 ). En contraste con la mayoría de los enfoques basados en el ADN, los datos de los lípidos a nivel de la comunidad pueden analizarse como abundancias relativas o absolutas. Si la biomasa total difiere sustancialmente entre las muestras, estos dos enfoques darán resultados muy diferentes; Las preguntas ecológicas subyacentes al experimento deben determinar qué enfoque se utiliza.
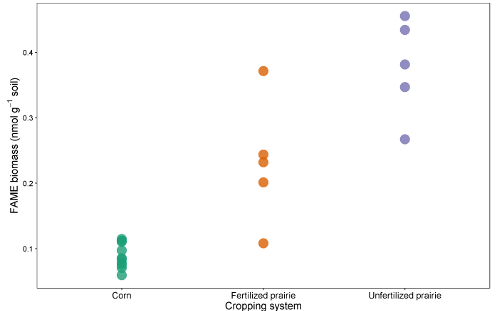
F Igura 1 : biomasa total de FAME. Home Idiomas Ingresar a Epistemonikos Búsqueda avanzada Comparación de la biomasa total de biomarcadores FAME (nmol g -1 de suelo) de maíz continuo, pradera fertilizada y pradera no fertilizada. Haga clic aquí para ver una versión más grande de esta figura.
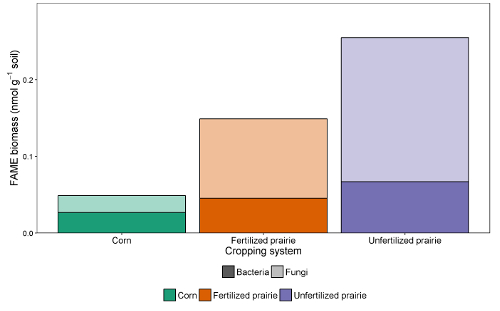
Figura 2 : Biomasa FAME de bacterias y hongos. Comparación del tratamiento de la biomasa de biomarcadores FAME (nmol g -1 ) bacteriana y fúngica de maíz continuo, pradera fertilizada y pradera no fecundada. Masa fúngica y bacteriana de abundancia absoluta. Promedio (suma f / suma b). Haga clic aquí para ver una versión más grande de esta figura.
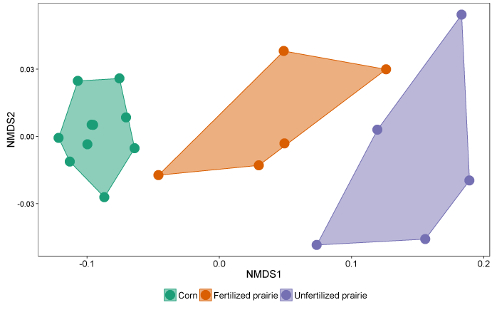
Figura 3 : Escalamiento dimensional no métrico de biomarcadores lipídicos FAME. Home Idiomas Ingresar a Epistemonikos Búsqueda avanzada Comparación de la comunidad microbiana general con perfiles de abundancia absoluta de todos los biomarcadores microbianos FAME lípidos. En este ejemplo, el maíz continuo y las comunidades de praderas no fertilizadas se separan y están muy separadas, mientras que algunas muestras de praderas fertilizadas tienen comunidades microbianas que se asemejan a las del maíz y otras se asemejan a la pradera no fertilizada. Haga clic aquí para ver una versión más grande de esta figura.
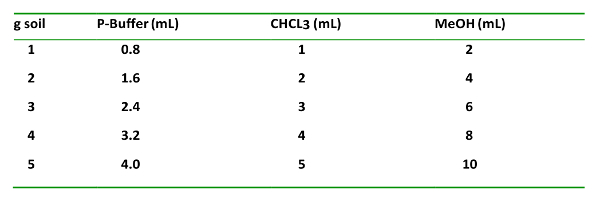
Tabla 1: T h e tanl ven tipo t, ti proporcionalidad en s añade g -1 suelo, un d t h EIR orde r o f añadir ITI o n. Esto es importante para la correcta extracción y separación de las fases orgánica y acuosa.
Discusión
Para el examen de múltiples muestras de experimentos con muchas repeticiones y / o unidades experimentales, los investigadores pueden encontrar el análisis de ácidos grasos fosfolípidos (PLFA) a ser prohibitivo en términos de tiempo y materiales [ 25] . Con el método PLFA, los fosfolípidos de la membrana celular se extraen, purifican e identifican usando la extracción acuosa-orgánica de dos fases biológica modificada de Bligh y Dyer. Esto es seguido por cromatografía de sílice en fase sólida para separar los lípidos por polaridad, y una metilación alcalina de ácidos grasos fosfolípidos en ésteres metílicos de ácidos grasos. En el perfil de PLFA el rendimiento lipídico puede ser bajo pero de una pureza muy alta. Microbial ID introdujo un método alternativo, el procedimiento de metil-éster de ácidos grasos (MIDI-FA). En el método MIDI-FA, todos los lípidos se extraen directamente de cultivos puros o muestras de suelo / sedimento 11 , 12 , 27 aSaponificación. Este método tiene menor pérdida de lípidos y es rápido porque no tiene ninguna de las etapas de concentración o purificación del método PLFA. Sin embargo, aunque el método MIDI-FA es rápido y menos costoso, ya que fue diseñado originalmente para identificar organismos en cultivo puro, no hay etapas de extracción o purificación iniciales. Por lo tanto, puede incluir compuestos similares a los lípidos co-extraídos de la materia orgánica del suelo que distorsionan la firma de la comunidad 27 , 28 , 29 . Debido a que esta inclusión también puede distorsionar las mediciones de biomasa, MIDI-FA se ha utilizado normalmente sólo para describir de manera grosera cualitativamente los lípidos del suelo [ 13] .
El procedimiento que describimos aquí combina lo mejor de los dos procedimientos de extracción separados: 1) una extracción y concentración de lípidos usando el método de Bligh y Dyer 26 modificado, y 2) el éster metílico de ácido graso saPontificación, metilación, extracción y lavado de base desarrollado comercialmente. Este método fue desarrollado para lograr los beneficios de ambos protocolos, mientras que minimizar las desventajas [ 15] . Al realizar la extracción inicial y aislar los componentes orgánicos solubles ( por ejemplo lípidos) antes de realizar MIDI-FA, y completar esto con un paso de purificación, este protocolo ofrece un equilibrio entre velocidad y precisión. Aunque este método puede no ser adecuado cuando se requiere alta pureza ( es decir , para el análisis de 13 C de PLFA) o cuando se analizan por separado los fosfolípidos y los lípidos neutros, en muchos casos permite detectar respuestas de la comunidad microbiana a las condiciones ambientales con mayor sensibilidad que las de ADN Métodos 30 , 31 , 32 , 33 . Los lípidos de la membrana se descomponen rápidamente después de la muerte celular,Reflejan la comunidad microbiana viviente en el momento del muestreo 5 , 7 , en contraste con el ADN ambiental en el que gran parte de la información proviene de organismos muertos o inactivos [ 34] . Dada la alta tasa de latencia observada entre los microorganismos del suelo 35 , la caracterización de la biomasa viva puede utilizarse para comprender las interacciones temporales planta-microbio a una escala temporal relativamente fina y los biomarcadores lipídicos pueden utilizarse para analizar el estado fisiológico de la comunidad microbiana 7 . Se ha demostrado que los métodos de alto rendimiento son necesarios para evaluar la respuesta microbiana en grandes campos de configuración [ 25] , y mientras que el método que aquí se propone no reproduce la exactitud de perfiles biomarcadores PLFA, aumenta el rendimiento, al tiempo que minimizar la variabilidad realizada con el MIDI- procedimiento. El método ha demostrado ser una herramienta eficaz paraPreguntas relacionadas con la dinámica de comunidades microbianas en una amplia gama de suelos en estudios agrícolas y de ecosistemas a gran escala 36 , 37 , 38 , 42 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 .
Las clases de lípidos se combinan con este método y puede haber una pérdida de la información contenida en esas clases separadas 22 , 39 , pero la combinación de las clases de lípidos puede fortalecer la potencia para detectar el origen de hongos micorrízicos arbusculares del ω5c 16: 1 de ambos fosfo - y los lípidos neutros 40 . Además, mientras que tEl número de ácidos grasos desconocidos (que podrían derivarse de materias orgánicas no vivas) puede ser mayor con este método, se demostró que es inferior a la FA-MIDI y permite la comparación de los perfiles lipídicos de los estudios con muchas muestras donde la muestra La capacidad de producción es una preocupación 15 . Se cree que la fracción neutra se deriva principalmente de los lípidos almacenados producidos por los hongos, aunque también puede haber alguna contribución menor de la fauna del suelo 41 . A la luz de esto, el método descrito aquí puede producir resultados que muestran una mayor contribución de los lípidos fúngicos 18: 2 ω 6,9c y 18: 1 ω 9c que PLFA. Los otros lípidos que tienden a aparecer en la fracción neutra incluyen algunos de los ácidos grasos saturados, por ejemplo 16: 0, 18: 0, 20: 0.
Hay diferentes maneras en que los datos de lípidos pueden ser expresados y analizados. Las representaciones más comunes son la abundancia (nmol g -1 suelo), mole fractioN (nmol lıpido individual nmol -1 lıpido total) y porcentaje molar (fracción molar * 100). Normalizados por los lípidos totales en una muestra, la fracción molar y el porcentaje molar son medidas de la abundancia relativa de un lípido dado. Después de una transformación apropiada, por ejemplo , arcosina de raíz cuadrada, la fracción molar es apropiada para su uso en análisis por componentes principales o ordenación de análisis de redundancia. Abundancia es la cantidad absoluta de un determinado lípido extraído por gramo de suelo. Debido a que la cantidad de lípidos por célula es razonablemente constante y la extracción de lípidos es altamente eficiente y completa, la abundancia total es una buena estimación de los lípidos totales y la abundancia de indicadores clave refleja la biomasa del grupo ecológico que representa 17 . Finalmente, una buena manera de ver la composición de la comunidad microbiana es utilizar métodos de análisis multivariante 16 , por ejemplo , métodos de ordenación tales como escalamiento multidimensional no métrico (NMDS -Que no necesita transformación de datos) o análisis de componentes principales (PCA), puede ser útil para comparar la abundancia relativa de todos los biomarcadores de lípidos.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado en parte por el Centro de Investigación de la Bioenergía de los Grandes Lagos del DOE (Oficina de Ciencia de la DOE BER DE-FC02-07ER64494) y la Oficina de Eficiencia Energética y Energía Renovable de DOE OBP (DE-AC05-76RL01830). Los autores desean reconocer a Anders Gurda por su paciente y hábil aportación a la videografía y edición.
Materiales
| Name | Company | Catalog Number | Comments |
| RapidVap | Labconco | 7900000 | Evaporative system |
| RapidVap | Labconco | 7491400 | Sample block |
| Chem-resistant vacuum pump | Labconco | 7584000 | Vacuum pump |
| Chemical trap cannister | Labconco | 7815300 | Trap cannister |
| Solvent insert | Labconco | 7515200 | Solvent trap insert |
| Diaphragm pump | Welch | 7398000 | DryFast vacuum pump |
| Water bath | Fisher | 15-462-15Q | 2 well water bath |
| Gas chromatograph | Various | N/A | For sample analysis |
| Centrifuge | Various | N/A | For sample separation |
| Freeze Dryer | Various | N/A | For sample lyophilization |
| Repipet (6) | BrandTech | 4720440 | For dispensing reagents |
| Vortex | Fisher | 02-215-365 | Analog vortex mixer |
| Teflon centrifuge tubes | Thermo/Nalgene | 3114-0030 | Teflon sample tubes |
| Caps | Thermo/Nalgene | DS3131-0020 | Caps for teflon tubes |
| Test tube | Corning | 9825-16 | 16x100mm tubes |
| Test tube | Corning | 9825-16xx | 16x150mm tubes |
| Caps | Corning | 9998-15 | 15-415 thread black phenolic caps w/PTFE liner |
| Pasteur pipets | Fisher | 13-678-20B | 14.6cm |
| 500 uL glass syringe | Fisher | 13684106LC | Hamilton 81217 |
| Amber vials | Agilent | 5182-0716 | 4mL Amber vials |
| Caps | Agilent | 5182-0717 | Blue screw caps |
| Inserts | Agilent | 5181-3377 | 400uL flat bottom glass inserts |
| Standards | |||
| 19:0 ethyl nonadecanoate (Ethyl nonadecanoate) | VWR | TCN0459-5G | Analytical standard |
| Chemicals | |||
| Dipotassium phosphate (K2HPO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Potassium phosphate monobasic (KH2PO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Methanol (CH3OH - HPLC grade or better) | Various | N/A | For making Reagents 1 & 2 |
| Sodium hydroxide (NaOH - ACS grade or better) | Various | N/A | For making Reagents 1 & 4 |
| Hydrochloric acid (6N HCL) | Various | N/A | For making Reagent 2 |
| Hexane (HPLC grade or better) | Various | N/A | For making Reagent 3 |
| MTBE (Methyl tert-butyl ether - HPLC grade or better) | Various | N/A | For making Reagent 3 |
Referencias
- Van Der Heijden, M. G. a., Bardgett, R. D., Van Straalen, N. M. The unseen majority: Soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecol Lett. 11 (3), 296-310 (2008).
- Bever, J. D., et al. Rooting theories of plant community ecology in microbial interactions. Trends Ecol Evol. 25 (8), 468-478 (2010).
- Suleiman, A. K. A., Manoeli, L., Boldo, J. T., Pereira, M. G., Roesch, L. F. W. Shifts in soil bacterial community after eight years of land-use change. Syst Appl Microbiol. 36 (2), 137-144 (2013).
- Sayer, E. J., et al. Grassland management influences spatial patterns of soil microbial communities. Soil Biol Biochem. 61, 61-68 (2013).
- Fernandes, M. F., Saxena, J., Dick, R. P. Comparison of Whole-Cell Fatty Acid (MIDI) or Phospholipid Fatty Acid (PLFA) Extractants as Biomarkers to Profile Soil Microbial Communities. Microbial Ecol. 66 (1), 145-157 (2013).
- van der Heijden, M. G. A., Wagg, C. Soil microbial diversity and agro-ecosystem functioning. Plant Soil. 363 (1-2), 1-5 (2013).
- Allison, V. J., Miller, R. M., Jastrow, J. D., Matamala, R., Zak, D. R. Changes in soil microbial community structure in a tallgrass prairie chronosequence. Soil Sci Soc Am J. 69 (5), 1412-1421 (2005).
- Kowalchuk, G. A., Buma, D. S., de Boer, W., Klinkhamer, P. G. L., van Veen, J. A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. A van Leeuw. 81 (1-4), 509-520 (2002).
- Bardgett, R. D., Hobbs, P. J., Frostegard, A. Changes in soil fungal:bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biol Fert Soils. 22 (3), 261-264 (1996).
- Frostegård, &. #. 1. 9. 7. ;., Tunlid, A., Bååth, E. Use and misuse of PLFA measurements in soils. Soil Biol Biochem. 43 (8), 1621-1625 (2011).
- Haack, S. K., Garchow, I. H., Odelson, D. A., Forney, L. J., Klug, M. J. Accuracy , Reproducibility , and Interpretation of Fatty Acid Methyl Ester Profiles of Model Bacterial Communitiest. Appl Environ Microbiol. 60 (7), 2483-2493 (1994).
- Drenovsky, R. E., Elliott, G. N., Graham, K. J., Scow, K. M. Comparison of phospholipid fatty acid ( PLFA ) and total soil fatty acid methyl esters ( TSFAME ) for characterizing soil microbial communities. Soil Biol Biochem. 36, 1793-1800 (2004).
- Cavigelli, M. A., Robertson, G. P., Klug, M. Fatty acid methyl ester ( FAME ) profiles as measures of soil microbial community structure. Plant Soil. 170, 99-113 (1995).
- Determination of microbial community structure using phospholipid fatty acid profiles. Molecular microbial ecology manual Available from: https://www.researchgate.net/profile/Robert_Findlay2/publication/281208148_Determination_of_microbial_community_structure_using_phospholipid_fatty_acid_profiles/links/55db4f0b08aec156b9afe776.pdf (2004)
- Balser, T. C., Liang, C., Gutknecht, J. L. Linking microbial community analysis and ecosystem studies: A rapid lipd analysis protocol for high throughput. Biol Fert Soils. , (2017).
- Findlay, R. H., King, G. M., Watling, L., Watling, L. E. S. Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sediments Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sedimentst. Appl Environ Microbiol. 55 (11), 2888-2893 (1989).
- Willers, C., Jansen van Rensburg, P. J., Claassens, S. Microbial signature lipid biomarker analysis - an approach that is still preferred, even amid various method modifications. J Appl Microbiol. 118 (6), 1251-1263 (2015).
- Zelles, L., Bai, Q. Y., Beck, T., Beese, F. Signature fatty-acids in phospholipids and lipopolysachharides as indicators of microbial biomass and community structure in agricultural soils. Soil Biol Biochem. 24 (4), 317-323 (1992).
- Frostegard, A., Tunlid, A., Baath, E. Phospholipid fatty-acid composition, biomass, and activity of microbial communities from 2 soil types experimentally exposed to different heavy-metals. Appl Environ Microbiol. 59 (11), 3605-3617 (1993).
- Federle, T. W., Dobbins, D. C., Thorntonmanning, J. R., Jones, D. D. Microbial biomass, activity, and community structure in subsurface soils. Ground Water. 24 (3), 365-374 (1986).
- Vestal, J. R., White, D. C. Lipid analysis in microbial ecology - quantitative approaches to the study of microbial communities. BioScience. 39 (8), 535-541 (1989).
- Wilkinson, S. G. Gram negative bacteria. Microbial Lipids. , 299-488 (1988).
- Balser, T. C., Treseder, K., Ekenler, M. Using lipid analysis and hyphal length to quantify AM and saprotrophic fungal abundance along a soil chronosequence. Soil Biol Biochem. 37 (3), 601-604 (2005).
- Buyer, J. S., Sasser, M. High throughput phospholipid fatty acid analysis of soils. Appl Soil Ecol. 61, 127-130 (2012).
- Bligh, E. G., Dyer, W. J. A rapid method of total lipid extraction and purification. Can J Biochem Phys. (37), 911-917 (1959).
- Schutter, M. E., Dick, R. P. Comparison of Fatty Acid Methyl Ester (FAME) Methods for Characterizing Microbial Communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Jandl, G., Leinweber, P., Schulten, H., Ekschmitt, K. Contribution of primary organic matter to the fatty acid pool in agricultural soils. Soil Biol Biochem. 37, 1033-1041 (2005).
- Nielsen, P., Petersen, S. O. Ester-linked polar lipid fatty acid profiles of soil microbial communities: a comparison of extraction methods and evaluation of interference from humic acids. Soil Biol Biochem. 32, 1241-1249 (2000).
- Duncan, D. S., Jewell, K. A., Suen, G., Jackson, R. D. Detection of short-term cropping system-induced changes to soil bacterial communities differs among four molecular characterization methods. Soil Biol Biochem. 96, 160-168 (2016).
- Liang, C., et al. Switchgrass rhizospheres stimulate microbial biomass but deplete microbial necromass in agricultural soils of the upper Midwest, USA. Soil Biol Biochem. 94, 173-180 (2016).
- Jangid, K., et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties. Soil Biol Biochem. 43 (10), 2184-2193 (2011).
- Ritz, K., et al. Spatial structure in soil chemical and microbiological properties in an upland grassland. FEMS Microbiol Ecol. 49 (2), 191-205 (2004).
- Carini, P., et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. J Chem Inf Model. 53 (9), 1689-1699 (2013).
- Lennon, J. T., Jones, S. E. Microbial seed banks: The ecological and evolutionary implications of dormancy. Nat Rev Microbiol. 9 (2), 119-130 (2011).
- Gutknecht, J. L. M., Field, C. B., Balser, T. C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years. Glob Change Biol. 18, 225-269 (2012).
- Pei, Z., et al. Soil and tree species traits both shape soil microbial communities during early growth of Chinese subtropical forests. Soil Biol Biochem. 96, 180-190 (2016).
- Oates, L. G., Duncan, D. S., Sanford, G. R., Liang, C., Jackson, R. D. Bioenergy cropping systems that incorporate native grasses stimulate growth of plant-associated soil microbes in the absence of nitrogen fertilization. Ag Ecosys Environ. 233, 396-403 (2016).
- Bååth, E. The use of neutral lipid fatty acids to indicate the physiological conditions of soil fungi. Microbial Ecol. 45 (4), 373-383 (2003).
- Ngosong, C., Gabriel, E., Ruess, L. Use of the signature Fatty Acid 16:1ω5 as a tool to determine the distribution of arbuscular mycorrhizal fungi in soil. J Lipids. 2012, 236807 (2012).
- Sharma, M. P., Buyer, J. S. Comparison of biochemical and microscopic methods for quantification of arbuscular mycorrhizal fungi in soil and roots. Appl Soil Ecol. 95, 86-89 (2015).
- Oates, L. G., Balser, T. C., Jackson, R. D. Subhumid pasture soil microbial communities affected by presence of grazing, but not grazing management. Appl Soil Ecol. 59, 20-28 (2012).
- Liang, C. Potential legacy effects of biofuel cropping systems on soil microbial communities in southern Wisconsin, USA. Ag Sci. 2 (2), 131-137 (2011).
- Herzberger, A. J., Duncan, D. S., Jackson, R. D. Bouncing Back Plant-Associated Soil Microbes Respond Rapidly to Prairie Establishment. PloS One. 9 (12), 1-14 (2014).
- Fraterrigo, J. M., Balser, T. C., Turner, M. G. Microbial community variation and its relationship with nitrogen mineralization in historically altered forests. Ecology. 87 (3), 570-579 (2006).
- Kao-Kniffin, J., Balser, T. C. Elevated CO2 differentially alters belowground plant and soil microbial community structure in reed canary grass-invaded experimental wetlands. Soil Biol Biochem. 39 (2), 517-525 (2007).
- Mentzer, J. L., Goodman, R. M., Balser, T. C. Microbial response over time to hydrologic and fertilization treatments in a simulated wet prairie. Plant Soil. 284 (1-2), 85-100 (2006).
- Ushio, M., Wagai, R., Balser, T. C., Kitayama, K. Variations in the soil microbial community composition of a tropical montane forest ecosystem: Does tree species matter. Soil Biol Biochem. 40 (10), 2699-2702 (2008).
- Bartelt-Ryser, J., Joshi, J., Schmid, B., Brandl, H., Balser, T. Soil feedbacks of plant diversity on soil microbial communities and subsequent plant growth. Perspect Plant Ecol. 7 (1), 27-49 (2005).
- Fichtner, A., von Oheimb, G., Härdtle, W., Wilken, C., Gutknecht, J. L. M. Effects of anthropogenic disturbances on soil microbial communities in oak forests persist for more than 100 years. Soil Biol Biochem. 70, 79-87 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados