Method Article
Méthode d'extraction et d'analyse de lipides pour caractériser les microbes du sol dans les expériences avec de nombreux échantillons
Dans cet article
Résumé
L'article décrit une méthode qui augmente le débit tout en équilibrant l'effort et la précision pour l'extraction des lipides à partir des membranes cellulaires des microorganismes à utiliser pour caractériser les lipides totaux et l'abondance relative des lipides indicateurs pour déterminer la structure de la communauté microbienne du sol dans les études avec de nombreux échantillons.
Résumé
Les communautés microbiennes sont des moteurs importants et des régulateurs des processus écosystémiques. Pour comprendre comment la gestion des écosystèmes peut affecter les communautés microbiennes, une technique relativement précise mais intensive en effort pour analyser la composition de la communauté microbienne est l'analyse des acides gras phospholipides (PLFA). Le PLFA a été développé pour analyser les biomarqueurs de phospholipides, qui peuvent être utilisés comme indicateurs de la biomasse microbienne et la composition de larges groupes fonctionnels de champignons et de bactéries. Il a été habituellement utilisé pour comparer les sols dans les communautés végétales alternatives, l'écologie et les régimes de gestion. La méthode PLFA s'est avérée être sensible à la détection des décalages dans la composition de la communauté microbienne.
Une autre méthode, l'extraction et l'analyse de l'ester méthylique d'acides gras (MIDI-FA) a été développée pour l'extraction rapide des lipides totaux, sans séparation de la fraction phospholipide, des cultures pures comme technique d'identification microbienne. Cette méthode estRapide mais moins adapté pour les échantillons de sol car il manque une étape initiale séparant les particules de sol et commence à la place avec une réaction de saponification qui produit vraisemblablement des artefacts de la matière organique de fond dans le sol.
Cet article décrit une méthode qui augmente le débit tout en équilibrant l'effort et la précision pour l'extraction des lipides à partir des membranes cellulaires des microorganismes à utiliser pour caractériser les lipides totaux et l'abondance relative des lipides indicateurs pour déterminer la structure de la communauté microbienne du sol dans les études avec de nombreux échantillons. La méthode combine la précision obtenue grâce au profilage PLFA en extrayant et en concentrant les lipides du sol comme première étape et en réduisant l'effort en saponisant la matière organique extraite et en procédant avec la méthode MIDI-FA en deuxième étape.
Introduction
Compte tenu du rôle clé des microorganismes dans le cyclage des nutriments 1 , la modification de la composition 2 de la communauté végétale, la régulation de la productivité végétale 3 et la décomposition de la matière organique 4 , la compréhension des communautés microbiennes du sol est essentielle à la compréhension des écosystèmes terrestres.
En raison de leur abondance relativement élevée dans le sol et de leur signature chimique, les biomarqueurs lipidiques peuvent être utilisés pour évaluer les groupes écologiques dominants comprenant les communautés microbiennes du sol 5 . En quantifiant les biomarqueurs lipidiques caractéristiques de différents groupes microbiens, nous pouvons estimer les lipides totaux, puis séparer ces lipides en groupes écologiquement pertinents tels que les bactéries Gram-positives (Gm +) et Gram négatif (Gm-), les mycorhizes arbusculaires (AM) et saprotrophiques Champignons et actinomycètes 5 , Ss = "xref"> 6 , 7 , 8 .
Il existe de nombreuses méthodes pour caractériser les aspects des communautés microbiennes. La méthode PLFA est utilisée habituellement pour comprendre la structure de base de la communauté microbienne. C'est un moyen efficace d'évaluer l'abondance relative des groupes microbiens ainsi que la biomasse microbienne totale. En raison du renouvellement rapide des lipides, le profilage PLFA permet également une détection relativement rapide des changements dans la communauté microbienne du sol et donne des informations qui permettent une comparaison de la fonction de l'écosystème, p. Ex. Les rapports fongiques: bactériens pour évaluer les taux de cyclage des éléments nutritifs 1 , 9 , 10 . Toutefois, bien que la méthode d'extraction PLFA soit respectée et respectée, elle prend aussi beaucoup de temps et ne se prête pas bien aux études à l'échelle de l'écosystème qui nécessitent un grand nombre d'échantillons provenant de répétitions d'échelle de terrainF "> 11 , 12 .
En revanche, la méthode d'extraction d'ester méthylique d'acide gras (MIDI-FA) a le potentiel de permettre un débit rapide. Dans cette méthode, les échantillons sont saponifiés, transformés en FAME, extraits, puis analysés. La méthode MIDI-FA est rapide mais moins discriminante que le PLFA, qui combine l'extraction des lipides avec la séparation des différentes classes lipidiques 13 (phospholipides, lipides neutres et glycolipides).
Dans ce protocole, nous décrivons une méthode qui combine des éléments du profilage lipidique de PLFA et MIDI-FA. Il utilise l'extraction des lipides en utilisant les étapes initiales d'extraction du chloroforme de la méthode de Bligh et Dyer modifiée, puis la saponification et la conversion en FAME. Cela fournit un moyen solide de détecter la structure de la communauté microbienne tout en excluant une grande partie du bruit de fond provenant de matériel non microbien 5 , 14 . Cette méthode a été développée pour atteindre un équilibre entre les protocoles PLFA et MIDI-FA, c'est-à-dire conserver la plus grande précision tout en augmentant le débit afin de rendre logistique et économiquement possible d'analyser les lipides à partir d'études à grande échelle avec de nombreux échantillons 15 . En effectuant l'extraction initiale et en isolant les composants organiques solubles ( p. Ex. Les lipides) avant d'effectuer MIDI-FA, et en terminant avec une étape de purification, le protocole offre un équilibre entre la vitesse et la précision.
Protocole
REMARQUE: Portez toujours un équipement de protection individuelle approprié (EPI) tout au long de la procédure. Pour éviter une contamination potentielle de l'échantillon, ne pas toucher les verreries à mains nues. Portez des gants appropriés lors de l'exécution des étapes de protocole nécessitant une manipulation du chloroforme.
1. Préparations (2 jours pour ~ 40 échantillons)
- Recueillir du sol dans des sacs stériles et transporter du champ dans un récipient contenant de la glace. S'il n'est pas possible de tamiser du sol frais et de se figer immédiatement, rangez les échantillons dans un congélateur de -80 ° C jusqu'à ce que vous puissiez commencer l'analyse.
- Préparer les sols en homogénéisant. Retirer les racines et les pierres et briser les mottes par tamisage grossier ( par ex. 2 mm).
- Préparez-vous pour le lyophilisation en mettant les sous-échantillons dans un récipient approprié conformément aux instructions du manuel du séchoir à glaçons et séchez-le dès que possible. Une fois que les sols sont lyophilisés, entreposer dans un récipient scellé avec du dessiccateur jusqu'à l'extraction. Il est préférable de sA déchiré des sols lyophilisés à un minimum de -20 ° C mais de préférence à -80 ° C.
- En préparation pour l'extraction, éliminer les sols lyophilisés de l'entreposage et broyer jusqu'à une consistance de type farine. Les méthodes de broyage comprennent le broyeur à billes, le batteur à bille et le mortier et le pilon. Après avoir broyé le sol, entreposer dans un congélateur (voir 1.3).
NOTE: La quantité d'échantillon utilisée pour l'extraction dépend de sa teneur en matière organique. Une ligne directrice générale consiste à utiliser 0,5 à 1 g d'un sol de 12 à 18% en poids de carbone et 3 à 5 g pour un sol de 1 à 3% de carbone en poids. - Préventez les tubes de centrifugation de 30 ml avec de l'hexane. Ajouter environ 2 à 3 mL d'hexane aux tubes et vortex pendant 5 sec. Décanter l'hexane vers un autre tube, et le vortex. L'hexane (2 à 3 ml) peut être utilisé pour rincer en série six tubes. Stocker les tubes rincés à l'hexane dans la hotte aspirante et éliminer l'hexane utilisé dans un récipient approprié.
- Enveloppez la verrerie dans 2 à 3 couches de papier d'aluminium et placez-les dans le four à moufle. Cuire(Muffle) à 450 ° C pendant 4,5 h.
- Préparer les réactifs.
- Pour la quantité de sol utilisée, ajouter des produits chimiques dans un rapport volumique 0,8: 1: 2 pour le tampon phosphate (tampon P): CHCl 3 : MeOH.
- Préparer une solution de tampon P: Phosphate-tampon: 0,1 M, pH 7,0.
- Ajouter 61 mL de stock de 1M K 2 HPO 4 , stérile (le produit chimique doit être certifié ACS ou mieux). Ajouter 39 ml de stock de 1 M KH 2 PO 4 , stérile (le produit chimique doit être certifié ACS ou mieux). Remplir à 1000 mL avec de l'eau de type 1. Ajuster le pH à 7,0 avec NaOH ou HCl. Conservez la solution inutilisée jusqu'à 7 jours à température ambiante ou 30 jours au réfrigérateur.
- Alternativement, peser 10,62 g de K 2 HPO 4 et 5,31 g de KH 2 PO 4 ; Diluer à 1 L avec de l'eau de type 1. Vérifier le pH, régler si nécessaire.
- Préparer le réactif 1, Réactif de saponification.
- Distribuer 300 ml d'eau de type 1.Ajouter 300 ml de CH 3 OH (classe HPLC ou supérieure). Ajouter 90,00 g de NaOH (certifié ACS ou mieux). Ajouter des pastilles de NaOH à la solution tout en agitant. Remuer jusqu'à dissolution des pastilles. Il est recommandé de stocker cette solution ne dépassant pas 30 d.
- Préparer le réactif 2, réactif de méthylation.
- Distribuer 275 ml de CH 3 OH (HPLC grade ou plus haut). Ajouter 325 mL de marque certifiée 6.00 N HCL en remuant. Il est recommandé de stocker cette solution ne dépassant pas 30 d.
- Préparer le réactif 3, Réactif d'extraction: 50% C 6 H 14 (Hexane), 50% C 5 H 12 O (MTBE).
- Dans la hotte aspirante, mélanger 200 mL de C 6 H 14 (HPLC grade ou plus haut) et 200 mL de C 5 H 12 O (HPLC ou plus). Bien mélanger; Capuchons fermement. Entreposer dans une armoire ou une hotte inflammable pour un maximum de 1 an.
- Préparer le réactif 4, réactif de lavage de base.
- Dispense 900Ml d'eau de type 1 au bécher. Ajouter 10,80 g de NaOH (Certified ACS ou better) en remuant. Remuer jusqu'à dissolution des pastilles. Il est recommandé de stocker cette solution ne dépassant pas 30 d.
- Préparer une solution stock standard interne.
- Peser 100 mg de norme EEE 19: 0. Ajouter à 50 ml de flacon volumétrique rincé à l'hexane. Ajouter ~ 15 mL de solution hexane-MTBE (réactif 3) dans un flacon, tourbillonner pour dissoudre. Compléter à 50 mL avec une solution hexane-MTBE (réactif 3). Capuchon et tourbillon pour mélanger. Transférer dans des tubes en verre rincé à l'hexane avec des bouchons doublés en PTFE et entreposer à -20 ° C.
2. JOUR 1 - Extraction des acides gras du sol (4 à 5 h pour ~ 40 échantillons)
- Préparez trois pipettes de distribution de 5 à 10 ml pour le tampon P, le chloroforme et le méthanol.
NOTE: Des alternatives moins dangereuses au chloroforme ont été proposées comme agents d'extraction. Cela peut entraîner des taux équivalents de récupération des lipides 16 , 17 , mais ne sont pas évalués dans ce protocole. - Peser les sols lyophilisés dans des tubes de centrifugation de 30 ml et enregistrer la masse du sol.
- Faites deux flans et incluez un standard de contrôle (un sol qui a déjà été extrait).
- Dans la hotte aspirante, ajouter les réactifs au sol dans le tube centrifuge dans l'ordre suivant: tampon P, CHCl 3 et MeOH (Tableau 1). Permettre aux sols de mouiller après l'addition du tampon P avant d'ajouter le CHCl 3 . Enlevez fermement les tubes de centrifugeuse et couvrez-les pour protéger contre la lumière.
- Placez-les sur le shaker horizontalement en vous assurant qu'ils sont bien sécurisés. Avec le réglage de la vitesse sur 280 tr / min, secouer pendant 1 h 18 .
- Préparez deux tubes en verre de 16 mm x 150 mm pour chaque échantillon comme suit: Étiqueter le tube et ajouter le même volume de CHCl 3 et un volume égal de tampon P.
- Retirer les tubes centrifuges du agitateur et centrifuger pendant 10 min à 1 430Xg et 25 ° C. La séparation des phases doit être visible dans le tube de verre.
- Dans la hotte aspirante, décanter le surnageant du tube centrifuge dans l'un des tubes préparés à l'étape 2.6. Répétez les étapes 2.4 à 2.5 et décantez le surnageant dans le second tube.
- Fixez fermement tous les tubes en verre de 16 mm x 150 mm avec des doublures doublées de PTFE et inversez 10 x pour mélanger.
- Permettre aux échantillons de rester intacts pendant la nuit pour séparer complètement les deux phases. Pour ce faire, conservez les échantillons dans une armoire ou un espace noir ou recouvert de papier d'aluminium à température ambiante. Il est acceptable de permettre aux extraits de se séparer pendant le week-end.
- Alternativement, si l'on souhaite passer directement à l'étape suivante, ou si les échantillons sont dérangés le lendemain, centrifuger les échantillons pendant 10 min à 1000 xg et 25 ° C.
REMARQUE: Suivez tous les échantillons dans un groupe de comparaison au même temps de repos de séparation de phase.
- Alternativement, si l'on souhaite passer directement à l'étape suivante, ou si les échantillons sont dérangés le lendemain, centrifuger les échantillons pendant 10 min à 1000 xg et 25 ° C.
3 JOURS2 - Isolation des lipides (3 à 4 h pour ~ 40 échantillons)
- Allumez l'appareil d'évaporation et le piège à solvants. Réglez l'appareil d'évaporation à 33 ° C et préchauffez.
- Mettre en place un aspirateur aspirant dans le capot: un ballon latéral relié à une pompe à vide, une longueur de tube de haute pureté et une pipette en verre.
- Aspirez la couche supérieure et l'interface (environ 2/3 de la descente) des deux tubes en verre, en conservant la couche aqueuse. Répétez le processus en utilisant une pipette propre pour chaque échantillon.
- Mélanger les couches aqueuses d'extrait du second tube avec celle du premier tube en décantant soigneusement. Assurez-vous que le tube à décanter est exempt de rayures ou de fissures.
- Une fois que les extraits de CHCl 3 sont combinés, inspecter le liquide - il devrait être clair. Sinon, ajouter MeOH jusqu'à ce qu'il soit dégagé.
- Sécher tous les échantillons en utilisant l'appareil d'évaporation sous vide. Commencez par des réglages de température 33 ° C, vitesse de vortex 26% et pression de 400 mbar.
- PDentelez les échantillons dans le système d'évaporation et, après 10 min, abaissez lentement la pression à 350 mbar.
- Surveillez la progression et, lorsque le niveau de liquide dans le tube atteint le niveau du bloc chauffant, diminuez la pression à 300 mbar.
- Continuez à surveiller la hauteur du liquide restant et quand il mesure environ 1 cm de profondeur, diminuez la pression à 200 mbar.
- Après avoir séché les échantillons, retirer les échantillons et faire fonctionner la pompe pendant 10 minutes supplémentaires pour éliminer les vapeurs.
- Captez les tubes de manière étanche et rangez les lipides dans un congélateur à -80 ° C.
- Éliminer le liquide aspiré conformément aux règles de sécurité chimique applicables.
4. JOUR 3 - Saponification et méthylation (6 à 7 h pour ~ 40 échantillons)
- Allumez les bains d'eau. Mettre le bain 1 à 95 ° C et le bain 2 à 80 ° C.
- Réglez les distributeurs de pipettes et assurez-vous qu'ils distribuent le bon volume.
Réactif 1: 1 mL par échantillon
Réactif 2: 2 mL par échantillon
Réactif 3: 1,25 mL par échantillon
Réactif 4: 3 mL par échantillon
REMARQUE: Le processus de chauffage entraînera une pression dans le tube. N'utilisez pas de tubes rayés, craqués ou ébréchés. - À l'aide d'un distributeur de pipettes, ajouter 1,0 mL de réactif 1 aux lipides séchés. Capot hermétiquement, vortex pendant 5 s et placez-le dans un rack.
- Placez le rack de tubes à échantillon dans le bain-marie à 95 ° C pendant 5 minutes. Retirez le rack de tubes du bain et vérifiez les fuites dans les tubes, indiqués par des bulles montant dans le tube. Resserrez ou remplacez les capuchons des tubes qui fuient, vérifiez à nouveau les fissures ou les copeaux. Continuer à chauffer les tubes dans le bain d'eau pendant 10 minutes supplémentaires.
- Retirez les échantillons du bain 1 et placez-les dans le bain 2 (80 ° C) et continuez l'incubation pendant 15 minutes supplémentaires.
- Retirez les tubes et refroidissez les échantillons en plaçant le rack dans une casserole d'eau du robinet pour le refroidir.
- Ajouter 2 ml de réactif 2 à chaque échantillon. Capuchons fermement et vortex pour 5 à 10 s.
- Placez la crémaillère dans le bain-marie à 80 ° C et incubez pendant 10 min.
- Retirez le rack de tubes du bain-marie et placez-le dans une casserole d'eau du robinet pour le refroidir. Agiter le rack de tubes pour accélérer le processus de refroidissement.
REMARQUE: Ne pas dépasser le temps et la température. Trop de chauffage pourrait dégrader les FAME. - En utilisant le distributeur de pipettes, ajouter 1,25 ml de réactif 3 à chaque tube d'échantillon pour extraire les esters méthyliques d'acides gras. Capuchon hermétiquement et mettre les tubes sur le shaker pendant 10 min.
- Après avoir secoué, permettez que le rack de tubes soit assis pendant 10 minutes pour que les phases se séparent. Transférer la phase organique (couche supérieure) vers un tube à essai en verre de 16 mm x 100 mm à l'aide d'une pipette en verre.
- Répétez l'extraction en ajoutant à nouveau le Réactif 3, secouant, en séparant les phases et en transférant la phase supérieure.
REMARQUE Ne transférez aucune des phases inférieures. Il est bon de laisser une petite quantité de phase supérieure dans le tube. - À l'aide du distributeur de pipettes, ajouter 3 ml de réactif 4 à la thE tubes de 16 mm x 100 mm.
- Capter les tubes à essai de manière étanche et tourbillonner pendant 20 à 30 s.
- Après vortex, centrifuger pendant 3 min à 1000 × g.
- À l'aide d'une pipette en verre propre, aspirer la phase organique supérieure et transférer dans un flacon d'ambre de 4 ml.
- Allumez l'appareil d'évaporation et le piège à solvants. Réglez l'appareil d'évaporation à 30 ° C, la vitesse du vortex à 26%, le vide à 200 mbar, et appuyez sur le préchauffage.
- Surveillez les progrès réalisés. Après avoir séché les échantillons, retirer les échantillons et faire fonctionner la pompe pendant 10 minutes supplémentaires pour éliminer les vapeurs.
- Retirer la pipette des bouteilles de réactif 1 et 4 et pomper à travers de l'acide dilué ( par exemple, 1% de HCl), puis l'eau de DI. Rincer la pipette utilisée pour le réactif 2 en pompant à travers de l'eau DI. Dans le capot, égoutter le solvant de la pipette utilisée pour le réactif 3 dans un récipient approprié et le maintenir dans le capot jusqu'à évaporation des résidus du solvant.
- Conservez les pipettes inversées, les pistons enlevés pour éviter de collerG des clapets anti-retour.
- Utilisez des pipettes en verre une fois, puis jetez-les dans un récipient approprié.
- Dans le capot, laisser les résidus de solvants sur la verrerie s'évaporer.
- Rincer la verrerie avec de l'eau propre et une solution de détergent.
5. JOUR 4 - Préparation de la solution de travail et transfert des FAME aux flacons GC (2-3 h pour ~ 40 échantillons)
- Rassembler les matériaux: 4 mL de flacons ambrés avec des échantillons séchés; Ampoules à ampoules de 2 ml GC; Plaques et embouts en verre à fond plat de 400 μL; Seringue de verre de 500 μL; Flacon volumétrique de 50 ml; Solution de stock - Nonadécanoate d'éthyle (norme interne 19: 0 EE) dans 50/50 hexane / MTBE (Réactif 3).
- À l'aide de la seringue en verre, ajouter 500 μL de solution mère à l'éthyl nonadécanoate (19: 0 EE) à une fiole à vapeur de 50 ml.
- Remplir le ballon au score volumétrique avec le réactif 3.
- Passez le ballon et inversez 5x pour le mélanger.
- Transférer 3 mL dans un flacon propre de 4 mL pour un réservoir de solution de travail.
- En utilisantE une seringue en verre, ajouter 300 μL de la solution de travail à chacun des flacons de 4 mL contenant les esters méthyliques et le capuchon d'acides gras séchés.
- Vortez l'échantillon pendant 15 s et placez-le pour 15 minutes.
- À l'aide d'une pipette Pasteur de verre, transférer soigneusement les esters méthyliques d'acides gras en suspension dans un flacon GC de 2 ml contenant l'insert de 400 μl.
- Conserver les flacons GC scellés dans un congélateur de -20 ° C avant l'analyse.
- Soumettre des échantillons pour l'analyse GC.
REMARQUE: l'analyse doit être effectuée à l'aide d'une colonne GC spécifique et des conditions décrites dans le Fichier supplémentaire S1 . Il est préférable que l'analyse GC soit complétée dans les 2 semaines suivant la méthylation.
Résultats
Les tableaux de données des rapports peuvent être regroupés dans une feuille de calcul ou une base de données. Après ajustement pour le facteur de réponse (un facteur de correction qui normalise la réponse pour différentes longueurs de chaîne), les zones de pointe peuvent être comparées avec la zone de pointe des normes externes ou internes pour arriver à une concentration dans l'extrait. En se divisant par la masse de sol extraite, les données peuvent être exprimées en masse de FAME par gramme de sol ou, en utilisant le poids moléculaire de chaque FAME, le nmol le plus communément rapporté par gramme de sol. La somme des FAME microbiens est révélatrice de la biomasse microbienne totale et peut être comparée entre les traitements ( figure 1 ). Certains FAME peuvent également être associés à des groupes microbiens particuliers tels que des bactéries Gram-positives ou Gram-négatives, des actinomycètes, des champignons mycorhiziens arbusculaires et des champignons saprotrophiques 19 , 20 , 21 , 22 , 23 , 24 . Les ratios de la masse des biomarqueurs spécifiques peuvent refléter l'abondance relative de ces groupes ( figure 2 ). Les modèles d'abondance globale de FAME créent des empreintes au niveau communautaire, qui permettent de comparer la dissemblance de la communauté microbienne avec des techniques multivariées telles que l'ordination ( figure 3 ). Contrairement à la plupart des approches basées sur l'ADN, les données lipidiques au niveau communautaire peuvent être analysées soit en abondance absolue soit en abondance absolue. Si la biomasse totale diffère sensiblement des échantillons, ces deux approches donneront des résultats très différents; Les questions écologiques sous-jacentes à l'expérience devraient déterminer quelle approche est utilisée.
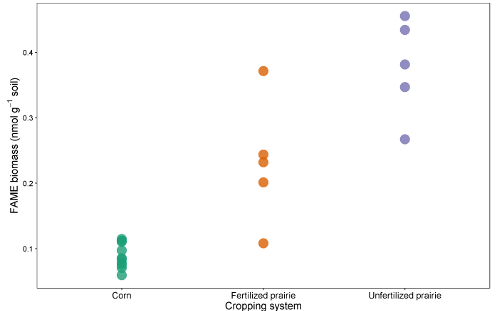
F Figure 1 : biomasse totale de FAME. Comparaison de la biomasse totale du biomarqueur FAME (sol nmol g -1 ) du maïs continu, de la prairie fécondée et de la prairie non fertilisée. Cliquez ici pour voir une version plus grande de ce chiffre.
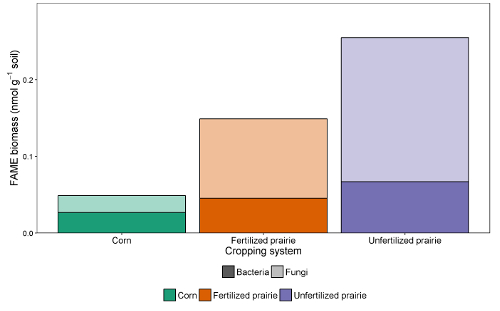
Figure 2 : biomasse FAME de bactéries et de champignons. Comparaison du traitement de la biomasse bactérienne et fongique du biomarqueur FAME (sol nmol g -1 ) du maïs continu, de la prairie fécondée et de la prairie non fertilisée. Masse fétide et bactérienne de l'abondance absolue. Moyenne (somme f / somme b). Cliquez ici pour voir une version plus grande de ce chiffre.
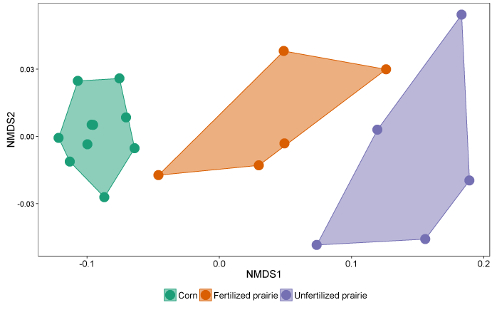
Figure 3 : Mise à l'échelle dimensionnelle non métrique des biomarqueurs lipidiques FAME. Comparaison de la communauté microbienne globale en utilisant des profils d'abondance absolue de tous les biomarqueurs lipidiques microfiens FAME. Dans cet exemple, les cultures continuelles de maïs et de prairies non fécondées se séparent et sont très éloignées tandis que certains échantillons de prairies fécondées ont des communautés microbiennes qui ressemblent à celles du maïs, et d'autres ressemblent à la prairie non fertilisée. Cliquez ici pour voir une version plus grande de ce chiffre.
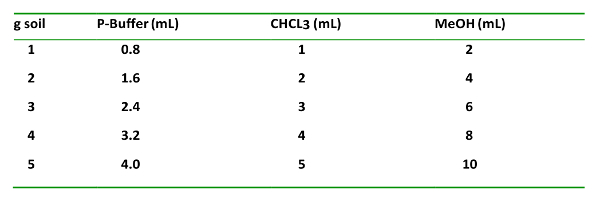
Tableau 1: T h e afinl ven type t, propor ti on s a ajouté g -1 sol, un d t h eir orde r o f o n iti ajouter. Ceci est important pour une extraction et une séparation appropriées des phases organique et aqueuse.
Discussion
Pour l'examen d'échantillons multiples provenant d'expériences avec de nombreuses unités réelles et / ou expérimentales, les chercheurs peuvent considérer l'analyse des acides gras phospholipides (PLFA) comme prohibitive en termes de temps et de matériaux 25 . Avec la méthode PLFA, les phospholipides de la membrane cellulaire sont extraits, purifiés et identifiés en utilisant l'extraction Bio-organique bifilaire Bligh et Dyer 26 modifiée. Ceci est suivi d'une Chromatographie de silice en phase solide pour séparer les lipides par polarité et une méthylation alcaline d'acides gras phospholipides en esters méthyliques d'acides gras. Dans le profilage PLFA, le rendement lipidique peut être faible mais d'une pureté très élevée. L'identification microbienne a introduit une autre méthode, la procédure d'ester méthylique d'acide gras (MIDI-FA). Dans la méthode MIDI-FA, tous les lipides sont extraits directement de cultures pures ou échantillons de sol / sédiments 11 , 12 , 27 à traversSaponification. Cette méthode a une perte de lipide inférieure et est rapide car elle n'a aucune des étapes de concentration ou de purification de la méthode PLFA. Cependant, alors que la méthode MIDI-FA est rapide et moins coûteuse, car elle a été conçue à l'origine pour identifier des organismes en culture pure, il n'y a pas d'étapes initiales d'extraction ou de purification. Ainsi, il peut inclure des composés semblables à des lipides co-extraits de la matière organique du sol qui déforment la signature de la communauté 27 , 28 , 29 . Parce que cette inclusion peut également fausser les mesures de la biomasse, MIDI-FA a généralement été utilisé uniquement pour décrire qualitativement les lipides du sol 13 .
La procédure que nous décrivons ici combine la meilleure des deux procédures d'extraction séparées: 1) une extraction et une concentration de lipides en utilisant la méthode de Bligh et Dyer 26 modifiée, et 2) l'ester de méthyle d'acide gras saPonification, méthylation, extraction et procédé de lavage de base développé commercialement. Cette méthode a été développée pour atteindre les avantages de ces deux protocoles tout en minimisant les inconvénients 15 . En effectuant l'extraction initiale et en isolant les composants organiques solubles ( p. Ex. Lipides) avant d'effectuer MIDI-FA, et en complétant cette étape de purification, ce protocole offre un équilibre entre la vitesse et la précision. Bien que cette méthode ne soit peut-être pas appropriée lorsqu'une pureté élevée est requise ( c.-à - d . Pour l'analyse PLFA 13 C) ou lors de l'analyse séparée des phospholipides et des lipides neutres, cela permet souvent de détecter les réponses de la communauté microbienne aux conditions environnementales avec une plus grande sensibilité que l'ADN Méthodes 30 , 31 , 32 , 33 . Les lipides membranaires se décomposent rapidement après la mort de la cellule, ce qui leur permet deReflètent la communauté microbienne vivante au moment de l'échantillonnage 5 , 7 , contrairement à l'ADN environnemental dans lequel une grande partie de l'information provient d'organismes morts ou inactifs 34 . Compte tenu du taux élevé de dormance observé chez les microorganismes du sol 35 , la caractérisation de la biomasse vivante peut être utilisée pour comprendre les interactions temporelles entre les plantes et les microbes à une échelle temporelle relativement fine et les biomarqueurs lipidiques peuvent être utilisés pour évaluer le statut physiologique de la communauté microbienne 7 . Il a été démontré que des méthodes à haut débit sont nécessaires pour évaluer la réponse microbienne dans les grands paramètres de terrain 25 et, bien que la méthode que nous proposons ici ne réplique pas la précision du profilage biomarker PLFA, elle augmente le débit tout en minimisant la variabilité réalisée avec le MIDI-FA procédure. La méthode s'est avérée être un outil efficace pour traiterQuestions relatives à la dynamique de la communauté microbienne sur un large éventail de sols dans des études agricoles et écosystémiques à grande échelle 36 , 37 , 38 , 42 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 .
Les classes de lipides sont combinées avec cette méthode et il peut y avoir une perte de l'information contenue dans ces classes séparées 22 , 39 , mais la combinaison des classes de lipides peut renforcer la puissance pour détecter l'origine des champignons mycorhizques arbusculaires des 16: 1 ω5c des deux phospho - et lipides neutres 40 . De plus, alors que tLe nombre d'acides gras inconnus (qui pourrait être dérivé de la matière organique non vivante) peut être plus élevé avec cette méthode, il a été démontré être inférieur à MIDI-FA et permet une comparaison du traitement des profils lipidiques des études avec de nombreux échantillons où l'échantillon La capacité de production est une préoccupation 15 . On pense généralement que la fraction neutre provient principalement des lipides de stockage produits par les champignons, bien qu'il puisse y avoir une plus petite contribution de la faune du sol 41 . À la lumière de cela, la méthode décrite ici peut donner des résultats montrant une plus grande contribution des lipides fongiques 18: 2 ω 6,9c et 18: 1 ω 9c que le PLFA. Les autres lipides qui ont tendance à apparaître dans la fraction neutre comprennent certains des acides gras saturés, par exemple 16: 0, 18: 0, 20: 0.
Il existe différentes façons d'exprimer et d'analyser les données lipidiques. Les représentations les plus courantes sont l'abondance (nmol g -1 sol), mole fractioN (nmol lipide individuel nmol -1 lipide total) et pourcentage en moles (fraction molaire * 100). Normalisé par les lipides totaux dans un échantillon, la fraction molaire et le pourcentage molaire sont des mesures de l'abondance relative d'un lipide donné. Après une transformation appropriée, par exemple , la racine carrée arcsine, la fraction molaire est appropriée pour une analyse par composantes principales ou une ordonnance d'analyse de redondance. L'abondance est la quantité absolue d'un lipide donné extrait par gramme de sol. Parce que la quantité de lipide par cellule est raisonnablement constante et que l'extraction des lipides est très efficace et complète, l'abondance totale est une bonne estimation des lipides totaux et l'abondance des indicateurs clés reflète la biomasse du groupe écologique qu'il représente 17 . Enfin, une bonne façon d'examiner la composition de la communauté microbienne est d'utiliser des méthodes d'analyse multivariée 16 , par exemple , des méthodes d'ordination telles que la mise à l'échelle multidimensionnelle non métrique (NMDS -Qui n'a pas besoin de transformation de données) ou l'analyse des composants principaux (PCA), peut être utile pour comparer l'abondance relative de tous les biomarqueurs lipidiques.
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Ce travail a été financé en partie par le Centre de recherche sur la bioénergie de Great Lakes (DOE BER Office of Science DE-FC02-07ER64494) et le bureau de l'efficacité énergétique et des énergies renouvelables de DOE OBP (DE-AC05-76RL01830). Les auteurs souhaitent remercier Anders Gurda pour sa contribution patiente et habile à la vidéographie et à l'édition.
matériels
| Name | Company | Catalog Number | Comments |
| RapidVap | Labconco | 7900000 | Evaporative system |
| RapidVap | Labconco | 7491400 | Sample block |
| Chem-resistant vacuum pump | Labconco | 7584000 | Vacuum pump |
| Chemical trap cannister | Labconco | 7815300 | Trap cannister |
| Solvent insert | Labconco | 7515200 | Solvent trap insert |
| Diaphragm pump | Welch | 7398000 | DryFast vacuum pump |
| Water bath | Fisher | 15-462-15Q | 2 well water bath |
| Gas chromatograph | Various | N/A | For sample analysis |
| Centrifuge | Various | N/A | For sample separation |
| Freeze Dryer | Various | N/A | For sample lyophilization |
| Repipet (6) | BrandTech | 4720440 | For dispensing reagents |
| Vortex | Fisher | 02-215-365 | Analog vortex mixer |
| Teflon centrifuge tubes | Thermo/Nalgene | 3114-0030 | Teflon sample tubes |
| Caps | Thermo/Nalgene | DS3131-0020 | Caps for teflon tubes |
| Test tube | Corning | 9825-16 | 16x100mm tubes |
| Test tube | Corning | 9825-16xx | 16x150mm tubes |
| Caps | Corning | 9998-15 | 15-415 thread black phenolic caps w/PTFE liner |
| Pasteur pipets | Fisher | 13-678-20B | 14.6cm |
| 500 uL glass syringe | Fisher | 13684106LC | Hamilton 81217 |
| Amber vials | Agilent | 5182-0716 | 4mL Amber vials |
| Caps | Agilent | 5182-0717 | Blue screw caps |
| Inserts | Agilent | 5181-3377 | 400uL flat bottom glass inserts |
| Standards | |||
| 19:0 ethyl nonadecanoate (Ethyl nonadecanoate) | VWR | TCN0459-5G | Analytical standard |
| Chemicals | |||
| Dipotassium phosphate (K2HPO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Potassium phosphate monobasic (KH2PO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Methanol (CH3OH - HPLC grade or better) | Various | N/A | For making Reagents 1 & 2 |
| Sodium hydroxide (NaOH - ACS grade or better) | Various | N/A | For making Reagents 1 & 4 |
| Hydrochloric acid (6N HCL) | Various | N/A | For making Reagent 2 |
| Hexane (HPLC grade or better) | Various | N/A | For making Reagent 3 |
| MTBE (Methyl tert-butyl ether - HPLC grade or better) | Various | N/A | For making Reagent 3 |
Références
- Van Der Heijden, M. G. a, Bardgett, R. D., Van Straalen, N. M. The unseen majority: Soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecol Lett. 11 (3), 296-310 (2008).
- Bever, J. D., et al. Rooting theories of plant community ecology in microbial interactions. Trends Ecol Evol. 25 (8), 468-478 (2010).
- Suleiman, A. K. A., Manoeli, L., Boldo, J. T., Pereira, M. G., Roesch, L. F. W. Shifts in soil bacterial community after eight years of land-use change. Syst Appl Microbiol. 36 (2), 137-144 (2013).
- Sayer, E. J., et al. Grassland management influences spatial patterns of soil microbial communities. Soil Biol Biochem. 61, 61-68 (2013).
- Fernandes, M. F., Saxena, J., Dick, R. P. Comparison of Whole-Cell Fatty Acid (MIDI) or Phospholipid Fatty Acid (PLFA) Extractants as Biomarkers to Profile Soil Microbial Communities. Microbial Ecol. 66 (1), 145-157 (2013).
- van der Heijden, M. G. A., Wagg, C. Soil microbial diversity and agro-ecosystem functioning. Plant Soil. 363 (1-2), 1-5 (2013).
- Allison, V. J., Miller, R. M., Jastrow, J. D., Matamala, R., Zak, D. R. Changes in soil microbial community structure in a tallgrass prairie chronosequence. Soil Sci Soc Am J. 69 (5), 1412-1421 (2005).
- Kowalchuk, G. A., Buma, D. S., de Boer, W., Klinkhamer, P. G. L., van Veen, J. A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. A van Leeuw. 81 (1-4), 509-520 (2002).
- Bardgett, R. D., Hobbs, P. J., Frostegard, A. Changes in soil fungal:bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biol Fert Soils. 22 (3), 261-264 (1996).
- Frostegård, Å, Tunlid, A., Bååth, E. Use and misuse of PLFA measurements in soils. Soil Biol Biochem. 43 (8), 1621-1625 (2011).
- Haack, S. K., Garchow, I. H., Odelson, D. A., Forney, L. J., Klug, M. J. Accuracy , Reproducibility , and Interpretation of Fatty Acid Methyl Ester Profiles of Model Bacterial Communitiest. Appl Environ Microbiol. 60 (7), 2483-2493 (1994).
- Drenovsky, R. E., Elliott, G. N., Graham, K. J., Scow, K. M. Comparison of phospholipid fatty acid ( PLFA ) and total soil fatty acid methyl esters ( TSFAME ) for characterizing soil microbial communities. Soil Biol Biochem. 36, 1793-1800 (2004).
- Cavigelli, M. A., Robertson, G. P., Klug, M. Fatty acid methyl ester ( FAME ) profiles as measures of soil microbial community structure. Plant Soil. 170, 99-113 (1995).
- Findlay, R. Determination of microbial community structure using phospholipid fatty acid profiles. Molecular microbial ecology manual. , Available from: https://www.researchgate.net/profile/Robert_Findlay2/publication/281208148_Determination_of_microbial_community_structure_using_phospholipid_fatty_acid_profiles/links/55db4f0b08aec156b9afe776.pdf 2885-2906 (2004).
- Balser, T. C., Liang, C., Gutknecht, J. L. Linking microbial community analysis and ecosystem studies: A rapid lipd analysis protocol for high throughput. Biol Fert Soils. , Forthcoming (2017).
- Findlay, R. H., King, G. M., Watling, L., Watling, L. E. S. Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sediments Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sedimentst. Appl Environ Microbiol. 55 (11), 2888-2893 (1989).
- Willers, C., Jansen van Rensburg, P. J., Claassens, S. Microbial signature lipid biomarker analysis - an approach that is still preferred, even amid various method modifications. J Appl Microbiol. 118 (6), 1251-1263 (2015).
- Balser, T. C. Phospholipid Fatty-acid Analysis (PLFA). Protocol. , Available from: http://nature.berkeley.edu/soilmicro/methods/BalserPLFA.pdf (2017).
- Zelles, L., Bai, Q. Y., Beck, T., Beese, F. Signature fatty-acids in phospholipids and lipopolysachharides as indicators of microbial biomass and community structure in agricultural soils. Soil Biol Biochem. 24 (4), 317-323 (1992).
- Frostegard, A., Tunlid, A., Baath, E. Phospholipid fatty-acid composition, biomass, and activity of microbial communities from 2 soil types experimentally exposed to different heavy-metals. Appl Environ Microbiol. 59 (11), 3605-3617 (1993).
- Federle, T. W., Dobbins, D. C., Thorntonmanning, J. R., Jones, D. D. Microbial biomass, activity, and community structure in subsurface soils. Ground Water. 24 (3), 365-374 (1986).
- Vestal, J. R., White, D. C. Lipid analysis in microbial ecology - quantitative approaches to the study of microbial communities. BioScience. 39 (8), 535-541 (1989).
- Wilkinson, S. G. Gram negative bacteria. Microbial Lipids. , 299-488 (1988).
- Balser, T. C., Treseder, K., Ekenler, M. Using lipid analysis and hyphal length to quantify AM and saprotrophic fungal abundance along a soil chronosequence. Soil Biol Biochem. 37 (3), 601-604 (2005).
- Buyer, J. S., Sasser, M. High throughput phospholipid fatty acid analysis of soils. Appl Soil Ecol. 61, 127-130 (2012).
- Bligh, E. G., Dyer, W. J. A rapid method of total lipid extraction and purification. Can J Biochem Phys. (37), 911-917 (1959).
- Schutter, M. E., Dick, R. P. Comparison of Fatty Acid Methyl Ester (FAME) Methods for Characterizing Microbial Communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Jandl, G., Leinweber, P., Schulten, H., Ekschmitt, K. Contribution of primary organic matter to the fatty acid pool in agricultural soils. Soil Biol Biochem. 37, 1033-1041 (2005).
- Nielsen, P., Petersen, S. O. Ester-linked polar lipid fatty acid profiles of soil microbial communities: a comparison of extraction methods and evaluation of interference from humic acids. Soil Biol Biochem. 32, 1241-1249 (2000).
- Duncan, D. S., Jewell, K. A., Suen, G., Jackson, R. D. Detection of short-term cropping system-induced changes to soil bacterial communities differs among four molecular characterization methods. Soil Biol Biochem. 96, 160-168 (2016).
- Liang, C., et al. Switchgrass rhizospheres stimulate microbial biomass but deplete microbial necromass in agricultural soils of the upper Midwest, USA. Soil Biol Biochem. 94, 173-180 (2016).
- Jangid, K., et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties. Soil Biol Biochem. 43 (10), 2184-2193 (2011).
- Ritz, K., et al. Spatial structure in soil chemical and microbiological properties in an upland grassland. FEMS Microbiol Ecol. 49 (2), 191-205 (2004).
- Carini, P., et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. J Chem Inf Model. 53 (9), 1689-1699 (2013).
- Lennon, J. T., Jones, S. E. Microbial seed banks: The ecological and evolutionary implications of dormancy. Nat Rev Microbiol. 9 (2), 119-130 (2011).
- Gutknecht, J. L. M., Field, C. B., Balser, T. C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years. Glob Change Biol. 18, 225-269 (2012).
- Pei, Z., et al. Soil and tree species traits both shape soil microbial communities during early growth of Chinese subtropical forests. Soil Biol Biochem. 96, 180-190 (2016).
- Oates, L. G., Duncan, D. S., Sanford, G. R., Liang, C., Jackson, R. D. Bioenergy cropping systems that incorporate native grasses stimulate growth of plant-associated soil microbes in the absence of nitrogen fertilization. Ag Ecosys Environ. 233, 396-403 (2016).
- Bååth, E. The use of neutral lipid fatty acids to indicate the physiological conditions of soil fungi. Microbial Ecol. 45 (4), 373-383 (2003).
- Ngosong, C., Gabriel, E., Ruess, L. Use of the signature Fatty Acid 16:1ω5 as a tool to determine the distribution of arbuscular mycorrhizal fungi in soil. J Lipids. 2012, 236807(2012).
- Sharma, M. P., Buyer, J. S. Comparison of biochemical and microscopic methods for quantification of arbuscular mycorrhizal fungi in soil and roots. Appl Soil Ecol. 95, 86-89 (2015).
- Oates, L. G., Balser, T. C., Jackson, R. D. Subhumid pasture soil microbial communities affected by presence of grazing, but not grazing management. Appl Soil Ecol. 59, 20-28 (2012).
- Liang, C. Potential legacy effects of biofuel cropping systems on soil microbial communities in southern Wisconsin, USA. Ag Sci. 2 (2), 131-137 (2011).
- Herzberger, A. J., Duncan, D. S., Jackson, R. D. Bouncing Back Plant-Associated Soil Microbes Respond Rapidly to Prairie Establishment. PloS One. 9 (12), 1-14 (2014).
- Fraterrigo, J. M., Balser, T. C., Turner, M. G. Microbial community variation and its relationship with nitrogen mineralization in historically altered forests. Ecology. 87 (3), 570-579 (2006).
- Kao-Kniffin, J., Balser, T. C. Elevated CO2 differentially alters belowground plant and soil microbial community structure in reed canary grass-invaded experimental wetlands. Soil Biol Biochem. 39 (2), 517-525 (2007).
- Mentzer, J. L., Goodman, R. M., Balser, T. C. Microbial response over time to hydrologic and fertilization treatments in a simulated wet prairie. Plant Soil. 284 (1-2), 85-100 (2006).
- Ushio, M., Wagai, R., Balser, T. C., Kitayama, K. Variations in the soil microbial community composition of a tropical montane forest ecosystem: Does tree species matter. Soil Biol Biochem. 40 (10), 2699-2702 (2008).
- Bartelt-Ryser, J., Joshi, J., Schmid, B., Brandl, H., Balser, T. Soil feedbacks of plant diversity on soil microbial communities and subsequent plant growth. Perspect Plant Ecol. 7 (1), 27-49 (2005).
- Fichtner, A., von Oheimb, G., Härdtle, W., Wilken, C., Gutknecht, J. L. M. Effects of anthropogenic disturbances on soil microbial communities in oak forests persist for more than 100 years. Soil Biol Biochem. 70, 79-87 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon