Method Article
Un metodo di estrazione e analisi lipidica per la caratterizzazione dei microbi del suolo negli esperimenti con molti campioni
In questo articolo
Riepilogo
L'articolo descrive un metodo che aumenta il throughput, mentre equilibra lo sforzo e la precisione per l'estrazione dei lipidi dalle membrane cellulari di microrganismi per l'uso nella caratterizzazione di entrambi i lipidi totali e l'abbondanza relativa dei lipidi indicatori per determinare la struttura della comunità microbica del suolo negli studi con molti campioni.
Abstract
Le comunità microbiche sono importanti driver e regolatori dei processi ecosistemici. Per capire come la gestione degli ecosistemi possa influenzare le comunità microbiche, una tecnica relativamente precisa ma intensa per la valutazione della composizione microbica della comunità è l'analisi dell'acido grasso fosfolipidico (PLFA). PLFA è stato sviluppato per analizzare i biomarkers di fosfolipidi, che possono essere usati come indicatori della biomassa microbica e la composizione di ampie funzionalità di funghi e batteri. È stato comunemente utilizzato per confrontare i terreni in comunità vegetali alternative, ecologia e regimi di gestione. Il metodo PLFA è stato dimostrato sensibile alla rilevazione degli spostamenti nella composizione microbica della comunità.
Un metodo alternativo, l'estrazione e l'analisi del metil estere di acidi grassi (MIDI-FA) è stata sviluppata per l'estrazione rapida dei lipidi totali, senza separazione della frazione fosfolipidica, dalle culture pura come tecnica di identificazione microbica. Questo metodo èRapida ma meno adatta per i campioni di terreno perché manca di un passo iniziale che separa le particelle del terreno e inizia invece con una reazione di saponificazione che probabilmente produce manufatti della materia organica di fondo nel terreno.
Questo articolo descrive un metodo che aumenta il throughput, mentre equilibra lo sforzo e la precisione per l'estrazione dei lipidi dalle membrane cellulari di microrganismi per l'uso nella caratterizzazione di entrambi i lipidi totali e l'abbondanza relativa dei lipidi indicatori per determinare la struttura della comunità microbica del suolo negli studi con molti campioni. Il metodo combina l'accuratezza raggiunta attraverso la profilazione PLFA estraendo e concentrando i lipidi del suolo come primo passo e riducendo lo sforzo saponendo il materiale organico estratto e lavorando con il metodo MIDI-FA come un secondo passo.
Introduzione
Dato il ruolo fondamentale dei microrganismi nel ciclo nutrizionale 1 , la modifica della composizione della comunità vegetali 2 , la regolazione della produttività vegetale 3 e la decomposizione della materia organica 4 , la comprensione delle comunità microbiche del suolo è fondamentale per la comprensione degli ecosistemi terrestri.
A causa della loro abbondanza relativamente elevata nel suolo e della loro firma chimica, i biomarkers lipidici possono essere utilizzati per profilare i gruppi ecologici dominanti che comprendono le comunità microbiche del suolo 5 . Quantificando i biomarkers lipidici che sono caratteristici di diversi gruppi microbici, possiamo stimare i lipidi totali e quindi separare questi lipidi in gruppi ecologicamente rilevanti come i batteri Gram positivi (Gm +) e Gram negativi (Gm-), i micoresi arbuscolari (AM) e saprotrofici Funghi e actinomycetes 5 , Ss = "xref"> 6 , 7 , 8 .
Ci sono molti metodi per caratterizzare gli aspetti delle comunità microbiche. Il metodo PLFA è comunemente usato per comprendere la struttura comunitaria microbica di base. È un modo efficace per valutare l'abbondanza relativa dei gruppi microbici e la biomassa microbica totale. A causa del rapido turnover dei lipidi, la profilazione PLFA consente anche una rilevazione relativamente veloce dei cambiamenti nella comunità microbica del suolo e fornisce informazioni che consentono di confrontare la funzione dell'ecosistema, ad esempio, i rapporti batterici con funghi per valutare i tassi di ciclaggio dei nutrienti 1 , 9 , 10 . Tuttavia, mentre il metodo di estrazione PLFA è rispettato e rispettato, è anche un tempo che richiede tempo e non si presta a studi su scala ecosistemica che richiedono un gran numero di campioni provenienti da repliche di scala di campoF "> 11 , 12 .
Al contrario, il metodo di estrazione del metil estere dell'acido grasso (MIDI-FA) ha il potenziale per consentire un rapido throughput. In questo metodo i campioni vengono saponificati, convertiti in FAME, estratti e quindi analizzati. Il metodo MIDI-FA è rapido ma meno discriminante di PLFA, che combina l'estrazione dei lipidi con la separazione delle diverse classi lipidiche 13 (fosfolipidi, lipidi neutri e glicolipidi).
In questo protocollo, descriviamo un metodo che combina gli elementi di PLFA e di profilassi lipidico MIDI-FA. Utilizza l'estrazione dei lipidi usando le fasi iniziali di estrazione del cloroformio del metodo Bligh e Dyer modificato e quindi la saponificazione e la conversione a FAMEs. Questo fornisce un modo robusto per rilevare la struttura della comunità microbica escludendo gran parte del rumore di fondo da materiale non microbico 5 , 14 . Questo metodo è stato sviluppato per ottenere un equilibrio tra i protocolli PLFA e MIDI-FA, vale a dire mantenere la maggior parte della precisione, aumentando il throughput per renderlo logistico ed economicamente fattibile per analizzare i lipidi da studi su larga scala con molti campioni 15 . Eseguendo l'estrazione iniziale e isolando i componenti solubili in organico ( ad esempio i lipidi) prima di eseguire MIDI-FA e completandolo con una fase di purificazione, il protocollo offre un equilibrio tra velocità e precisione.
Protocollo
NOTA: indossare sempre adeguati dispositivi di protezione individuale (PPE) durante tutta la procedura. Per evitare possibili contaminazioni del campione, non toccare le vetrine con le mani nude. Indossare guanti appropriati quando eseguono passaggi di protocollo che richiedono la manipolazione di cloroformio.
1. Preparativi (2 giorni per ~ 40 campioni)
- Raccogliere il suolo in sacchetti sterili e trasportare dal campo in un contenitore contenente ghiaccio. Se non è possibile schiarire il terreno fresco e lasciarli congelare immediatamente, conservare i campioni in un congelatore da -80 ° C fino a quando non sarà pronto per iniziare l'analisi.
- Preparare il terreno mediante omogeneizzazione. Rimuovere le radici e le pietre e rompere i tondini da una setacciatura grossolana ( es. 2 mm).
- Preparare l'essiccazione congelante inserendo i sottoinsiemi in un contenitore appropriato come indicato nel manuale del congelatore e congelare asciutto il più presto possibile. Una volta che i suoli vengono congelati, conservare in un contenitore sigillato con essiccante fino all'estrazione. È meglio sTritare il suolo congelato a un minimo di -20 ° C ma preferibilmente a -80 ° C.
- In preparazione all'estrazione, rimuovere i terreni secchi di liofilizzazione dalla conservazione e macinare a una consistenza di farina. I metodi di macinazione includono il mulino a sfera, il battitore a sfere e il mortaio e il pestello. Dopo aver macinato il terreno, conservare in un congelatore (vedere 1.3).
NOTA: la quantità di campione utilizzata per l'estrazione dipende dal suo contenuto di materia organica. Un orientamento generale è quello di utilizzare 0,5-1 g di un terreno che è dal 12 al 18% in peso di carbonio e da 3 a 5 g per un terreno dal 1 al 3% in peso di carbonio. - Pre-risciacquare tubi centrifuga da 30 ml con esano. Aggiungere circa 2 a 3 ml di esano ai tubi e vortex per 5 sec. Decanta l'esano ad un altro tubo e vortice. L'esano (da 2 a 3 ml) può essere utilizzato per risciacquare seriamente sei tubi. Conservare i tubi esauriti esagonali invertiti nel cappuccio e smaltire l'esano utilizzato in un contenitore di rifiuti appropriato.
- Avvolgere le vetrerie in 2 o 3 strati di fogli di alluminio e collocare nella fornace a muffola. Infornare(Muffola) a 450 ° C per 4,5 ore.
- Preparare reagenti.
- Per la quantità di terreno impiegata, aggiungete sostanze chimiche in un rapporto di volume 0,8: 1: 2 per il tampone fosfato (P-buffer): CHCl 3 : MeOH.
- Preparare la soluzione P-tampone: Fosfato-Buffer: 0,1 M, pH 7,0.
- Aggiungere 61 ml di 1M K 2 HPO 4 , sterile (chimico deve essere certificato ACS o meglio). Aggiungere 39 ml di 1 M KH 2 PO 4 , sterile (chimico deve essere certificato ACS o meglio). Riempire a 1000 mL con acqua tipo 1. Regolare il pH a 7,0 con NaOH o HCl. Conservare la soluzione inutilizzata per un massimo di 7 giorni a temperatura ambiente o 30 giorni in frigorifero.
- In alternativa, pesare 10,62 g di K 2 HPO 4 e 5,31 g di KH 2 PO 4 ; Diluire a 1 L con acqua tipo 1. Controllare il pH, regolare se necessario.
- Preparare il reagente 1, il reagente di saponificazione.
- Dispensare 300 ml di acqua tipo 1.Aggiungere 300 ml di CH 3 OH (grado HPLC o superiore). Aggiungere 90,00 g di NaOH (ACS certificato o meglio). Aggiungere la pellicola di NaOH alla soluzione mescolando. Mescolare finché le pellet si dissolvono. Si raccomanda di memorizzare questa soluzione non più di 30 d.
- Preparare il reagente 2, il reagente per la metilazione.
- Dispensare 275 ml di CH 3 OH (grado HPLC o superiore). Aggiungere 325 ml di certificato 6,00 N HCL mescolando. Si raccomanda di memorizzare questa soluzione non più di 30 d.
- Preparare il reagente 3, il reagente di estrazione: 50% C 6 H 14 (esano), 50% C 5 H 12 O (MTBE).
- Nella cappa fumo, combinare 200 mL di C 6 H 14 (grado HPLC o superiore) e 200 mL di C 5 H 12 O (HPLC grado o superiore). Mescolare bene; Cappuccio strettamente. Conservare in armadio infiammabile o cappuccio per almeno 1 anno.
- Preparare il reagente 4, il reagente di lavaggio base.
- Dispensare 900ML di acqua di tipo 1 al bicchiere. Aggiungere 10,80 g di NaOH (ACS certificato o meglio) mescolando. Mescolare finché le pellet si dissolvono. Si raccomanda di memorizzare questa soluzione non più di 30 d.
- Preparare la soluzione di magazzino di standard interno.
- Ponderate 100 mg di standard 19: 0 EE. Aggiungere a 50 ml di pallone volumetrico lavato con esano. Aggiungere 15 ml di soluzione di Hexane-MTBE (reagente 3) a pallone, turbine per sciogliere. Portare a 50 mL con soluzione di Hexane-MTBE (reagente 3). Cappa e turbinio per mescolare. Trasferire in tubi di vetro espanso con tappi foderati in PTFE e conservare a -20 ° C.
2. GIORNO 1 - Estrazione degli acidi grassi dal suolo (da 4 a 5 ore per ~ 40 campioni)
- Preparare tre pipette di erogazione da 5 a 10 mL per P-buffer, cloroformio e metanolo.
NOTA: sono state proposte meno alternative pericolose per il cloroformio come estrattori. Questi possono determinare tassi equivalenti di recupero dei lipidi 16 , 17 , ma non sono valutati in questo protocollo. - Pesare i suoli congelati in tubi di centrifuga da 30 ml e registrare la massa del suolo.
- Effettuare due spazi vuoti e includere uno standard di controllo (un terreno estratto in precedenza).
- Nella cappa, aggiungere i reagenti al terreno in provetta da centrifuga nel seguente ordine: P-tampone, CHCl 3, e MeOH (Tabella 1). Lasciare il tempo terreni a umido dopo l'aggiunta P-tampone prima di aggiungere il CHCl 3. Coprire i tubi della centrifuga e coprire per proteggere dalla luce.
- Metterli sullo scuotitore in senso orizzontale assicurandoti che siano ben fissati. Con la velocità impostata su 280 giri / min, agitare per 1 h 18 .
- Preparare due 16 mm x 150 mm tubi di vetro per ogni campione come segue: Etichettare la provetta e aggiungere lo stesso volume di CHCl 3 e un volume uguale di P-buffer.
- Rimuovere i tubi della centrifuga dall'agitatore e centrifugare per 10 minuti a 1.430Xg e 25 ° C. La separazione di fase deve essere visibile nel tubo di vetro.
- Nella cappa di fumo, decantare il surnatante dal tubo di centrifuga in uno dei tubi preparati al punto 2.6. Ripetere i passaggi da 2.4 a 2.5 e decantare il surnatante nel secondo tubo.
- Bloccare saldamente tutti i tubi di vetro 16 mm × 150 mm con tappi in PTFE e invertiti 10 x per mescolare.
- Consentire ai campioni di rimanere indisturbati durante la notte per completare la separazione delle due fasi. A tal fine, conservare i campioni in un armadietto scuro o in uno strato di alluminio a temperatura ambiente. È accettabile permettere che gli estratti si separino durante il fine settimana.
- In alternativa, se si desidera passare direttamente al passo successivo o se i campioni vengono disturbati il giorno successivo, centrifugare campioni per 10 minuti a 1.000 xg e 25 ° C.
NOTA: Soggetti tutti i campioni all'interno di un gruppo di confronto allo stesso tempo di separazione di fase.
- In alternativa, se si desidera passare direttamente al passo successivo o se i campioni vengono disturbati il giorno successivo, centrifugare campioni per 10 minuti a 1.000 xg e 25 ° C.
3. GIORNO2 - Isolamento dei lipidi (da 3 a 4 ore per ~ 40 campioni)
- Accendere l'apparecchio di evaporazione e la trappola solvente. Impostare l'apparecchio di evaporazione a 33 ° C e preriscaldare.
- Impostare un aspiratore di vuoto nel cofano: un braccio laterale collegato ad una pompa a vuoto, una lunghezza di tubo di alta purezza e una pipetta di vetro.
- Aspirare lo strato superiore e l'interfaccia (circa 2/3 del senso di discesa) dei due tubi di vetro, mantenendo lo strato acquoso. Ripetere il processo utilizzando una pipetta pulita per ogni campione.
- Combinare i strati acquosi di estratto dal secondo tubo con quello nel primo tubo attentamente decantando. Assicurarsi che il tubo in decantazione sia privo di graffi o crepe.
- Una volta che i CHCl 3 estratti vengono combinati, ispezionare il liquido - dovrebbe essere chiaro. Se non aggiungete MeOH finché non sia chiaro.
- Asciugare tutti i campioni utilizzando l'apparecchio di evaporazione sotto vuoto. Iniziare con impostazioni della temperatura di 33 ° C, velocità vortice 26% e pressione 400 mbar.
- PPigiate i campioni nel sistema di evaporazione e dopo 10 minuti lentamente abbassate la pressione a 350 mbar.
- Monitorare il progresso e quando il livello del liquido nel tubo ha raggiunto il livello del blocco di riscaldamento, diminuire la pressione a 300 mbar.
- Continuare a monitorare l'altezza del liquido residuo e quando è di circa 1 cm di profondità, diminuire la pressione a 200 mbar.
- Dopo aver asciugato i campioni, rimuovere i campioni e far funzionare la pompa per altri 10 minuti per eliminare i vapori.
- Bloccare i tubi saldamente e conservare i lipidi in un congelatore a -80 ° C.
- Smaltire il liquido aspirato in conformità alle vigenti norme in materia di sicurezza chimica.
4. GIORNO 3 - Saponificazione e metilazione (da 6 a 7 ore per ~ 40 campioni)
- Accendere i bagni d'acqua. Impostare il bagno da 1 a 95 ° C e bagnare da 2 a 80 ° C.
- Impostare i distributori della pipetta e assicurarsi di distribuire il volume corretto.
Reagente 1: 1 mL per campione
Reagente 2: 2 mL per campione
Reagente 3: 1,25 mL per campione
Reagente 4: 3 mL per campione
NOTA: Il processo di riscaldamento creerà pressione nel tubo. Non utilizzare tubi graffiati, incrinati o tagliati. - Utilizzando un distributore di pipetta aggiungere 1.0 mL di Reagente 1 ai lipidi secchi. Capeggiare bene, vortexare per 5 s e collocare in un rack.
- Mettere il rack dei tubi di campionamento nel bagno d'acqua di 95 ° C per 5 minuti. Rimuovere il rack dei tubi dalla vasca e controllare le tubazioni per le perdite, indicati da bolle che aumentano nel tubo. Riavvitare o sostituire i tappi di tubi perdenti e controllare nuovamente le crepe oi chip. Continuare a riscaldare i tubi nel bagno d'acqua per altri 10 minuti.
- Rimuovere i campioni dal bagno 1 e collocare nel bagno 2 (80 ° C) e continuare l'incubazione per altri 15 minuti.
- Rimuovere i tubi e raffreddare i campioni posizionando il rack in una padella di acqua del rubinetto per raffreddare.
- Aggiungere 2 mL di Reagente 2 ad ogni campione. Capeggiare bene e vortex per 5 a 10s.
- Mettere il rack nell'acqua da 80 ° C e incubare per 10 min.
- Rimuovere il rack dei tubi dal bagno d'acqua e metterlo in una padella di acqua del rubinetto per raffreddare. Agitare la cremagliera dei tubi per accelerare il processo di raffreddamento.
NOTA: Non superare il tempo e la temperatura. Troppo il riscaldamento potrebbe degradare FAMEs. - Utilizzando il dispensatore di pipetta aggiungere 1,25 ml di reagente 3 ad ogni tubo di campione per estrarre gli esteri metilici dell'acido grasso. Cappare bene e mettere i tubi sull'agitatore per 10 min.
- Dopo la scossa, lasciare riposare il rack dei tubi per 10 minuti per separare le fasi. Trasferire la fase organica (top layer) ad un tubo di prova di vetro da 16 mm x 100 mm usando una pipetta di vetro.
- Ripetere l'estrazione aggiungendo aggiungendo il reagente 3, scuotendo, consentendo la separazione delle fasi e il trasferimento della fase superiore.
NOTA Non trasferire alcuna parte inferiore. È bene lasciare una piccola quantità di fase superiore nel tubo. - Utilizzando l'erogatore della pipetta, aggiungete 3 mL di Reagente 4 a thTubi da 16 mm x 100 mm.
- Bloccare i tubi di prova strettamente e vorticarsi per 20-30 s.
- Dopo vortexing, centrifugare per 3 min a 1000 × g.
- Usando una pipetta di vetro pulita, aspirare la fase organica superiore e trasferire in una fiala ambrata da 4 ml.
- Accendere l'apparecchio di evaporazione e la trappola solvente. Impostare l'apparecchio di evaporazione a 30 ° C, la velocità del vortice fino al 26%, il vuoto a 200 mbar e premere il preriscaldamento.
- Monitorare il progresso. Dopo aver asciugato i campioni, rimuovere i campioni e far funzionare la pompa per altri 10 minuti per eliminare i vapori.
- Rimuovere la pipetta dalle bottiglie reagenti 1 e 4 e pompare attraverso un po 'di acido diluito ( es. 1% HCl), seguita da acqua DI. Sciacquare la pipetta utilizzata per il reagente 2 pompando attraverso l'acqua di DI. Nel cappuccio, scolare il solvente dalla pipetta utilizzata per il reagente 3 in un contenitore appropriato e tenerlo nella cappa finché i residui di solvente non sono evaporati.
- Conservare le pipette invertite, con i pistoncini rimossi per impedire il bastoneG delle valvole di ritegno.
- Usare pipette di vetro una volta e poi smaltire in un contenitore appropriato.
- Nel cappuccio, lasciare residui di solvente sul vetro per evaporare.
- Sciacquare le vetrerie con acqua pulita e soluzione detergente.
5. GIORNO 4 - Preparazione della soluzione di lavoro e trasferimento di FAME ai flaconi GC (2-3 ore per ~ 40 campioni)
- Raccogliere i materiali: 4 ml di fiale ambrato con campioni secchi; Ambrati 2 ml di fiale GC; Inserti e capsule in vetro a fondo piatto da 400 μL; 500 μL siringa di vetro; 50 mL pallone volumetrico; Soluzione in titanio - Nonadecanoato etilico (19: 0 EE standard interno) in 50/50 esano / MTBE (Reagente 3).
- Utilizzando la siringa di vetro, aggiungere 500 μL di soluzione di base di nonadecanoato etilico (19: 0 EE) a una fiala volumetrica da 50 ml.
- Riempire il pallone al punteggio volumetrico con il reagente 3.
- Tappo di protezione e invertito 5x per mescolare.
- Trasferire 3 mL in un flacone pulito da 4 mL per un serbatoio di soluzione di lavoro.
- Usando thS, aggiungere 300 μL della soluzione di lavoro a ciascuna delle fiale da 4 ml contenenti gli esteri metilici dell'acido grasso secchi e il cappuccio.
- Vortex il campione per 15 s e mettere da parte stand per 15 minuti.
- Usando una pipetta di Pasteur di vetro, trasferire accuratamente gli esteri metilici dell'acido grasso sospeso in un flaconcino da 2 ml di GC contenente l'inserto da 400 μL.
- Conservare i flaconi sigillati GC in un congelatore -20 ° C prima dell'analisi.
- Invia campioni per l'analisi GC.
NOTA: L'analisi deve essere eseguita utilizzando una specifica colonna GC e le condizioni descritte nel file aggiuntivo S1 . È meglio che l'analisi del GC sia completata entro 2 settimane dalla metilazione.
Risultati
Le tabelle di dati dai report possono essere raggruppate in un foglio di calcolo o database. Dopo aver regolato il fattore di risposta (un fattore di correzione che normalizza la risposta per diverse lunghezze di catena), le aree di picco possono essere confrontate con l'area di picco degli standard esterni o interni per arrivare ad una concentrazione nell'estratto. Dividendo attraverso la massa del suolo estratto, i dati possono essere espressi come massa di FAME per grammo di terreno o, utilizzando il peso molecolare di ogni FAME, il più comunemente riportato nmol per grammo di terreno. La somma dei FAME microbici è indicativa della biomassa microbica totale e può essere confrontata tra i trattamenti ( Figura 1 ). Alcuni FAME possono anche essere associati a particolari gruppi microbici come batteri Gram-positivi o Gram-negativi, Actinomycetes, funghi micorrizali arbuscolari e funghi saprotrofici 19 , 20 , 21 , 22 , 23 , 24 . I rapporti della massa di biomarcatori specifici possono riflettere l'abbondanza relativa di questi gruppi ( Figura 2 ). Complessivamente i modelli di abbondanza FAME creano impronte digitali a livello comunitario, che consentono di confrontare la dissimilarità delle comunità microbiche attraverso tecniche multivariate come l'ordinazione ( Figura 3 ). In contrasto con la maggior parte degli approcci basati sul DNA, i dati lipidici a livello comunitario possono essere analizzati sia come abitudini relative o assolute. Se la biomassa totale differisce sostanzialmente tra i campioni, questi due approcci daranno risultati molto diversi; Le domande ecologiche sottostanti all'esperimento dovrebbero determinare quale approccio viene utilizzato.
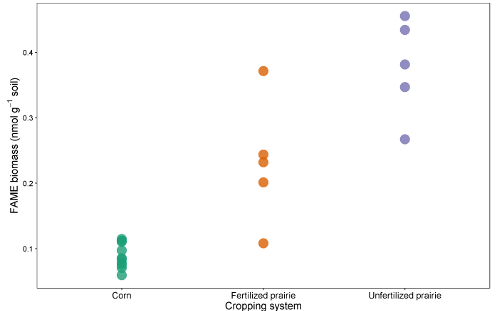
F Igure 1 : biomassa totale FAME. Confronto tra la biomassa totale biomarker FAME (nmol g -1 terreno) dal grano continuo, prateria fecondata e prateria non fertilitata. Clicca qui per visualizzare una versione più grande di questa figura.
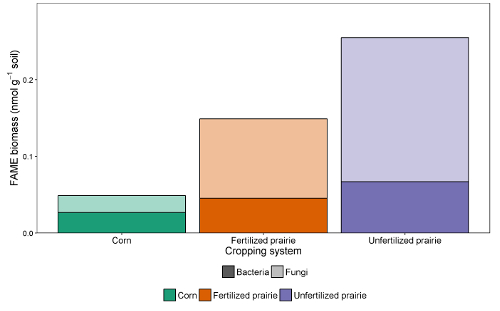
Figura 2 : FOME biomassa di batteri e funghi. Trattamento confronto di biomassa biomarker FAME batterica e fungina (nmol g -1 terreno) dal mais continuo, prateria fecondata e prateria non fertilitata. Massa fungine e batteriche da abbondanza assoluta. Media (somma f / somma b). Clicca qui per visualizzare una versione più grande di questa figura.
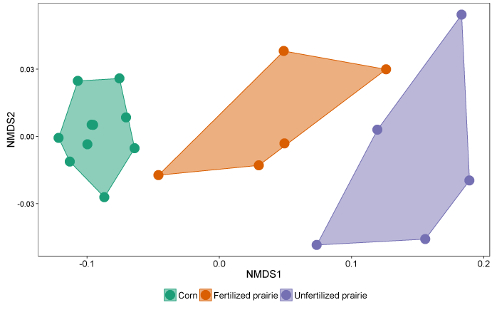
Figura 3 : Scala dimensionale non metrica dei biomarker lipidici FAME. Confronto della comunità microbica globale usando profili assoluti di abbondanza di tutti i biomarkers lipidi microbici FAME. In questo esempio, il mais continuo e le comunità di prateria non fertili si sono separati e sono molto distanti, mentre alcuni campioni di prateria fecondata hanno comunità microbiche che somigliano a quelle del mais e altri somigliano alla prateria non fertile. Clicca qui per visualizzare una versione più grande di questa figura.
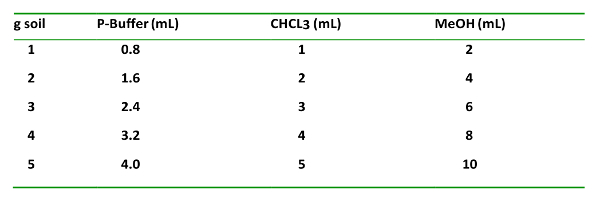
Tabella 1: T h e cosìl ven tipo t, propor zi s aggiunti g -1 terreno, uno d t h EIR orde r o f inserirlo iti o n. Ciò è importante per una corretta estrazione e separazione delle fasi organiche e acquose.
Discussione
Per l'esame di campioni multipli provenienti da esperimenti con molti replicati e / o unità sperimentali, i ricercatori possono trovare l'analisi di acido grasso-acido fosfolipidico (PLFA) in modo proibitivo in termini di tempo e di materiali 25 . Con il metodo PLFA, i fosfolipidi della membrana cellulare vengono estratti, purificati e identificati utilizzando l'estrazione acquosa-organica bidimensionale Bligh e Dyer 26 . Ciò è seguita da una cromatografia silica a fase solida per separare i lipidi dalla polarità e una metilazione alcalina di acidi grassi di fosfolipidi in esteri metilici dell'acido grasso. Nella profilazione PLFA la resa lipidica può essere bassa ma di purezza molto alta. ID Microbial ha introdotto un metodo alternativo, la procedura di metil-estere dell'acido grasso (MIDI-FA). Nel metodo MIDI-FA, tutti i lipidi vengono estratti direttamente da colture pura o campioni di terreno / sedimento 11 , 12 , 27 attraversosaponificazione. Questo metodo ha una minore perdita di lipidi ed è rapido perché non ha nessuna delle fasi di concentrazione o di purificazione del metodo PLFA. Tuttavia, mentre il metodo MIDI-FA è veloce e meno costoso, perché è stato originariamente progettato per identificare gli organismi nella cultura pura, non ci sono iniziali operazioni di estrazione o di purificazione. Così può includere composti lipidici co-estratti dalla materia organica del suolo che distorcono la firma comunitaria 27 , 28 , 29 . Poiché questa inclusione può anche distorcere le misure della biomassa, MIDI-FA è stata usata tipicamente per descrivere qualitativamente i lipidi del suolo 13 .
La procedura che descriviamo qui combina il meglio delle due procedure di estrazione separate: 1) un'estrazione e una concentrazione di lipidi usando il metodo Bligh e Dyer 26 modificato e 2) l'estere metilico dell'acido grassoLa silificazione, la metilazione, l'estrazione e la procedura di lavaggio base sviluppata in commercio. Questo metodo è stato sviluppato per ottenere i vantaggi di entrambi questi protocolli, minimizzando gli svantaggi 15 . Eseguendo l'estrazione iniziale e isolando i componenti solubili in organico ( ad es. Lipidi) prima di eseguire MIDI-FA e completandolo con una fase di purificazione, questo protocollo offre un equilibrio tra velocità e precisione. Anche se questo metodo potrebbe non essere adatto quando richiede purezza elevata ( cioè per 13 C PLFA) o quando analizza separatamente i fosfolipidi ei lipidi neutri, in molti casi consente di individuare le risposte microbiche della comunità a condizioni ambientali con maggiore sensibilità rispetto a quella del DNA Metodi 30 , 31 , 32 , 33 . I lipidi a membrana si decompongono rapidamente dopo la morte cellulare che li ha permessiRiflettono la comunità microbica vivente al momento del campionamento 5 , 7 , contrariamente al DNA ambientale in cui gran parte delle informazioni provengono da organismi morti o inattivi 34 . Data l'elevata percentuale di dormienza osservata tra i microrganismi del suolo 35 , la caratterizzazione della biomassa viva può essere utilizzata per comprendere le interazioni temporanee tra pianta e microbi a una scala temporale relativamente fine e i biomarkers lipidici possono essere utilizzati per analizzare lo stato fisiologico della comunità microbica 7 . È stato dimostrato che sono necessari metodi di trasmissione elevata per valutare la risposta microbica in grandi impostazioni di campo 25 e, sebbene il metodo che proponiamo qui non replica l'accuratezza della profilazione dei biomarker PLFA, aumenta il throughput minimizzando la variabilità realizzata con la MIDI-FA procedura. Il metodo si è rivelato uno strumento efficace nell'affrontareDomande relative alla dinamica delle comunità microbiche su un'ampia gamma di suoli in studi su vasta scala agricoli e ecosistemi 36 , 37 , 38 , 42 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 .
Le classi lipidiche sono combinate con questo metodo e ci può essere una perdita delle informazioni contenute in quelle classi separate 22 , 39 , ma combinando le classi lipidiche può rafforzare il potere di rilevare l'origine arbuscolare dei funghi micorisici del 16: 1 ω5c da entrambi i fosfori - e lipidi neutri 40 . Inoltre, mentre tIl numero di acidi grassi sconosciuti (che potrebbero derivare da materia organica non vivente) può essere più elevato con questo metodo, è stato dimostrato inferiore a quello di MIDI-FA e consente il confronto tra i profili lipidici da studi con molti campioni in cui il campione La capacità di trasmissione è una preoccupazione 15 . La frazione neutrale è generalmente pensata derivare principalmente da lipidi di deposito prodotti dai funghi, anche se può esserci anche un minor contributo della fauna del suolo 41 . Alla luce di questo, il metodo descritto qui può dare risultati che mostrano un maggior contributo dei lipidi fungini 18: 2 ω 6,9c e 18: 1 ω 9c rispetto a PLFA. Gli altri lipidi che tendono ad apparire nella frazione neutrale includono alcuni degli acidi grassi saturi, ad es. 16: 0, 18: 0, 20: 0.
Esistono diversi modi in cui i dati lipidici possono essere espressi e analizzati. Le rappresentazioni più comuni sono l'abbondanza (nmol g -1 terreno), mole fractioN (nmol singolo lipid nmol -1 totale lipid), e mole percentuale (frazione mole * 100). Normalizzati dai lipidi totali in un campione, la frazione molare e il mole percentuale sono misure della relativa abbondanza di un dato lipide. Dopo una trasformazione appropriata, ad esempio , la frazione quadrata di radice quadrata di arcsine è appropriata per l'uso in analisi da parte dei componenti principali o dell'ordinazione di analisi di ridondanza. L'abbondanza è la quantità assoluta di un dato lipide estratto per grammo di terreno. Poiché la quantità di lipidi per cellula è ragionevolmente costante e l'estrazione lipidica è altamente efficiente e completa, l'abbondanza totale è una buona stima dei lipidi totali e l'abbondanza di indicatori chiave riflette la biomassa del gruppo ecologico che rappresenta 17 . Infine, un buon modo per esaminare la composizione microbica della comunità è quello di utilizzare metodi di analisi multivariata 16 , ad esempio metodi di ordinazione come la scala multimetrica nonmetrica (NMDS -Che non necessita di trasformazione dei dati) o analisi dei componenti principali (PCA), può essere utile per confrontare l'abbondanza relativa di tutti i biomarker lipidici.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato in parte dal DOE BER Ufficio di Scienza DE-FC02-07ER64494 e DOE OBP Ufficio di Efficienza Energetica e Energia Rinnovabile (DE-AC05-76RL01830). Gli autori vorrebbero riconoscere Anders Gurda per il suo paziente e competente contributo alla videografia e al montaggio.
Materiali
| Name | Company | Catalog Number | Comments |
| RapidVap | Labconco | 7900000 | Evaporative system |
| RapidVap | Labconco | 7491400 | Sample block |
| Chem-resistant vacuum pump | Labconco | 7584000 | Vacuum pump |
| Chemical trap cannister | Labconco | 7815300 | Trap cannister |
| Solvent insert | Labconco | 7515200 | Solvent trap insert |
| Diaphragm pump | Welch | 7398000 | DryFast vacuum pump |
| Water bath | Fisher | 15-462-15Q | 2 well water bath |
| Gas chromatograph | Various | N/A | For sample analysis |
| Centrifuge | Various | N/A | For sample separation |
| Freeze Dryer | Various | N/A | For sample lyophilization |
| Repipet (6) | BrandTech | 4720440 | For dispensing reagents |
| Vortex | Fisher | 02-215-365 | Analog vortex mixer |
| Teflon centrifuge tubes | Thermo/Nalgene | 3114-0030 | Teflon sample tubes |
| Caps | Thermo/Nalgene | DS3131-0020 | Caps for teflon tubes |
| Test tube | Corning | 9825-16 | 16x100mm tubes |
| Test tube | Corning | 9825-16xx | 16x150mm tubes |
| Caps | Corning | 9998-15 | 15-415 thread black phenolic caps w/PTFE liner |
| Pasteur pipets | Fisher | 13-678-20B | 14.6cm |
| 500 uL glass syringe | Fisher | 13684106LC | Hamilton 81217 |
| Amber vials | Agilent | 5182-0716 | 4mL Amber vials |
| Caps | Agilent | 5182-0717 | Blue screw caps |
| Inserts | Agilent | 5181-3377 | 400uL flat bottom glass inserts |
| Standards | |||
| 19:0 ethyl nonadecanoate (Ethyl nonadecanoate) | VWR | TCN0459-5G | Analytical standard |
| Chemicals | |||
| Dipotassium phosphate (K2HPO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Potassium phosphate monobasic (KH2PO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Methanol (CH3OH - HPLC grade or better) | Various | N/A | For making Reagents 1 & 2 |
| Sodium hydroxide (NaOH - ACS grade or better) | Various | N/A | For making Reagents 1 & 4 |
| Hydrochloric acid (6N HCL) | Various | N/A | For making Reagent 2 |
| Hexane (HPLC grade or better) | Various | N/A | For making Reagent 3 |
| MTBE (Methyl tert-butyl ether - HPLC grade or better) | Various | N/A | For making Reagent 3 |
Riferimenti
- Van Der Heijden, M. G. a, Bardgett, R. D., Van Straalen, N. M. The unseen majority: Soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecol Lett. 11 (3), 296-310 (2008).
- Bever, J. D., et al. Rooting theories of plant community ecology in microbial interactions. Trends Ecol Evol. 25 (8), 468-478 (2010).
- Suleiman, A. K. A., Manoeli, L., Boldo, J. T., Pereira, M. G., Roesch, L. F. W. Shifts in soil bacterial community after eight years of land-use change. Syst Appl Microbiol. 36 (2), 137-144 (2013).
- Sayer, E. J., et al. Grassland management influences spatial patterns of soil microbial communities. Soil Biol Biochem. 61, 61-68 (2013).
- Fernandes, M. F., Saxena, J., Dick, R. P. Comparison of Whole-Cell Fatty Acid (MIDI) or Phospholipid Fatty Acid (PLFA) Extractants as Biomarkers to Profile Soil Microbial Communities. Microbial Ecol. 66 (1), 145-157 (2013).
- van der Heijden, M. G. A., Wagg, C. Soil microbial diversity and agro-ecosystem functioning. Plant Soil. 363 (1-2), 1-5 (2013).
- Allison, V. J., Miller, R. M., Jastrow, J. D., Matamala, R., Zak, D. R. Changes in soil microbial community structure in a tallgrass prairie chronosequence. Soil Sci Soc Am J. 69 (5), 1412-1421 (2005).
- Kowalchuk, G. A., Buma, D. S., de Boer, W., Klinkhamer, P. G. L., van Veen, J. A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. A van Leeuw. 81 (1-4), 509-520 (2002).
- Bardgett, R. D., Hobbs, P. J., Frostegard, A. Changes in soil fungal:bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biol Fert Soils. 22 (3), 261-264 (1996).
- Frostegård, Å, Tunlid, A., Bååth, E. Use and misuse of PLFA measurements in soils. Soil Biol Biochem. 43 (8), 1621-1625 (2011).
- Haack, S. K., Garchow, I. H., Odelson, D. A., Forney, L. J., Klug, M. J. Accuracy , Reproducibility , and Interpretation of Fatty Acid Methyl Ester Profiles of Model Bacterial Communitiest. Appl Environ Microbiol. 60 (7), 2483-2493 (1994).
- Drenovsky, R. E., Elliott, G. N., Graham, K. J., Scow, K. M. Comparison of phospholipid fatty acid ( PLFA ) and total soil fatty acid methyl esters ( TSFAME ) for characterizing soil microbial communities. Soil Biol Biochem. 36, 1793-1800 (2004).
- Cavigelli, M. A., Robertson, G. P., Klug, M. Fatty acid methyl ester ( FAME ) profiles as measures of soil microbial community structure. Plant Soil. 170, 99-113 (1995).
- Findlay, R. Determination of microbial community structure using phospholipid fatty acid profiles. Molecular microbial ecology manual. , Available from: https://www.researchgate.net/profile/Robert_Findlay2/publication/281208148_Determination_of_microbial_community_structure_using_phospholipid_fatty_acid_profiles/links/55db4f0b08aec156b9afe776.pdf 2885-2906 (2004).
- Balser, T. C., Liang, C., Gutknecht, J. L. Linking microbial community analysis and ecosystem studies: A rapid lipd analysis protocol for high throughput. Biol Fert Soils. , Forthcoming (2017).
- Findlay, R. H., King, G. M., Watling, L., Watling, L. E. S. Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sediments Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sedimentst. Appl Environ Microbiol. 55 (11), 2888-2893 (1989).
- Willers, C., Jansen van Rensburg, P. J., Claassens, S. Microbial signature lipid biomarker analysis - an approach that is still preferred, even amid various method modifications. J Appl Microbiol. 118 (6), 1251-1263 (2015).
- Balser, T. C. Phospholipid Fatty-acid Analysis (PLFA). Protocol. , Available from: http://nature.berkeley.edu/soilmicro/methods/BalserPLFA.pdf (2017).
- Zelles, L., Bai, Q. Y., Beck, T., Beese, F. Signature fatty-acids in phospholipids and lipopolysachharides as indicators of microbial biomass and community structure in agricultural soils. Soil Biol Biochem. 24 (4), 317-323 (1992).
- Frostegard, A., Tunlid, A., Baath, E. Phospholipid fatty-acid composition, biomass, and activity of microbial communities from 2 soil types experimentally exposed to different heavy-metals. Appl Environ Microbiol. 59 (11), 3605-3617 (1993).
- Federle, T. W., Dobbins, D. C., Thorntonmanning, J. R., Jones, D. D. Microbial biomass, activity, and community structure in subsurface soils. Ground Water. 24 (3), 365-374 (1986).
- Vestal, J. R., White, D. C. Lipid analysis in microbial ecology - quantitative approaches to the study of microbial communities. BioScience. 39 (8), 535-541 (1989).
- Wilkinson, S. G. Gram negative bacteria. Microbial Lipids. , 299-488 (1988).
- Balser, T. C., Treseder, K., Ekenler, M. Using lipid analysis and hyphal length to quantify AM and saprotrophic fungal abundance along a soil chronosequence. Soil Biol Biochem. 37 (3), 601-604 (2005).
- Buyer, J. S., Sasser, M. High throughput phospholipid fatty acid analysis of soils. Appl Soil Ecol. 61, 127-130 (2012).
- Bligh, E. G., Dyer, W. J. A rapid method of total lipid extraction and purification. Can J Biochem Phys. (37), 911-917 (1959).
- Schutter, M. E., Dick, R. P. Comparison of Fatty Acid Methyl Ester (FAME) Methods for Characterizing Microbial Communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Jandl, G., Leinweber, P., Schulten, H., Ekschmitt, K. Contribution of primary organic matter to the fatty acid pool in agricultural soils. Soil Biol Biochem. 37, 1033-1041 (2005).
- Nielsen, P., Petersen, S. O. Ester-linked polar lipid fatty acid profiles of soil microbial communities: a comparison of extraction methods and evaluation of interference from humic acids. Soil Biol Biochem. 32, 1241-1249 (2000).
- Duncan, D. S., Jewell, K. A., Suen, G., Jackson, R. D. Detection of short-term cropping system-induced changes to soil bacterial communities differs among four molecular characterization methods. Soil Biol Biochem. 96, 160-168 (2016).
- Liang, C., et al. Switchgrass rhizospheres stimulate microbial biomass but deplete microbial necromass in agricultural soils of the upper Midwest, USA. Soil Biol Biochem. 94, 173-180 (2016).
- Jangid, K., et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties. Soil Biol Biochem. 43 (10), 2184-2193 (2011).
- Ritz, K., et al. Spatial structure in soil chemical and microbiological properties in an upland grassland. FEMS Microbiol Ecol. 49 (2), 191-205 (2004).
- Carini, P., et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. J Chem Inf Model. 53 (9), 1689-1699 (2013).
- Lennon, J. T., Jones, S. E. Microbial seed banks: The ecological and evolutionary implications of dormancy. Nat Rev Microbiol. 9 (2), 119-130 (2011).
- Gutknecht, J. L. M., Field, C. B., Balser, T. C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years. Glob Change Biol. 18, 225-269 (2012).
- Pei, Z., et al. Soil and tree species traits both shape soil microbial communities during early growth of Chinese subtropical forests. Soil Biol Biochem. 96, 180-190 (2016).
- Oates, L. G., Duncan, D. S., Sanford, G. R., Liang, C., Jackson, R. D. Bioenergy cropping systems that incorporate native grasses stimulate growth of plant-associated soil microbes in the absence of nitrogen fertilization. Ag Ecosys Environ. 233, 396-403 (2016).
- Bååth, E. The use of neutral lipid fatty acids to indicate the physiological conditions of soil fungi. Microbial Ecol. 45 (4), 373-383 (2003).
- Ngosong, C., Gabriel, E., Ruess, L. Use of the signature Fatty Acid 16:1ω5 as a tool to determine the distribution of arbuscular mycorrhizal fungi in soil. J Lipids. 2012, 236807(2012).
- Sharma, M. P., Buyer, J. S. Comparison of biochemical and microscopic methods for quantification of arbuscular mycorrhizal fungi in soil and roots. Appl Soil Ecol. 95, 86-89 (2015).
- Oates, L. G., Balser, T. C., Jackson, R. D. Subhumid pasture soil microbial communities affected by presence of grazing, but not grazing management. Appl Soil Ecol. 59, 20-28 (2012).
- Liang, C. Potential legacy effects of biofuel cropping systems on soil microbial communities in southern Wisconsin, USA. Ag Sci. 2 (2), 131-137 (2011).
- Herzberger, A. J., Duncan, D. S., Jackson, R. D. Bouncing Back Plant-Associated Soil Microbes Respond Rapidly to Prairie Establishment. PloS One. 9 (12), 1-14 (2014).
- Fraterrigo, J. M., Balser, T. C., Turner, M. G. Microbial community variation and its relationship with nitrogen mineralization in historically altered forests. Ecology. 87 (3), 570-579 (2006).
- Kao-Kniffin, J., Balser, T. C. Elevated CO2 differentially alters belowground plant and soil microbial community structure in reed canary grass-invaded experimental wetlands. Soil Biol Biochem. 39 (2), 517-525 (2007).
- Mentzer, J. L., Goodman, R. M., Balser, T. C. Microbial response over time to hydrologic and fertilization treatments in a simulated wet prairie. Plant Soil. 284 (1-2), 85-100 (2006).
- Ushio, M., Wagai, R., Balser, T. C., Kitayama, K. Variations in the soil microbial community composition of a tropical montane forest ecosystem: Does tree species matter. Soil Biol Biochem. 40 (10), 2699-2702 (2008).
- Bartelt-Ryser, J., Joshi, J., Schmid, B., Brandl, H., Balser, T. Soil feedbacks of plant diversity on soil microbial communities and subsequent plant growth. Perspect Plant Ecol. 7 (1), 27-49 (2005).
- Fichtner, A., von Oheimb, G., Härdtle, W., Wilken, C., Gutknecht, J. L. M. Effects of anthropogenic disturbances on soil microbial communities in oak forests persist for more than 100 years. Soil Biol Biochem. 70, 79-87 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon