Method Article
Um método de extração e análise de lipídios para caracterizar micróbios de solo em experimentos com muitas amostras
Neste Artigo
Resumo
O artigo descreve um método que aumenta o rendimento enquanto equilibra esforço e precisão para a extração de lipídios das membranas celulares de microorganismos para uso na caracterização de lipídios totais e abundância relativa de lípidos indicadores para determinar a estrutura da comunidade microbiana do solo em estudos com muitas amostras.
Resumo
As comunidades microbianas são impulsionadores e reguladores importantes dos processos ecossistêmicos. Para entender como a gestão dos ecossistemas pode afetar as comunidades microbianas, uma técnica relativamente precisa, intensiva e intensiva para análise da composição da comunidade microbiana é a análise de ácidos gordos fosfolipídicos (PLFA). O PLFA foi desenvolvido para analisar biomarcadores de fosfolípidos, que podem ser utilizados como indicadores de biomassa microbiana e a composição de amplos grupos funcionais de fungos e bactérias. Geralmente, ele usou-se para comparar solos em comunidades alternativas de plantas, ecologia e regimes de gestão. O método PLFA mostrou-se sensível à detecção de mudanças na composição da comunidade microbiana.
Um método alternativo, extração e análise de éster metílico de ácidos gordos (MIDI-FA) foi desenvolvido para extração rápida de lipídios totais, sem separação da fração de fosfolípidos, de culturas puras como técnica de identificação microbiana. Este método éRápido, mas é menos adequado para amostras de solo porque falta um passo inicial separando as partículas do solo e começa em vez disso com uma reação de saponificação que provavelmente produz artefatos da matéria orgânica de fundo no solo.
Este artigo descreve um método que aumenta o rendimento enquanto equilibra esforço e precisão para a extração de lipídios das membranas celulares de microorganismos para uso na caracterização de lipídios totais e na abundância relativa de lípidos indicadores para determinar a estrutura da comunidade microbiana do solo em estudos com muitas amostras. O método combina a precisão obtida através do perfil PLFA, extraindo e concentrando os lipídios do solo como um primeiro passo e uma redução de esforço ao saponificar o material orgânico extraído e processando com o método MIDI-FA como um segundo passo.
Introdução
Dado o papel fundamental dos microrganismos no ciclismo nutritivo 1 , a modificação da composição da comunidade vegetal 2 , a regulação da produtividade vegetal 3 e a decomposição da matéria orgânica 4 , a compreensão das comunidades microbianas do solo é vital para a compreensão dos ecossistemas terrestres.
Devido à sua abundância relativamente elevada no solo e à sua assinatura química, os biomarcadores lipídicos podem ser utilizados para perfis dos grupos ecológicos dominantes que compõem comunidades microbianas do solo 5 . Ao quantificar os biomarcadores lipídicos que são característicos de diferentes grupos microbianos, podemos estimar os lipídios totais e, em seguida, separamos esses lipídios em grupos ecologicamente relevantes, como bactérias Gram-positivas (Gm +) e Gram-negativas (Gm-), micorrízicas arbusculares (AM) e saprótroficas Fungos e actinomicetos 5 , Ss = "xref"> 6 , 7 , 8 .
Existem muitos métodos para caracterizar aspectos das comunidades microbianas. O método PLFA é comumente usado para compreender a estrutura básica da comunidade microbiana. É uma maneira eficaz de avaliar a abundância relativa de grupos microbianos, bem como a biomassa microbiana total. Devido ao rápido turnover de lipídios, o perfil de PLFA também permite a detecção relativamente rápida de mudanças na comunidade microbiana do solo e fornece informações que permitem a comparação da função do ecossistema, por exemplo, fúngicas: índices bacterianos para avaliar as taxas de ciclagem de nutrientes 1 , 9 , 10 . No entanto, enquanto o método de extração de PLFA é honrado e bem respeitado, também é demorado e não se presta bem aos estudos de escala ecossistêmica que requerem um grande número de amostras de repetições de escala de campoF "> 11 , 12 .
Em contraste, o método de extração de éster metílico de ácidos gordos (MIDI-FA) tem o potencial de permitir um rendimento rápido. Neste método, as amostras são saponificadas, convertidas em FAMEs, extraídas e depois analisadas. O método MIDI-FA é rápido, mas menos discriminatório do que o PLFA, que combina a extração de lipídios com a separação de diferentes classes lipídicas 13 (fosfolípidos, lipídios neutros e glicolípidos).
Neste protocolo, descrevemos um método que combina elementos de perfil de lipídeos de PLFA e MIDI-FA. Ele emprega extração de lipídios usando as etapas iniciais de extração de clorofórmio do método de Bligh e Dyer modificado, e depois saponificação e conversão para FAMEs. Isso fornece uma maneira robusta de detectar a estrutura da comunidade microbiana, excluindo grande parte do ruído de fundo do material não microbiano 5 , 14 . Este método foi desenvolvido para alcançar um equilíbrio entre os protocolos PLFA e MIDI-FA, ou seja, reter a maior parte da precisão ao mesmo tempo em que aumenta a taxa de transferência para tornar logistica e economicamente viável a análise de lipídios em estudos de larga escala com muitas amostras 15 . Ao realizar a extração inicial e isolar os componentes orgânicos solúveis ( por exemplo, lipídios) antes de executar MIDI-FA, e completando isso com um passo de purificação, o protocolo oferece um equilíbrio entre velocidade e precisão.
Protocolo
NOTA: Use sempre o equipamento de proteção pessoal apropriado (PPE) durante todo o procedimento. Para evitar a potencial contaminação da amostra, não toque no vidro com as mãos nuas. Use luvas apropriadas quando estiver executando etapas de protocolo que exigem manipulação de clorofórmio.
1. Preparações (2 dias para ~ 40 amostras)
- Coloque o solo em sacos estéreis e transporte do campo em um refrigerador contendo gelo. Se não for possível peneirar o solo fresco e congelar imediatamente, armazene as amostras em um congelador de -80 ° C até estar pronto para iniciar a análise.
- Prepare o solo homogeneizando. Remova as raízes e as pedras e separe os camadas por uma peneiração grosseira ( por exemplo, 2 mm).
- Prepare-se para a liofilização colocando subconjuntos em um recipiente apropriado de acordo com as instruções do manual do congelador seco e congelar o mais rápido possível. Uma vez que os solos são liofilizados, guarde em um recipiente selado com dessecante até a extração. O melhor é sRasgou o solo liofilizado a um mínimo de -20 ° C, mas de preferência a -80 ° C.
- Em preparação para a extração, remova os solos liofilizados do armazenamento e moer até uma consistência semelhante à farinha. Os métodos para moagem incluem moinho de bolas, batedores de bola e argamassa e pilão. Após triturar o solo, guarde em um freezer (ver 1.3).
NOTA: A quantidade de amostra utilizada para extração depende do seu teor de matéria orgânica. Uma orientação geral é usar 0,5 a 1 g de um solo que seja de 12 a 18% de carbono em peso e 3 a 5 g para um solo de 1 a 3% de carbono em peso. - Pré-enxaguar tubos de centrífuga de 30 mL com hexano. Adicione aproximadamente 2 a 3 mL de hexano aos tubos e vortex por 5 s. Decantar hexano para outro tubo e vortex. Hexano (2 a 3 mL) pode ser usado para enxaguar em série seis tubos. Armazene os tubos enxaguados com hexano invertidos na chaminé e descarte o hexano usado em um recipiente de lixo apropriado.
- Enrole o vidro em 2 a 3 camadas de papel alumínio e coloque no forno de mufla. Assar(Muffle) a 450 ° C durante 4,5 h.
- Prepare reagentes.
- Para a quantidade de solo utilizado, adicionar produtos químicos em um 0,8: 1: 2 proporção em volume em tampão fosfato (P-tampão): CHCl 3: MeOH.
- Preparar solução de tampão P: fosfato-tampão: 0,1 M, pH 7,0.
- Adicione 61 mL de estoque de K 2 HPO 4 1M, estéril (o produto químico deve ser certificado com qualidade ACS ou melhor). Adicione 39 mL de estoque de 1 M KH 2 PO 4 , estéril (o produto químico deve ser certificado ACS ou melhor). Preencha para 1000 mL com água de Tipo 1. Ajuste o pH para 7,0 com NaOH ou HCl. Armazene a solução não utilizada por até 7 d à temperatura ambiente ou 30 dias no refrigerador.
- Alternativamente, pesar 10,62 g de K 2 HPO 4 e 5,31 g de KH 2 PO 4 ; Diluir para 1 L com água de Tipo 1. Verifique o pH, ajuste se necessário.
- Prepare o Reagente 1, Reagente de Saponificação.
- Distribuir 300 mL de água de Tipo 1.Adicionar 300 mL de CH 3 OH (grau HPLC ou superior). Adicione 90,00 g de NaOH (certificado ACS ou melhor). Adicione pellets de NaOH à solução enquanto se agita. Mexa até que as pastilhas se dissolvam. Recomenda-se que esta solução não armazene mais de 30 d.
- Preparar Reagente 2, Reagente de Metilação.
- Dispensar 275 mL de CH 3 OH (grau HPLC ou superior). Adicione 325 mL de CHC 6.00 N certificado enquanto se mexa. Recomenda-se que esta solução não armazene mais de 30 d.
- Preparar o Reagente 3, Reagente de Extração: 50% C 6 H 14 (Hexano), 50% C 5 H 12 O (MTBE).
- Na exaustão, combine 200 mL de C 6 H 14 (grau HPLC ou superior) e 200 mL de C 5 H 12 O (grau HPLC ou superior). Misture bem; Capture firmemente. Armazene no armário inflamável ou na chaminé durante mais de 1 ano.
- Prepare Reagente 4, Reagente de lavagem base.
- Dispense 900ML de água tipo 1 para copo. Adicione 10,80 g de NaOH (Certified ACS ou melhor) enquanto mexa. Mexa até que as pastilhas se dissolvam. Recomenda-se que esta solução não armazene mais de 30 d.
- Prepare a solução-mãe do padrão interno.
- Pesar 100 mg de padrão 19: 0 EE. Adicionar a 50 mL de balão volumétrico enxaguado com hexano. Adicione ~ 15 mL de solução de Hexano-MTBE (reagente 3) ao balão, faça um redemoinho para dissolver. Desligue para 50 mL com solução de Hexano-MTBE (reagente 3). Cap e redemoinho para misturar. Transfira para tubos de vidro enxaguados com hexano com tampas revestidas com PTFE e guarde-se a -20 ° C.
2. DIA 1 - Extração de Ácidos Graxos do Solo (4 a 5 h para ~ 40 Amostras)
- Prepare três pipetas de distribuição de 5 a 10 mL para tampão P, clorofórmio e metanol.
NOTA: alternativas alternativas menos perigosas ao clorofórmio foram propostas como extractantes. Estes podem resultar em taxas equivalentes de recuperação de lipídios 16 , 17 , mas não são avaliadas neste protocolo. - Pesar solos liofilizados em tubos de centrífuga de 30 mL e registrar a massa do solo.
- Faça dois espaços em branco e inclua um padrão de verificação (um solo que foi extraído anteriormente).
- Na hotte, adicionar os reagentes para o solo no tubo de centrifugação na seguinte ordem: P-tampão, CHCl 3 e MeOH (Tabela 1). Permitir que o tempo para molhar o solo após a adição de P-tampão antes de adicionar o CHCI3. Tire os tubos da centrífuga firmemente e cubra para proteger da luz.
- Coloque-os no agitador horizontalmente, assegurando-se de que estão bem protegidos. Com a velocidade ajustada em 280 rpm, agite durante 1 h 18 .
- Preparar dois 16 milímetros x 150 mm tubos de vidro para cada amostra como se segue: Rotular o tubo e adicionar o mesmo volume de CHCl3 e um volume igual de P-tampão.
- Remova os tubos da centrífuga do agitador e centrifugue por 10 min a 1.430Xg e 25 ° C. A separação de fases deve ser visível no tubo de vidro.
- Na exaustão, decanta o sobrenadante do tubo de centrífuga para um dos tubos preparados no passo 2.6. Repita os passos 2.4 a 2.5 e decanta o sobrenadante para o segundo tubo.
- Tire firmemente todos os tubos de vidro de 16 mm x 150 mm com tampas revestidas com PTFE e inverta 10 x para misturar.
- Permita que as amostras permaneçam intactas durante a noite para completar a separação das duas fases. Para fazer isso, mantenha as amostras em um armário / espaço escuro ou cobertas de papel alumínio à temperatura ambiente. É aceitável permitir que os extratos se separem durante o fim de semana.
- Alternativamente, se alguém deseja mover-se diretamente para o próximo passo, ou se as amostras forem perturbadas no dia seguinte, centrifugar as amostras por 10 min a 1.000 xg e 25 ° C.
NOTA: Assine todas as amostras dentro de um grupo de comparação para o mesmo tempo de parada de separação de fase.
- Alternativamente, se alguém deseja mover-se diretamente para o próximo passo, ou se as amostras forem perturbadas no dia seguinte, centrifugar as amostras por 10 min a 1.000 xg e 25 ° C.
3 DIAS2 - Isolamento de Lípidos (3 a 4 h para ~ 40 Amostras)
- Ligue o aparelho de evaporação e a armadilha de solvente. Coloque o aparelho de evaporação a 33 ° C e aquecer o pré-aquecimento.
- Configure um aspirador de vácuo no capô: um balão de braço lateral conectado a uma bomba de vácuo, um comprimento de tubulação de alta pureza e uma pipeta de vidro.
- Aspirar a camada superior e a interface (aproximadamente 2/3 do caminho para baixo) dos dois tubos de vidro, mantendo a camada aquosa. Repita o processo usando uma pipeta limpa para cada amostra.
- Combine as camadas aquosas de extração do segundo tubo com a do primeiro tubo com uma decantação cuidadosa. Certifique-se de que o tubo que está decantando está livre de arranhões ou rachaduras.
- Uma vez que os extratos de CHCl 3 são combinados, inspecione o líquido - deve ser claro. Se não adicionar MeOH até ficar limpo.
- Secar todas as amostras usando o aparelho de evaporação a vácuo. Comece com configurações de temperatura 33 ° C, velocidade do vortex 26% e pressão 400 mbar.
- PEncaixe as amostras no sistema de evaporação e, após 10 min, baixe lentamente a pressão para 350 mbar.
- Monitorize o progresso e, quando o nível de líquido no tubo atingiu o nível do bloco de aquecimento, diminua a pressão para 300 mbar.
- Continue a monitorar a altura do líquido restante e, quando tiver aproximadamente 1 cm de profundidade, diminua a pressão para 200 mbar.
- Depois que as amostras secarem, remova as amostras e execute a bomba por 10 minutos adicionais para limpar os vapores.
- Capte os tubos firmemente e armazene os lípidos em um congelador a -80 ° C.
- Descarte o líquido aspirado de acordo com as normas de segurança química aplicáveis.
4. DIA 3 - Saponificação e Metilação (6 a 7 h para ~ 40 Amostras)
- Ligue os banhos de água. Coloque o banho 1 a 95 ° C e banhe 2 a 80 ° C.
- Coloque os distribuidores de pipeta e assegure-se de que estão distribuindo o volume correto.
Reagente 1: 1 mL por amostra
Reagente 2: 2 mL por amostra
Reagente 3: 1,25 mL por amostra
Reagente 4: 3 mL por amostra
NOTA: O processo de aquecimento irá aumentar a pressão no tubo. Não use tubos arranhados, rachados ou lascados. - Usando um distribuidor de pipeta adicione 1,0 mL de Reagente 1 aos lipídios secos. Capture firmemente, vortex por 5 s e coloque em um rack.
- Coloque a cremalheira de tubos de amostra no banho de água a 95 ° C durante 5 min. Retire a cremalheira dos tubos do banho e verifique se os tubos estão vazados, indicados por bolhas que se elevam no tubo. Aperte ou substitua as tampas dos tubos com vazamento, e verifique novamente as rachaduras ou as aparas. Continue a aquecer os tubos no banho de água por mais 10 min.
- Remova as amostras do banho 1 e coloque no banho 2 (80 ° C) e continue a incubação por mais 15 min.
- Retire os tubos e esfriar as amostras, colocando o rack em uma panela de água da torneira para o resfriamento.
- Adicione 2 mL de Reagente 2 a cada amostra. Capa bem e vortex por 5 a 10s.
- Coloque o suporte no banho-maria a 80 ° C e incube durante 10 min.
- Remova a grelha de tubos do banho de água e coloque em uma panela de água da torneira para resfriamento. Agite o rack de tubos para acelerar o processo de resfriamento.
NOTA: Não exceda o tempo ea temperatura. Muito aquecimento pode degradar FAMEs. - Usando o distribuidor de pipeta, adicione 1,25 mL de Reagente 3 a cada tubo de amostra para extrair os ésteres metílicos de ácidos gordos. Prenda firmemente e coloque os tubos no agitador durante 10 min.
- Depois de agitar, permita que a cremalheira de tubos se assente durante 10 minutos para que as fases se separem. Transfira a fase orgânica (camada superior) para um tubo de teste de vidro de 16 mm x 100 mm usando uma pipeta de vidro.
- Repita a extração adicionando adicionalmente o Reagente 3, agitando, permitindo que as fases se separem e a transferência da fase superior.
NOTA Não transfira nenhuma fase inferior. É bom deixar uma pequena quantidade de fase superior no tubo. - Usando o distribuidor de pipeta, adicione 3 mL de Reagent 4 to thTubos de 16 mm x 100 mm.
- Coloque os tubos de ensaio firmemente e vortex por 20 a 30 s.
- Após o vortex, centrifugar durante 3 min a 1000 × g.
- Usando uma pipeta de vidro limpo, aspirar a fase orgânica superior e transferir para um frasco de âmbar de 4 mL.
- Ligue o aparelho de evaporação e a armadilha de solvente. Ajuste o aparelho de evaporação a 30 ° C, a velocidade do vortex para 26%, o vácuo para 200 mbar e pressione o pré-aquecimento.
- Monitorize o progresso. Depois que as amostras secarem, remova as amostras e execute a bomba por 10 minutos adicionais para limpar os vapores.
- Remova a pipeta das garrafas de reagente 1 e 4 e bombeie através de um ácido diluído ( por exemplo, 1% de HCl), seguido de água DI. Enxaguar a pipeta utilizada no Reagente 2 por bombeamento através da água DI. No capô, drenar o solvente da pipeta usada para o Reagente 3 em um recipiente adequado e mantê-lo no capô até os resíduos do solvente terem evaporado.
- Armazene as pipetas invertidas, com os êmbolos removidos para evitar a colocaçãoG das válvulas de retenção.
- Use pipetas de vidro uma vez e depois descarte em um recipiente apropriado.
- No capô, permita que os resíduos de solventes em produtos de vidro se evaporem.
- Enxaguar produtos de vidro com água limpa e solução detergente.
5. DIA 4 - Preparação da Solução de Trabalho e Transferência de FAMEs para os Frascos GC (2-3 h para ~ 40 Amostras)
- Reúna materiais: 4 mL de frascos âmbar com amostras secas; Frascos amber de 2 mL GC; Inserções e tampas de vidro de fundo plano de 400 μL; Seringa de vidro de 500 μL; Balão volumétrico de 50 mL; Solução de estoque - Nonadecanoato de etilo (padrão interno de 19: 0 EE) em 50/50 de hexano / MTBE (Reagente 3).
- Usando a seringa de vidro, adicione 500 μL de solução de reserva de nonadecanoato de etilo (19: 0 EE) a um balão volumétrico de 50 mL.
- Encha o balão para a pontuação volumétrica com o Reagente 3.
- Coloque o balão e inverta 5x para misturar.
- Transfira 3 mL para um frasco limpo de 4 mL para um reservatório de solução de trabalho.
- Usando oE uma seringa de vidro, adicione 300 μL da solução de trabalho a cada um dos frascos de 4 mL contendo os ésteres metílicos e tampões de ácidos gordos secos.
- Vorteie a amostra por 15 s e reserve para 15 minutos.
- Usando uma pipeta Pasteur de vidro, transferir cuidadosamente os ésteres metílicos de ácidos gordos suspensos para um frasco GC de 2 mL contendo a inserção de 400 μL.
- Armazene frascos de GC selados em um congelador de -20 ° C antes da análise.
- Envie amostras para análise GC.
NOTA: A análise deve ser realizada usando uma coluna GC específica e condições descritas no Arquivo Suplementar S1 . É melhor que a análise GC seja completada dentro de 2 semanas de metilação.
Resultados
As tabelas de dados dos relatórios podem ser agrupadas em uma planilha ou banco de dados. Depois de ajustar o fator de resposta (um fator de correção que normaliza a resposta para diferentes comprimentos de cadeia), as áreas de pico podem ser comparadas com a área de pico de padrões externos ou internos para chegar a uma concentração no extrato. Ao dividir-se pela massa de solo extraída, os dados podem ser expressos como massa de FAME por grama de solo ou, usando o peso molecular de cada FAME, o nmol mais comummente relatado por grama de solo. A soma dos FAMEs microbianas é indicativa da biomassa microbiana total e pode ser comparada entre os tratamentos ( Figura 1 ). Certos FAMEs também podem ser associados a grupos microbianas particulares, como bactérias Gram-positivas ou Gram-negativas, actinomicetos, fungos micorrízicos arbusculares e fungos saprótrofos 19 , 20 , 21 , 22 , 23 , 24 . As proporções da massa de biomarcadores específicos podem refletir a abundância relativa desses grupos ( Figura 2 ). Os padrões de abundância de FAME globais criam impressões digitais a nível comunitário, que permitem comparar a dissimilaridade da comunidade microbiana através de técnicas multivariadas como a ordenação ( Figura 3 ). Em contraste com a maioria das abordagens baseadas em DNA, os dados lipídicos a nível comunitário podem ser analisados como abundâncias relativas ou absolutas. Se a biomassa total difere substancialmente entre as amostras, essas duas abordagens darão resultados muito diferentes; As questões ecológicas subjacentes ao experimento devem determinar qual abordagem é usada.
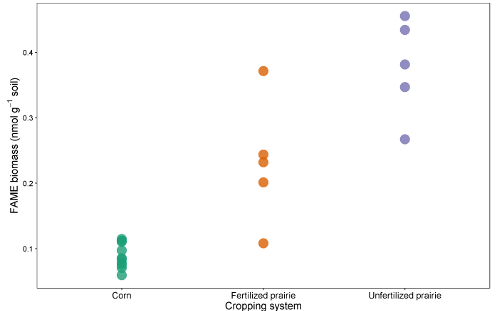
F Figura 1 : biomassa total FAME. Comparação da biomassa total do biomarcador FAME (solo nmol g -1 ) de milho contínuo, pradaria fertilizada e pradaria não fertilizada. Clique aqui para ver uma versão maior dessa figura.
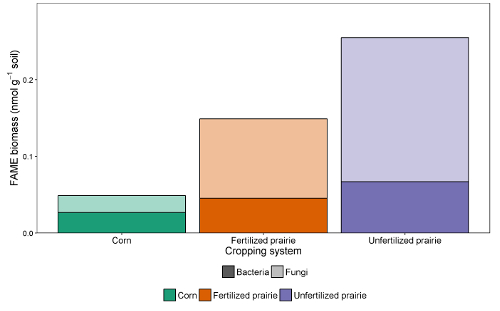
Figura 2 : biomassa FAME de bactérias e fungos. Comparação de tratamento de biomassa biomarcadora bacteriana e fúngica FAME (solo nmol g -1 ) de milho contínuo, pradaria fertilizada e pradaria não fertilizada. Massa fúngica e bacteriana da abundância absoluta. Média (soma f / soma b). Clique aqui para ver uma versão maior dessa figura.
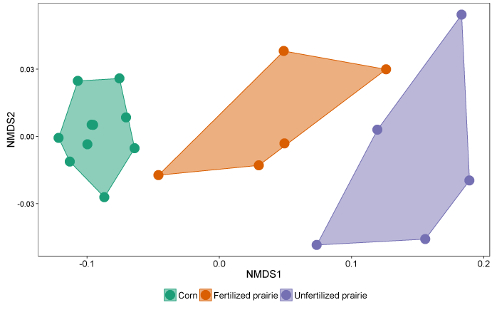
Figura 3 : Escala dimensional não métrica de biomarcadores lipídicos FAME. Comparação da comunidade microbiana global usando perfis de abundância absoluta de todos os biomarcadores microbianas de lipídios FAME. Neste exemplo, o milho contínuo e as comunidades de pradarias não fertilizadas se separam e estão muito distantes, enquanto algumas amostras de pradaria fertilizadas têm comunidades microbianas que se assemelham às do milho e outras se assemelham à pradaria não fertilizada. Clique aqui para ver uma versão maior dessa figura.
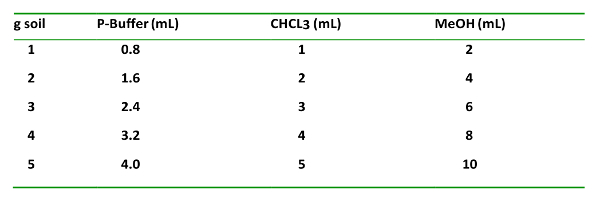
Tabela 1: T h e assiml ven tipo t, ti proporcio na s adicionados g -1 solo, um d t h eir orde r f iti adicionar o n. Isto é importante para a extração e separação adequadas das fases orgânica e aquosa.
Discussão
Para o exame de múltiplas amostras de experiências com muitas repetições e / ou unidades experimentais, os pesquisadores podem achar que a análise de ácidos gordurosos fosfolipídicos (PLFA) seja proibitiva em termos de tempo e materiais 25 . Com o método PLFA, os fosfolípidos das membranas celulares são extraídos, purificados e identificados usando a extração aquosa e orgânica bifásica Bligh e Dyer 26 modificada. Isto é seguido por cromatografia de sílica em fase sólida para separar os lípidos por polaridade e uma metilação alcalina de ácidos gordos fosfolípidos em ésteres metílicos de ácidos gordos. No perfil de PLFA, o rendimento lipídico pode ser baixo, mas de uma pureza muito alta. ID microbiana introduziu um método alternativo, o procedimento de éster metílico de ácido gordo (MIDI-FA). No método MIDI-FA, todos os lipídios são extraídos diretamente de culturas puras ou amostras de solo / sedimento 11 , 12 , 27 atéSaponificação. Este método tem menor perda de lipídios e é rápido porque não tem nenhuma das etapas de concentração ou purificação do método PLFA. No entanto, enquanto o método MIDI-FA é rápido e menos caro, porque foi originalmente projetado para identificar organismos em cultura pura, não há etapas iniciais de extração ou purificação. Assim, pode incluir compostos semelhantes a lipídeos co-extraídos da matéria orgânica do solo que distorcem a assinatura da comunidade 27 , 28 , 29 . Como essa inclusão também pode distorcer as medidas de biomassa, MIDI-FA geralmente foi usado apenas para descrever qualitativamente grosseiramente os lipídios do solo 13 .
O procedimento que descrevemos aqui combina o melhor dos dois procedimentos de extração separados: 1) uma extração e concentração de lipídios usando o método de Bligh e Dyer 26 modificado, e 2) o éster metílico do ácido gordo saProcedimento de ponificação, metilação, extração e lavagem base desenvolvido comercialmente. Este método foi desenvolvido para alcançar os benefícios de ambos os protocolos, minimizando as desvantagens 15 . Ao realizar a extração inicial e isolar os componentes orgânicos solúveis ( por exemplo, lipídios) antes de executar MIDI-FA, e completando isso com um passo de purificação, este protocolo oferece um equilíbrio entre velocidade e precisão. Embora este método possa não ser adequado quando a alta pureza é necessária ( ou seja , para análise PLFA de 13 C) ou ao analisar separadamente os fosfolípidos e lipídios neutros, em muitos casos permite a detecção de respostas comunitárias microbianas a condições ambientais com maior sensibilidade do que a base de DNA Métodos 30 , 31 , 32 , 33 . Os lipídios da membrana se decompõem rapidamente após a morte celular, permitindo-lhesReflete a comunidade microbiana viva no momento da amostragem 5 , 7 , em contraste com o DNA ambiental em que grande parte da informação vem de organismos mortos ou inativos 34 . Dada a alta taxa de dormência observada entre os microorganismos do solo 35 , a caracterização da biomassa viva pode ser utilizada para compreender as interações temporais de plantas e micróbios em uma escala temporal relativamente fina e os biomarcadores lipídicos podem ser utilizados para avaliar o estado fisiológico da comunidade microbiana 7 . Foi demonstrado que são necessários métodos de alto rendimento para avaliar a resposta microbiana em grandes configurações de campo 25 e, embora o método que propomos aqui não reproduza a precisão do perfilamento de biomarcadores PLFA, ele aumenta o rendimento e minimiza a variabilidade realizada com o MIDI-FA procedimento. O método provou ser uma ferramenta eficaz no endereçamentoQuestões relacionadas à dinâmica da comunidade microbiana em uma ampla gama de solos em estudos agrícolas e de ecossistemas em larga escala 36 , 37 , 38 , 42 , 43 , 44 , 45 , 46 , 47 , 48 , 49 , 50 .
As classes de lípidos são combinadas com este método e pode haver uma perda da informação contida nessas classes separadas 22 , 39 , mas combinar as classes de lipídeos pode fortalecer o poder para detectar a origem dos fungos micorrízicos arbusculares do 16: 1 ω5c de ambos os fosfatos - e lipídios neutros 40 . Além disso, enquanto tO número de ácidos gordurosos desconhecidos (que poderia ser derivado de matéria orgânica não viva) pode ser maior com este método, mostrou-se menor do que MIDI-FA e permite comparação de perfis lipídicos com estudos com muitas amostras onde a amostra A capacidade de produção é uma preocupação 15 . Considera-se que a fração neutra deriva principalmente dos lipídios de armazenamento produzidos por fungos, embora também possa haver alguma contribuição menor da fauna do solo 41 . À luz disso, o método descrito aqui pode produzir resultados que mostram uma maior contribuição dos lipídios fúngicos 18: 2 ω 6,9c e 18: 1 ω 9c do que PLFA. Os outros lipídios que tendem a aparecer na fração neutra incluem alguns dos ácidos gordurosos saturados, por exemplo , 16: 0, 18: 0, 20: 0.
Existem diferentes maneiras pelas quais os dados lipídicos podem ser expressos e analisados. As representações mais comuns são a abundância (nmol g -1 solo), mole fractioN (nmol lípido individual nmol -1 lipídio total) e percentual molar (fração molar * 100). Normalizado pelos lipídios totais em uma amostra, fração mole e porcentagem molar são medidas da abundância relativa de um dado lípido. Após uma transformação adequada, por exemplo , a raíz quadrada arcsine, fração mole é apropriada para uso em análise por componentes principais ou ordenação de análise de redundância. Abundância é a quantidade absoluta de um dado lípido extraído por grama de solo. Como a quantidade de lipídeo por célula é razoavelmente constante e a extração lipídica é altamente eficiente e abrangente, a abundância total é uma boa estimativa dos lipídios totais e a abundância de indicadores-chave reflete a biomassa do grupo ecológico que representa 17 . Finalmente, uma boa maneira de analisar a composição da comunidade microbiana é usar métodos de análise multivariada 16 , por exemplo , métodos de ordenação, como a escala multidimensional não-métrica (NMDS -Que não precisa de transformação de dados) ou análise de componentes principais (PCA), pode ser útil para comparar a abundância relativa de todos os biomarcadores de lipídios.
Divulgações
Os autores não têm nada a divulgar.
Agradecimentos
Este trabalho foi financiado em parte pelo Centro de Pesquisa de Bioenergia DOE Great Lakes (DOE BER Office of Science DE-FC02-07ER64494) e DOE OBP Escritório de Eficiência Energética e Energia Renovável (DE-AC05-76RL01830). Os autores gostariam de reconhecer Anders Gurda por sua contribuição paciente e habilidosa para videografia e edição.
Materiais
| Name | Company | Catalog Number | Comments |
| RapidVap | Labconco | 7900000 | Evaporative system |
| RapidVap | Labconco | 7491400 | Sample block |
| Chem-resistant vacuum pump | Labconco | 7584000 | Vacuum pump |
| Chemical trap cannister | Labconco | 7815300 | Trap cannister |
| Solvent insert | Labconco | 7515200 | Solvent trap insert |
| Diaphragm pump | Welch | 7398000 | DryFast vacuum pump |
| Water bath | Fisher | 15-462-15Q | 2 well water bath |
| Gas chromatograph | Various | N/A | For sample analysis |
| Centrifuge | Various | N/A | For sample separation |
| Freeze Dryer | Various | N/A | For sample lyophilization |
| Repipet (6) | BrandTech | 4720440 | For dispensing reagents |
| Vortex | Fisher | 02-215-365 | Analog vortex mixer |
| Teflon centrifuge tubes | Thermo/Nalgene | 3114-0030 | Teflon sample tubes |
| Caps | Thermo/Nalgene | DS3131-0020 | Caps for teflon tubes |
| Test tube | Corning | 9825-16 | 16x100mm tubes |
| Test tube | Corning | 9825-16xx | 16x150mm tubes |
| Caps | Corning | 9998-15 | 15-415 thread black phenolic caps w/PTFE liner |
| Pasteur pipets | Fisher | 13-678-20B | 14.6cm |
| 500 uL glass syringe | Fisher | 13684106LC | Hamilton 81217 |
| Amber vials | Agilent | 5182-0716 | 4mL Amber vials |
| Caps | Agilent | 5182-0717 | Blue screw caps |
| Inserts | Agilent | 5181-3377 | 400uL flat bottom glass inserts |
| Standards | |||
| 19:0 ethyl nonadecanoate (Ethyl nonadecanoate) | VWR | TCN0459-5G | Analytical standard |
| Chemicals | |||
| Dipotassium phosphate (K2HPO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Potassium phosphate monobasic (KH2PO4 - ACS grade or better) | Various | N/A | For making Phosphate-Buffer |
| Methanol (CH3OH - HPLC grade or better) | Various | N/A | For making Reagents 1 & 2 |
| Sodium hydroxide (NaOH - ACS grade or better) | Various | N/A | For making Reagents 1 & 4 |
| Hydrochloric acid (6N HCL) | Various | N/A | For making Reagent 2 |
| Hexane (HPLC grade or better) | Various | N/A | For making Reagent 3 |
| MTBE (Methyl tert-butyl ether - HPLC grade or better) | Various | N/A | For making Reagent 3 |
Referências
- Van Der Heijden, M. G. a, Bardgett, R. D., Van Straalen, N. M. The unseen majority: Soil microbes as drivers of plant diversity and productivity in terrestrial ecosystems. Ecol Lett. 11 (3), 296-310 (2008).
- Bever, J. D., et al. Rooting theories of plant community ecology in microbial interactions. Trends Ecol Evol. 25 (8), 468-478 (2010).
- Suleiman, A. K. A., Manoeli, L., Boldo, J. T., Pereira, M. G., Roesch, L. F. W. Shifts in soil bacterial community after eight years of land-use change. Syst Appl Microbiol. 36 (2), 137-144 (2013).
- Sayer, E. J., et al. Grassland management influences spatial patterns of soil microbial communities. Soil Biol Biochem. 61, 61-68 (2013).
- Fernandes, M. F., Saxena, J., Dick, R. P. Comparison of Whole-Cell Fatty Acid (MIDI) or Phospholipid Fatty Acid (PLFA) Extractants as Biomarkers to Profile Soil Microbial Communities. Microbial Ecol. 66 (1), 145-157 (2013).
- van der Heijden, M. G. A., Wagg, C. Soil microbial diversity and agro-ecosystem functioning. Plant Soil. 363 (1-2), 1-5 (2013).
- Allison, V. J., Miller, R. M., Jastrow, J. D., Matamala, R., Zak, D. R. Changes in soil microbial community structure in a tallgrass prairie chronosequence. Soil Sci Soc Am J. 69 (5), 1412-1421 (2005).
- Kowalchuk, G. A., Buma, D. S., de Boer, W., Klinkhamer, P. G. L., van Veen, J. A. Effects of above-ground plant species composition and diversity on the diversity of soil-borne microorganisms. A van Leeuw. 81 (1-4), 509-520 (2002).
- Bardgett, R. D., Hobbs, P. J., Frostegard, A. Changes in soil fungal:bacterial biomass ratios following reductions in the intensity of management of an upland grassland. Biol Fert Soils. 22 (3), 261-264 (1996).
- Frostegård, Å, Tunlid, A., Bååth, E. Use and misuse of PLFA measurements in soils. Soil Biol Biochem. 43 (8), 1621-1625 (2011).
- Haack, S. K., Garchow, I. H., Odelson, D. A., Forney, L. J., Klug, M. J. Accuracy , Reproducibility , and Interpretation of Fatty Acid Methyl Ester Profiles of Model Bacterial Communitiest. Appl Environ Microbiol. 60 (7), 2483-2493 (1994).
- Drenovsky, R. E., Elliott, G. N., Graham, K. J., Scow, K. M. Comparison of phospholipid fatty acid ( PLFA ) and total soil fatty acid methyl esters ( TSFAME ) for characterizing soil microbial communities. Soil Biol Biochem. 36, 1793-1800 (2004).
- Cavigelli, M. A., Robertson, G. P., Klug, M. Fatty acid methyl ester ( FAME ) profiles as measures of soil microbial community structure. Plant Soil. 170, 99-113 (1995).
- Findlay, R. Determination of microbial community structure using phospholipid fatty acid profiles. Molecular microbial ecology manual. , Available from: https://www.researchgate.net/profile/Robert_Findlay2/publication/281208148_Determination_of_microbial_community_structure_using_phospholipid_fatty_acid_profiles/links/55db4f0b08aec156b9afe776.pdf 2885-2906 (2004).
- Balser, T. C., Liang, C., Gutknecht, J. L. Linking microbial community analysis and ecosystem studies: A rapid lipd analysis protocol for high throughput. Biol Fert Soils. , Forthcoming (2017).
- Findlay, R. H., King, G. M., Watling, L., Watling, L. E. S. Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sediments Efficacy of Phospholipid Analysis in Determining Microbial Biomass in Sedimentst. Appl Environ Microbiol. 55 (11), 2888-2893 (1989).
- Willers, C., Jansen van Rensburg, P. J., Claassens, S. Microbial signature lipid biomarker analysis - an approach that is still preferred, even amid various method modifications. J Appl Microbiol. 118 (6), 1251-1263 (2015).
- Balser, T. C. Phospholipid Fatty-acid Analysis (PLFA). Protocol. , Available from: http://nature.berkeley.edu/soilmicro/methods/BalserPLFA.pdf (2017).
- Zelles, L., Bai, Q. Y., Beck, T., Beese, F. Signature fatty-acids in phospholipids and lipopolysachharides as indicators of microbial biomass and community structure in agricultural soils. Soil Biol Biochem. 24 (4), 317-323 (1992).
- Frostegard, A., Tunlid, A., Baath, E. Phospholipid fatty-acid composition, biomass, and activity of microbial communities from 2 soil types experimentally exposed to different heavy-metals. Appl Environ Microbiol. 59 (11), 3605-3617 (1993).
- Federle, T. W., Dobbins, D. C., Thorntonmanning, J. R., Jones, D. D. Microbial biomass, activity, and community structure in subsurface soils. Ground Water. 24 (3), 365-374 (1986).
- Vestal, J. R., White, D. C. Lipid analysis in microbial ecology - quantitative approaches to the study of microbial communities. BioScience. 39 (8), 535-541 (1989).
- Wilkinson, S. G. Gram negative bacteria. Microbial Lipids. , 299-488 (1988).
- Balser, T. C., Treseder, K., Ekenler, M. Using lipid analysis and hyphal length to quantify AM and saprotrophic fungal abundance along a soil chronosequence. Soil Biol Biochem. 37 (3), 601-604 (2005).
- Buyer, J. S., Sasser, M. High throughput phospholipid fatty acid analysis of soils. Appl Soil Ecol. 61, 127-130 (2012).
- Bligh, E. G., Dyer, W. J. A rapid method of total lipid extraction and purification. Can J Biochem Phys. (37), 911-917 (1959).
- Schutter, M. E., Dick, R. P. Comparison of Fatty Acid Methyl Ester (FAME) Methods for Characterizing Microbial Communities. Soil Sci Soc Am J. 64 (5), 1659-1668 (2000).
- Jandl, G., Leinweber, P., Schulten, H., Ekschmitt, K. Contribution of primary organic matter to the fatty acid pool in agricultural soils. Soil Biol Biochem. 37, 1033-1041 (2005).
- Nielsen, P., Petersen, S. O. Ester-linked polar lipid fatty acid profiles of soil microbial communities: a comparison of extraction methods and evaluation of interference from humic acids. Soil Biol Biochem. 32, 1241-1249 (2000).
- Duncan, D. S., Jewell, K. A., Suen, G., Jackson, R. D. Detection of short-term cropping system-induced changes to soil bacterial communities differs among four molecular characterization methods. Soil Biol Biochem. 96, 160-168 (2016).
- Liang, C., et al. Switchgrass rhizospheres stimulate microbial biomass but deplete microbial necromass in agricultural soils of the upper Midwest, USA. Soil Biol Biochem. 94, 173-180 (2016).
- Jangid, K., et al. Land-use history has a stronger impact on soil microbial community composition than aboveground vegetation and soil properties. Soil Biol Biochem. 43 (10), 2184-2193 (2011).
- Ritz, K., et al. Spatial structure in soil chemical and microbiological properties in an upland grassland. FEMS Microbiol Ecol. 49 (2), 191-205 (2004).
- Carini, P., et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity. J Chem Inf Model. 53 (9), 1689-1699 (2013).
- Lennon, J. T., Jones, S. E. Microbial seed banks: The ecological and evolutionary implications of dormancy. Nat Rev Microbiol. 9 (2), 119-130 (2011).
- Gutknecht, J. L. M., Field, C. B., Balser, T. C. Microbial communities and their responses to simulated global change fluctuate greatly over multiple years. Glob Change Biol. 18, 225-269 (2012).
- Pei, Z., et al. Soil and tree species traits both shape soil microbial communities during early growth of Chinese subtropical forests. Soil Biol Biochem. 96, 180-190 (2016).
- Oates, L. G., Duncan, D. S., Sanford, G. R., Liang, C., Jackson, R. D. Bioenergy cropping systems that incorporate native grasses stimulate growth of plant-associated soil microbes in the absence of nitrogen fertilization. Ag Ecosys Environ. 233, 396-403 (2016).
- Bååth, E. The use of neutral lipid fatty acids to indicate the physiological conditions of soil fungi. Microbial Ecol. 45 (4), 373-383 (2003).
- Ngosong, C., Gabriel, E., Ruess, L. Use of the signature Fatty Acid 16:1ω5 as a tool to determine the distribution of arbuscular mycorrhizal fungi in soil. J Lipids. 2012, 236807(2012).
- Sharma, M. P., Buyer, J. S. Comparison of biochemical and microscopic methods for quantification of arbuscular mycorrhizal fungi in soil and roots. Appl Soil Ecol. 95, 86-89 (2015).
- Oates, L. G., Balser, T. C., Jackson, R. D. Subhumid pasture soil microbial communities affected by presence of grazing, but not grazing management. Appl Soil Ecol. 59, 20-28 (2012).
- Liang, C. Potential legacy effects of biofuel cropping systems on soil microbial communities in southern Wisconsin, USA. Ag Sci. 2 (2), 131-137 (2011).
- Herzberger, A. J., Duncan, D. S., Jackson, R. D. Bouncing Back Plant-Associated Soil Microbes Respond Rapidly to Prairie Establishment. PloS One. 9 (12), 1-14 (2014).
- Fraterrigo, J. M., Balser, T. C., Turner, M. G. Microbial community variation and its relationship with nitrogen mineralization in historically altered forests. Ecology. 87 (3), 570-579 (2006).
- Kao-Kniffin, J., Balser, T. C. Elevated CO2 differentially alters belowground plant and soil microbial community structure in reed canary grass-invaded experimental wetlands. Soil Biol Biochem. 39 (2), 517-525 (2007).
- Mentzer, J. L., Goodman, R. M., Balser, T. C. Microbial response over time to hydrologic and fertilization treatments in a simulated wet prairie. Plant Soil. 284 (1-2), 85-100 (2006).
- Ushio, M., Wagai, R., Balser, T. C., Kitayama, K. Variations in the soil microbial community composition of a tropical montane forest ecosystem: Does tree species matter. Soil Biol Biochem. 40 (10), 2699-2702 (2008).
- Bartelt-Ryser, J., Joshi, J., Schmid, B., Brandl, H., Balser, T. Soil feedbacks of plant diversity on soil microbial communities and subsequent plant growth. Perspect Plant Ecol. 7 (1), 27-49 (2005).
- Fichtner, A., von Oheimb, G., Härdtle, W., Wilken, C., Gutknecht, J. L. M. Effects of anthropogenic disturbances on soil microbial communities in oak forests persist for more than 100 years. Soil Biol Biochem. 70, 79-87 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados